基于2D DenseU-net的核磁共振海马体分割
时佳丽, 郭立君, 张 荣, 高琳琳, 李小宝
(宁波大学 信息科学与工程学院, 浙江 宁波 315211)
阿尔兹海默症是一种常发病于老年人群的痴呆症状,即老年痴呆症[1]。早期临床表现为脑部海马体萎缩,医生可通过核磁共振技术对患者脑部进行三维成像,继而基于影像分析结果进行诊断并制定相关治疗方案。在判断海马体是否萎缩时,医生通常需要对磁共振成像(magnetic resonance imaging,MRI)中的海马体结构进行分割,并进行形状和体积分析。由于海马体属于灰质结构,在MRI图像中与周围组织对比度低,而且海马的形状不规则、体积较小、边缘无明显界线、个体差异性大,从磁共振图像上自动分割海马体结构仍是一项具有挑战性的任务。目前,海马体的手动分割结果仍然被认为是海马体积形态分析的金标准。手动分割一对左右海马体至少需要2~3 h[2],该过程枯燥费时、主观性强且不具有可重复性[3]。因此,实现海马体的自动分割,对研究海马体的结构和形态具有现实意义。
随着2012年 AlexNet 在ILSVRC(ImageNet Large Scale Visual Recognition Challenge)上获得优胜后,关于深度神经网络的研究掀起了新的浪潮。由于医学图像分割问题是为图像中的像素或体素指定具有生物学意义的标记,因而,可将分割问题转化为分类问题,即对图像中的像素或体素进行特征提取,然后基于特征将各个像(体)素分类[4,5]。卷积神经网络(CNN)作为深度神经网络的一种网络结构,不仅在以自然图像为分析处理对象的计算机视觉任务中展示了卓越性能,同时也在医学图像分析领域取得了突破性进展[6,7]。CNN可直接应用于分类图像中的每个像素或体素,典型的像素级分类处理是以滑窗方式处理,即以某个像素点为中心取一个图像块(或立方体块)作为样本训练深度学习模型,使深度学习模型能够提取到深层特征,然后基于特征训练分类器。基于深度学习的像素级分类早期主要采用这种方式。如Kim等[8]采用无监督的训练方式,利用深度网络从图像块中提取特征,代替传统标记融合方法中人工提取的特征,完成海马体整体的分割,提升了分割准确率。Pereira等[9]针对大脑MRI图像,采用较小的卷积核,设计较深的卷积神经网络用于大脑图像分割。然而,这种逐像素取图像块的分类方法非常耗时,且相邻像素块之间存在重叠、运算重复、效率低下的问题,此外,受图像块大小固定限制,此方法无法建模较大的上下文信息,从而影响分类性能[10,11]。针对这种滑窗式分类方法中因相邻输入图像块之间的重叠而导致重复卷积计算的问题,Long等[12]提出了FCN进行像素级分类,将全连接层转换成卷积运算,可以接受任意尺寸的输入图像,并通过浅层特征和高层特征相融合的方式兼顾局部和全局信息,进行像素到像素的语义分割。完全卷积神经网络(FCNs)已经在图像分割领域取得了巨大成功,基于该网络的脑部图像分割中大致分为两类:一是2D FCNs,例如U-net框架[13]、多通道FCN[14]和VGG-16[15]模型;二是3D FCNs[16,17],将2D卷积替换为3D卷积。其中,基于2D FCNs的方法忽略了z轴的上下文信息[18,19],即使采用相邻的切片,检测仍然不能沿着第三维的信息获取空间结构特征,从而降低了分割性能;3D FCN方法虽然可以通过建模3D信息提供更加完整的结构特征,但具有高计算成本和高内存消耗的3D卷积会限制网络深度以及卷积核的感受野,进而限制3D FCN的使用[20,21]。
为了解决这种情况,很多学者尝试加深2D网络层次,以达到更准确、更高效的训练结果。然而,深度卷积网络结构容易出现梯度消失的现象,且当下一些主流深度模型存在较低的特征重用率,导致网络的学习效率难以提高。针对该问题,本文在现有医学图像分割模型U-net基础上,提出一种2D DenseU-net模型,融合了DenseNet[22]和U-net两种网络结构,在U-net中构建当前层与前面所有层的密集连接,缓解梯度消失的同时,加强了特征传播与特征复用。经ADNI库实验表明,本文算法显著提高了海马体分割精度。具体而言,本文主要在数据预处理和网络模型两个方面做了以下的创新和改进:
(1) 数据预处理:宏观角度分析,由于海马体在原始的三维MRI图像中所占比例很小,即含有海马体结构的切片数量过少,造成网络训练出现正负样本失衡问题。本文采用正样本增强技术平衡正负样本;微观角度分析,即使在含有海马体结构的二维MRI切片中,与脑室等脑部其它结构相比,海马体尺寸小且形状不规则,将整幅MRI切片数据直接输入到现有的分割模型中,不能充分学习海马体特征信息。本文通过尺寸剪切及去除无效样本技术放大海马体结构特征并加快模型收敛速度;针对海马体结构边缘不清晰甚至不连续的特点,本文提出一种基于随机数的边缘采样方法,强化海马体边缘细节信息的同时增多了样本个数。
(2) 网络模型:融合DenseNet和U-net两种网络结构,在U-net模型中引入当前层与前面所有层的密集连接,有助于训练过程中梯度的反向传播,缓解梯度消失问题,进而搭建更深层次的网络,并加强特征传播与特征复用;DenseU-net在发挥密集连接优势的同时,保持了U-net网络中下采样和上采样的长连接结构,有助于保存浅层信息。经ADNI库实验证明这种改进显著提升了海马体分割精度,并且加快了训练速度。
1 方法
目前U-net模型已广泛应用于医学图像分割,如脑肿瘤MRI图像、肺部CT图像以及细胞结构等领域。与医学图像的其它组织相比,海马体具有以下特点:尺寸小、形状不规则,且在常规MRI中,其与周边组织结构对比度低,导致边界不清晰甚至不连续。这些特点导致将整幅MRI图像直接输入原始的U-net模型时,海马无法得到有效分割。为此,需要搭建深层次的网络结构提高训练精度和效率。然而,深度卷积神经网络容易出现梯度消失现象,针对该问题,本文对现有U-net模型作出改进,提出一种2D DenseU-net算法框架,并对原有的样本数据进行有针对性的预处理,保证充分提取到完整有效的海马特征信息用于网络训练,最终在ADNI库中取得了显著的分割效果。
1.1 分割海马体的2D DenseU-net算法框架
目前广泛应用于医学图像分割领域的U-net模型只是前向地从上一层较低层次特征中学习出更高层次的特征,此后就不再利用较低层次的特征。换言之,每一层的特征在整个网络中通常只使用一次。特征的重用率低下导致网络的学习效率难以提高。虽然研究人员一直致力于层数更深的网络研究,但一味地提升网络深度而没有提高特征的使用率意义不大。为了提高特征的使用效率,本文借助DenseNet的思想,尝试对网络学习到的每层特征进行重用,建立了当前层与前面所有层的密集连接。目前比较流行的连接方式主要有以下两种,如图1所示。

图1 两种连接方式Two connection methods
图1(a)是ResNet模型的核心,通过建立前面层与后面层之间的“短路连接”(shortcuts,skip connection),有助于训练过程中梯度的反向传播,从而能训练出更深的CNN网络;图1(b)是DenseNet模型的核心,它建立的是当前层与前面所有层的密集连接(dense connection),通过特征在channel上的连接来实现特征重用,加强特征传播。
本文提出的2D DenseU-net算法框架,是在U-net模型基础上建立当前层与前面所有层的密集连接,具体结构如图2所示。其整体网络分为下采样层和上采样层两部分,其中每部分均由3个类似图1(b)结构的密集连接块组成。图2中分别给出了首个下采样密集连接块(Downsampled DenseBlock)与最后一个上采样密集连接块(Upsampled DenseBlock)的具体结构。
在下采样密集连接块中,包含两次3×3的卷积重复应用提取特征以及步幅为2的2×2的最大池化降低分辨率,两次卷积由原来的直接连接变为当前层与前面所有层的密集连接。每一次卷积后通过引入BN(Batch Normalization)提高模型收敛速度,同时在池化操作前利用dropout防止模型过拟合,提高网络泛化能力。此外,每一次下采样均增加特征通道的数量即卷积核个数,保证充分提取海马体结构的特征信息。
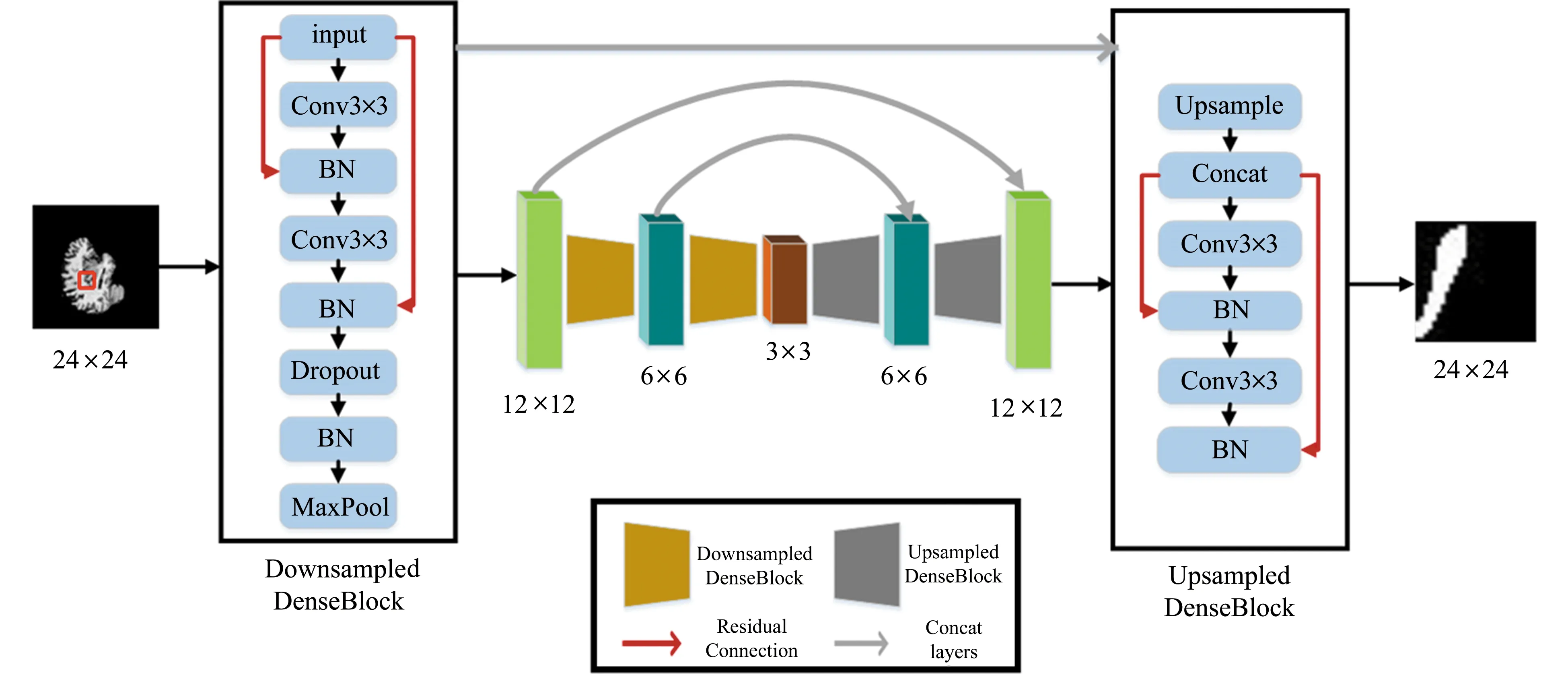
图2 2D DenseU-net模型2D DenseU-net model
在上采样密集连接块中,首先利用2×2的反卷积提高分辨率,接着是两次3×3的卷积重复应用提取特征,这里同样采用当前层与前面所有层的密集连接。与此同时,每一步的上采样都会加入来自对应下采样过程的特征图。
本文算法在上采样和下采样的卷积过程中选用修正线性函数ReLU[23],加速模型收敛,增强网络稀疏性,减少参数间的依赖关系,有效防止了Sigmoid 激活函数在反向传播过程中出现梯度消失的情况;在网络模型的最后一层使用Sigmoid函数作为神经元的激活函数,并设置交叉熵代价函数作为损失函数,避免均方误差损失函数学习速率降低的问题;此外,本文采用自适应矩估计(adaptive moment estimation,Adam)算法作为最佳的梯度下降方法。上述模型框架中的密集连接有助于训练过程中梯度的反向传播,缓解梯度消失并加强特征传播与特征复用;DenseU-net在发挥密集连接优势的同时,保持了U-net中下采样和上采样的长连接结构,有助于保存浅层信息。经ADNI库实验,证明这种改进显著提升了海马体分割精度,并且加快了训练速度。
1.2 图像处理
在输入网络训练模型前,如果只对原始图像进行简单的预处理,输出结果将含有大量噪声。本文针对海马体结构特点,对原始MRI图像进行特殊处理,增强数据样本中的细节部分,保证充分提取完整的海马体特征信息用于网络训练。
1.2.1正样本数据增强
由于磁共振成像中含有大量的与海马体结构无关的体素数据,导致在利用MRI切片数据训练模型时存在严重的正负样本失衡问题,即含有海马体的切片数量远少于不含海马体的切片个数,使得训练模型过度倾向负样本,进而造成图像分割偏差。针对此问题,本文采用一种正样本数据增强的方法来平衡正负样本数量。和以往的数据增强目的方法不同,原有的数据增强是对所有数据进行统一处理以达到增多训练样本的目的,而本文的出发点是为了平衡正负样本,只对正样本数据和对应标签即含有海马体的MRI切片通过弹性形变、随机旋转、通道移动、随机剪切、随机缩放捆绑进行5次变形,以获取更多的正样本数据来提高训练效果,从而达到精准分割。图3是不同MRI切片经过正样本数据增强的示例。
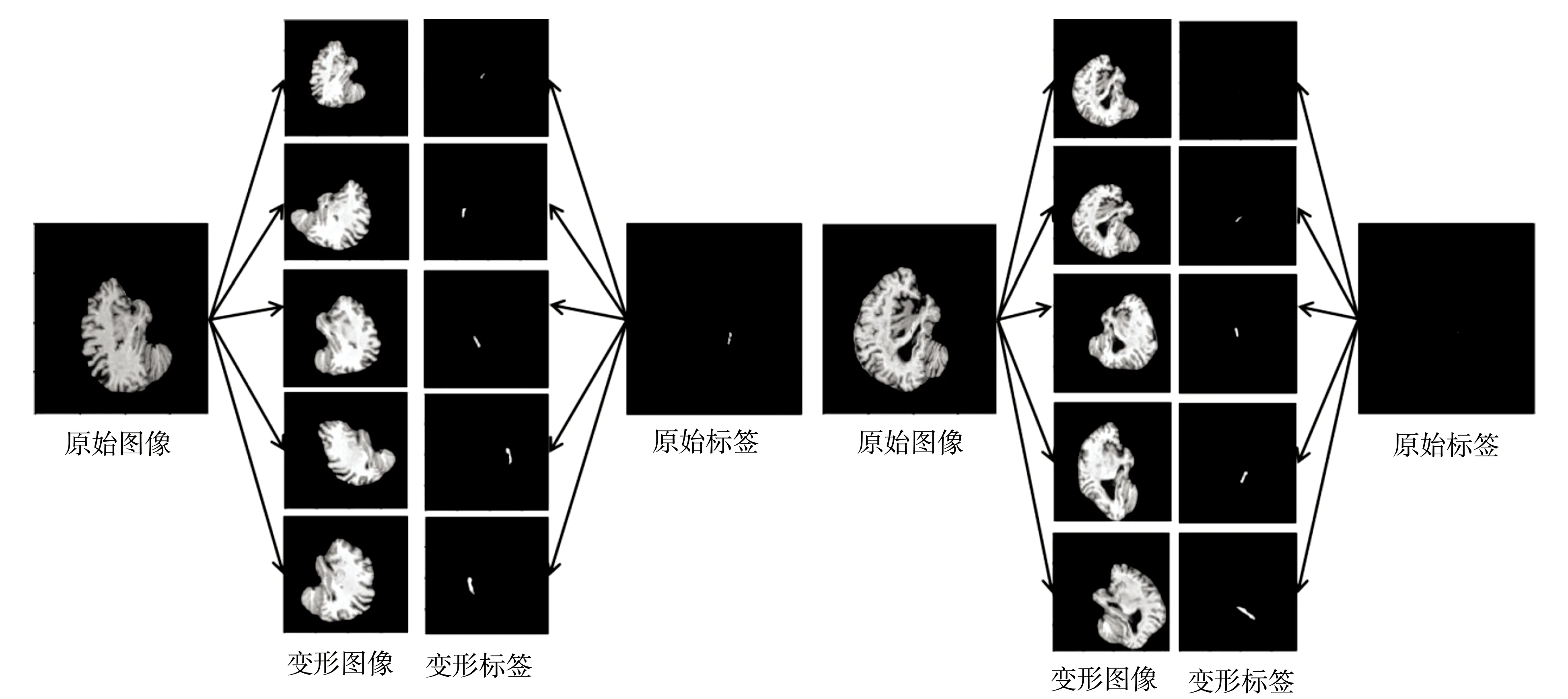
图3 正样本数据增强技术Positive sample data enhancement technique
1.2.2尺寸均等剪切及筛选处理
在含有海马体结构的MRI切片样本中,由于海马体尺寸小且形状不规则,导致不能充分完整地学习海马体特征信息。此外,原始图像数据的尺寸较大,影响训练速度。本文通过将192×192尺寸的数据尝试划分为96×96、48×48及24×24三种尺寸,经大量对比实验表明,24×24的尺寸可以取得最高的分割效果。因此与传统方法将整幅图像作为网络的输入不同,本文通过将每幅图像划分为若干小尺寸的稠密图像块,放大海马体结构细节信息,并以此作为网络的输入,同时也增加了数据样本个数;并在此基础上利用标签信息手动去除大量无效数据,从而简化了训练模型,加快了训练速度。
1.2.3边缘采样
海马体与其它的医学图像组织结构相比,存在边界不清淅、不连续的特点。然而图像边缘含有图像形状的丰富信息,由于海马体自身结构的特点,在提取特征时可能无法完整获取边缘信息,虽然经过特殊的处理已经取得有效的分割效果,但可视化本文结果与标签发现边缘部分仍然比较模糊。因此为了获取更多的边缘细节信息,保证能够充分提取完整的特征用于网络训练,本文增加了边缘采样的处理,即在原有数据基础上,相继补充了各尺寸图像对应的以包含海马信息像素点为中心所依次截取的边缘图像块一起再次投入训练集。本文对边缘采样技术作了以下详细说明。
关于中心点位置的选择:由于目的是增加边缘信息,本文首先将中心点设置在海马边缘处。为了丰富采集的边缘样本,这里对中心点位置的选取增加了随机性和多样性。以边缘位置的像素点为基准,随机选取其指向海马内部一定范围内的点作为新的中心点,并依次提取96×96、48×48、24×24的样本与之前对应尺寸的数据一起再次投入训练集。虽然中心点的选择限制在海马区域内,但每次提取的数据是以这些点为中心的一定范围内的图像,所以实际提取的特征是包含海马及其周围较大区域的信息,该操作达到强化边缘细节的同时也增多了数据样本的个数。如图4所示,白点为实验过程中选取的中心点位置。实验结果证明了该处理对自动精准分割海马的有效性。
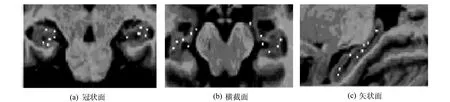
图4 边缘采样技术Edge sampling technique
2 实验结果与分析
2.1 实验数据
本文的实验数据均来自ADNI(Alzheimer′s Disease Neuroimaging Initiative)数据库,它是由美国国家生物医学成像和生物工程研究所(NIBIB)、美国食品和药物管理局(FDA)、民营医药企业与非营利组织组成的研究协会所提供的,其网站上的数据集十分庞大。ADNI收集了超过800名来自全球59个地区的测试者,为阿尔兹海默症(Alzheimer′s Disease,AD)和轻度认识功能障碍(mild cognitive impariment,MCI)的研究提供了丰富的数据支持和诊断决策[3]。参考了文献[3]获取数据的方法,本文实验选择ADNI网站上的数据集是使用快速梯度回波成像技术(MPRAGE)采集并跟踪2年的1.5T T1相,详细参数为:TR为2400 ms;层厚为1.2 mm;图片尺寸为192×192×160。实验共使用140组来自真实患者和健康对比人群的人脑核磁共振影像数据,并由多位资深影像科医生使用专业高纬脑部绘图工具进行海马体分割标注。该数据集为MRI影像数据,每一组数据是一个nii格式的文件,原始图像为三维图像,每个影像包含一系列大脑横截面、冠状面和矢状面的断层图,每一个三维图像由不同数量的二维切片组成,本文利用ITK-SNAP可视化了一组MRI影像数据,如图5所示为有海马体标注的脑部结构图,其中的红色和绿色标注分别为左右脑的海马体结构。
2.2 训练过程
数据划分:本文将140组原始MRI数据划分 为两份,100组用于模型训练,其余40组用于测试。100组数据经过归一化、正样本数据增强、尺寸剪切及去除无效样本、边缘采样等一系列预处理,最终输入网络的为含有海马体的27412组24×24尺寸的有效MRI切片数据样本,40组测试数据仅做了相同的尺寸剪切处理。
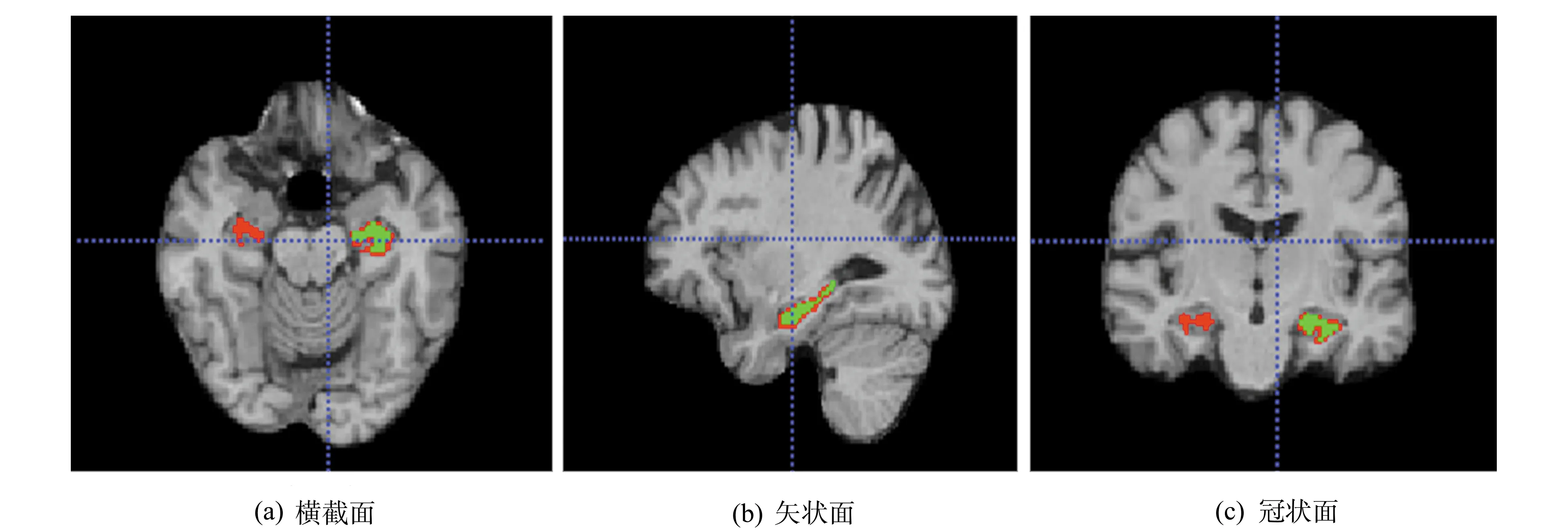
图5 脑部MRI结构Brain MRI structure
参数设置:本文算法通过交叉验证,将用于模型训练的100组数据分为训练集和验证集,提高训练精度,用于防止模型过于复杂而引起的过拟合。与十折交叉验证不同,本文主要设置一个0.2的概率值,将每一份训练集按照一定的比例划分出一部分作为验证集,然后再根据每一次测试结果迭代更新训练参数,达到最优的分割精度。整个训练过程使用Tensorflow和Keras混合深度框架,在GPU环境中训练,训练过程中的参数设置为:训练循环次数epoch为100;batch size为32;卷积层的激活函数选择Relu; dropout层设置比例为0.5;初始学习率设置为0.001,当验证集准确率停滞时,迭代调整降低学习率。最后将训练好的模型推理测试集,二维可视化本文算法的分割结果,与groundturth对比分析,并给出定量结果。
2.3 实验结果定量分析
本文采用Dice系数评价分割结果,该评价标准一般用于医学图像分割领域,是分割结果与专家手动分割金标准的相似度表示,计算方法如式(1)所示。
(1)
式中,Vfcn表示网络分割结果,Vmanu表示对应的标签信息。
2.3.1不同U-net网络模型以及不同尺寸样本对比分析
为了评估不同网络结构改进方法以及不同尺寸样本对海马体分割精度的影响,本文采用96×96、48×48、24×24三种大小的样本分别在不同的U-net网络中训练。这里尝试两种连接方式改进U-net模型:(1)在U-net基础上建立类似图1(a)结构的当前层与前面层的 “短路连接”,得到ResU-net模型;(2)在U-net基础上建立类似图1(b)结构的当前层与前面所有层的密集连接,得到DenseU-net模型。分析比较三种尺寸的图像块在U-net、ResU-net、DenseU-net网络中训练的分割效果,每一次训练均采用正样本数据增强、尺寸剪切及去除无效样本、边缘采样三种数据预处理方式。表1总结了各方法在ADNI库上的海马体分割Dice精度。
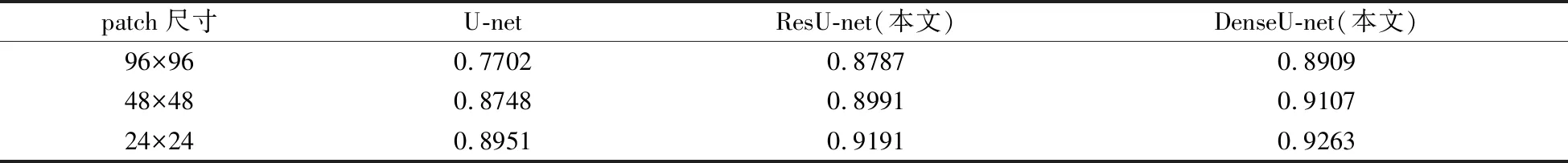
表1 不同改进网络模型以及不同尺寸样本分割结果
从表中得到以下两个结论:(1)用一定尺寸的图像块训练网络模型时,对特征进行重用可以有效提高分割精度,且DenseU-net与ResU-net两种改进模型的对比结果表明“密集连接”优于“短路连接”。这是由于当前层与前面所有层的密集连接方式使得梯度和特征的传递更加有效,网络更加容易训练,从而可以搭建更深层次的网络,且特征复用实现了网络中所有层之间最大的信息流动;(2)网络模型一定时,采用小尺寸的图像块训练效果高于大尺寸的图像块。这是由于海马体本身体积较小,在整个脑部组织中所占比例很少,小尺寸的图像块更有利于提取有效的特征信息。因此总体而言24×24尺寸的样本在本文提出的2D DenseU-net海马体分割算法中取得了最佳分割结果。
2.3.2不同数据预处理对比分析
为了评估正样本数据增强、尺寸剪切及去除无效样本、边缘采样3种数据预处理方式对提高海马体分割精度的有效性,本文统一采用24×24尺寸的图像块在DenseU-net模型中做了以下3组对比实验,分析比较不同预处理技术对分割结果的影响。其中正样本数据增强用a表示,尺寸剪切及去除无效样本用b表示,边缘采样用c表示,表2总结了各方法在ADNI库上的海马体分割Dice精度。
表2的第二列表示3种方式都采用;第三列只采用尺寸剪切及去除无效样本、边缘采样;第四列只采用正样本数据增强、边缘采样;第五列只采用正样本数据增强、尺寸剪切及去除无效样本。分析表2可得:(1)第三列与第二列对比可知,正样本数据增强技术可以平衡正负样本,有效提升模型泛化能力;(2)第四列与第二列对比可知,尺寸剪切可以放大海马体结构细节,并利用标签信息去除大量无效数据简化训练模型,加快训练速度;(3)第五列与第二列对比可知,边缘采样技术增多了数据样本,强化边缘信息。综上所述,3种数据预处理方式均显著提升了分割效果。
2.3.3不同海马体分割方法对比分析
为进一步评估所提方法在海马体分割任务中的分割效果,将本文方法与已有的海马体分割方法——多数表决算法[24]、SCOSD算法[25]、基于相似度的标记方法[26]、基于字典学习的标记方法[27]、联合标记融合算法[26]、nnU-Net[28]、基于稀疏表示和判别性字典学习的标记方法[29]、串行处理卷积神经网络[14]、U-net[13]模型以及本文采用“短路连接”改进的ResU-net模型进行了比较。表3总结了各方法在ADNI库上的海马体分割Dice精度,数据来源于相关论文和代码。
从表3中可以观察到,相比其它方法,本文提出的2D DenseU-net海马体分割算法实现了最高分割效果,原因主要是:(1)相比于人工设定的特征模型,通过有监督的训练,深度网络能自主从图像中提取本质特征,更适合于对比度低、边界不清晰的海马图像;(2)DenseU-net算法中的密集连接方式缓解梯度消失的同时,加强特征传播与特征复用,且保留了U-net上采样和下采样中的长连接结构,有助于保存浅层信息;(3)由于海马体与其它医学组织相比存在较大差别,因此有效的预处理技术在海马体分割任务中显得尤为重要,即本文采用的3种预处理方法保证了充分提取到海马体细节特征用于网络训练。

表2 不同数据预处理方法分割结果

表3 不同海马体分割方法结果比较
2.4 实验过程中loss曲线图对比分析
为了观察不同尺寸样本在不同U-net网络模型中训练的稳定性,本文依次绘制了96×96、48×48、24×24三种尺寸图像块分别在U-net、ResU-net、DenseU-net网络中训练的loss变化趋势图,每一次训练均采用正样本数据增强、尺寸剪切及去除无效样本、边缘采样3种数据预处理方式。图6展示了各方法在ADNI库上实验的loss曲线图。
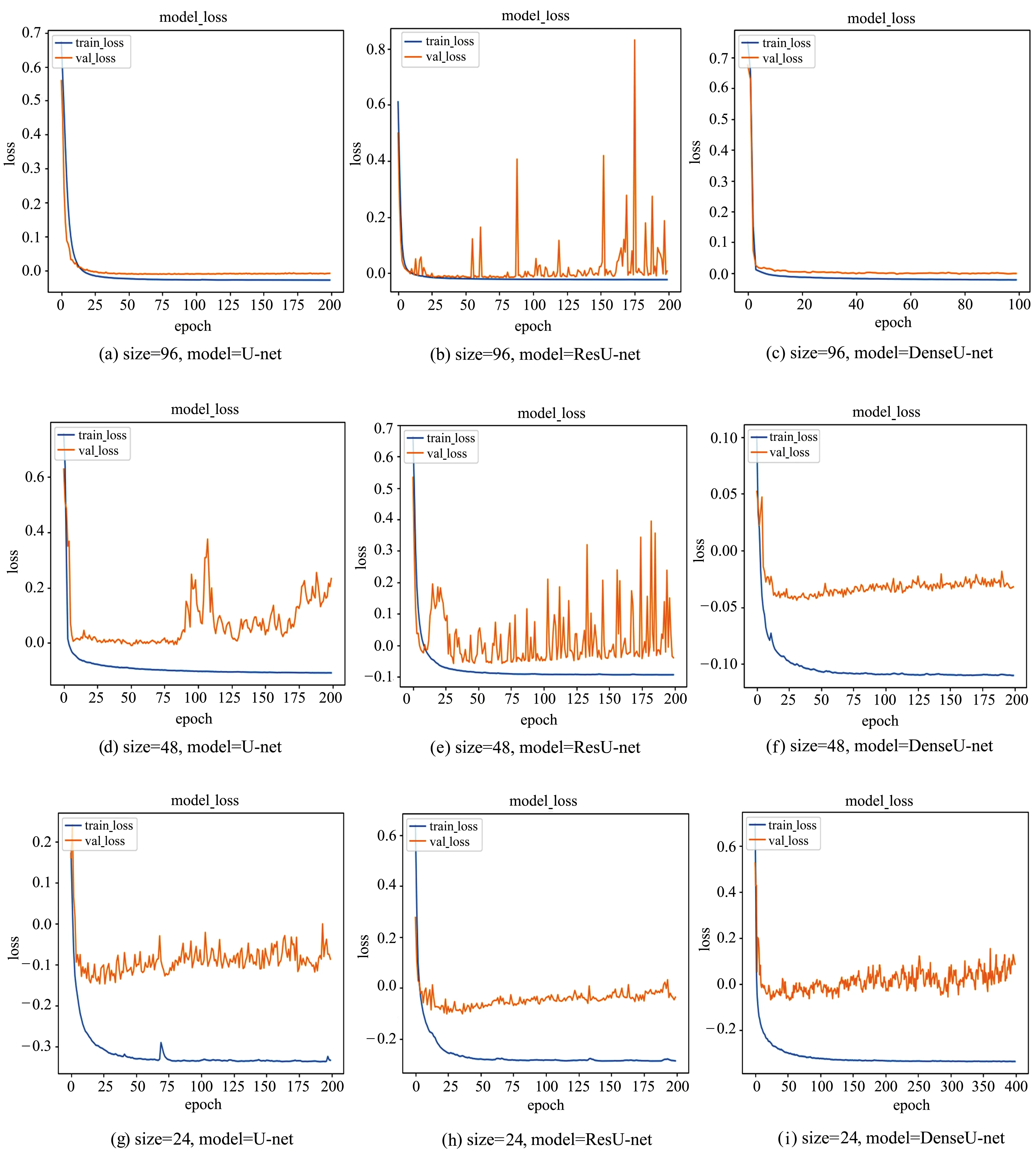
图6 不同patch和模型的训练loss图The training loss map of different patches and different models
从图6可以观察到,当尺寸一定时,采用图1(a)结构的“短路连接”方式导致网络训练过程中震动较大,模型稳定性差;在同一个网络中,虽然大尺寸的图像块训练过程和验证过程拟合度很好,但是分割精度较低。因此总体而言24×24尺寸的图像块在本文提出的2D DenseU-net海马体分割算法中取得了最佳分割结果。
2.5 实验结果定性分析
为了更加直观地评估不同网络结构改进方法以及不同尺寸样本对提高海马体分割精度的有效性,本文将96×96、48×48、24×24三种尺寸图像块分别在U-net、ResU-net、DenseU-net网络中的分割结果可视化,每一次训练均采用正样本数据增强、尺寸剪切及去除无效样本、边缘采样3种数据预处理方式。图7展示了各方法在ADNI库上的海马体分割结果与对应的标签。其中每一行表示输入图像块尺寸一定时,不同U-net网络的海马体分割结果;每一列表示在一定的网络结构中,采用不同尺寸的图像块对海马体分割结果的影响。此外,用黄框、蓝框和红框分别粗略标记了96、48以及24三种尺寸在3个网络模型中的分割结果与原始标签存在的差异。
从图7可以观察到,在每一列中,网络相同时红色框差异性最小即小尺寸效果最好,说明对于海马体这种体积小、对比度低的分割目标,若采用的图像块尺寸过大,图像块之间的信息差别在整个图像块信息中占比极低,即图像块之间十分相 似,使网络很难区分不同类别的目标点,导致网络在海马体上的分割效果较差;在每一行中,尺寸相同时即相同颜色框下DenseU-net模型分割效果最显著,说明密集连接方式优于“短路连接”,且特征重用明显提高了海马体分割精度。因此整体而言24×24尺寸的图像块在本文提出的2D DenseU-net算法中取得了最佳分割结果。

图7 不同patch和模型的海马体分割结果可视化a.不同尺寸样本的U-net分割结果;b.不同尺寸样本的ResU-net分割结果;c.不同尺寸样本的DenseU-net分割结果Visualization of hippocampus segmentation results of different patches and different modelsa. The segmentation results of different size samples in U-net model; b. the segmentation results of different size samples in ResU-net model; c. the segmentation results of different size samples in DenseU-net model
3 结论
为提高人脑核磁共振影像中海马体结构的自动化精准分割,本文提出一种针对海马体结构的2D DenseU-net算法框架。经过大量对比实验,证明了在U-net基础上通过建立当前层与前面所有层的密集连接,有助于训练过程中梯度的反向传播,缓解梯度消失,进而搭建更深层次的网络,并加强特征传播与特征复用;DenseU-net在发挥密集连接优势的同时,保持了U-net网络中下采样和上采样的长连接结构,有助于保存浅层信息;此外,本文的数据预处理技术虽然均为现有的常用方法,但针对海马的特殊结构,本文依次验证了三种预处理方法对提高海马分割精度的有效性,且与以往基于感兴趣区域的海马分割方法不同,本文通过切块增多了数据样本,更有利于深度模型的训练。针对MRI是3D图像,2D单尺度网络不能有效结合上下文信息,下一步会利用多尺度思想提升网络性能,进一步提高分割精度。

