环状RNA的产生、研究方法及功能
刘旭庆,高宇帮,2,赵良真,蔡宇晨,王汇源,苗苗,顾连峰,张航晓
环状RNA的产生、研究方法及功能
刘旭庆1,高宇帮1,2,赵良真1,蔡宇晨1,王汇源1,苗苗1,顾连峰1,张航晓1
1. 福建农林大学林学院,基础林学与蛋白质组学中心,福州 350002 2. 福建农林大学生命科学学院,福州 350002
随着高通量测序技术的发展,环状RNA (circular RNAs, circRNAs)逐渐成为非编码RNA研究领域的热点。本文系统综述了环状RNA侧翼内含子自身互补配对驱动、RNA结合蛋白驱动以及套索驱动这3种环状RNA形成模型,并从高通量文库构建、生物信息学鉴别和常用的实验验证等3个方面对环状RNA的研究方法进行了介绍。同时,本文详细归纳了环状RNA作为microRNA (miRNA)或蛋白的海绵体、调控宿主基因的选择性剪接和表达、翻译成多肽等多种功能。最后通过系统综述植物环状RNA的特征及最新研究进展,为环状RNA在植物学中的进一步研究提供了新的视野。
环状RNA;反向剪接;选择性剪接;海绵体
环状RNA(circular RNAs, circRNAs)是一类由mRNA前体(pre-mRNA)经反向剪接(back-splicing)形成的共价闭合环状非编码RNA[1]。1976年,Sanger等[2]首先在植物类病毒中发现了共价闭合的环状RNA。Hsu等[3]和Arnberg等[4]分别通过电镜在HeLa细胞和酵母()线粒体中也发现了类似的环状转录本。1981年,在四膜虫()中发现一种核糖体RNA (rRNA)基因含有一段能在剪接后形成环状RNA的内含子序列[5]。1988年,在古细菌()中发现23S rRNA的内含子序列在特定位点发生剪接形成稳定的环状RNA并发挥类似转座子的功能[6]。1991年,Nigro等[7]在研究人类()抑癌基因()时发现了由于不同剪接方式形成的几种环状转录本,随后陆续在人类()基因[8,9]、小鼠()()基因[10]、大鼠()细胞色素基因[11]及人类基因[12]中都发现环状RNA的存在。2010年,Burd等[13]发现环状RNA(cANRIL)会影响抑癌基因的表达,表明其可能具有生化和表型效应。
虽然早在几十年前就已发现环状RNA,但由于环状RNA不具有游离的3′和5′末端[14],无法通过依赖于富集poly (A)的分子技术检测到;同时,可环化外显子是经反向剪接接合的,异于经典的线性剪接,早期转录组分析的映射算法无法直接将测序得到的片段联配到基因组,使得人们一度认为环状RNA只是错误剪接的副产物。随着高通量测序和生物信息学分析技术的发展,2012年,Salzman等[15]首次指出环状RNA是由mRNA前体经反向剪接产生的环状转录本,并且发现其大量存在于人类的不同类型细胞中;Hansen等[16]发现环状RNA可以作为microRNA (miRNA)的海绵体,调控生物体的生长发育过程。自此,环状RNA的研究迅速成为研究的焦点(图1)。本文系统综述了环状RNA的形成机制、研究方法及主要功能,概述了植物环状RNA的主要特征及最新研究进展,并对环状RNA研究有待进一步提高的几个方面进行了展望。
1 环状RNA的形成
根据基因结构注释信息,环状RNA的来源主要有:(1)仅外显子来源的环状RNA (exon circRNA)[1,15];(2)上下游外显子反向剪接且内含子保留形成的外显子-内含子环状RNA (exon-intron circRNA, EIciRNA)[17];(3)仅内含子来源的环状RNA (circular intronic RNA, ciRNA)[18];(4)融合基因来源的环状RNA(fusion-circRNA, f-circRNA)[19,20];(5)聚合酶Ⅱ (Pol Ⅱ)的转录通读形成的通读环状RNA (read-through circRNA, rt-circRNA)[21]。尽管环状RNA的来源不同,但主要是通过头对尾的反向剪接方式产生的,其形成受到顺式和反式作用元件的调控[22],但对其生物形成机制的研究并不完善。目前报道的驱动环状RNA形成的方式主要有:内含子配对驱动、RNA结合蛋白(RBPs)驱动以及套索驱动环化这3种模型(图2),除此之外,Pol Ⅱ的转录、剪接体的活性及Poly (A)的延伸等因素也会影响环状RNA的形成。

图1 环状RNA研究的关键事件
1.1 环化驱动模型
1.1.1 内含子配对驱动环化
由于环化外显子两侧的内含子序列互补配对(图2A),使得mRNA前体下游5′剪接供体位点直接与上游3′剪接受体位点接合形成环状RNA,所以内含子配对驱动又叫做直接反向剪接模型[23]。研究发现,含30~40 nt的反向互补重复序列的微型内含子(<100 nt)就可促进外显子的环化[24]。RCMs (reverse complementary matches)、ICSs (intronic complementary sequences)及元件等是可环化外显子侧翼内含子上含量丰富的互补配对序列,能有效促进侧翼内含子配对进而环化。例如秀丽隐杆线虫()侧翼内含子上的RCMs可促进转录本形成发夹结构,从而促进外显子的环化[25]。来源于的融合基因f-circM9,是由于和基因上存在ICSs而有效环化的[19];在人类circGCN1L1的研究中,通过CRISPR/Cas9敲除ICS序列,则无法检测到circGCN1L1的表达[26],暗示该基序对circGCN1L1的生成至关重要。元件是哺乳动物尤其是人基因中驱动成环的主要方式,已成为分析和预测环状RNA形成的重要基础[1,27,28]。长侧翼内含子有助于外显子环状RNA的环化[17,27],原因可能是等互补元件的数量随着内含子长度的增加而增加。
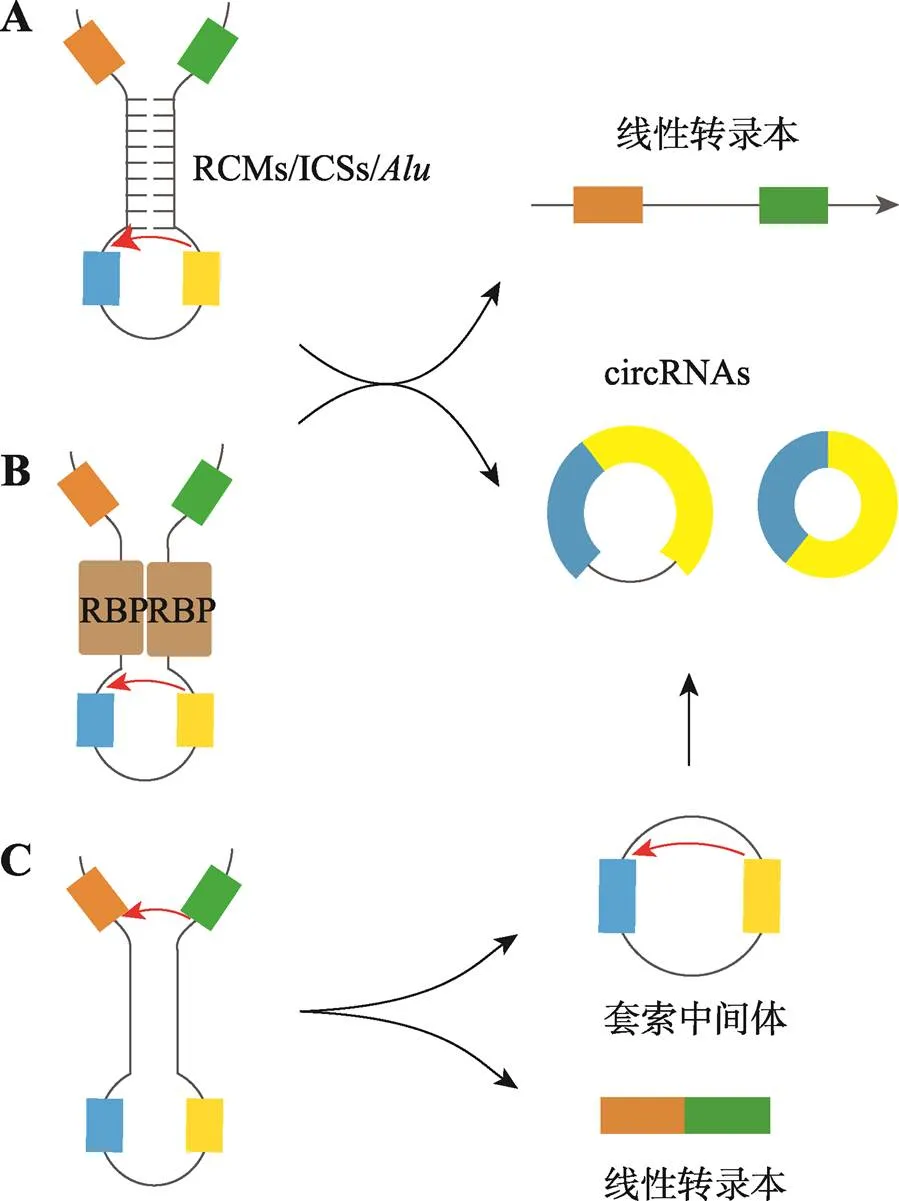
图2 环状RNA形成的环化模型
A:内含子自身互补配对。RCMs/ICSs/等互补配对元件促进侧翼内含子配对进而环化。B:RNA结合蛋白驱动。大多数RNA结合蛋白通过结合到可环化外显子的侧翼内含子上来促进环化。C:套索驱动。mRNA前体剪接时会发生外显子跳读事件,产生包含内含子-外显子的套索中间体,随后该中间体发生反向剪接形成环状RNA。
1.1.2 RNA结合蛋白驱动环化
RNA结合蛋白伴随RNA的生命始终[29],是促进组织特异性环状RNA形成的关键[30]。RNA结合蛋白通过结合侧翼内含子序列中的特殊基序参与环状RNA的形成(图2B)。在果蝇()和人基因中,基因的第2个外显子可形成环状RNA,其形成依赖于侧翼内含子上MBL的特异性结合基序[31]。NF90/NF110 (nuclear factor 90/ 110)具有双链RNA结合域(double-strand RNA-binding domain, dsRBD),通过结合至等互补重复序列而促进环化[32~34]。HRNPL (heterogeneous nuclear ribonucleoprotein L)通过与可环化外显子的侧翼序列结合来促进环状RNA的形成[35]。相似的,FUS (fused in sarcoma)倾向于结合到环化接合位点两侧的内含子序列来影响环状RNA的形成[36]。QKI (quaking)是一个二聚体,通过与可环化外显子的侧翼内含子结合而促进两翼内含子互相靠近,进而连接成环[37,38]。RBM20 (RNA-binding motif protein 20)参与小鼠基因的I-band区域的选择性剪接,敲除基因位点后,无法形成与RBM20相关的环状RNA[39]。hnRNP (heterogenous nucear ribonuceoprotein)和SR (serine–arginine)蛋白通过与内含子重复序列协作,共同调节环状RNA的表达[40]。上述RNA结合蛋白对环状RNA的生成起到促进作用,也有RNA结合蛋白抑制环化的报道。例如ADAR1 (adenosine deaminase 1 acting on RNA)通过与双链RNA相结合,能将腺嘌呤核苷编辑为次黄嘌呤核苷(即A-I RNA编辑)[41]。元件是ADAR的主要结合靶位点,A-I RNA编辑可打乱等配对元件的稳定性进而抑制环状RNA的形成[25,42,43]。除此之外,DHX9 (DEAH-box helicase 9)是通过与反向互补元件相结合,导致元件的解旋进而抑制了环状RNA的形成[44]。
1.1.3 套索驱动环化
当mRNA前体进行经典的GU/AG剪接时,可以发生跨外显子的剪接方式,即外显子跳读(exon skipping),产生包含内含子-外显子的套索中间体,随后该中间体发生反向剪接形成环状RNA[23](图2C)。在人体中普遍存在GU/AG经典剪接信号,也存在如GC/AG或AU/AC等的非经典剪接信号[21]。然而,这种依赖于GU/AG经典剪接信号的驱动模式在水稻()中却非常少,其普遍存在如GC/ GG、CA/GC及GG/AG等非经典剪接信号[45]。内含子环状RNA是套索驱动模型中的特殊方式,其依赖于5′剪接位点的7 nt GU-rich基序和分支位点(branchpoint site)的11 nt C-rich基序,通过聚合酶Ⅱ的转录形成一个套索内含子,最终通过2′, 5′-磷酸二酯键共价连接而环化,接着从内含子3′端到分支位点的多余序列被降解[18]。在人和小鼠中发现,内含子环状RNA的数量要远远超过外显子环状RNA的数量[46],并且在其他脊椎动物细胞的细胞质中也发现了稳定的内含子环状RNA[47]。目前在拟南芥()、番茄()、水稻和玉米()中的研究表明这种套索结构在植物中也是广泛存在的[48]。
1.2 其他调控因子
Pol Ⅱ的转录伸长率(transcription elongation rates, TER)能影响剪接体的组装和特异性序列的剪接调控位点,进而影响了剪接事件的发生[49]。研究发现,环状RNA的形成与Pol Ⅱ的转录伸长率成正相关[26],并且抑制Pol Ⅱ的转录终止会促进转录通读进而促进了环状RNA的生成[50],通读环状RNA就是由于Pol Ⅱ的通读而产生的[21]。值得一提的是,另一种常见的聚合酶,即聚合酶Ⅲ(Pol Ⅲ),具有催化合成如tRNA,5S rRNA及snRNA等小分子RNA的特性[51],于是Schmidt等[52]运用Pol Ⅲ型启动子构建了一种高效的环化体系,该体系的环化效率要明显优于运用Pol Ⅱ型启动子构建的环化体系。最近,Litke等[53]基于核酶(ribosome)自剪切作用将Pol Ⅲ型环化体系的环化效率大大提高,实现了环状RNA的适体(aptamer)过表达。研究还发现,抑制核心剪接体如SF3b/SF3a复合体的活性,可促进mRNA前体反向剪接形成环状RNA[50];再者,存在于内含子中的G-U摆动配对及poly (A)的延伸也会抑制环状RNA的形成[24]。在细菌和古细菌中缺少剪接体,其通过特殊的内切酶识别切割BHB (bulge-helix-bulge)结构,然后通过连接酶形成环状RNA[54]。在真核生物如真菌、脊椎动物、植物及人体中,tRNA前体在成熟过程中通过特异性蛋白也可形成环状RNA[55,56]。
2 环状RNA的研究方法
2.1 环状RNA的鉴别
2.1.1 生物信息学鉴别方法
早期普通RNA-Seq文库构建方法无法揭示环状RNA的全貌,只能捕捉少量的环状RNA[21]。当前采用的环状RNA建库方法是:(1)不经过poly (A)筛选的文库构建方法,去除rRNA后进行转录组测序;(2) rRNA去除并结合RNase R处理的文库构建方法[1,57](图3A)。然而,一些不含poly (A)尾且RNase R处理后未完全降解的线性转录本通常会干扰环状RNA的下游分析[58]。为解决这一问题,Panda等[46]利用RPAD (RNase R treatment followed by Polyadenylation and poly (A) + RNA Depletion)这一新方法,鉴定到了大量高富集的环状RNA。另外,研究者根据不同的实验目的,会采用不同的经过优化的RNA- Seq文库构建方法。例如Vo等[21]采用的外显子捕获技术(exome capture RNA-seq),利用探针准确捕获到了大量的环状RNA,而且保留了线性mRNA。另外,超深度非poly (A) RNA-seq (ultra-deep non-poly- A RNA-seq)能深层次鉴别到更多的环状RNA,包括大量融合环状RNA[59]。

图3 环状RNA的文库构建和生物信息学研究方法
A:RNase R处理的高通量测序文库构建。B:基于反向剪接位点测序片段的环状RNA识别方法。
环状RNA的反向剪接位点(back-splicing junction, BSJ)是鉴别和定量环状RNA的关键[60],测序后获得的读段(reads)需经过环状RNA识别软件进行基因组比对和校正[61](图3B)。Hansen等[62]对多种环状RNA检测工具进行了比较,发现单个软件往往因为算法的设计问题在某些方面存在着一定的局限性,建议同时使用2个及以上的软件进行环状RNA的预测。例如Ji等[30]在识别人、猕猴()及小鼠的环状RNA时,同时使用CIRI2、DCC、MapSplice、CircExplore2及CIRI-full等5种软件。值得一提的是,研究者虽然可以通过二代测序获得大量的反向剪接位点,却无法高通量获得环状RNA的完整内部信息,也无法对不同的选择性剪接产物进行精准定量。于是,Zheng等[63]提出了基于环状转录本测序中的反向重叠区特征获取全长序列的方法CIRI-full,其不仅可以判断双端测序获得的环状序列是否覆盖整个环状RNA,而且可以重组每个反向剪接位点产生的多个选择性剪接产物的全长序列,并精准预测选择性剪接产物的相对丰度。另外,对于环状RNA的选择性剪接事件研究的匮乏,Feng等[64]开发的软件CircSplice,可以在短时间内特异性地识别环状RNA的内部选择性剪接事件,并发现这些由于选择性剪接产生的环状RNA异构体对癌症有一定的调控功能。目前已知的环状RNA识别软件超过20种,应用于识别、可视化及组装人、动物、植物等生物体中的环状RNA[61,65](表1)。
随着大量的环状RNA被鉴定出来,急需界面友好的数据库存储并可视化这些环状RNA数据,目前已公布了超过15种环状RNA的数据库,包括人、小鼠、拟南芥和水稻等动植物(表2)。其中与植物环状RNA相关的数据库有5个,本实验室建立的ASmiR数据库存储了目前常见的几种植物的环状RNA数据,并呈现了miRNA靶位点和线性RNA及环状RNA的选择性剪接之间的相互调控[80]。
2.1.2 分子生物学鉴别和验证方法
由于环状RNA没有游离的3′和5′端,因而无法应用基于poly (A)的分子生物学技术,如RACE (rapid amplification of cDNA end),也无法通过普通的电泳迁移率和片段大小来区分。目前在实验室中主要有以下几种鉴别方法:(1) RT-PCR定量法:针对环状RNA的反向剪接位点设计一对发散引物(divergent primer),只有环状RNA才可得到预期扩增片段。并进一步对该扩增产物进行Sanger测序确认环状RNA的真实性[93]。但由于PCR方法可能会发生模板的切换或mRNA之间的反式剪接(trans-splicing)等情况,一般采用RNase R消化线性转录本,富集环状RNA的定量PCR方法来鉴别环状转录本[94]。(2)探针杂交法:RNase H是一个可以降解RNA-DNA中RNA分子的核酸内切酶。当两个短的DNA探针杂交至目的RNA分子时,若该RNA分子为线性,则在RNase H消化后电泳出现三条带;若该RNA分子为环状,则为两条带(一个DNA探针时,同理推测)[95]。RNA印迹[10,95]也是高频率使用的一种方法,值得注意的是,探针设计要跨越反向剪接位点(内含子环状RNA的探针根据内含子序列设计)。(3)二维变性聚丙烯酰胺凝胶(2D-PAGE)电泳:环状RNA的迁移率比线性RNA分子要慢,在2D-PAGE中线性RNA沿对角线迁移而环状RNA则弧向迁移[96]。在研究中往往结合以上多种方法来验证环状RNA。如Ghorbani等[97]在研究受胁迫下玉米的环状RNA的表达情况时,同时使用了RT-PCR、RNase R、2D-PAGE及RNA印迹等多种方法来鉴别环状RNA。

表1 环状RNA的识别软件
2.2 环状RNA的功能验证方法
2.2.1 构建环状RNA缺陷型突变体
突变体的构建主要有物理诱导、化学诱变及基因编辑等,CRISPR/Cas9就是基因编辑中高效又低成本的一种方法,已大量运用于人[98]、动物[99]、植物[100]及果蝇[101]基因的定向精准修饰。Cdrlas是哺乳动物大脑中高表达的一种环状RNA,能吸附miR-7和miR-671。Piwecka等[102]利用CRISPR/Cas9在小鼠基因组中敲除基因座,获得了缺陷型突变体(-KO)。研究发现敲除后,miR-7的表达量显著降低,而miR-671显著上调。在- KO小鼠大脑中的进一步研究发现,由miR-7调控的靶基因如、及等的表达水平升高,这暗示了Cdrlas在行为应激方面的作用。有趣的是,Csy4是CRISPR家族中Cas9的同源蛋白,能切割RNA分子且维持5′端产物的稳定性,Borchardt等[103]利用该特性构建了具有Csy4识别位点的环状RNA过表达体系。目前针对环状RNA基因编辑技术在植物中的应用还比较少。

表2 环状RNA存放数据库
2.2.2 环状RNA过表达载体
根据内源外显子环化的原理,构建环状RNA过表达载体,研究侧翼内含子序列、蛋白因子等对环化的促进或抑制作用,探索环状RNA的功能[22]。环状RNA过表达载体包含可环化外显子和具有反向互补序列的侧翼内含子来促使环化剪接,其转染到细胞中可以主动促进成环。Ashwal-Fluss等[31]为了探究环状RNA的形成机制,构建了不同长度侧翼内含子序列的circLuna和circMbl的环状RNA过表达载体,发现侧翼内含子对环状RNA的形成具有重要作用。Conn等[37]通过研究QKI对基因的某一区段形成环状RNA的影响时,构建具有荧光蛋白GFP和mCherry的环状RNA过表达载体,探究QKI对环状RNA形成的影响。
2.2.3 RNA干扰技术
为验证目的环状RNA的功能,可以使用RNA干扰技术来有效沉默或抑制环状RNA的表达,即由siRNA/shRNA与环状RNA结合并使之降解[104,105]。首先在不影响线性mRNA的基础上,针对特定的环状RNA序列设计跨越反向剪接位点的siRNA/shRNA,以敲低环状RNA的表达,观察其产生的生物效应或表型变化。同时需要针对环状RNA的线性宿主基因设计专门的siRNA/shRNA进行敲除,确保该生物效应或表型是由所对应的环状RNA被干扰后所引起的,经过这两步得到环状RNA特有的调控功能的结论。例如,Legnini等[106]用RNA干扰技术敲低circZNF609,发现明显抑制了肌细胞的增殖。Zhu等[107]通过构建shRNA载体敲除与肠道干细胞(intestinal stem cells, ISCs)器官形成密切相关的circPan3,发现circPan3在体内参与了免疫细胞介导的肠道干细胞自我更新。
3 环状RNA的功能
环状RNA在早期被认为是一种剪接副产物,不具有任何功能。随着人们的深入研究,发现环状RNA并非是剪接副产物,其来源广泛、保守、稳定、具有组织特异性,并且在生物体的生长发育过程中扮演着多种功能角色[1,22,108]。本文从环状RNA作为miRNA或蛋白的海绵体、调控宿主基因的选择性剪接和表达、翻译成多肽等几个方面描述其功能(图4)。
3.1 作为miRNA或蛋白的海绵体
环状RNA上存在miRNA的结合位点,可作为miRNA的海绵体[109](图4A)。例如小鼠上有16个miR-138的结合位点;ciRS-7上含有超过70个miR-7的结合位点,通过AGO2蛋白实现竞争性吸附miR-7[110]。在癌症中越来越多的报道表明环状RNA充当miRNA的海绵体并参与转录调控的功能,如circRNA-cTFRC[111]、circPSMC3[112]和circSETD3[113]等,其中circRNA-cTFRC充当miR-107的海绵体,促进宿主基因的表达。研究者分别在水稻[114]、小麦(L)[115]、番茄[116]、枸橘(L)[117]及拟南芥[114]等植物中也发现了一些具有miRNA海绵体功能的环状RNA。但是,Ye等[114]发现拟南芥中仅约5.0%的环状RNA可以预测到miRNA的靶位点,而在水稻中该比例也仅为6.6%。可能原因是,植物中环状RNA内部要有足够的miRNA结合位点或环状RNA的表达量足够高,才能实现对靶基因的调控功能[114]。另外在水稻中发现Os08circ16564存在OsmiR172的结合位点,当过表达Os08circ16564并保持OsmiR172的表达水平不变的情况下,其线性基因的表达水平降低[93]。
有些环状RNA上有一个或者多个RNA结合蛋白的结合位点,可作为蛋白分子的海绵体(图4A)。MBL能促进circMbl的合成,且合成的circMbl上存在特异性的MBL结合位点[31]。当MBL高表达时,会促进circMbl的生成而抑制了线性转录本的表达;并且circMbl会与过量的MBL结合,使其含量趋于稳定[31]。circPABPN1来源于基因,HuR是调控线性转录本合成的蛋白,对circPABPN1的丰度无影响。有趣的是,circPABPN1能与HuR结合,进而显著影响了HuR与的结合,使得的表达水平降低[118]。CircFoxo3上同时存在MDM2 (mouse double-minute 2)和p53的结合位点,circFoxo3能促使MDM2诱导的p53的泛素化,导致p53蛋白的整体降解[119]。最新研究发现,m6A (N6-methyladenosine)修饰的环状RNA能与m6A识别蛋白YTHDF2结合,YTHDF2通过募集HRSP12,介导RNA内切核酸酶RNase P/MRP复合物降解环状RNA[120]。另外,在拟南芥中发现,内含子套索RNA能与切割复合物DCL1/HYL1结合,进而抑制了miRNA的合成[121]。
3.2 调控宿主基因的选择性剪接
拟南芥()基因的第6外显子可生成环状RNA,该环状RNA可强烈结合宿主基因的DNA基因座,形成一个RNA:DNA杂合体的R-loop结构(图4B)。该R-loop结构可抑制该区段的转录,于是发生了跨外显子的选择性剪接事件,促进产生选择性剪接转录本变体,影响其开花表型[122]。在毛竹中的数据表明,在产生环状RNA的基因中发生选择性剪接事件的频率显著高于随机选择的基因,说明环状RNA可能调控其线性基因的选择性剪接过程[123]。
3.3 调控宿主基因的表达
环状RNA除了作为miRNA或蛋白的海绵体参与调控宿主基因的表达外[68,110],还能通过与RNA互作参与转录后调控[17]。Li等[17]在人类细胞核中发现EIciRNA能与U1 snRNA互作形成复合物EIciRNA-U1 snRNP,该复合物再与Pol Ⅱ转录复合物相互作用而促进宿主基因的表达(图4C,左图)。另外,Zhang等[18]通过敲低ci-ankrd52、ci-mcm5和ci-sirt7这3种内含子环状RNA的表达,发现相应的宿主基因表达量都降低了,说明内含子环状RNA能顺式调控宿主基因的表达。环状RNA还可以通过与线性转录本竞争性剪接,从而影响宿主基因的表达[31]。Lu等[93]通过构建水稻过表达遗传转化体系,发现环状RNA及它的线性异构体可能抑制宿主基因的转录后表达。环状RNA还能通过表观遗传修饰来调控宿主基因的表达。m6A是腺嘌呤重要的甲基化修饰[124],Zhou等[125]在人类细胞中发现了大量m6A修饰的环状RNA,该环状RNA会影响宿主基因的稳定性(图4C,右图)。来源于基因的环状RNA(FECR1),能特异性地与启动子区域结合,并招募去甲基酶TET1诱导该区域的去甲基化,进而调控靶基因表达[126]。DNMT1是哺乳动物DNA甲基化维持所必需的关键甲基转移酶,研究者检测到DNMT1参与了FECR1诱导启动子的去甲基化过程,还发现FECR1能与启动子结合,从而下调的转录[126]。另外,Wei等[127]在拟南芥中鉴定了首个m6A结合蛋白ECT2,ECT2能在细胞核中调节3′ UTR的修饰,亦可在细胞质中调节线性RNA的稳定性。

图4 环状RNA的功能
A:充当miRNA或蛋白的海绵体。B:通过R-loop介导调控宿主基因的选择性剪接事件。C:调控宿主基因的表达。左图为环状RNA通过与U1 snRNA及Pol Ⅱ转录复合物互作,进而调控宿主基因的表达;右图为m6A修饰的环状RNA会影响宿主基因的稳定性。D:翻译功能。环状RNA能翻译多肽。E:衍生假基因。环状RNA经反转录衍生假基因,并整合进基因组。
3.4 环状RNA潜在的翻译功能
环状RNA不仅能调控线性转录本的转录,还能翻译产生蛋白[106,128](图4D)。m6A修饰的环状RNA可能在转录后调控水平上参与靶基因的转录。Yang等[129]发现在人类细胞中的环状RNA上存在大量一致性的m6A基序,并且在多种蛋白的参与下,一个m6A位点便可启动环状RNA的翻译。环状RNA不仅可以通过m6A修饰进行翻译,有研究表明含有IRES (internal ribosomal entry site)的环状RNA也可以在体内或体外翻译[130],在果蝇中还发现一类可利用宿主基因的起始子与核糖体结合而行使翻译功能的环状RNA[128]。Zhang等[131]发现p53诱导转录的长链非编码RNA ()的第2个外显子可形成环状RNA (circPINTexon2),circPINTexon2可翻译87个氨基酸的多肽(PINT87aa),该多肽能直接与转录伸长因子PAF1复合物互作而阻止了mRNA的转录伸长。最新研究发现,高富集的体外环化的环状RNA可在小鼠体内稳定地表达蛋白,说明环状RNA可作为一种有效的蛋白表达工具[132]。
3.5 环状RNA的其他功能
线性mRNA会经过反转录衍生得到与外显子先后顺序相同的假基因,稳定的环状RNA也可以经过反转录后衍生出倒序的假基因并整合进入基因组[133](图4E)。Dong等[133]在小鼠参考基因组中通过CIRCpseudo鉴别到至少33个来源于同一环状RNA (circRFWD2)的假基因,还鉴别到了来源于circSATB1和circDIAP3的假基因;还发现来源于人类circPRKDC和circCAMSAP1的假基因在大猩猩()和黑猩猩()的基因组中也存在。更有趣的是,在来源于小鼠circSATB1的假基因上发现了能影响染色体构型和调控基因表达的CTCF结合位点,而在对应的的外显子上却没有该结合位点,这预示着环状RNA可能通过衍生假基因对生物体的生长发育有着一定的生物效应。然而,这些假基因是如何被反转录并整合到基因组中仍然是未知的。
4 环状RNA在植物中的研究
4.1 植物环状RNA的特征及形成
目前对环状RNA的研究大部分集中在动物上,植物环状RNA的研究还不够广泛。在拟南芥[134]、水稻[93]、玉米[135,136]、番茄[137]、大麦[138]、小麦[115]、猕猴桃()[139]、沙棘()[140]和毛竹[123]等植物中都鉴别到了环状RNA。与动物中环状RNA一样,植物中的环状RNA也会由于选择性反向剪接及选择性剪接而产生多种类型[141]。并且,植物中的环状RNA也具有保守性[114]、发育阶段特异性[114]和物种特异性[142]。
在动物中的大多数研究表明环状RNA的形成与内含子反向互补序列、RNA结合蛋白、碱基配对的稳定性和侧翼内含子的长度等密切相关[17,22,27]。植物中环状RNA的形成与动物是有相似之处的。在玉米中发现,可环化外显子的侧翼内含子上的反向互补序列参与环状RNA的形成[135,136]。在水稻和拟南芥中发现可环化外显子的侧翼内含子序列普遍长于非可环化外显子的侧翼内含子序列[93,114],说明长侧翼内含子也是促进植物环状RNA形成的因素。然而,研究发现影响环状RNA形成的重复序列及反向互补序列在水稻和拟南芥中都是非常少量的[93,114,134],这说明在植物中除了内含子反向互补序列外,可能还有其他的因素影响植物环状RNA的形成。另外,在水稻中鉴别到将近2806个全长环状RNA,但仅有206个拥有经典的GU/AG剪接信号[45],说明在水稻中大多数环状RNA的形成可能并不依赖于经典的GU/AG剪接信号。
4.2 环状RNA在植物发育过程中的作用
环状RNA对生物体的生长发育研究大多数集中在动物上,随着环状RNA研究的深入,植物环状RNA的研究也越来越多。Chen等[135]在玉米中发现,重复序列LLEs (LINE1-like element)与其反向互补配对序列在环状RNA侧翼序列中显著富集,并且发现这些序列与circ1690的表达紧密相关,并且circ1690通过与线性RNA相互作用可影响玉米的穗高。当玉米和拟南芥在受到不同程度的干旱胁迫时,环状RNA发生了不同程度的变化,说明环状RNA可以作为植物干旱响应的有效分子标记信号[136]。另外,研究者在拟南芥中过表达circGORK,发现转基因植株的种子萌发对脱落酸(ABA)超敏感,且植株表现出更加耐旱的表型,这为环状RNA直接调控干旱胁迫应答提供了有力证据[136]。植物环状RNA不仅能在干旱胁迫下起作用,而且Ye等[114]发现水稻中的环状RNA与其宿主基因能协作响应磷胁迫。随着番茄的成熟,反向剪接位点数量逐渐上升,并且过表达circPSY1使得番茄红素和β-胡萝卜素降低,说明环状RNA对果实成熟也有潜在的功能[141]。Zhou等[142]在马铃薯细菌性软腐病(,)研究中发现,在mRNA、环状RNA及lincRNA (long intergenic non-coding RNA)之间存在一定的共表达关系,说明环状RNA在植物体的疾病响应过程中对转录组重编程发挥着一定的作用。相信随着研究的深入化,更多的植物环状RNA及其功能将被揭示。
5 结语与展望
得益于二代高通量测序技术的不断改进(图3),环状RNA的神秘面纱逐渐被揭开。目前检测环状RNA普遍采用二代短片段高通量测序方法,如需要获得全长的环状RNA,研究者需要根据短片段进行RNA组装,然而组装是个易错过程,并存在线性转录本的污染问题,所以开发基于Pacific BioSciences (PacBio)和Oxford Nanopore Technologies (ONT)的环状RNA长片段测序方法是未来研究复杂环状RNA的关键技术。获得全长环状RNA将打开研究其内部复杂选择性剪接类型的大门,更多环状RNA的调控功能将被揭示。
与环状RNA形成机制研究相比较,环状RNA如何被降解的相关报道十分匮乏。Hansen等[104]发现Cdrlas上有miR-671的互补序列,Cdr1as通过吸附miR-671而导致了Ago2介导的Cdr1as的降解,那么其他的环状RNA是否也以相同的机制被降解以维持环状RNA在生物体内的表达平衡?常规的降解组测序只适用于线性转录本,需要开发针对环状RNA的特异降解组文库构建方法来大规模解析环状RNA的降解机制。m6A修饰的环状RNA会影响相应宿主基因的稳定性[125],那么m6A修饰是否会影响环状RNA自身的稳定性?这些都是环状RNA降解需要解决的问题。但目前环状RNA的修饰检测仍然处于瓶颈之中,已有报道用基于m6A抗体免疫沉淀(immunoprecipitation, IP)后深度测序的方法来检测环状RNA修饰[129],通过反向剪接位点来识别环状RNA的序列,但是由于线性转录本很难用RNase R去除完全,所以如何识别环状RNA反向剪接位点外的序列修饰依然是一个急需解决的问题。
在环状RNA的功能方面,环状RNA在生物体中动态表达,与miRNA、mRNA及lncRNA等形成互作网络,参与调控宿主基因的转录及转录后表达,环状RNA并非错误剪接的副产物,而是具有重要调控功能的RNA分子[22]。目前大部分研究集中于环状RNA的海绵体功能,而其翻译功能是一个重要的被忽视的方向。现有的大部分研究是基于预测的方法来研究环状RNA的翻译功能,但在准确性上还有待进一步提高。基于蛋白质组学的方法将是未来研究环状RNA的高效而准确的技术手段,通过构建反向剪接位点的特征氨基酸数据库,重新进行蛋白搜库,可能会发现非常有价值的环状RNA翻译调控问题。
尽管环状RNA在生物体的不同组织或细胞中都广泛存在,但事实上形成环状RNA的反向剪接效率是远低于经典线性剪接的[26,31,95,130],导致大多数环状RNA的表达丰度较线性基因低,然而细胞是通过何种信号及何种方式调节同一基因座上的环状RNA与线性转录本的比例仍需进一步地研究;并且,选择性反向剪接会形成不同的环状RNA异构体[72],但是何种类别的剪接因子通过特异性结合何种类别的基序来进行精准剪接形成环状RNA的仍需进一步地研究。随着多组学的结合使用,相信在将来人们对表观遗传学所涉及的长链环状RNA会有全新的认知。
[1] Jeck WR, Sorrentino JA, Wang K, Slevin MK, Burd CE, Liu J, Marzluff WF, Sharpless NE. Circular RNAs are abundant, conserved, and associated with ALU repeats., 2013, 19(2): 426–426.
[2] Sanger HL, Klotz G, Riesner D, Gross HJ, Kleinschmidt AK. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures., 1976, 73(11): 3852–3856.
[3] Hsu MT, Coca-Prados M. Electron microscopic evidence for the circular form of RNA in the cytoplasm of eukaryotic cells., 1979, 280(5720): 339–340.
[4] Arnberg AC, Van Ommen GJ, Grivell LA, Van Bruggen EF, Borst P. Some yeast mitochondrial RNAs are circular., 1980, 19(2): 313–319.
[5] Grabowski PJ, Zaug AJ, Cech TR. The intervening sequence of the ribosomal RNA precursor is converted to a circular RNA in isolated nuclei of tetrahymena., 1981, 23(2): 467–476.
[6] Kjems J, Garrett RA. Novel splicing mechanism for the ribosomal RNA intron in the archaebacterium desulfurococcus mobilis., 1988, 54(5): 693–703.
[7] Nigro JM, Cho K, Fearon ER, Kern SE, Ruppert JM, Oliner JD, Kinzler KW, Vogelstein B. Scrambled exons., 1991, 64(3): 607–613.
[8] Cocquerelle C, Daubersies P, Majérus MA, Kerckaert JP, Bailleul B. Splicing with inverted order of exons occurs proximal to large introns., 1992, 11(3): 1095– 1098.
[9] Cocquerelle C, Mascrez B, Hétuin D, Bailleul B. Mis-splicing yields circular RNA molecules., 1993, 7(1): 155–160.
[10] Capel B, Swain A, Nicolis S, Hacker A, Walter M, Koopman P, Goodfellow P, Lovell-Badge R. Circular transcripts of the testis-determining gene sry in adult mouse testis., 1993, 73(5): 1019–1030.
[11] Zaphiropoulos PG. Circular RNAs from transcripts of the rat cytochromegene: correlation with exon skipping., 1996, 93(13): 6536–6541.
[12] Zaphiropoulos PG. Exon skipping and circular RNA formation in transcripts of the human cytochromegene in epidermis and of the rat androgen binding protein gene in testis., 1997, 17(6): 2985– 2993.
[13] Burd CE, Jeck WR, Liu Y, Sanoff HK, Wang Z, Sharpless NE. Expression of linear and novel circular forms of an-associated non-coding RNA correlates with atherosclerosis risk., 2010, 6(12): e1001233.
[14] Luo J, Wang XL, Sun ZC, Wu D, Zhang W, Wang ZJ. Progress in circular RNAs of plants., 2018, 40(6): 467–477.骆甲, 王型力, 孙志超, 吴迪, 张玮, 王正加. 植物环状RNA研究进展. 遗传, 2018, 40(6): 467–477.
[15] Salzman J, Gawad C, Wang PL, Lacayo N, Brown PO. Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types., 2012, 7(2): e30733.
[16] Hansen TB, Venø MT, Damgaard CK, Kjems J. Comparison of circular RNA prediction tools., 2015, 44(6): e58.
[17] Li Z, Huang C, Bao C, Chen L, Lin M, Wang X, Zhong G, Yu B, Hu W, Dai L, Zhu P, Chang Z, Wu Q, Zhao Y, Jia Y, Xu P, Liu H, Shan G. Exon-intron circular RNAs regulate transcription in the nucleus., 2015, 22(3): 256–264.
[18] Zhang Y, Zhang XO, Chen T, Xiang JF, Yin QF, Xing YH, Zhu S, Yang L, Chen LL. Circular intronic long noncoding RNAs., 2013, 51(6): 792–806.
[19] Guarnerio J, Bezzi M, Jeong JC, Paffenholz SV, Berry K, Naldini MM, Lo-Coco F, Tay Y, Beck AH, Pandolfi PP. Oncogenic role of fusion-circRNAs derived from cancer- associated chromosomal translocations., 2016, 165(2): 289–302.
[20] Tan S, Sun D, Pu W, Gou Q, Guo C, Gong Y, Li J, Wei YQ, Liu L, Zhao Y. Circular RNA F-circEA-2a derived fromfusion gene promotes cell migration and invasion in non-small cell lung cancer., 2018, 17(1):138.
[21] Vo JN, Cieslik M, Zhang Y, Shukla S, Xiao L, Zhang Y, Wu YM, Dhanasekaran SM, Engelke CG, Cao X, Robinson DR, Nesvizhskii AI, Chinnaiyan AM. The landscape of circular RNA in cancer., 2019, 176(4): 869–881.e13.
[22] Li X, Yang L, Chen LL. The biogenesis, functions, and challenges of circular RNAs., 2018, 71(3): 428–442.
[23] Chen LL, Yang L. Regulation of circRNA biogenesis., 2015, 12(4): 381–388.
[24] Liang D, Wilusz JE. Short intronic repeat sequences facilitate circular RNA production., 2014, 28(20): 2233–2247.
[25] Ivanov A, Memczak S, Wyler E, Torti F, Porath HT, Orejuela MR, Piechotta M, Levanon EY, Landthaler M, Dieterich C, Rajewsky N. Analysis of intron sequences reveals hallmarks of circular RNA biogenesis in animals., 2014, 10(2): 170–177.
[26] Zhang Y, Xue W, Li X, Zhang J, Chen S, Zhang JL, Yang L, Chen LL. The biogenesis of nascent circular RNAs., 2016, 15(3): 611–624.
[27] Zhang XO, Wang HB, Zhang Y, Lu X, Chen LL, Yang L. Complementary sequence-mediated exon circularization., 2014, 159(1): 134–147.
[28] Dong R, Ma XK, Chen LL, Yang L. Increased complexity of circRNA expression during species evolution., 2016, 14(8): 1064–1074.
[29] Hafner M, Landthaler M, Burger L, Khorshid M, Hausser J, Berninger P, Rothballer A, Ascano M Jr, Jungkamp A, Munschauer M, Ulrich A, Wardle GS, Dewell S, Zavolan M, Tuschl T. Transcriptome-wide identification of RNA-binding protein and microRNA target sites by PAR-CLIP., 2010, 141(1): 129–141.
[30] Ji P, Wu W, Chen S, Zheng Y, Zhou L, Zhang J, Cheng H, Yan J, Zhang S, Yang P, Zhao F. Expanded expressionlandscape and prioritization of circular RNAs in mammals., 2019, 26(12): 3444–3460.e5.
[31] Ashwal-Fluss R, Meyer M, Pamudurti NR, Ivanov A, Bartok O, Hanan M, Evantal N, Memczak S, Rajewsky N, Kadener S. CircRNA biogenesis competes with pre-mRNA splicing., 2014, 56(1): 55–66.
[32] Duchange N, Pidoux J, Camus E, Sauvaget D. Alternative splicing in the human interleukin enhancer binding factor 3 (ILF3) gene., 2000, 261(2): 345–353.
[33] Urcuqui-Inchima S, Castaño ME, Hernandez-Verdun D, St-Laurent G, Kumar A. Nuclear factor 90, a cellular dsRNA binding protein inhibits the HIV Rev-export function., 2006, 3: 83.
[34] Li X, Liu CX, Xue W, Zhang Y, Jiang S, Yin QF, Wei J, Yao RW, Yang L, Chen LL. Coordinated circRNA biogenesis and function with NF90/NF110 in viral infection., 2017, 67(2): 214–227.e7.
[35] Fei T, Chen Y, Xiao T, Li W, Cato L, Zhang P, Cotter MB, Bowden M, Lis RT, Zhao SG, Wu Q, Feng FY, Loda M, He HH, Liu XS, Brown M. Genome-wide CRISPR screen identifies HNRNPL as a prostate cancer dependency regulating RNA splicing., 2017, 114(26): E5207–E5215.
[36] Errichelli L, Dini Modigliani S, Laneve P, Colantoni A, Legnini I, Capauto D, Rosa A, De Santis R, Scarfò R, Peruzzi G, Lu L, Caffarelli E, Shneider NA, Morlando M, Bozzoni I. FUS affects circular RNA expression in murine embryonic stem cell-derived motor neurons., 2017, 8: 14741.
[37] Conn SJ, Pillman KA, Toubia J, Conn VM, Salmanidis M, Phillips CA, Roslan S, Schreiber AW, Gregory PA, Goodall GJ. The RNA binding protein quaking regulates formation of circRNAs., 2015, 160(6): 1125–1134.
[38] Teplova M, Hafner M, Teplov D, Essig K, Tuschl T, Patel DJ. Structure-function studies of STAR family Quaking proteins bound to theirRNA target sites., 2013, 27(8): 928–940.
[39] Khan MA, Reckman YJ, Aufiero S, van den Hoogenhof MM, van der Made I, Beqqali A, Koolbergen DR, Rasmussen TB, van der Velden J, Creemers EE, Pinto YM. RBM20 regulates circular RNA production from the titin gene., 2016, 119(9): 996–1003.
[40] Kramer MC, Liang D, Tatomer DC, Gold B, March ZM, Cherry S, Wilusz JE. Combinatorial control ofcircular RNA expression by intronic repeats, hnRNPs, and SR proteins., 2015, 29(20): 2168–2182.
[41] Nishikura K. Functions and regulation of RNA editing by ADAR deaminases., 2010, 79: 321–349.
[42] Kim DD, Kim TT, Walsh T, Kobayashi Y, Matise TC, Buyske S, Gabriel A. Widespread RNA editing of embedded alu elements in the human transcriptome., 2004, 14(9): 1719–1725.
[43] Rybak-Wolf A, Stottmeister C, Glažar P, Jens M, Pino N, Giusti S, Hanan M, Behm M, Bartok O, Ashwal-Fluss R, Herzog M, Schreyer L, Papavasileiou P, Ivanov A, Öhman M, Refojo D, Kadener S, Rajewsky N. Circular RNAs in the mammalian brain are highly abundant, conserved, and dynamically expressed., 2015, 58(5): 870–885.
[44] Aktaş T, Avşar Ilık İ, Maticzka D, Bhardwaj V, Pessoa Rodrigues C, Mittler G, Manke T, Backofen R, Akhtar A. DHX9 suppresses RNA processing defects originating from the Alu invasion of the human genome., 2017, 544(7648): 115–119.
[45] Ye CY, Zhang X, Chu Q, Liu C, Yu Y, Jiang W, Zhu QH, Fan L, Guo L. Full-length sequence assembly reveals circular RNAs with diverse non-GT/AG splicing signals in rice., 2016, 14(8): 1055–1063.
[46] Panda AC, De S, Grammatikakis I, Munk R, Yang X, Piao Y, Dudekula DB, Abdelmohsen K, Gorospe M. High-purity circular RNA isolation method (RPAD) reveals vast collection of intronic circRNAs., 2017, 45(12): e116.
[47] Talhouarne GJS, Gall JG. Lariat intronic RNAs in the cytoplasm of vertebrate cells., 2018, 115(34): E7970–E7977.
[48] Zhang X, Zhang Y, Wang T, Li Z, Cheng J, Ge H, Tang Q, Chen K, Liu L, Lu C, Guo J, Zheng B, Zheng Y. A comprehensive map of intron branchpoints and lariat RNAs in plants., 2019, 31(5): 956–973.
[49] Bentley DL. Coupling mRNA processing with transcription in time and space., 2014, 15(3): 163–175.
[50] Liang D, Tatomer DC, Luo Z, Wu H, Yang L, Chen LL, Cherry S, Wilusz JE. The output of protein-coding genes shifts to circular RNAs when the pre-mRNA processing machinery is limiting., 2017, 68(5): 940–954.e3.
[51] Abascal-Palacios G, Ramsay EP, Beuron F, Morris E, Vannini A. Structural basis of RNA polymerase Ⅲ transcription initiation., 2018, 553(7688): 301– 306.
[52] Schmidt CA, Noto JJ, Filonov GS, Matera AG. A method for expressing and imaging abundant, stable, circular RNAsusing tRNA splicing., 2016, 572: 215–236.
[53] Litke JL, Jaffrey SR. Highly efficient expression of circular RNA aptamers in cells using autocatalytic transcripts., 2019, doi: 10.1038/s41587- 019-0090-6.
[54] Salgia SR. Two reactions of Haloferax volcanii RNA splicing enzymes: joining of exons and circularization of introns., 2003, 9(3): 319–330.
[55] Englert M, Sheppard K, Gundllapalli S, Beier H Sölla D. Branchiostoma floridae has separate healing and sealing enzymes for 5'-phosphate RNA ligation., 2010, 107(39): 16834–16839.
[56] Noto JJ, Schmidt CA, Matera AG. Engineering and expressing circular RNAs via tRNA splicing., 2017, 14(8): 978–984.
[57] Szabo L, Salzman J. Detecting circular RNAs: bioinformatic and experimental challenges., 2016, 17(11): 679–692.
[58] Vincent HA, Deutscher MP. Substrate recognition and catalysis by the exoribonuclease RNase R., 2006, 281(40): 29769–29775.
[59] Chen S, Huang V, Xu X, Livingstone J, Soares F, Jeon J, Zeng Y, Hua JT, Petricca J, Guo H, Wang M, Yousif F, Zhang Y, Donmez N, Ahmed M, Volik S, Lapuk A, Chua MLK, Heisler LE, Foucal A, Fox NS, Fraser M, Bhandari V, Shiah YJ, Guan J, Li J, Orain M, Picard V, Hovington H, Bergeron A, Lacombe L, Fradet Y, Têtu B, Liu S, Feng F, Wu X, Shao YW, Komor MA, Sahinalp C, Collins C, Hoogstrate Y, de Jong M, Fijneman RJA, Fei T, Jenster G, van der Kwast T, Bristow RG, Boutros PC, He HH. Widespread and functional RNA circularization in localized prostate cancer., 2019, 176(4): 831–843.e22.
[60] Pandey PR, Rout PK, Das A, Gorospe M, Panda AC. RPAD (RNase R treatment, polyadenylation, and poly(A)+RNA depletion) method to isolate highly pure circular RNA., 2018, 155: 41–48.
[61] Jakobi T, Dieterich C. Computational approaches for circular RNA analysis., 2019, 10(3): e1528.
[62] Hansen TB. Improved circRNA identification by combining prediction algorithms., 2018, 6: 20.
[63] Zheng Y, Ji P, Chen S, Hou L, Zhao F. Reconstruction of full-length circular RNAs enables isoform-level quantification., 2019, 11(1): 2.
[64] Feng J, Chen K, Dong X, Xu X, Jin Y, Zhang X, Chen W, Han Y, Shao L, Gao Y, He C. Genome-wide identification of cancer-specific alternative splicing in circRNA., 2019, 18(1): 35.
[65] Sharma D, Sehgal P, Hariprakash J, Sivasubbu S, Scaria V. Methods for annotation and validation of circular RNAs from RNAseq data., 2019, 1912: 55–76.
[66] Wang K, Singh D, Zeng Z, Coleman SJ, Huang Y, Savich GL, He X, Mieczkowski P, Grimm SA, Perou CM, Macleod JN, Chiang DY, Prins JF, Liu J. MapSplice: accurate mapping of RNA-seq reads for splice junction discovery., 2010, 38(18): e178.
[67] Chen L, Yu Y, Zhang X, Liu C, Ye C, Fan L. PcircRNA_finder: a software for circRNA prediction in plants., 2016, 32(22): 3528–3529.
[68] Memczak S, Jens M, Elefsinioti A, Torti F, Krueger J, Rybak A, Maier L, Mackowiak SD, Gregersen LH, Munschauer M, Loewer A, Ziebold U, Landthaler M, Kocks C, le Noble F, Rajewsky N. Circular RNAs are a large class of animal RNAs with regulatory potency., 2013, 495(7441): 333–338.
[69] Metge F, Czaja-Hasse LF, Reinhardt R, Dieterich C. FUCHS-towards full circular RNA characterization using RNAseq., 2017, 5: e2934.
[70] Westholm JO, Miura P, Olson S, Shenker S, Joseph B, Sanfilippo P, Celniker SE, Graveley BR, Lai EC. Genome-wide analysis of drosophila circular RNAs reveals their structural and sequence properties and age-dependent neural accumulation., 2014, 9(5): 1966–1980.
[71] Hoffmann S, Otto C, Doose G, Tanzer A, Langenberger D, Christ S, Kunz M, Holdt LM, Teupser D, Hackermüller J. A multi-split mapping algorithm for circular RNA, splicing, trans-splicing and fusion detection., 2014, 15(2): R34.
[72] Zhang XO, Dong R, Zhang Y, Zhang JL, Luo Z, Zhang J, Chen LL, Yang L. Diverse alternative back-splicing and alternative splicing landscape of circular RNAs., 2016, 26(9): 1277–1287.
[73] Szabo L, Morey R, Palpant NJ, Wang PL, Afari N, Jiang C, Parast MM, Murry CE, Laurent LC, Salzman J. Statistically based splicing detection reveals neural enrichment and tissue-specific induction of circular RNA during human fetal development., 2015, 16: 126.
[74] Gao Y, Zhang J, Zhao F. Circular RNA identification based on multiple seed matching., 2018, 19(5): 803–810.
[75] Cheng J, Metge F, Dieterich C. Specific identification and quantification of circular RNAs from sequencing data., 2016, 32(7): 1094–1096.
[76] Feng J, Xiang Y, Xia S, Liu H, Wang J, Ozguc FM, Lei L, Kong R, Diao L, He C, Han L. Circview: a visualization and exploration tool for circular RNAs., 2018, 19(6): 1310–1316.
[77] You X, Conrad TO. Acfs: accurate circRNA identification and quantification from RNA-Seq data., 2016, 6(1): 38820.
[78] Gao Y, Wang H, Zhang H, Wang Y, Chen J, Gu L. PRAPI: post-transcriptional regulation analysis pipeline for Iso-Seq., 2018, 34(9): 1580–1582.
[79] Song X, Zhang N, Han P, Moon BS, Lai RK, Wang K, Lu W. Circular RNA profile in gliomas revealed by identification tool UROBORUS., 2016, 44(9): e87.
[80] Wang H, Wang H, Zhang H, Liu S, Wang Y, Gao Y, Xi F, Zhao L, Liu B, Reddy ASN, Lin C, Gu L. The interplay between microRNA and alternative splicing of linear and circular RNAs in eleven plant species., 2019, doi: 10.1093/bioinformatics/btz038.
[81] Xia S, Feng J, Chen K, Ma Y, Gong J, Cai F, Jin Y, Gao Y, Xia L, Chang H, Wei L, Han L, He C. CSCD: a database for cancer-specific circular RNAs., 2018, 46(D1): D925–D929.
[82] Glažar P, Papavasileiou P, Rajewsky N. CircBase: a database for circular RNAs., 2014, 20(11): 1666– 1670.
[83] Fan C, Lei X, Fang Z, Jiang Q, Wu FX. CircR2Disease: a manually curated database for experimentally supported circular RNAs associated with various diseases., 2018, 2018: bay044.
[84] Xia S, Feng J, Lei L, Hu J, Xia L, Wang J, Xiang Y, Liu L, Zhong S, Han L, He C. Comprehensive characterization of tissue-specific circular RNAs in the human and mouse genomes., 2017, 18(6): 984–992.
[85] Ye J, Wang L, Li S, Zhang Q, Zhang Q, Tang W, Wang K, Song K, Sablok G, Sun X, Zhao H. AtCircDB: a tissue-specific database for arabidopsis circular RNAs., 2019, 20(1): 58–65.
[86] Ghosal S, Das S, Sen R, Basak P, Chakrabarti J. Circ2Traits: a comprehensive database for circular RNA potentially associated with disease and traits., 2013, 4: 283.
[87] Zhang P, Meng X, Chen H, Liu Y, Xue J, Zhou Y, Chen M. PlantCircNet: a database for plant circRNA–miRNA– mRNA regulatory networks.,2017, 2017: bax089.
[88] Dudekula DB, Panda AC, Grammatikakis I, De S, Abdelmohsen K, Gorospe M. CircInteractome: a web tool for exploring circular RNAs and their interacting proteins and microRNAs., 2016, 13(1): 34–42.
[89] Chu Q, Zhang X, Zhu X, Liu C, Mao L, Ye C, Zhu QH, Fan L. PlantcircBase: a database for plant circular RNAs., 2017, 10(8): 1126–1128.
[90] Liu YC, Li JR, Sun CH, Andrews E, Chao RF, Lin FM, Weng SL, Hsu SD, Huang CC, Cheng C, Liu CC, Huang HD. CircNet: a database of circular RNAs derived from transcriptome sequencing data., 2015, 44(D1): D209–D215.
[91] Meng X, Hu D, Zhang P, Chen Q, Chen M. CircFunBase: a database for functional circular RNAs., 2019, 2019: baz003.
[92] Chen X, Han P, Zhou T, Guo X, Song X, Li Y. CircRNADb: a comprehensive database for human circular RNAs with protein-coding annotations., 2016, 6: 34985.
[93] Lu T, Cui L, Zhou Y, Zhu C, Fan D, Gong H, Zhao Q, Zhou C, Zhao Y, Lu D, Luo J, Wang Y, Tian Q, Feng Q, Huang T, Han B. Transcriptome-wide investigation of circular RNAs in rice., 2015, 21(12): 2076–2087.
[94] Suzuki H, Zuo Y, Wang J, Zhang MQ, Malhotra A, Mayeda A. Characterization of RNase R-digested cellular RNA source that consists of lariat and circular RNAs from pre-mRNA splicing., 2006, 34(8): e63.
[95] Starke S, Jost I, Rossbach O, Schneider T, Schreiner S, Hung LH, Bindereif A. Exon circularization requires canonical splice signals., 2014, 10(1): 103– 111.
[96] Awan AR, Manfredo A, Pleiss JA. Lariat sequencing in a unicellular yeast identifies regulated alternative splicing of exons that are evolutionarily conserved with humans., 2013, 110(31): 12762–12767.
[97] Ghorbani A, Izadpanah K, Peters JR, Dietzgen RG, Mitter N. Detection and profiling of circular RNAs in uninfected and maize Iranian mosaic virus-infected maize., 2018, 274: 402–409.
[98] Cong L, Zhang F. Genome engineering using CRISPR- Cas9 system., 2015, 1239: 197–217.
[99] Wang H, Yang H, Shivalila CS, Dawlaty MM, Cheng AW, Zhang F, Jaenisch R. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas- mediated genome engineering., 2013, 153(4): 910– 918.
[100] Feng Z, Zhang B, Ding W, Liu X, Yang DL, Wei P, Cao F, Zhu S, Zhang F, Mao Y, Zhu JK. Efficient genome editing in plants using a CRISPR/Cas system., 2013, 23(10): 1229–1232.
[101] Wang Y, Huang J, Xu R. Seamless genome editing inby combining CRISPR/Cas9 and piggyBac technologies., 2019, 41 (5): 422– 429.王珏, 黄娟, 许蕊. 利用CRISPR/Cas9和piggyBac实现果蝇基因组无缝编辑. 遗传, 2019, 41 (5): 422–429.
[102] Piwecka M, Glažar P, Hernandez-Miranda LR, Memczak S, Wolf SA, Rybak-Wolf A, Filipchyk A, Klironomos F, Cerda Jara CA, Fenske P, Trimbuch T, Zywitza V, Plass M, Schreyer L, Ayoub S, Kocks C, Kühn R, Rosenmund C, Birchmeier C, Rajewsky N. Loss of a mammalian circular RNA locus causes miRNA deregulation and affects brain function., 2017, 357(6357): eaam8526.
[103] Borchardt EK, Meganck RM, Vincent HA, Ball CB, Ramos SBV, Moorman NJ, Marzluff WF, Asokan A. Inducing circular RNA formation using the CRISPR endoribonuclease Csy4., 2017, 23(5): 619–627.
[104] Hansen TB, Wiklund ED, Bramsen JB, Villadsen SB, Statham AL, Clark SJ, Kjems J. MiRNA-dependent gene silencing involving Ago2-mediated cleavage of a circular antisense RNA., 2011, 30(21): 4414– 4422.
[105] O’keefe E. SiRNAs and shRNAs: tools for protein knockdown by gene silencing., 2013, 3: 197.
[106] Legnini I, Di Timoteo G, Rossi F, Morlando M, Briganti F, Sthandier O, Fatica A, Santini T, Andronache A, Wade M, Laneve P, Rajewsky N, Bozzoni I. Circ-ZNF609 is a circular RNA that can be translated and functions in myogenesis., 2017, 66(1): 22–37.e9.
[107] Zhu P, Zhu X, Wu J, He L, Lu T, Wang Y, Liu B, Ye B, Sun L, Fan D, Wang J, Yang L, Qin X, Du Y, Li C, He L, Ren W, Wu X, Tian Y, Fan Z. IL-13 secreted by ILC2s promotes the self-renewal of intestinal stem cells through circular RNA circPan3., 2019, 20(2): 183–194.
[108] Salzman J, Chen RE, Olsen MN, Wang PL, Brown PO. Cell-type specific features of circular RNA expression., 2013, 9(9): e1003777.
[109] Guo JU, Agarwal V, Guo H, Bartel DP. Expanded identification and characterization of mammalian circular RNAs., 2014, 15(7): 409.
[110] Hansen TB, Jensen TI, Clausen BH, Bramsen JB, Finsen B, Damgaard CK, Kjems J. Natural RNA circles function as efficient microRNA sponges., 2013, 495(7441): 384–388.
[111] Su H, Tao T, Yang Z, Kang X, Zhang X, Kang D, Wu S, Li C. Circular RNA cTFRC acts as the sponge of MicroRNA-107 to promote bladder carcinoma progression., 2019, 18(1): 27.
[112] Rong D, Lu C, Zhang B, Fu K, Zhao S, Tang W, Cao H. CircPSMC3 suppresses the proliferation and metastasis of gastric cancer by acting as a competitive endogenous RNA through sponging miR-296-5p., 2019, 18(1): 25.
[113] Xu L, Feng X, Hao X, Wang P, Zhang Y, Zheng X, Li L, Ren S, Zhang M, Xu M. CircSETD3 (Hsa_circ_0000567) acts as a sponge for microRNA-421 inhibiting hepatocellular carcinoma growth., 2019, 38(1): 98.
[114] Ye CY, Chen L, Liu C, Zhu QH, Fan L. Widespread noncoding circular RNAs in plants., 2015, 208(1): 88–95.
[115] Wang Y, Yang M, Wei S, Qin F, Zhao H, Suo B. Identification of circular RNAs and their targets in leaves ofLunder dehydration stress., 2016, 7: 2024.
[116] Yin J, Liu M, Ma D, Wu J, Li S, Zhu Y, Han B. Identification of circular RNAs and their targets during tomato fruit ripening., 2018, 136: 90–98.
[117] Zeng RF, Zhou JJ, Hu CG, Zhang JZ. Transcriptome- wide identification and functional prediction of novel and flowering-related circular RNAs from trifoliate orange (L. Raf.)., 2018, 247(5): 1191–1202.
[118] Abdelmohsen K, Panda AC, Munk R, Grammatikakis I, Dudekula DB, De S, Kim J, Noh JH, Kim KM, Martindale JL, Gorospe M . Identification of HuR target circular RNAs uncovers suppression oftranslation by circPABPN1., 2017, 14(3): 361–369.
[119] Du WW, Fang L, Yang W, Wu N, Awan FM, Yang Z, Yang BB. Induction of tumor apoptosis through a circular RNA enhancing Foxo3 activity., 2016, 24(2): 357.
[120] Park OH, Ha H, Lee Y, Boo SH, Kwon DH, Song HK, Kim YK. Endoribonucleolytic cleavage of m6A-containing RNAs by RNase P/MRP complex., 2019, 74(3): 494–507.e8.
[121] Li Z, Wang S, Cheng J, Su C, Zhong S, Liu Q, Fang Y, Yu Y, Lv H, Zheng Y, Zheng B. Intron lariat RNA inhibits microRNA biogenesis by sequestering the dicing complex in., 2016, 12(11): e1006422.
[122] Conn VM, Hugouvieux V, Nayak A, Conos SA, Capovilla G, Cildir G, Jourdain A, Tergaonkar V, Schmid M, Zubieta C, Conn SJ. A circRNA fromregulates splicing of its cognate mRNA through R-loop formation., 2017, 3: 17053.
[123] Wang Y, Gao Y, Zhang H, Wang H, Liu X, Xu X, Zhang Z, Markus VK, Kaiqiang H, Wang H, Xi F, Zhao L, Lin C, Gu L. Genome-wide profiling of circular RNAs in the rapidly growing shoots of moso bamboo ()., 2019,doi: 10.1093/pcp/ pcz043.
[124] Roundtree IA, Evans ME, Pan T, He C. Dynamic RNA modifications in gene expression regulation., 2017, 169(7): 1187–1200.
[125] Zhou C, Molinie B, Daneshvar K, Pondick JV, Wang J, Van Wittenberghe N, Xing Y, Giallourakis CC, Mullen AC. Genome-wide maps of m6A circRNAs identify widespread and cell-type-specific methylation patterns that are distinct from mRNAs., 2017, 20(9): 2262–2276.
[126] Chen N, Zhao G, Yan X, Lv Z, Yin H, Zhang S, Song W, Li X, Li L, Du Z, Jia L, Zhou L, Li W, Hoffman AR, Hu JF, Cui J. A novelexonic circular RNA promotes metastasis in breast cancer by coordinately regulating TET1 and DNMT1., 2018, 19(1): 218.
[127] Wei LH, Song P, Wang Y, Lu Z, Tang Q, Yu Q, Xiao Y, Zhang X, Duan HC, Jia G. The m6A reader ECT2 controls trichome morphology by affecting mRNA stability in., 2018, 30(5): 968–985.
[128] Pamudurti NR, Bartok O, Jens M, Ashwal-Fluss R, Stottmeister C, Ruhe L, Hanan M, Wyler E, Perez- Hernandez D, Ramberger E, Shenzis S, Samson M, Dittmar G, Landthaler M, Chekulaeva M, Rajewsky N, Kadener S. Translation of circRNAs., 2017, 66(1): 9–21.e7.
[129] Yang Y, Fan X, Mao M, Song X, Wu P, Zhang Y, Jin Y, Yang Y, Chen LL, Wang Y, Wong CC, Xiao X, Wang Z. Extensive translation of circular RNAs driven by N6-methyladenosine., 2017, 27(5): 626–641.
[130] Wang Y, Wang Z. Efficient backsplicing produces translatable circular mRNAs., 2014, 21(2): 172– 179.
[131] Zhang M, Zhao K, Xu X, Yang Y, Yan S, Wei P, Liu H, Xu J, Xiao F, Zhou H, Yang X, Huang N, Liu J, He K, Xie K, Zhang G, Huang S, Zhang N. A peptide encoded by circular form of LINC-PINT suppresses oncogenic transcriptional elongation in glioblastoma., 2018, 9(1): 4475.
[132] Wesselhoeft RA, Kowalski PS, Parker-Hale FC, Huang Y, Bisaria N, Anderson DG. RNA circularization diminishes immunogenicity and can extend translation duration., 2019, 74(3): 508–529.e4.
[133] Dong R, Zhang XO, Zhang Y, Ma XK, Chen LL, Yang L. CircRNA-derived pseudogenes., 2016, 26(6): 747–750.
[134] Sun X, Wang L, Ding J, Wang Y, Wang J, Zhang X, Che Y, Liu Z, Zhang X, Ye J, Wang J, Sablok G, Deng Z. Integrative analysis oftranscriptomics reveals intuitive splicing mechanism for circular RNA., 2016, 590(20): 3510–3516.
[135] Chen L, Zhang P, Fan Y, Lu Q, Li Q, Yan J, Muehlbauer GJ, Schnable PS, Dai M, Li L. Circular RNAs mediated by transposons are associated with transcriptomic and phenotypic variation in maize., 2017, 217(3): 1292–1306.
[136] Zhang P, Fan Y, Sun X, Chen L, Terzaghi W, Bucher E, Li L, Dai M. A large-scale circular RNA profiling reveals universal molecular mechanisms responsive to drought stress in maize and., 2019, 98(4): 697–713.
[137] Zuo J, Wang Q, Zhu B, Luo Y, Gao L. Deciphering the roles of circRNAs on chilling injury in tomato., 2016, 479(2): 132–138.
[138] Darbani B, Noeparvar S, Borg S. Identification of circular RNAs from the parental genes involved in multiple aspects of cellular metabolism in barley., 2016, 7: 776.
[139] Wang Z, Liu Y, Li D, Li L, Zhang Q, Wang S, Huang H. Identification of circular RNAs in kiwifruit and their species-specific response to bacterial canker pathogen invasion., 2017, 8: 413.
[140] Zhang G, Duan A, Zhang J, He C. Genome-wide analysis of long non-coding RNAs at the mature stage of sea buckthorn () fruit., 2016, 596: 130–136.
[141] Tan J, Zhou Z, Niu Y, Sun X, Deng Z. Identification and functional characterization of tomato circRNAs derived from genes involved in fruit pigment accumulation., 2017, 7(1): 8594.
[142] Zhou R, Zhu Y, Zhao J, Fang Z, Wang S, Yin J, Chu Z, Ma D. Transcriptome-wide identification and characterization of potato circular RNAs in response to pectobacterium carotovorum subspecies brasiliense infection., 2018, 19(1): 71.
Biogenesis, research methods, and functions of circular RNAs
Xuqing Liu1, Yubang Gao1,2, Liangzhen Zhao1, Yuchen Cai1, Huiyuan Wang1, Miao Miao1, Lianfeng Gu1, Hangxiao Zhang1
The field of circular non-coding RNAs have been gradually attracted wide attention with the developments of high-throughput sequencing. In this review, we systematically summarize three driving models for circRNAs biogenesis: intron-pairing-driven, RNA binding protein-driven and lariat-driven. In addition, we also briefly introduce the current research methods of circRNAs, which include high-throughput library construction methods, identification through bioinformatics and common experimental verification. Here, we also systematically summarize the functions of circRNAs, including microRNA (miRNA) or protein sponges, regulating the alternative splicing (AS) and expression of host genes, and extensive translation. Finally, we provide a systematic characterization and the latest research progress of circRNAs, which provide a new perspective for further studies of circRNAs in plants.
circular RNAs; back-splicing; alternative splicing; sponge
2019-04-09;
2019-05-07
国家自然科学基金项目(编号:31800566),福建省自然科学基金项目(编号:2018J01608),国家重点研发计划项目(编号:2016YFD0600106、2018YFD0600101), 校国际合作(编号:KXGH17016)和福建省科技创新团队项目(编号:118/KLA18069A)资助[Supported by the National Natural Science Foundation of China (No. 31800566), Fujian Provincial Natural Science Foundation (No. 2018J01608), the National Key R&D Program of China (Nos. 2016YFD0600106, 2018YFD0600101), the International Science and Technology Cooperation and Exchange Fund (No. KXGH17016), and Fujian Provincial Science and Technology Innovation Team Project (No. 118/KLA18069A)]
刘旭庆,硕士研究生,专业方向:林木遗传育种。E-mail: 1752501050@qq.com
顾连峰,博士,教授,博士生导师,研究方向:生物信息学和林木遗传改良。E-mail: lfgu@fafu.edu.cn
张航晓,博士,讲师,研究方向:林木遗传改良。E-mail: hxzhang@fafu.edu.cn
10.16288/j.yczz.19-061
2019/5/8 13:48:21
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20190508.1346.004.html
(责任编委: 赵方庆)

