牡蛎诺如病毒多样性研究中病毒富集方法的评估
郭 萍, 潘迎捷,2, 喻勇新,2
(1. 上海海洋大学 食品学院, 上海 201306;2. 农业部水产品贮藏保鲜质量安全风险评估实验室, 上海 201306)
诺如病毒(Norovirus,NoV),又称为诺瓦克病毒,于1968年在美国俄亥俄州诺瓦克地区的一所学校中暴发的一起流行性腹泻的患者粪便中发现,因此而得名[1-4]。诺如病毒是单股正链的RNA病毒,属于杯状病毒科,主要分为7个基因类群,其中GI、GII、GIV型诺如病毒可以感染人类[5-6],由于GIV型诺如病毒没有GI和GII型流行,故一般着重于GI和GII型诺如病毒的研究,在GI和GII基因类群中,又可以分别细分出至少9和21种基因型。
诺如病毒在环境中传播的主要载体是以牡蛎为代表的双壳软体动物[7]。贝类中NoV污染不但危害人体健康,还严重影响我国的水产贸易[8-11]。研究发现,牡蛎中几乎含有所有的GI型和大部分GII型诺如病毒[18],通过对牡蛎中诺如病毒多样性的探究,可以更加了解牡蛎中诺如病毒的感染情况,有助于今后对海产品诺如病毒污染的监测和防控。
目前,对于牡蛎中RNA提取的方法有很多,但是大部分都关注诺如病毒检测工作,还没有一个较好的用于后期宏基因组测序的提取方法,这需要所提取的RNA尽可能覆盖整个生物群体,诺如病毒损失率尽可能低。因此本实验对4种常用宏基因组学研究中病毒富集方法(PEG6000沉淀法、超速离心法、过滤法和直接提取法)[12-15]进行评估,通过加入一定量含有GII.4型诺如病毒基因的慢病毒(Lentivirus)到预先检测为不含诺如病毒的牡蛎消化腺中,经过4种方法处理后提取RNA,利用一步法反转录荧光定量PCR对提取的RNA中Lentivirus进行定量,通过计算回收率来评估4种方法。同时为了进一步验证4种方法是否有利于去除微生物污染,采用16S rRNA通用引物对最后提取的RNA进行反转录PCR进行验证。目的是为了探寻一种最适用于后续牡蛎中诺如病毒多样性研究中RNA提取的方法。
1 材料与方法
1.1 材料
1.1.1 Lentivirus和GII型诺如病毒荧光定量PCR探针引物 带有GII.4 基因的Lentivirus 合成于深圳市百恩维生物科技有限公司;GII型诺如病毒荧光定量PCR探针(Ring2)引物(COG2F/COG2R)合成于宝生物工程(大连)有限公司,引物探针序列如表1所示。
1.1.2 试剂及仪器 1×PBS磷酸盐缓冲液、聚乙二醇6000(PEG6000)、三羟甲基氨基甲烷(Tris)购于上海生工生物公司;0.45 μm和0.22 μm滤膜滤器购于密理博中国有限公司;动物总RNA快速提取试剂盒(货号:GK3015)购于上海捷瑞生物工程公司;一步法反转录试剂盒(货号:RR055A),PCRTaq聚合酶购于宝生物工程(大连)有限公司;小型高速冷冻离心机、高速冷冻离心机、PCR仪、移液枪均采购于德国艾本德公司(Eppendorf); MP破碎仪购于美国MP生物公司; 一步法反转录q-PCR试剂盒(货号:4392656)购于美国ABI生物公司。
1.1.3 采样 新鲜太平洋牡蛎采集于上海市芦潮港水产市场(每只约150 g),处于低温条件立即带回实验室。
1.2 方法
1.2.1 RNA提取 按动物总RNA快速提取试剂盒说明书进行提取。
1.2.2 反转录巢氏PCR(Nested RT-PCR) 反转录巢氏PCR分为反转录PCR(RT-PCR)和巢氏PCR(nested PCR)。RT-PCR具体操作为:1 μL 的逆转录酶(酶保存在甘油中,吸取和吹打都要缓慢进行,以免造成人为误差),12.5 μL的2×RT-PCR 缓冲液,0.5 μmol/L的GI和GII型正反向引物(表1)于PCR小管中,加入30 ng的 RNA 模板后,最终以RNase-free ddH2O补足至25 μL。RT-PCR的程序如下:首先50℃下反转录30 min,在反转录酶的作用下,将单链 RNA 反转录成 cDNA(complementary-DNA),紧接着94℃预变性2 min;94℃变性30 s,55℃退火30 s,72℃延伸30 s,35个循环。Nested PCR具体操作为:将第一轮 RT-PCR的产物稀释10倍作为第二轮PCR的模板。第二轮PCR体具体操作为:将第一轮 RT-PCR的产物稀释10倍作为第二轮PCR的模板。第二轮PCR体系配方如下:分别取 12.5 μL的2×PCR缓冲液,1 μmol/L的GI和GII型正反向引物(表1),1 μL的模板于 PCR 小管中,最终用RNase-free ddH2O补足至25 μL。第二轮PCR的程序如下:94℃预变性5 min;94℃变性30 s,55℃退火保持30 s,72℃延伸30 s;35个循环。最后在72℃下保温15 min加poly-A 尾,以便后续的 TA 连接和测序。
1.2.3 牡蛎中诺如病毒检测 从水产市场采集完牡蛎后立即在超净工作台中对其解剖得到整只消化腺,用MP组织破碎仪将消化腺破碎均匀(转速:6 M/S;时间:30 s),平均分成几等分分装,每份50 mg,保存于-80℃冰箱。取一份牡蛎消化腺按照1.2.1所述的方法提取RNA。利用1.2.2的方法对所提 RNA进行诺如病毒检测,通过2%的琼胶电泳观察是否产生阳性GI和GII的PCR产物条带。
1.2.4 一步法反转录荧光定量PCR 通过反转录荧光定量PCR对Lentivirus拷贝数进行定量,20 μL反应体系为:10 μL 2×RT mix buffer, 1.8 μL 10 μmol/L的正反向引物(COG2F/COG2R), 0.5 μL 10 μmol/L的探针(Ring 2),0.5 μL的RT-Enzyme,2 μL RNA模板和3.4 μL的ddH2O。反应程序为:42℃预变性15 min;95℃变性10 min,95℃退火15 s,60℃延伸60 s,40个循环。在60℃收集荧光信号。
将108copies/μL GII型诺如病毒RNA标准品进行倍比稀释, 共稀释7个梯度,分别为107~101copies/μL。每个梯度的RNA标准品当作模板加入荧光定量PCR的反应体系中来制备绝对定量标准曲线。

表1 PCR引物
注:a为引物5′端在序列M87661的位置;b为引物5′端在序列X86557的位置
1.2.5 病毒富集方法比较 取2 g阴性牡蛎消化腺加入1 μL的Lentivirus,用18 mL的1×PBS将消化腺混匀,在4℃下8000 r/min离心20 min,取上清,并将上清均分为两部分。方法1(PEG6000沉淀法):取一部分的上清加入PEG6000至2%的终浓度,4℃孵育18 h,紧接着在4℃下8000 r/min离心20 min,弃上清,用100 r/min的Tris-HCl将沉淀重旋,提取RNA;方法2(过滤法):将另一部分的上清连续过0.45 μm和0.22 μm后,取滤液提取RNA;方法3(超速离心法):将方法2中过完膜的滤液9000 g超速离心4 h,弃上清,用100 μL的Tris-HCl将沉淀重旋,提取RNA;方法4(直接提取法):取2 g阴性牡蛎消化腺加入1 μL的Lentivirus后,直接提取RNA。图1为本过程的流程图,对4种方法提取的病毒RNA进行一步法反转录荧光定量PCR(如1.2.3所述)来计算Lentivirus的数量。

图1 4种方法流程图
1.2.6 16S PCR检测 以1.2.5提取的RNA为模板,用引物27F/1492R来进行RT-PCR反应,进行16S检测,将PCR反应产物通过2%的琼胶电泳来观察条带的亮弱。
25 μL PCR反应体系如下:12.5 μL的2×RT-PCR buffer, 27F(10 mmol/L)和1492R(10 mmol/L)各1 μL,30 ng RNA模板,RNase-free ddH2O补足至25 μL。
PCR反应程序如下:50℃反转录30 min;95℃预变性4 min;94℃变性30 s,50℃退火30 s,72℃延伸45 s,30个循环;最后再72℃延伸10 min。
2 结果与分析
2.1 牡蛎中诺如病毒检测
在2%的琼胶电泳观察是否产生阳性GI和GII的PCR产物(GI:330 bp,GII:343 bp)。如图2胶图所示,无诺如病毒GI型或者GII型目的条带产生,由此选择该只牡蛎消化腺为实验样品,定义为阴性牡蛎。
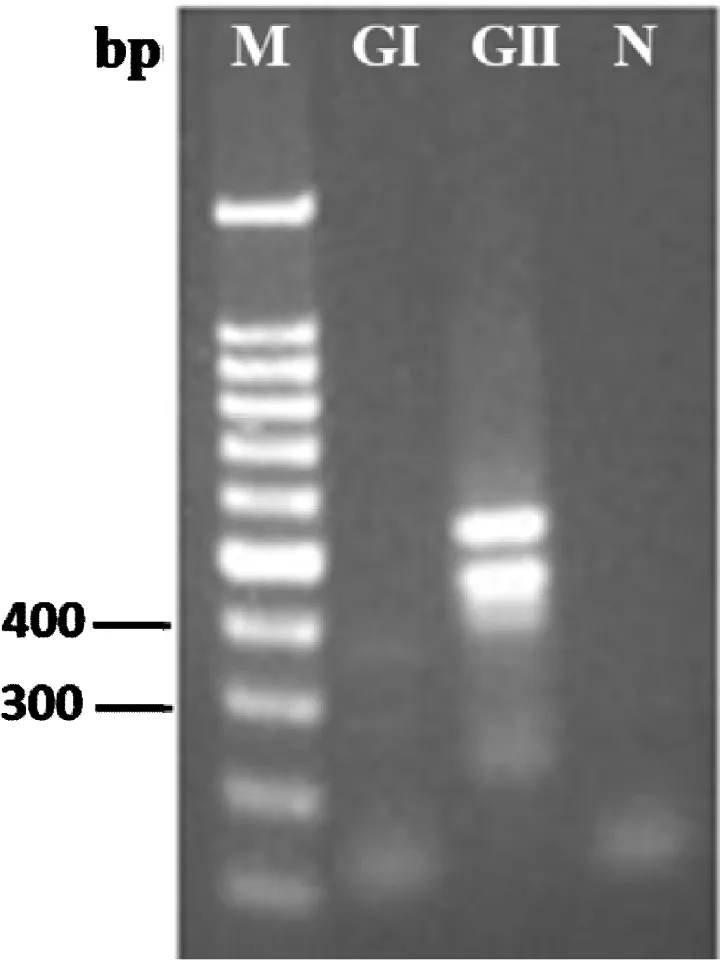
Nested RT-PCR的琼脂凝胶电泳图:M为DNA分子标尺(100 bp);GI为GI型诺如病毒检测结果;GII为GII型诺如病毒检测结果;N为阴性对照
图2 牡蛎检测结果
Figure 2 Result of oyster detection
2.2 反转录荧光定量PCR
Lentivirus的起始数量如图3-a、b显示,图3-a所示标准曲线R2为0.996,扩增效率为100.057%。图3-b所示原液和稀释10倍的Lentivirus都得到一个良好的扩增曲线,即Lentivirus的起始拷贝数为1.00×105copies/μL。
经过4种方法处理后得到的Lentivirus的数量如图3-c、d显示,图3-c所示标准曲线R2为0.999,扩增效率为100.889%。图3-d为4种不同处理后的扩增曲线,效果良好。由计算结果可知:方法1中Lentivirus的数量为2.17×103copies/μL;方法2为2.65×103copies/μL;方法3为1.03×103copies/μL;方法4为2.83×103copies/μL。图3-e、f为第2次重复性实验结果,图3-e所示标准曲线为0.997,扩增效率为100.125%。由图3-f的扩增曲线计算结果可知:方法1中Lentivirus的数量为2.20×103copies/μL;方法2为2.70×103copies/μL;方法3为1.27×103copies/μL,方法4为2.75×103copies/μL。由这两次重复性实验可知,计算结果相差不大,具有真实可靠性。
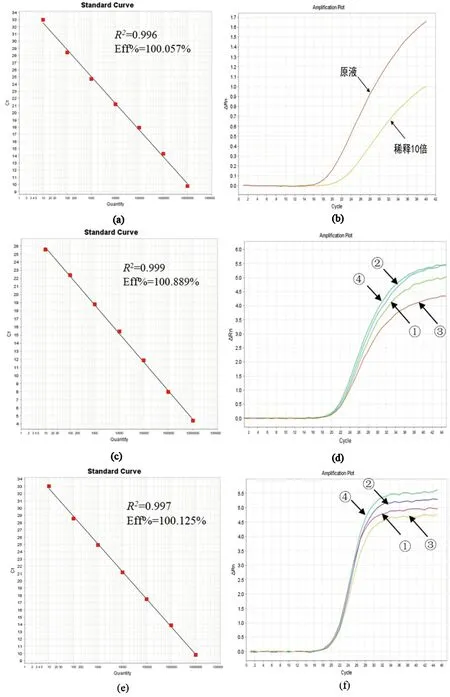
a、b:Lentivirus定量的标准曲线与扩增曲线;c、d:第一次方法评估结果的标准曲线与扩增曲线;e、f:第二次方法评估结果的标准曲线与扩增曲线
图3 荧光定量PCR结果
Figure 3 Result of RT-q PCR
利用荧光定量PCR的方法对Lentivirus起始拷贝数和经过4种方法提取后的Lentivirus进行定量,通过对比前后数量变化计算出Lentivirus回收率(表2),由两次重复实验可知这4种方法的Lentivirus回收率无明显区别,相比较而言方法1的回收率高于剩余其他3种方法,可初步判断方法1的效果优于另外3种方法。
2.3 16S PCR检测
将经过4种不同方法提取的RNA用16S RT-PCR的方法进行检测,通过2%的琼胶电泳条带的亮弱来反映细菌的污染程度,图4所示两次重复性实验结果,可看出经过这4种方法处理后都存在着一定程度的细菌污染,而方法4的条带稍亮于其他方法的条带,由此可说明方法4存在着相对较强的细菌污染。
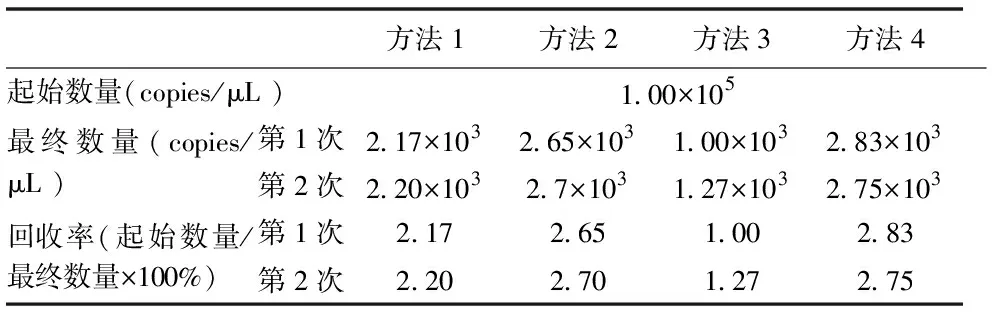
表2 4种方法回收率的计算
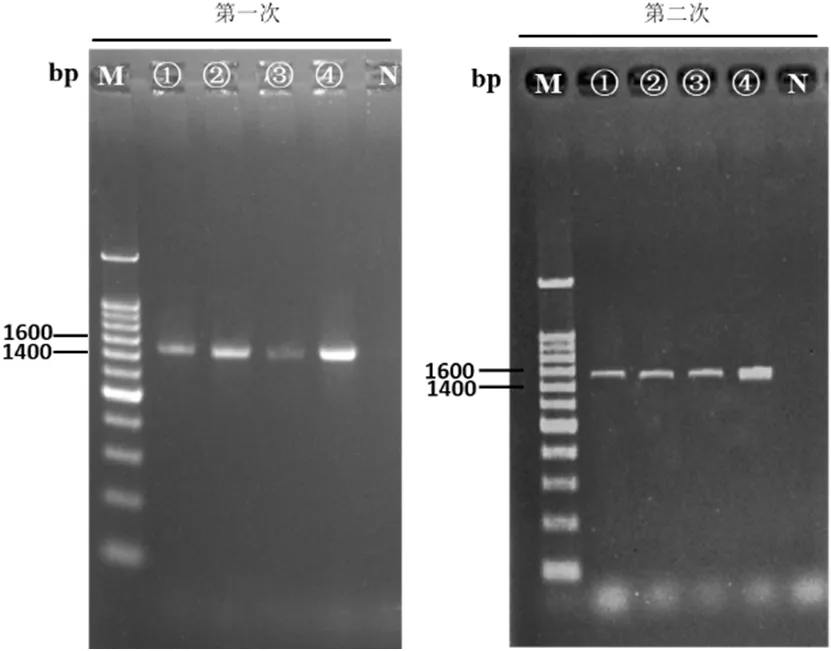
M:DNA分子标尺(200 bp);1~4:分别代表4种不同方法;N:阴性对照
图4 16S PCR检测结果
Figure 4 The result of 16S PCR
3 讨论
诺如病毒具有很强的感染能力,感染剂量大约为10到100个病毒颗粒,具有传播快的特点。而牡蛎作为诺如病毒环境传播的主要载体和滤食性动物,具有富集诺如病毒的作用,目前越来越多的人因为生食或半熟的牡蛎而感染诺如病毒。传统的RT-PCR方法并不能反映诺如病毒的流行病学[12],通过利用宏基因组学探究牡蛎中诺如病毒的多样性,不仅有助于监测海产品污染情况,还可以探寻诺如病毒与环境中潜在有助于感染的微生物的关系,更好地了解诺如病毒的传播[16]。因此探寻一种最适用于牡蛎诺如病毒多样性研究中RNA提取的方法则显得至为重要。
在本文中,为了更好地保证牡蛎RNA提取效果,我们针对目前市面上常有的3款RNA提取试剂盒(捷瑞,GK3015;天根,DP431;凯捷,74904)进行了比较,根据RNA琼脂糖凝胶电泳图发现捷瑞(GK3015)试剂盒提取的牡蛎RNA含量最高,且高浓度的proteinase K可以提高RNA的提取效率,凯捷试剂盒提取牡蛎中RNA的结果最差,因此将捷瑞试剂盒用于本实验中提取。此外,由Lentivirus回收率结果可知方法4>方法2>方法1>方法3, 而根据核酸凝胶电泳图条带亮弱可得细菌污染程度为方法4>方法2>方法1>方法3。可见在提取RNA时,对样品预处理过程越多会导致病毒损失率相应增加,但是这个富集过程在去除细菌污染方面具有一定的效果。对于后续多样性分析研究中,可能会由于缺少富集方法而导致它具有较为严重的细菌污染,对后期宏基因组测序产生一定的影响,但是更重要的是直接提取法相对较高的回收率可以更好地反映牡蛎中诺如病毒的多样性,尤其是在牡蛎样品中,诺如病毒颗粒数目与其他微生物相比只具有很小的比例,减少病毒损失就显得尤为重要。
通过比较4种目前常用于宏基因组测序的牡蛎中病毒富集方法,评价出一种效果相对较好的方法运用于牡蛎样品中,可能存在一定的实验偏差,但为后期顺利进行牡蛎中诺如病毒的多样性研究提供科学依据。

