酶解大豆分离蛋白的抗原活性变化及其抗原表位分析
王章存,袁路阳,张 露,胡金强,安广杰,赵学伟
(1.郑州轻工业大学食品与生物工程学院,河南 郑州 450001;2.食品生产与安全河南省协同创新中心,河南 郑州 450001)
大豆蛋白是最重要的植物蛋白来源,具有蛋白质含量高、氨基酸组成合理等优点,被广泛应用于食品工业中[1-2]。然而,大豆被公认为世界“八大”过敏原之一[3],目前已从大豆中确认出38 种蛋白质具有抗原性,其中大豆中的主要储藏蛋白β-伴大豆球蛋白(7S)和大豆球蛋白(11S)是主要的抗原性蛋白[4]。大豆导致的主要症状表现为胃部不适或过敏性皮炎,严重时导致人体和动物毛细血管渗透性增加、腹泻等,严重影响人体健康[5-6]。在世界范围内均对大豆蛋白的抗原性给予高度重视。
食品加工中最常用加热处理并不能有效消除大豆蛋白的抗原性,其原因是加热处理仅能破坏其构象型抗原表位(又称抗原决定簇),很难破坏其序列型抗原表位[7]。酶解处理不仅可破坏蛋白质的空间构象,也可水解序列型抗原表位,能有效降低甚至消除蛋白质的抗原性。近年来国内外对此进行了较多研究,发现用碱性蛋白酶水解降低抗原性的效果比其他蛋白酶较好[8-11]。但有研究表明,即使碱性蛋白酶也无法完全消除其抗原活性,而且酶解过程产生新的肽段对酶解具有一定的抵抗作用[12-15]。这些抗酶解肽段是否具有抗原活性、其氨基酸是否存在序列性抗原表位、它们来源于大豆蛋白的哪些亚基及其抗酶解的原因等问题,尚鲜见文献报道。
本研究用碱性蛋白酶水解大豆分离蛋白后,在分析蛋白质分子成分和抗原性变化的基础上,采用基质辅助激光解析串联飞行时间质谱(matrix-assisted laser desorption/ionization time of flight mass spectrometry,MALDI-TOF-MS)分析酶解后新产生的抗酶解成分的序列特征,并与抗原数据库数据及大豆分离蛋白的各亚基序列进行匹配,从分子水平上解释酶解不能完全消除大豆蛋白抗原性的原因,为有效酶解大豆蛋白去除其抗原性提供科学理论。
1 材料与方法
1.1 材料与试剂
大豆分离蛋白 河南阳光油脂公司。
碱性蛋白酶(2.4 AU/g) 丹麦诺维信公司;大豆酶联免疫吸附测定(enzyme-linked immunosorbent assay,ELISA)试剂盒 上海纪宁公司;蛋白胶微量回收试剂盒 北京百奥莱博科技有限公司;低分子质量标准蛋白(分子质量14~97 ku) 上海生物化学研究所;十二烷基硫酸钠 美国Sigma公司;三羟甲基氨基甲烷、甘氨酸、溴酚蓝、丙烯酰胺、二硫苏糖醇、N,N’-亚甲基双丙烯酰胺 生工生物工程(上海)股份有限公司。以上试剂均为分析纯。
1.2 仪器与设备
TDZ5-WS型低速离心机 湖南湘仪仪器公司;冷冻干燥机 北京博医康实验仪器公司;DYY-6C型电泳仪北京六一仪器厂;5800 MALDI-TOF-MS仪 美国AB SCIEX公司。
1.3 方法
1.3.1 大豆分离蛋白的水解
取10 g大豆分离蛋白加水配制成5%溶液,加热至55 ℃,用2 mol/L NaOH溶液调至pH 8.0。然后按照酶-底物为0.5%的比例加入碱性蛋白酶,反应开始并计时,整个反应过程中通过滴加2 mol/L NaOH溶液维持反应体系中pH值的稳定,反应达到一定时间后,取适量样品于沸水浴中保温10 min进行灭酶。然后将灭酶后的样品冷冻干燥并于-20 ℃保存。
1.3.2 十二烷基硫酸钠-聚丙烯酰氨凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDSPAGE)分析
根据Laemmli[16]的方法采用不连续垂直凝胶电泳系统。浓缩胶5%,电泳电压80V。分离胶12%,电泳电压110 V。采用14~97 ku低分子质量的标准蛋白,电泳结束后,采用考马斯亮蓝R-250进行染色,脱色后,采用Gel-PRO ANALYZER软件分析。
1.3.3 竞争法ELISA对酶解物抗原活性的测定[17]
采用竞争法ELISA检测酶解产物的抗原性,具体操作按照ELISA试剂盒说明书的步骤进行,计算出样品浓度,以抗原剩余率(%)表示酶解效果。每个样品重复3 次,数据采用Origin 8.0软件处理。
1.3.4 胰蛋白酶的胶内酶解
将21 ku和23 ku SDS-PAGE蛋白条带分别进行胶内酶解,方法参照文献[18]相关步骤进行操作。酶解后的样品经C18Cartridge脱盐后,冻干并于-20 ℃贮存备用。
1.3.5 MALDI-TOF-MS分析[19]
采用配有N2激光(337 nm,3 nm脉冲间隔)的MALDI-TOF-MS对所得肽段进行分析。取1 μL肽样品点加到专用不锈钢盘中,以α-氰基-4-羟基肉桂酸作为基质(用量1 μL,配制方法为取5 mg固体基质试剂,溶解到0.5 mL 0.1%三氟乙酸和50%乙腈溶液中),点样后室温下干燥。用Sigma公司标准分子质量肽进行外标分析。
采用正离子模式和自动获取数据的模式采集数据,一级质谱(MS)扫描范围为800~4 000 Da,选择信噪比大于50的母离子进行二级质谱(MS2)分析,二级质谱(MS2)累计叠加2 500 次。数据图谱由FlexControl软件采集,采用Data Explorer V4.5软件分析。
1.3.6 数据库检索
质谱测试原始文件用Mascot 2.2软件检索相应的数据库。检索参数如下:数据库:uniprot Glycine max;搜索类型:(MS+MS2)相结合;水解用酶:胰蛋白酶;质量值:单同位素;蛋白质质量:无限制;肽质量允许量:+100×10-6;肽片段质量允许误差范围:±0.4 Da;酶切位点数:1。
1.3.7 肽链抗原活性测定
SDS-PAGE胶片上的目的条带(未染色),采用蛋白胶微量回收试剂盒按照说明书步骤分别将胶中蛋白进行回收。回收的蛋白经C18Cartridge脱盐后用N2吹干于-20 ℃保存。将回收的样品用10 μL水复溶,按照1.3.3节方法进行抗原性的测定。
2 结果与分析
2.1 酶解过程中大豆分离蛋白成分的变化
大豆分离蛋白经碱性蛋白酶水解不同时间后,通过SDS-PAGE方法分析酶解物中组分的变化,结果如图1所示。
从图1可以看出,随着酶解时间的延长,蛋白质谱带发生了规律性的变化。酶解10 min 时,7S蛋白中的α’、α和β三个亚基都已完全消失,11S球蛋白中的38 ku酸性亚基(图1A)和21 ku碱性亚基(图1B)含量也明显减少,与此同时,新产生了大量23 ku组分及大量的分子质量小于14 ku组分。30 min时,21 ku不仅没有继续减少,而且相对于10 min时有少量增加的迹象,表明酶解过程中有新生成的21 ku组分。90 min时,38 ku的酸性亚基已完全消失。直到120 min时,21 ku和23 ku组分含量基本保持不变。从30~120 min酶解的趋势看,21 ku和23 ku组分始终没有将被酶解减少的迹象。
以上结果表明,大豆分离蛋白中的7S蛋白很容易被碱性蛋白酶水解,而11S球蛋白的碱性亚基不易被水解,新生成的21 ku和23 ku肽段对碱性蛋白酶的抗酶解现象更明显。
2.2 酶解物抗原活性分析
蛋白质的抗原性取决于其分子结构,酶解被认为是消除蛋白质抗原性的有效手段。但大量研究表明,抗原蛋白质分子中的抗原表位分为构象型和序列性两种类型[20-21]。构象型抗原表位随着蛋白质空间构象的变化消失明显,而序列性表位则相对稳定,即使蛋白质分子受到酶解处理,只要该氨基酸序列不被切断,则仍可能具有抗原活性。
为了解大豆分离蛋白酶解过程中抗原活性的变化,采用竞争法ELISA专用试剂盒测定不同酶解时间后大豆分离蛋白酶解物的抗原活性(图2)。之所以采用竞争法ELISA分析,是基于大豆蛋白经过酶解后分子质量降低,该法可以测定出样品中小分子抗原的存在,而且灵敏度很高。
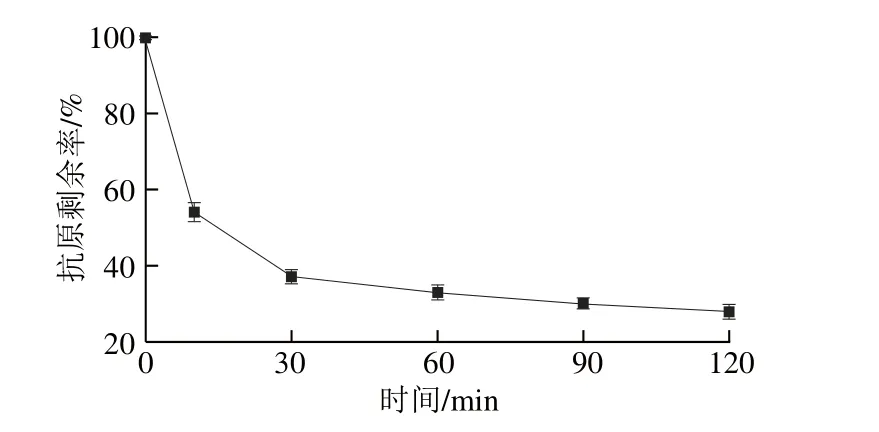
图2 不同酶解时间大豆分离蛋白抗原活性的变化Fig. 2 Changes in antigenicity during the hydrolysis of soybean protein isolate
从图2可以看出,在酶解开始10 min后,抗原活性有明显下降的趋势,此阶段也是7S蛋白中α’、α和β三个亚基被迅速酶解的时间(图1),可能在酶解的前10 min,抗原蛋白的空间结构发生明显变化,破坏了构象表位。在10~30 min时,抗原活性下降的趋势有所减弱,可能是随着构象表位的减少,酶与抗原蛋白的结合位点也随之减少,酶解速率下降减弱了抗原活性的下降趋势,也可能是在构象表位进一步遭到破坏后暴露出了抗原蛋白空间内部的线性表位,在一定程度上抑制了抗原活性下降的趋势[22]。在30~120 min时,抗原活性下降的趋势进一步减弱,并逐渐趋于平缓,此变化规律与酶解过程中蛋白质分子亚基的变化趋势相似,可能此时只剩余抗原蛋白中的线性表位,且这些线性表位不易被碱性蛋白酶快速水解破坏。直到酶解120 min时,大豆分离蛋白的抗原活性也未完全消失,意味着酶解物分子中可能仍有线性表位的存在。据报道,大豆11S球蛋白碱性亚基(图1中B条带)很少能与过敏者血清的IgE抗体发生结合而引起过敏反应[23-25]。所以酶解过程中抗原性的变化基本与该亚基没有关系,水解120 min后酶解物所残留的抗原活性都来源于新生成的组分。
2.3 MALDI-TOF-MS肽段分析
为进一步探讨酶解过程中新形成的具有抵抗碱性蛋白酶解特点的21 ku和23 ku肽分子特征与酶解物中残留抗原性的关系,分别对SDS-PAGE中21 ku和23 ku谱带(图1)进行切胶回收,并采用胰蛋白酶对回收的两个胶条分别进行胶内酶解,将胶内酶解物用MALDI-TOF-MS进行分析,见图3。将肽段的氨基酸序列与抗原数据库http://www.immuneepitope.org中抗原表位的氨基酸序列进行匹配,结果见表1、2。
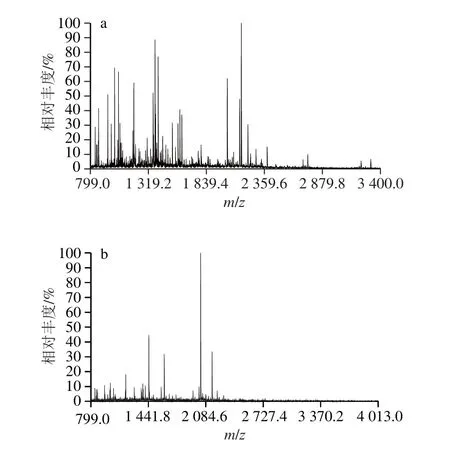
图3 酶解120 min时21 ku(a)和23 ku(b)的MALDI-TOF-MSFig. 3 MALDI-TOF-MS of 21 ku (a) and 23 ku (b) peptides obtained in the 120 min hydrolysate
从图3a可以看出,21 ku经胰蛋白酶胶内酶解后产生了很多肽段。通过Mascot软件将MALDI-TOF-MS检测到的肽段序列与抗原数据库中抗原表位序列匹配发现:MALDI-TOF-MS检测到21 ku中有16 条肽段(共检测到25 条)的氨基酸序列与抗原表位序列有部分序列重合,此结果说明:21 ku肽链经胰蛋白酶进行胶内酶解后,大部分完整的抗原表位都已被破坏。

表1 肽段21 ku中检测到的氨基酸序列和对应抗原序列Table 1 Identi fied peptide sequences in 21 ku fragment and corresponding antigen sequences

表2 肽段23 ku中检测到的氨基酸序列和对应抗原序列Table 2 Identifified peptides sequences in 23 ku fragment and corresponding antigen sequences
为进一步分析未经胰蛋白酶水解的21 ku肽链中是否存在完整的抗原表位序列,根据胰蛋白酶水解位点是赖氨酸(K)和精氨酸(R)羧基端的特点,将已检出抗原序列片段进行拼接,并与蛋白质数据库(http://www.uniprot.org)中大豆蛋白氨基酸序列进行匹配确定出其所在原大豆分离蛋白氨基酸序列的位置,再与抗原数据库的抗原表位氨基酸序列匹配,结果发现2 个完整的序列表位(图4):一个是表1中含有部分抗原序列的VLQRFNQR和SPQLQNLRDYR拼接后的氨基酸序列,与β-伴大豆球蛋白α-亚基中的VLQRFNQRSPQLQNLRDYR(序号为216~234)氨基酸序列重合。经抗原数据库查询,这个序列中包含有完整的抗原表位序列LQRFNQRSPQLQNLR。另一个是表1中TISSEDKPFNLR、SRDPIYSNK、LGKFFEITPEK和FFEITPEKNPQLR肽段经胰蛋白酶水解位点拼接后的氨基酸序列,与β-伴大豆球蛋白中α-亚基中的TISSEDKPFNLRSRDPIYSNKLGKFFEITPEK NPQLR(序列为398~434)的氨基酸序列重合。经抗原数据库查询,此序列中包含有完整的抗原表位序列SEDKPFNLRSRDPIYSNKLGKFFEITPEKN。以上结果说明:酶解物中仍残留抗原性的原因之一是酶解新产生的21 ku肽链中含有2 个完整的抗原表位,且此21 ku肽链来源于β-伴球蛋白的α-亚基。

图4 21 ku亚基中鉴定出包含完整抗原的肽段序列以及所在大豆分离蛋白的亚基位置Fig. 4 Complete antigen sequence in 21 ku peptide and its location in soybean protein
从图3b可以看出,23 ku肽链经胰蛋白酶胶内酶解后也产生了很多肽段。按照前述相同的分析方法处理后发现,有12 条肽段(共检测到17 条)含有抗原表位部分序列,其中有3 条肽段(表2中相对分子质量为1 010、1 051和1 618对应的肽段)均分别与两个以上的抗原表位部分序列重合。但是,将胰蛋白酶水解位点拼接后再与抗原数据库中的抗原序列进行匹配,未发现含有完整抗原表位序列的肽段。意味着未经胰蛋白酶水解的23 ku肽链中,没有与完整抗原表位序列相匹配的氨基酸序列。推测大豆分离蛋白经碱性蛋白酶水解120 min后所产生的23 ku组分中不存在抗原活性。
另外,在该蛋白酶解产物中还存在一些相对分子质量小于21 ku的组分(图1)。由于SDS-PAGE中这些组分的颜色较浅(意味着蛋白含量较少)且谱带特征不明显(分散),暂未对这些组分进行研究。这些组分是否含有序列型抗原表位,有待于进一步深入分析。
2.4 肽链抗原性分析
为验证21 ku和23 ku肽段是否存在抗原性,本研究采用蛋白胶微量回收试剂盒对胶内21 ku和23 ku蛋白分别回收,并采用竞争法ELISA试剂盒对其进行抗原活性的测定,计算其抗原剩余率分别为15.7%和2.1%。
21 ku组分有相对较高的抗原性(为大豆分离蛋白抗原性的15.7%),这与该组分中检测到存在完整抗原表位的结果一致。在23 ku组分的抗原剩余率仅为2.1%,而质谱分析中未检测出表位序列,这与两个因素有关:一是23 ku中可能存在未知的序列表位,相关数据库中没有列入,所以无法比对;二是可能与大豆蛋白分子中糖链的存在有关。β-伴大豆球蛋白是一类糖蛋白[26],有些糖蛋白的糖链本身就是抗原[27-29],且有研究表明大豆抗原蛋白经N-糖苷酶脱去糖链后其抗原性降低[30-31]。上述23 ku组分抗原性是否与糖链有关,本课题将进一步深入研究。
3 结 论
随着酶解时间的延长,大豆分离蛋白各组分及其酶解物的抗原活性发生规律性变化,且二者存在很大的相似性。酶解前30 min时,7S蛋白中α’、α和β三个亚基迅速被降解,且产生了21 ku和23 ku新组分,直至120 min,这些组分依然存在。竞争法ELISA分析表明:在酶解最初的30 min内抗原活性快速降低,之后降低趋势减缓,直到酶解120 min时,依然有28%的抗原剩余率。将21 ku和23 ku的电泳谱带分别经胰蛋白酶水解后采用MALDITOF-MS分析,其结果与抗原数据库、大豆蛋白数据库中相关氨基酸序列进行匹配表明:21 ku组分中含有两个完整的序列型表位,且这两个序列型表位均来自β-伴大豆球蛋白的α-亚基;在23 ku经胰蛋白酶水解后虽有片段与抗原表位部分序列相同,但未发现有完整的序列型表位存在。经对21 ku和23 ku组分单独进行抗原性测定发现,21 ku组分中有相对较高的抗原性,与上述21 ku中存在完整抗原表位的实验结果基本吻合,而23 ku依然表现出微弱的抗原性,是否与糖蛋白中糖链有关,有待进一步研究。上述结果一定程度解释了酶解物仍有抗原活性存在的部分原因,也揭示了新生成的具有抗原活性表位肽链的分子来源。

