八肋游仆虫组织蛋白酶B-1的克隆、表达和性质分析
刘婧妮,王软林,梁爱华
(山西大学 生物技术研究所,化学生物学与分子工程教育部重点实验室,山西 太原 030006)
0 引言
组织蛋白酶B(Cathepsin B,CTSB)是一类木瓜蛋白酶类半胱氨酸蛋白水解酶[1],广泛存在于从病毒到哺乳动物和人等生物体内。CTSB 的合成与其他蛋白酶相似[2],首先由糙面内质网上的核糖体合成无活性的前体酶原,随后信号肽在内质网腔中被水解掉,形成只含前体肽和催化结构域的酶原;接着酶原经过糖基化和磷酸化等修饰,与高尔基体上的甘露糖-6-磷酸受体结合,包裹在分泌小泡中转移至溶酶体;在溶酶体的酸性环境下,前体肽在自身或其他酶的催化作用下被水解,从而形成有活性的成熟 CTSB。
CTSB 的催化作用由天冬酰胺残基、半胱氨酸残基以及组氨酸残基组成的活性中心实现,除了内肽酶活性外,大部分的 CTSB 具有一个约由20个氨基酸组成的封闭环 (occluding loop) 结构[3],这使得 CTSB 具有除肽链内切酶以外的羧基二肽酶(外肽酶)活性,在封闭环结构中,有两个起关键作用的组氨酸残基,这两个残基能够直接影响组织蛋白酶 B 对酸碱环境的偏好性[4],如若封闭环缺失将导致内切酶活性上升而外肽酶活性下降,也会增加该酶与大分子抑制剂结合的能力[5]。此外,CTSB 还具有底物特异性,与组织蛋白酶L(CTSL)相比,CTSB 除可以催化 Z-Phe-Arg-AMC 底物的水解外,还可以催化 Z-Arg-Arg-AMC 底物的水解[6-7],这与 C 端 S2 domain 特殊氨基酸组成密切相关[8]。在高等真核生物的 CTSB 中,该位点为酸性氨基酸,能够催化 Z-Arg-Arg-AMC 和 Z-Phe-Arg-AMC 两种底物的水解。
目前关于 CTSB 的研究主要集中在抗寄生虫药物研发及癌症治疗[9]等方面。CTSB 除具有蛋白水解功能外,有研究已证明 CTSB 在寄生虫的免疫逃避[10]、营养摄取、包囊的形成和脱包囊等过程中也发挥着重要的作用。在大量寄生虫病的治疗中,CTSB 也被证实可以作为寄生虫病药物治疗的候选疫苗[11-12]。CTSB 还参与肿瘤的发生、血管生成、侵袭及转移等过程,被用作检测肿瘤恶性程度的标志物[13-14]。此外,CTSB 还参与寄生虫体内卵细胞的成熟过程中,它可以参与卵黄蛋白的水解[15],导致胚胎存活率下降。
八肋游仆虫(Euplotesoctocarinatus)是一种淡水纤毛虫,具有一个营养大核和一个生殖小核,其大核基因组高度片段化[16]。八肋游仆虫在蛋白质翻译水平上,存在着特殊的编程性核糖体移码现象[17-19],在遗传密码子的使用上,UGA 不是作为终止密码子而是重新编码为半胱氨酸,UAA 和UAG 作为终止密码子[20]。实验室前期通过对八肋游仆虫组织蛋白酶 B (EuplotesoctocarinatusCathepsinB,EoCTSB) 基因家族的分析,得到了6条可以编码EoCTSB的基因,且根据其与人CTSB基因相似性从低到高依次命名为EoCTSB-1、EoCTSB-2、EoCTSB-3、EoCTSB-4、EoCTSB-5、EoCTSB-6。本文选取EoCTSB-1进行表达纯化及其性质的研究,为进一步探讨八肋游仆虫中 6 个 EoCTSB 在细胞中的水解作用及其他功能奠定基础。
1 材料与方法
1.1 实验材料及主要生化试剂
八肋游仆虫由本课题组培养;大肠杆菌感受态细胞E.coliDH5α和E.coliBL21由本实验室制备保存;PCR引物由上海生工生物公司合成,DNA序列测定由华大基因公司完成;TaqDNA聚合酶、限制性内切酶、质粒抽提试剂盒、DNA胶回收试剂均购自TaKaRa公司;EasyScript®First-Strand cDNA Synthesis SuperMix试剂盒购自北京全式金公司;His亲和层析柱材料购于GE Healthcare公司;Anti-His抗体购于上海生工生物公司;荧光合成肽底物 N-苄氧羰基L苯丙氨酸L精氨酸-7-氨基-4-甲基香豆素(Z-Phe-Arg-AMC)购于peptide公司;其他生化试剂均购于山西鲲鹏公司。
1.2 实验方法
1.2.1 数据来源
EoCTSB-1的转录组数据由本实验室前期测得,嗜热四膜虫的数据下载自http:∥ciliate.org/index.php/home/welcome,人、小鼠及斑马鱼的CTSB序列分别下载自GenBank No:NP001899、No:AH001867.2、No:NP998501。
1.2.2 序列分析
利用MegAlign 程序中 Clustal W 算法对 EoCTSB-1 与其他生物 CTSB 的氨基酸序列进行比对;利用DNAStar软件包中的 SeqBuilder 程序对EoCTSB-1进行氨基酸翻译,利用在线软件ExPASy (http:∥web.expasy.org/compute-pi/) 对EoCTSB-1 的理论等电点及分子量进行分析。
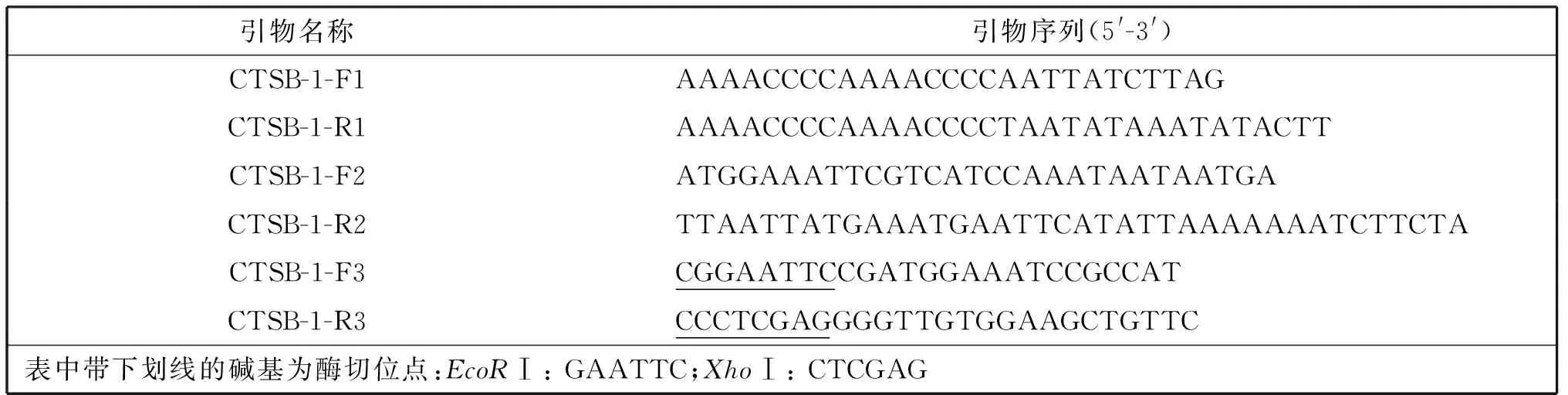
表1 实验所用引物序列
1.2.3 八肋游仆虫基因组 DNA 的提取及 cDNA 的合成
饥饿处理生长良好的八肋游仆虫5~7 d,然后室温下4 000 r/min离心5 min收集细胞,利用酚-氯仿法提取基因组DNA。利用miRNeasy Mini Kit (50)提取八肋游仆虫总RNA,随后利用EasyScript®First-Strand cDNA Synthesis SuperMix试剂盒合成cDNA的第一条链,并于-40℃保存备用。
1.2.4EoCTSB-1基因的克隆
根据八肋游仆虫基因组及转录组数据分析得到EoCTSB-1序列,设计引物CTSB-1-F1、CTSB-1-R1,CTSB-1-F2、CTSB-1-R2(表1),分别以八肋游仆虫基因组和cDNA为模板进行PCR扩增,利用质量分数1%琼脂糖凝胶电泳检测PCR产物。回收PCR产物,将其与pMD18-T过夜连接,连接产物转化至E.coliDH5α感受态细胞中,过夜培养后,挑取单菌落于5 ml LB培养基中过夜培养,提取质粒pMD18-T-EoCTSB-1,并测序。
1.2.5 重组质粒 pET-30a-EoCTSB-1 的构建
将测序正确的重组质粒pMD18-T-EoCTSB-1作为模板,利用特异性引物CTSB-1-F2、CTSB-1-R2进行PCR扩增,胶回收产物和载体pET-30a经EcoRⅠ和XhoⅠ双酶切。酶切产物回收后经T4连接酶连接,转化DH5α感受态,第二日裂菌筛选阳性克隆,提取重组质粒pET-30a-EoCTSB-1,酶切鉴定后送测序。
1.2.6 EoCTSB-1 蛋白的表达和纯化
将构建成功的重组表达质粒pET-30a-EoCTSB-1转化至E.coliBL21 感受态细胞中,于37℃培养箱中倒置过夜培养。首先进行小量诱导表达,从转化的平板挑单克隆到5 mL含相应抗性的LB液体培养基中,37℃培养,培养至OD=0.6-0.8,IPTG(0.5 mmol·L-1)诱导,37℃培养4 h,取1 mL诱导的菌液,12 000 r/mim,离心 1 min,超声破碎,分别取上清和沉淀(沉淀用50~100 μL 10 mmol·L-1Tris-HCl(pH 8.0)溶液吹散)加入与缓冲液等体积的2×loading buffer,100℃煮5 min,电泳检测。
小量表达的结果显示蛋白表达形式为包涵体,采用蛋白变复性方法获得可溶性蛋白。根据小量表达的最优条件,进行 EoCTSB-1蛋白的大量表达。8 000 r/min,离心6 min 收菌,弃上清。菌体用20~30 mL 10 mmol·L-1Tris-HCl(pH 8.0)溶液吹散,超声波破碎(500 W, 180次,每次5 s,间隔5 s)。13 000 r/min,离心20 min,弃去上清,沉淀用50 μL 10 mmol·L-1Tris-HCl(pH 8.0)溶液吹散。然后经过Ni柱纯化,用10 mmol·L-1Tris-HCl(pH 8.0)溶液平衡镍柱,约100 mL,再用含8 mol·L-1尿素、0.5 mol·L-1氯化钠的 10 mmol·L-1Tris-HCl(pH 8.0)溶液平衡镍柱,约50 mL。然后上样,上样结束后,用含 8 mol·L-1尿素、0.5 mol·L-1氯化钠的10 mmol·L-1Tris-HCl(pH 8.0)溶液洗柱。最后分别用含15 mmol·L-1咪唑、60 mmol·L-1咪唑、500 mmol·L-1咪唑的10 mmol·L-1Tris-HCl(pH 8.0)(含8 mol·L-1尿素、0.5 mol·L-1氯化钠)溶液洗脱,分别收集蛋白。最后利用western blot检测。
1.2.7 温度对 EoCTSB-1 活性的影响
1.2.7.1 最适反应温度的测定
以Z-Phe-Arg-AMC为底物,在pH 5.5的条件下,测定EoCTSB-1酶液在20℃~80℃的肽水解活性,每5℃为一个间隔,反应时间为10 min。
1.2.7.2 热稳定性的测定
将EoCTSB-1酶液放置在pH 5.5的条件下,在20℃~80℃的温度范围内,每5℃为一个间隔,放置60 min。然后用冰水冷却至4℃。以Z-Phe-Arg-AMC为底物,在pH 5.5的条件下,测定肽水解活性,反应时间为 10 min。实验重复3次。
1.2.8 pH 对 EoCTSB-1 活性的影响
pH范围:用0.2 mol·L-1Na2HPO4和 0.1 mol·L-1柠檬酸配成pH值为3.0、3.5、4.0、4.5、5.0、5.5、6.0、6.5、7.0、7.5、8.0的反应缓冲液。实验重复3次。
1.2.8.1 最适pH的测定
以ZPhe-Arg-AMC为底物,在37℃下,测定EoCTSB-1酶液在不同反应pH条件下的肽水解活性,反应时间为10 min。实验重复3次。
1.2.8.2 pH稳定性的测定
将 EoCTSB-1 酶液在 20℃,3.0~8.0的不同pH范围内,每0.5个pH单位为一个间隔,放置60 min,然后将pH调回至反应pH,pH为5.5,以Z-Phe-Arg-AMC为底物,测定肽水解活性,反应时间为 10 min。实验重复3次。
2 结果分析
2.1 八肋游仆虫EoCTSB-1基因的克隆与序列分析
分别利用特异性端粒引物CTSB-1-F1/R1和ORF引物CTSB-1-F2/R2(见表1),分别以基因组和cDNA为模板扩增EoCTSB-1的全长及ORF区。序列分析显示EoCTSB-1全长为980 bp,5′-Leader 区长31 bp,3′-Trailer 区长23 bp,ORF区为862 bp,无内含子,该基因在翻译过程中需要一次+1编程性移码[17],EoCTSB-1基因所编码蛋白产物的氨基酸数目为286个,分子量为32 570.66 D,理论等电点为6.22。
2.2 EoCTSB-1的氨基酸序列分析
利用DNAStar 软件中的 MegAlign 程序对EoCTSB-1及其他生物的CTSB进行氨基酸序列比对,结果显示(图1),EoCTSB-1在进化上既具有保守性,也具有特殊性。
EoCTSB-1具有保守的催化三联体结构(天冬酰胺残基N、半胱氨酸残基C以及组氨酸残基H),也具有前体肽结构,前体肽因其在空间结构会对催化活性中心有覆盖,降低底物和酶的结合能力,所以也被称作组织蛋白酶的内源抑制剂。在酸性条件下,前体酶原会发生自剪切,形成有活性的成熟蛋白。
EoCTSB-1的特殊性表现在不具有信号肽、封闭环结构及不能催化特异底物三个方面。信号肽的缺失推测EoCTSB-1不是分泌型蛋白,而是在胞内发挥催化功能。封闭环是CTSB催化活性部位一段额外的肽段,完整的封闭环结构由上下游两个保守的半胱氨酸和两个关键的组氨酸残基组成,序列比对结果显示,在人、斑马鱼、小鼠和四膜虫的CTSB中均具有完整的封闭环结构,EoCTSB-1缺失该结构,将导致外肽酶活性的消失。CTSB催化特异性底物的能力与S2 domain的特殊位点氨基酸有关,氨基酸序列比对结果显示,EoCTSB-1和四膜虫在S2 domain的特殊位点的氨基酸为中性氨基酸,推测它们均不具有催化特异性底物Z-Arg-Arg-AMC的能力。
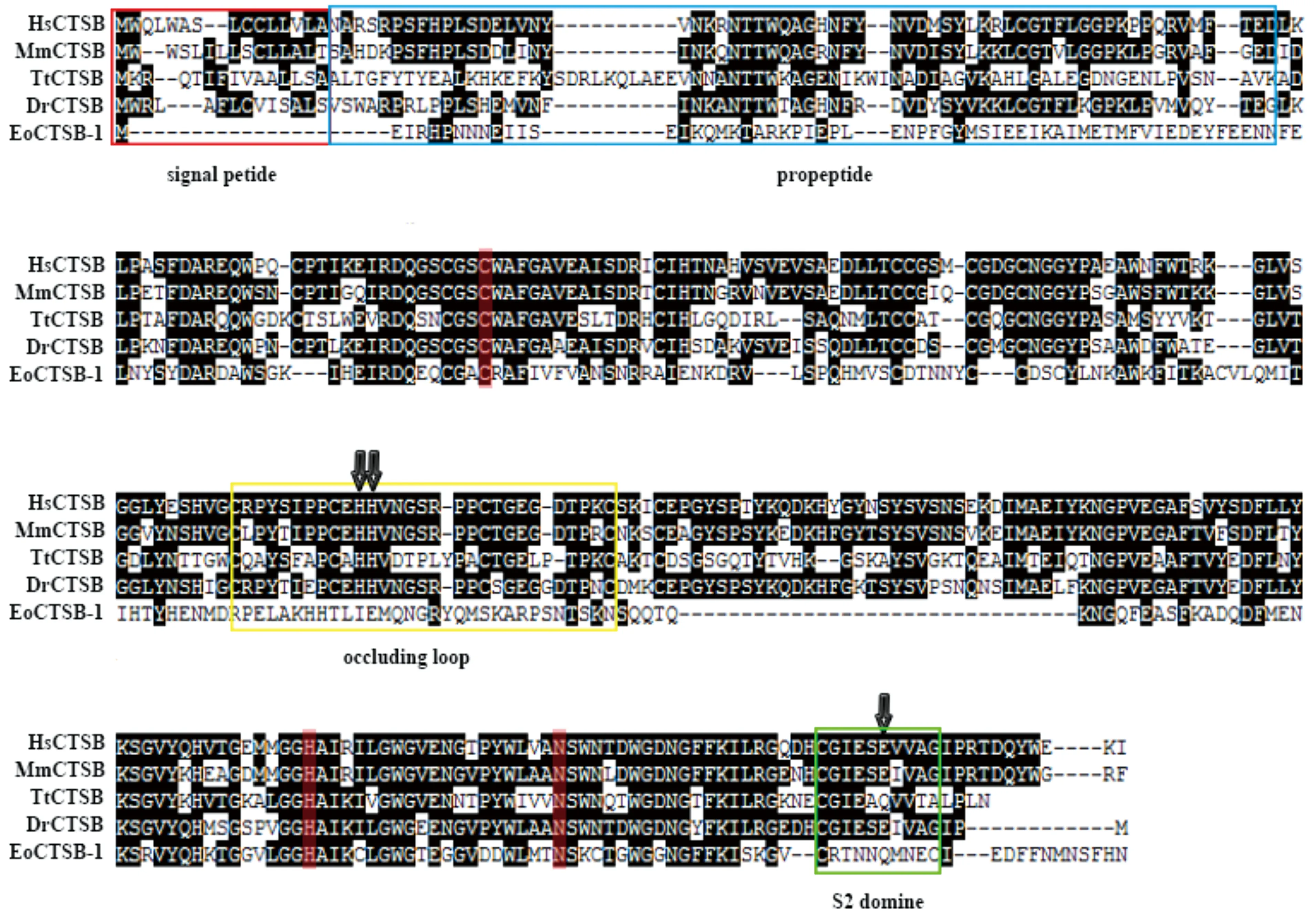
Three highly conserved CTSB active sites are indicated by red shadow.The signal peptide region is indicated by red shadow.The propeptite is showed by blue box. The occluding loop region is showed by yellow box,the histidine residues critical are indicated by the arrows. The S2 domain is indicated by green box,with the critical substrate specificity residue indicated by a filled arrow.Fig.1 Sequence alignment of CTSB fromEuplotes octocarinatusCTSB-1 with those from other organisms红色阴影表示三个保守的活性位点;红色框表示信号肽;蓝色框表示前体肽;黄色框表示封闭环;绿色框表示S2区域;黑色箭头表示关键性残基。图1 八肋游仆虫组织蛋白酶B-1与其他物种的CTSB 氨基酸序列比较
2.3 重组表达质粒pET-30a-EoCTSB-1的构建与EoCTSB-1蛋白的表达纯化
重组表达质粒pET-30a-EoCTSB-1经双酶切和测序鉴定构建成功(图2A),将构建成功的重组表达质粒pET-30a-EoCTSB-1转入感受态E.coliBL21中,进行小量试表达。结果显示在37℃诱导后的全菌中,出现新增蛋白条带且分子量约42 kD,这与预测融合蛋白的大小一致,在未加IPTG的全菌中没有此条带,分别对诱导后全菌的沉淀和上清提取物分析,结果显示新增蛋白条带在沉淀中出现,上清则检测不到,利用His-Tag抗体进行western blot检测,也表明该蛋白表达形式主要为包涵体(图2B)。之后大量表达目的蛋白,利用Ni柱亲核层析纯化目的蛋白(图2C),将500 mmol·L-1咪唑洗脱得到的变性蛋白,利用尿素浓度逐渐降低的缓冲液过夜透析,使蛋白逐渐复性,最后利用30 kD的浓缩管将目的蛋白浓缩。Western印迹分析表明,浓缩后可溶性EoCTSB-1蛋白可与Anti-His抗体产生特异性的反应(图2D),表明通过变复性获得了可溶的 EoCTSB-1。
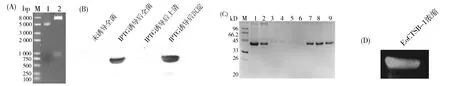
(A)Construction of recombinant plasmid pET-30a-EoCTSB-1 lane M:Trans2KTMPlusⅡ DNA Marker,lane1: Recombinant plasmid pET-30a-EoCTSB-1, lane2: Recombinant plasmid pET-30a-EoCTSB-1 was digested byEcoRⅠ andXhoⅠ. (B) Western Blotting was used to detect the expression of EoCTSB-1 protein;(C) Mass purification of EoCTSB-1,lane M: Marker, lane1 and 2: whole cell extract and supernatantprotein fraction ofE.coilpost IPTG induction; lane 3-4:15 mmol·L-1imidazole elution, lane 5-6:60 mmol·L-1imidazole elution, lane7-9:500 mmol·L-1imidazole elution;(D) Western Blotting detects the concentrated EoCTSB-1 protein.Fig.2 Construction of recombinant plasmid pET-30a-EoCTSB-1 and expression and purification of EoCTSB-1(A) 重组表达质粒pET30aEoCTSB-1的构建,lane M:Trans2KTMPlusⅡ DNA Marker,lane1:重组质粒pET-30a-EoCTSB-1,lane2:重组质粒pET-30a-EoCTSB-1经EcoRⅠ和XhoⅠ双酶切;(B) Western Blotting 检测 EoCTSB-1 蛋白的表达;(C) EoCTSB-1的大量纯化,lane M:Marker,lane1:蛋白原样,lane2:IPTG诱导后上清,lane3-4:15 mmol·L-1咪唑洗脱,lane5-6:60 mmol·L-1咪唑洗脱,lane7-9:500 mmol·L-1咪唑洗脱;(D) Western Blotting检测浓缩后的EoCTSB-1蛋白。图2 重组质粒pET-30a-EoCTSB-1构建及EoCTSB-1蛋白的表达与纯化
2.4 温度对 EoCTSB-1 活性的影响
对EoCTSB-1的最适反应温度及热稳定性进行测定,从图3A中可以看出EoCTSB-1的最适反应温度为35℃。从图3B中可以看出,EoCTSB-1在40℃放置1 h后,只残留18%的活性。

Fig.3 (A) Profile of temperature-dependent of EoCTSB-1; (B) Profile of thermal stability of EoCTSB-1图3 (A) EoCTSB-1的最适反应温度;(B) EoCTSB-1的热稳定性
2.5 pH 对 EoCTSB-1 活性的影响
对 EoCTSB-1 的最适反应 pH 及 pH 的稳定性进行测定,从图 4A 中可以得出 EoCTSB-1 的最适反应 pH 为 5.0。从图 4B 中可以得出,EoCTSB-1 在 pH 为 3.5 的缓冲液中放置1 h后,仍保持 85% 的活性,随着缓冲液 pH 的增加,酶活明显的下降,但EoCTSB-1在 pH 为 7.0 的缓冲液中处理1 h后,仍有 30% 的活性。

Fig.4 (A) Profile of pH-dependent of EoCTSB-1;(B) Profile of pH stability of EoCTSB-1图4 (A) EoCTSB-1的最适反应pH;(B)EoCTSB-1的pH稳定性
3 讨论
近年来,关于CTSB的报道主要集中于寄生虫病和人肿瘤细胞的治疗中,在其他生物中的研究较少。实验室前期对八肋游仆虫进行了基因组和转录组测序,通过分析,我们在八肋游仆虫数据库中找到6条可以编码EoCTSB的基因,而在高等真核生物和人体内,CTSB基因只有1条。为了进一步探讨纤毛虫中组织蛋白酶B的功能,本文选取EoCTSB-1进行进一步研究,首先扩增得到EoCTSB-1基因的全长序列,并对由其核酸序列推导出的氨基酸序列进行了分析,与其他真核生物的CTSB的氨基酸序列比对结果显示,EoCTSB-1含有保守的催化活性位点和前体肽结构,但缺乏信号肽结构,推测其不是分泌型蛋白,细胞内的具体功能还需进一步的研究。此外,EoCTSB-1还缺乏完整的封闭环结构,即没有外肽酶活性,通过查阅文献发现在一些藻类、寄生虫和原生动物中,封闭环结构也发生了不同程度的变化,包括氨基酸或小肽片段的插入、大片段的删减以及完全缺失[21],所以此现象并不是八肋游仆虫所特有的。有文献报道,封闭环的缺失虽然消除了外肽酶活性,但不影响内肽酶活性,且对其有促进作用[22]。
在序列分析基础上,本研究对EoCTSB-1蛋白进行了表达和纯化,在SDS-PAGE中显示纯化的蛋白大小约为42 kD,比前期预测的组织蛋白酶B分子量32 kD大了10 kD左右,这是因为pET-30a载体在N端除具有His标签外,还具大小约8 kD的N-Thrombin标签。小量预表达结果显示蛋白表达形式主要为包涵体,我们通过变复性的方法获得了可溶性蛋白,然后对其酶活性质进行了研究。结果分析显示,EoCTSB-1的最适温度是35℃,最适pH为5.0,与其他报道的CTSB的最适酶活一致[23]。与利用酵母表达系统表达纯化的蛋白热稳定性[24]相比,EoCTSB-1热稳定性较差,推测EoCTSB-1热稳定性较差可能与它在大肠杆菌中表达不能被糖基化有关。EoCTSB-1的pH稳定性测定结果显示,在pH 7.0的缓冲液处理1 h后,仍具有30%的活性,这与其他生物CTSB的报道是一致的[25]。分析EoCTSB-1的氨基酸序列,找到序列中存在3个可能的糖基化位点,下一步可尝试在酵母中表达EoCTSB-1,从而通过糖基化提高该蛋白的热稳定性以便开展更多的性质研究。总之,本研究为我们后续对其他5条EoCTSB基因的研究提供了实验数据,对下一步制备特异性抗体开展CTSB在八肋游仆虫中功能的研究奠定基础。

