水稻根系发育基因OsKSR7的克隆与功能分析
周佳琴,朱俊兆,杨思学,诸周洁,姚婕,郑文娟,朱世华,丁沃娜
水稻根系发育基因的克隆与功能分析
周佳琴1,朱俊兆2,杨思学2,诸周洁2,姚婕2,郑文娟2,朱世华2,丁沃娜2
(1宁波大学海洋学院,浙江宁波 315211;2宁波大学科学技术学院,浙江宁波 315212)
【目的】水稻根系是与地上部性状和产量密切相关的重要农艺性状。通过鉴定新的水稻根系发育相关基因,为深入解析水稻根系发育遗传机理奠定基础。【方法】从甲基磺酸乙酯(ethyl methane sulfonate,EMS)诱变的水稻Kasalath突变体库中筛选到1个根系发育缺陷的突变体()。通过溶液培养和田间种植,对该突变体进行苗期表型鉴定及成熟期主要农艺性状考察。将分别与野生型籼稻Kasalath和粳稻Nipponbare杂交,F2群体进行遗传分析和突变基因的图位克隆,对预测的候选基因进行测序验证。构建由35S启动子驱动的回复载体,通过农杆菌介导转入突变体成熟胚诱导的愈伤组织进行转基因互补验证。【结果】与野生型相比,幼苗期的主根、不定根、侧根和根毛的伸长都受到抑制,主根、不定根和侧根的长度分别只有野生型的33%、38.9%和35.3%,但不定根数有显著增加。农艺性状调查发现,的株高、穗数、茎秆粗细、结实率、千粒重和剑叶长宽等性状都受到显著影响,其中,穗数和结实率的差异极显著,分别只有野生型的56.3%和37.3%。遗传分析表明,突变体和籼稻Kasalath杂交的F1表型正常,F2群体中正常植株与短根突变植株的分离比符合3﹕1,表明突变体的突变性状受1对隐性核基因控制;利用SSR和InDel分子标记将突变基因定位在水稻第11染色体上IND1与IND2之间,物理距离约为143 kb的区间。在该区间有25个预测注释基因,候选基因测序比对发现,突变体中的一个编码转运蛋白的基因第一个外显子上ATG后73 bp处的T突变为A,导致编码的第25位氨基酸由色氨酸突变为精氨酸。生物信息学分析表明是介导蛋白质从内质网(ER)运向高尔基体(Golgi)的COPII有被小泡的SEC23亚基在水稻中的同源基因。RT-PCR表明的表达水平在野生型和突变体中无显著差异,35S启动子驱动的的回复载体能够使突变体的表型回复成野生型,证实的表型是由突变引起。【结论】是一个水稻短根突变体,其产量相关的几个重要农艺性状显著受抑制,突变基因为,编码COPII有被小泡的SEC23亚基,与已报道的水稻根系基因都不等位,是一个新的水稻根系发育调控基因。
水稻;短根突变体;遗传分析;图位克隆;功能互补
0 引言
【研究意义】根系是植物在长期进化过程中形成的重要营养器官,发达的根系为水稻生长发育提供充足的养分和水分,根系也是感知逆境信号的重要器官,与水稻抗逆性密切相关[1-3]。根系形态与产量的关系一直是水稻根系研究的热点,地下部根系性状与地上部性状具有联动关系,根长、根重、根直径等与地上部农艺性状密切相关[4-6]。但长期以来由于研究技术的局限性,水稻根系功能基因的研究相对落后。【前人研究进展】水稻是须根系单子叶作物,其根系由主根、不定根、侧根和根毛组成。目前,已克隆了40多个水稻根系发育调控基因[7-9],其中部分和产量相关。如,编码K+/Na+/Cl-协同转运蛋白的通过调控K+/Na+/Cl-平衡参与细胞伸长进程,突变体整个生育期的主根、侧根和冠根均变短,大田下产量下降,穗数、千粒重、每穗粒数均显著降低[10]。编码β-酮脂酰-酰基载体蛋白合酶的参与脂肪酸合成,突变体的主根、侧根和不定根都明显变短,分蘖数、花粉育性和千粒重均降低[11]。编码线粒体蛋白的与铁的动态平衡有关,胚后根变短,成熟期植株较野生型矮化,分蘖数显著减少[12]。编码中性/碱性转化酶的催化蔗糖水解成葡萄糖和果糖,在根系发育早期供碳源和能量突变体的主根、不定根和侧根均变短,花期延迟,花粉部分不育,结实率显著降低[13]。此外,编码WUSCHEL相关的同源异型框基因的可能是整合生长素和细胞分裂素信号传导的因子,突变体主根变小,发芽后冠状根数目减少甚至没有冠状根,成熟期株高变矮,分蘖数减少,结实率大幅下降[14]。参与调控根生长的基因编码蛋白的功能差异很大,表明多因素多信号途径调控水稻根生长发育的复杂性。【本研究切入点】通过水稻根系基因的功能鉴定,对水稻根系发育的分子调控模式有了一定程度的认识,但相对于其他器官的研究程度来说仍远远不够,需要分离和鉴定更多的根系调控基因。【拟解决的关键问题】本研究从EMS诱变的籼稻Kasalath突变体库中筛选获得1个短根突变体,通过对该突变体进行表型鉴定、突变基因的图位克隆和转基因互补验证,为深入解析该基因的功能和在水稻根系育种上的应用奠定基础。
1 材料与方法
1.1 试验材料
短根突变体来自浙江大学植物生理学与生物化学国家重点实验室构建的籼稻(Kasalath, indica)EMS诱变突变体库。2011—2015年,在宁波大学科学技术学院试验田连续自交5代,突变性状稳定且不分离。将突变体分别与Kasalath和粳稻Nipponbare杂交,观察F1的根系表型,F1自交获得的F2群体用于遗传分析和基因定位。
1.2 突变体的表型鉴定
采用溶液培养法培养水稻幼苗[15],观察生长7、14和21 d的野生型Kasalath和突变体表型并拍摄,对植株的重要性状(株高、种子根长、不定根长、不定根数和侧根长)进行测量,绘制根系生长曲线。样本统计数为20株,3次重复。
随机选取大田种植的野生型Kasalath和突变体各10株,成熟期测定主要农艺性状,包括株高、穗数、茎秆粗细、剑叶长宽、穗长、结实率和千粒重等。
1.3 分子标记的选择与设计
基因定位用的SSR(simple sequence repeat)分子标记来自Gramene网站(http://www.gramene.org/),在12条水稻染色体上按照遗传距离均匀分布的原则选择SSR标记进行初定位。精细定位中新的分子标记发展是根据已公布的Nipponbare全基因组序列和Kasalath全基因组重测序数据,在初定位区间内分析2个亲本间有差异的序列并设计引物,选取有多态性的InDel标记用于进一步定位。
1.4 突变基因的图位克隆
采用TPS法提取2个亲本、F1和F2分离群体的DNA[16]。在×Nipponbare的F2分离群体中随机挑选30个短根株系提取DNA后等量混合形成突变体基因池。PCR扩增挑选的有多态性的SSR标记,用非变性聚丙烯酰胺凝胶电泳进行检测,观察银染显色后的带型判断连锁关系,对疑似连锁的标记进行解包检测。在初定位区间内设计新的分子标记和扩大定位群体,逐渐缩小定位区间。通过水稻基因组数据库网站(http://rice.plantbiology.msu.edu/cgi-bin/gbrowse/ rice/)分析定位区间内的候选基因,测序验证确定突变位点。
1.5 转基因互补验证
根据的mRNA序列和带有35S启动子的pCAMBIA1300载体上的多克隆位点信息,设计带有酶切位点的引物OsKSR7-F(5-AAATCCC ATCCCCACCACCTCTC-3)和OsKSR7-R(5- AAAAATCTCCCACAACTCAGACCT-3)扩增的CDS序列,上下游引物分别引入Ⅰ和HⅠ酶切位点(下划线表示)。以野生型kasalash叶片的cDNA为模板进行PCR扩增,PCR产物和pCAMBIA1300载体用Ⅰ和HⅠ双酶切后连接,用热击法转化大肠杆菌DH5α感受态细胞。阳性克隆抽提质粒后用Ⅰ和HⅠ双酶切验证再测序确定。鉴定后的质粒用电击法转化农杆菌感受态EHA105,经农杆菌介导的水稻遗传转化体系转入突变体成熟胚诱导的愈伤组织[17]。用Trizol法分别提取T0代转基因植株、野生型和突变体叶片的总RNA,逆转录成cDNA后用引物RT-F(5-TAGGTTCTTGCTCCCGGTGTC-3)和RT-R(5-ATGGCCTTGACTGACCAATTGTT-3)进行RT-PCR鉴定,选取基因表达量高的转基因植株繁种获得纯合株系后进行表型分析。
2 结果
2.1 突变体表型鉴定
对溶液培养7 d的幼苗进行观察,与野生型相比,突变体植株整体矮小,地上部高度为野生型的75.9%(图1-A,表1),主根、不定根和侧根的长度分别只有野生型的33%、38.9%和35.3%,但不定根数有显著增加(图1-B,表1)。在体式镜下观察根部表型,发现根毛的发生和伸长也都受抑制(图1-C)。根系生长曲线表明突变体根系的伸长明显比野生型缓慢(图2)。
在成熟期考察重要的农艺性状,与野生型相比,突变体主要表现为穗数减少,为野生型的56.3%;茎秆变细,为野生型的70.8%;结实率明显降低,仅为野生型的37.3%。此外株高变矮,叶片变窄,叶长变短,千粒重显著降低(表2)。说明呈“一因多效”,不仅影响根系发育,而且影响水稻的产量。
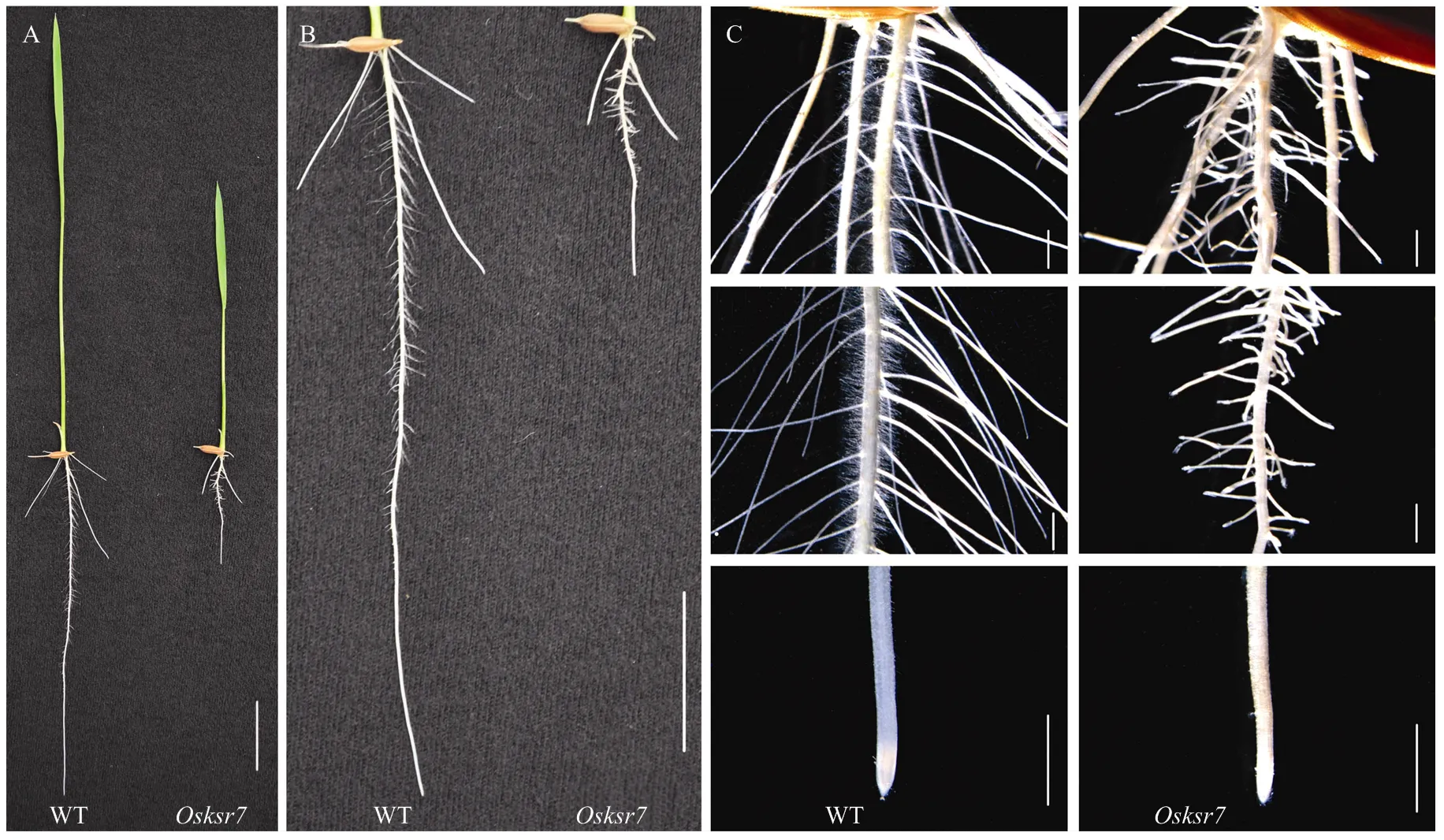
A:野生型(WT)和突变体Osksr7全株照,bar=2 cm;B:野生型(WT)和突变体Osksr7根部照,bar= 2 cm;C:野生型(WT)和突变体Osksr7主根的体视镜照片,bar=1 mm

图2 突变体Osksr7与野生型(WT)的主根(A)和不定根(B)的生长曲线
2.2 突变体的遗传分析
将短根突变体与籼稻Kasalath杂交获得F1,F1个体根系正常,溶液培养7 d的338个F2植株中,野生型表型的有257株,突变体表型的有81株(图1-B),经卡方检验符合孟德尔的单基因控制的遗传规律(χ2=0.19<χ20.05=3.84)(表3),表明的突变表型由1对隐性核基因控制,命名突变基因为。
2.3 OsKSR7的图位克隆
利用混合分离分析法(bulked segregant analysis,BSA)[18]进行的初定位。用均匀分布于水稻12条染色体的115对SSR引物进行初步定位,发现突变基因与第11染色体上的RM21标记连锁,再用30株F2单株对RM21进行连锁分析,确定了该分子标记与突变位点连锁。
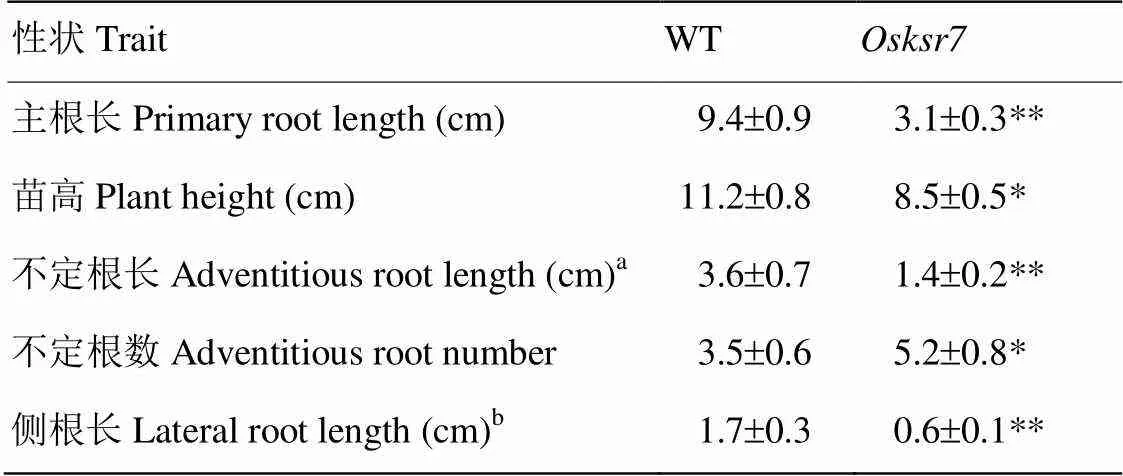
表1 野生型(WT)和突变体Osksr7生长7 d的植株表型比较(平均值±标准差)
a最长的3根不定根的平均长度;b主根上测量的最长10根侧根的平均长度;*表示在0.05水平差异显著;**表示在0.01水平差异显著。下同
aThe average length of three longest adventitious roots;bThe average length of ten longest lateral roots on each seminal root; *Significant at<0.05; ** Significant at<0.01. The same as below
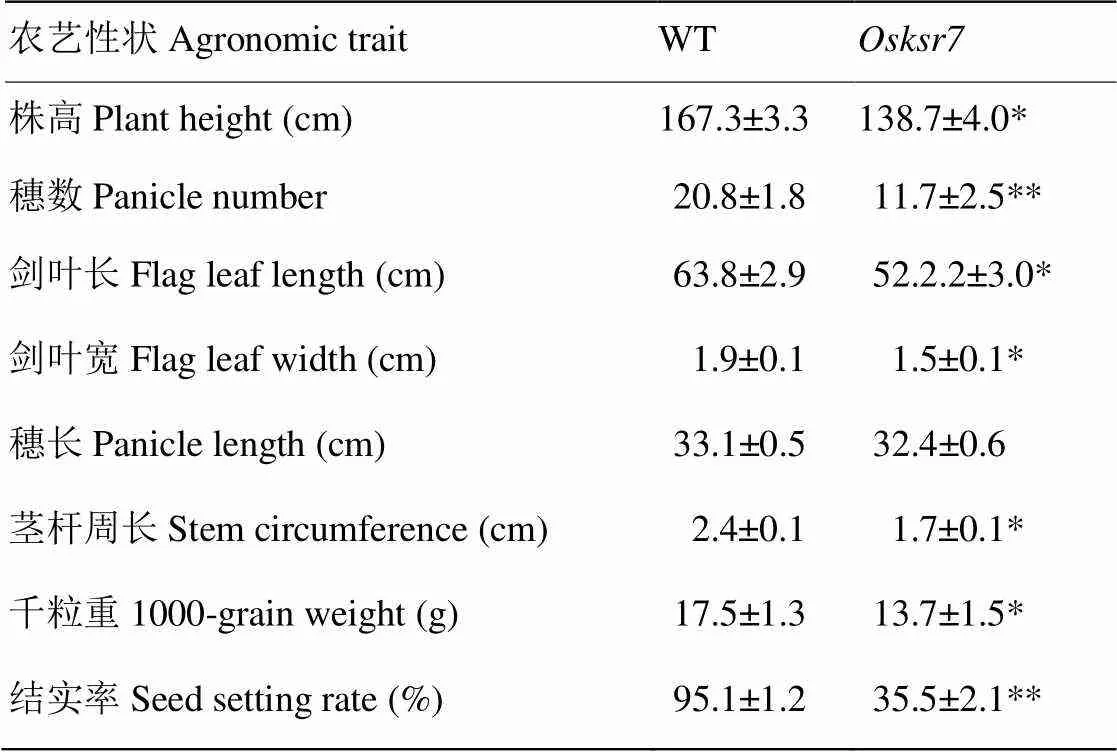
表2 野生型(WT)和突变体Osksr7的农艺性状比较(平均值±标准差)

表3 突变体Osksr7的遗传学分析
之后,扩大定位群体至1 587株,并在RM21的上下游设计新的InDel标记(表4),最终将基因锁定在分子标记为InD1和InD2之间的143 kb区间内,覆盖有2个BAC克隆:OSJNBb0013M05和OSJNBa0041K10(图3-A)。根据水稻基因组注释信息,该区间有25个预测基因,其中4个为功能基因,对这4个候选基因PCR扩增后测序,发现基因号为LOC_Os11g24560的第一个外显子上ATG后73 bp处的T突变为A,导致编码的氨基酸序列的25位色氨酸(Trp)突变为精氨酸(Arg)。生物信息学分析结果表明,该基因预测编码一个转运蛋白,是负责内质网到高尔基体蛋白运输的COPII有被小泡的SEC23亚基。LOC_Os11g24560基因全长为7 681 bp,编码区为2 382 bp,有12个外显子和11个内含子(图3-B),蛋白序列为793个氨基酸。

A:OsKSR7在第11染色体上的精细定位;B:OsKSR7的结构,黑框代表外显子,白框代表非翻译区

表4 用于Osksr7定位的分子标记序列
2.4 OsKSR7的互补验证分析
为了确认突变体表型是由的突变引起的,构建35S启动子驱动的的回复载体,以突变体成熟胚愈伤组织为受体,通过农杆菌EHA105介导,成功获得8个根系变长的T0转基因株系。T2代纯合株系溶液培养鉴定,发现突变体的表型回复至野生型状态(图4-A和图4-B),经RT-PCR验证在转基因株系中超表达,且该基因在野生型和突变体中的表达水平没有明显差异(图4-C)。说明突变体表型确实是由突变引起。
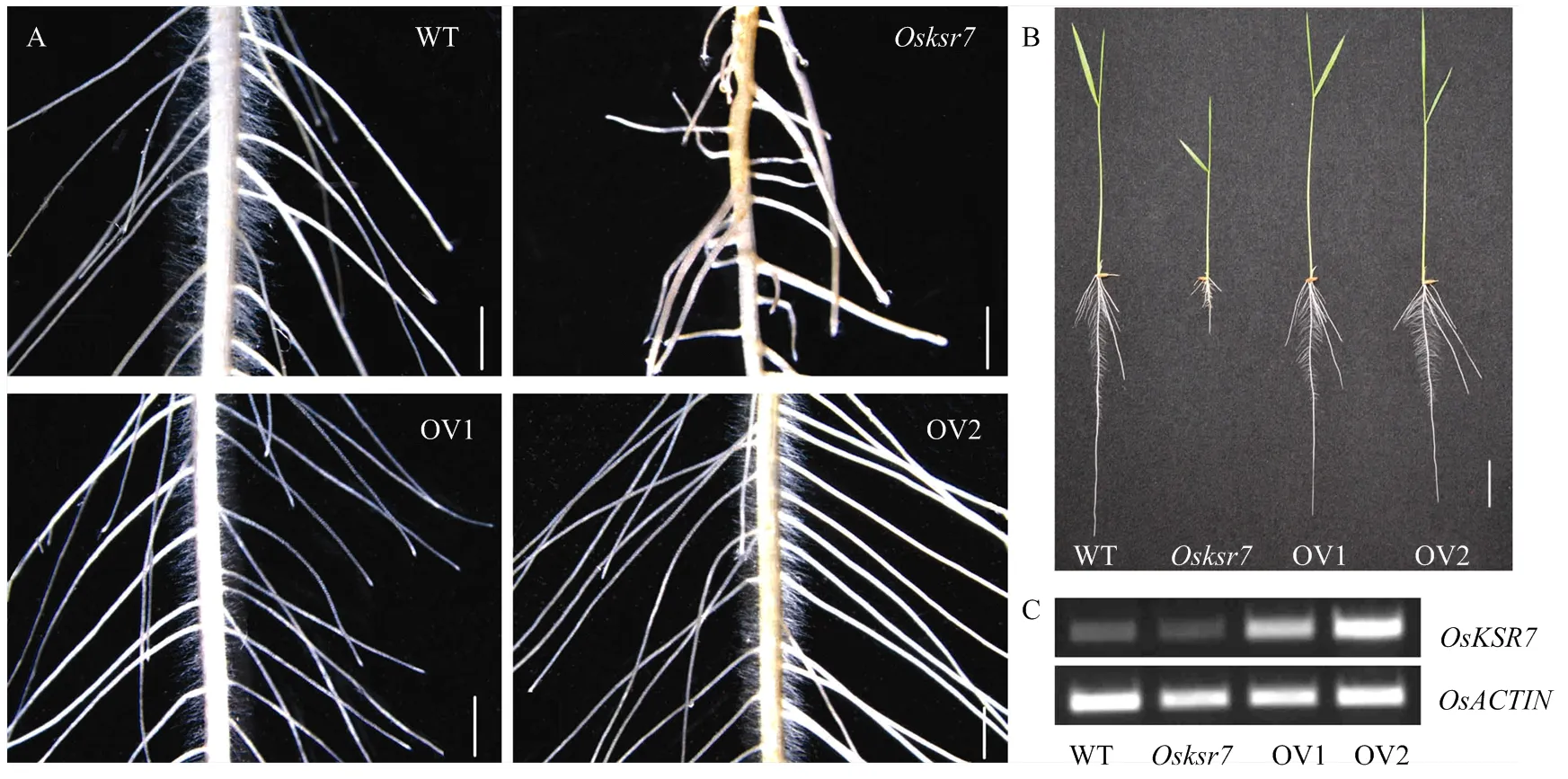
A:野生型(WT)、突变体Osksr7和2个超表达转基因回复株系(OV1、OV2)的主根体视镜照片,bar=1 mm;B:野生型(WT)、突变体Osksr7和2个超表达转基因回复株系(OV1、OV2)的全株表型,bar=2 cm;C:RT-PCR检测OsKSR7的表达
3 讨论
3.1 短根突变体Osksr7突变基因的确定
水稻根系的发育是一个复杂的生物学过程,参与该过程的基因很多。目前报道的根系发育调控基因,多具有“一因多效”性[7]。本研究报道的突变体的主根、不定根、侧根和根毛的生长发育均受到抑制,且成熟期穗数、茎秆粗细和结实率等重要农艺性状都受到显著影响,说明是一个与水稻生长和单株产量密切相关的重要功能基因。通过基因定位将定位在水稻第11染色体上,迄今为止,第11染色体上仅报道了一个编码谷氨酰tRNA酰胺基转移酶B亚基的基因(LOC_Os11g34210)参与根系发育,突变体主根生长迟缓,但是不定根、侧根以及根毛的生长与野生型相似,苗高也与野生型相似[19]。候选基因测序发现,突变体在LOC_Os11g24560第1个外显子上发生了一个T-A碱基替换,导致编码的Trp突变为Arg。RT-PCR表明突变基因的表达水平在野生型和突变体中无显著差异,35S启动子驱动的的回复载体能够使突变体的表型回复成野生型,证实的单碱基突变是造成突变表型的原因。
3.2 突变基因的功能分析
生物信息学分析表明LOC_Os11g24560是COPII有被小泡的SEC23亚基在水稻中的同源蛋白。COPII小泡是真核生物蛋白质分泌途径中的重要组分,介导蛋白质从ER运向Golgi,在保持细胞内各个细胞器动态平衡中发挥重要功能[20]。COPII小泡由5种蛋白亚基组成,分别是小G蛋白Sar1、内衣被组分SEC23和SEC24,外衣被组分SEC13和SEC31,这5个细胞质蛋白在所有真核生物中都高度保守[21]。SEC23作为COPII小泡的内衣被蛋白,折叠形成5个明显的结构域:α螺旋、β折叠、锌指结构、主干区域和C端区域[22]。研究发现不同的物种中存在不同数目的SEC23亚型,酵母中只有1个SEC23亚型(SEC23p)[23],哺乳动物有2个SEC23亚型(SEC23A和SEC23B)[24],拟南芥SEC23有7个亚型(AtSEC23A-G)[25]。SEC23将COPII小泡的各个亚基连接起来,同时也是SAR1的GTP酶激活蛋白(GAP)。研究还发现SEC23能够和其他蛋白相互作用,调控COPII小泡形成的多种过程[26-28]。
已有研究表明SEC23的部分保守氨基酸残基具有重要作用。酵母SEC23蛋白的第722位精氨酸是其GAP活性所必需的[22]。人SEC23A第382位苯丙氨酸发生突变会降低其对SEC31的亲和力并导致颅缝异型增生病[29-30]。斑马鱼Sec23A第402位亮氨酸突变后表现出和人颅缝异型增生病类似的颅面发育缺陷[31]。研究发现拟南芥AtSEC23A蛋白中保守天冬氨酸残基是其与AtSAR1特异性结合所必需的[32]。本研究的第25位色氨酸突变为精氨酸后即导致严重的根系发育和农艺性状缺陷,说明该氨基酸残基具有潜在的重要功能。
目前,关于SEC23参与调控植物生长发育的研究较少,主要在拟南芥中进行。Tanaka等[33]研究发现AtSEC24B和AtSEC24C参与雌雄胚子发育,且功能冗余。AtSEC24A对雄性育性[34]、内质网-高尔基体完整性[35]和维持萼片细胞大小分化[36]都是不可或缺的。Zeng等[32]研究表明AtSEC23A和AtSAR1发生特异性结合才能发挥输出内质网蛋白功能。最近研究发现AtSEC23A和AtSEC23D在调控花粉壁形成、花粉粒外壁分化和绒毡层发育中起重要作用[25]。水稻中还没有的相关研究报道。的克隆为揭示SEC23调控水稻根系性状的遗传机理奠定基础,也为水稻根系分子育种提供了基因资源。
4 结论
EMS诱变获得一个水稻短根突变体,其结实率等重要农艺性状受到显著影响。突变表型受1对隐性核基因控制,为COPII有被小泡的SEC23亚基突变所致。SEC23在调控水稻根系发育及单株产量中起重要作用。
[1] GIEHL R F, GRUBER B D, VON W N. It's time to make changes: modulation of root system architecture by nutrient signals., 2014,65(3): 769-778.
[2] MEISTER R, RAJANI M S, RUZICKA D, SCHACHTMAN D P. Challenges of modifying root traits in crops for agriculture., 2014, 19(12): 779-788.
[3] Villordon A Q, Ginzberg I, Firon N. Root architecture and root and tuber crop productivity., 2014, 19(7): 419-425.
[4] 石庆华, 黄英金, 李木英, 徐益群, 谭雪明, 张佩莲. 水稻根系性状与地上部的相关及根系性状的遗传研究. 中国农业科学, 1997, 30(4): 62-68.
SHI Q H, HUANG Y J, LI M Y, XU Y Q, TAN X M, ZHAGN P L. Studies on the heredity of root characteristics and correlation between the characteristics of roots and upperground parts in rice., 1997, 30(4): 62-68. (in Chinese)
[5] 洛育. 黑龙江省水稻根系性状与地上部性状的关系. 中国农学通报, 2010, 26(14): 165-168.
LUO Y. Dependence relation of aerial part traits and root traits of rice in heilongjiang province., 2010, 26(14): 165-168. (in Chinese)
[6] ROGERS E D, BENFEY P N. Regulation of plant root system architecture: implications for crop advancement., 2015, 32(32C): 93-98.
[7] REBOUILLAT J, DIEVART A, VERDEIL J L, ESCOUTE J, GIESE G, BREITLER J C, GANTET P, ESPEOUT S, GUIDERDONI E, PÉRIN C. Molecular genetics of rice root development., 2009, 2(1): 15-34.
[8] E Z-G, GE L, WANG L. Molecular mechanism of adventitious root formation in rice., 2012, 68(3): 325-331.
[9] YU P, GUTJAHR C, LI C, HOCHHOLDINGER F. Genetic control of lateral root formation in cereals., 2016, 21(1): 951-961.
[10] CHEN Z C, YAMAJI N, KASHINO-FUJII M, MA J F. A cation-chloride cotransporter gene is required for cell elongation and osmoregulation in rice., 2016, 171(1): 494-507.
[11] DING W, LIN L, ZHANG B,XIANG X, WU J, PAN Z, ZHU S. OsKASI, a β-ketoacyl-[acyl carrier protein] synthase I, is involved in root development in rice (L.)., 2015, 242(1): 203-213.
[12] JIA L, WU Z, HAO X, CARRIE C, ZHENG L, WHELAN J, WU Y, WANG S, WU P, MAO C. Identification of a novel mitochondrial protein, short postembryonic roots 1 (SPR1), involved in root development and iron homeostasis in., 2011, 189(3): 843-855.
[13] JIA L, ZHANG B, MAO C, LI J, WU Y, WU P, WU Z. OsCYT-INV1 for alkaline/neutral invertase is involved in root cell development and reproductivity in rice (L.)., 2008, 228(1): 51-59.
[14] ZHAO Y, HU Y F, DAI M Q, HUANG L M, ZHOU D X. The WUSCHEL-related homeobox gene WOX11 is required to activate shoot-borne crown root development in rice., 2009, 21(3): 736-748.
[15] DING W, TONG H, ZHENG W, YE J, PAN Z, ZHANG B, ZHU S. Isolation, characterization and transcriptome analysis of a cytokinin receptor mutant, 2017, 8: 88.
[16] 丁沃娜, 童艳丽, 吴晶, 朱世华. 一个水稻短根毛突变体的鉴定和基因定位. 中国农业科学, 2011, 44(21): 4333-4339.
DING W N, TONG Y L, WU J, ZHU S H. Identification and gene mapping of a novel short root hair mutant in rice., 2011, 44(21): 4333-4339. (in Chinese)
[17] CHEN S, JIN W, WANG M, ZHANG F, ZHOU J, JIA Q, WU Y, LIU F, WU P. Distribution and characterization of over 1000 T-DNA tags in rice genome., 2003, 36(1): 105-113.
[18] MICHELMORE R W, PAPAN I, KESSELI R V. Identification of markers linked to disease-resistance genes by bulked segregant analysis: a rapid method to detect markers in specific genomic regions by using segregating populations., 1991, 88(21): 9828-9832.
[19] QIN C, CHENG L, ZHANG H, HE M, SHEN J, ZHANG Y, WU P. OsGatB, the subunit of tRNA-dependent amidotransferase, is required for primary root development in rice., 2016, 7: 599.
[20] VENDITTI R, WILSON C, DE MATTEIS M A. Exiting the ER: what we know and what we don’t., 2014, 24(1): 9-18.
[21] MILLER E A, BARLOWE C. Regulation of coat assembly-sorting things out at the ER., 2010, 22(4): 447-453.
[22] BI X, CORPINA R A, GOLDBERG J. Structure of the Sec23/24-Sar1 pre-budding complex of the COPII vesicle coat., 2002, 419(6904): 271-277.
[23] ROBINSON D G, HERRANZ M C, BUBECK J, PEPPERKOK R, RITZENTHALER C. Membrane dynamics in the early secretory pathway., 2007, 26(4): 199-225.
[24] SCHWARZ K, IOLASCON A, VERISSIMO F, TREDE N S, HORSLEY W, CHEN W, PAW B H, HOPFNER K P, HOLZMANN K, RUSSO R, ESPOSITO M R, SPANO D, DE FALCO L, HEINRICH K, JOGGERST B, ROJEWSKI M T, PERROTTA S, DENECKE J, PANNICKE U, DELAUNAY J, PEPPERKOK R, HEIMPEL H. Mutations affecting the secretory COPII coat component SEC23B cause congenital dyserythropoietic anemia type II., 2009, 41(8): 936-940.
[25] ABOULELA M, NAKAGAWA T, OHSHIMA A, NISHIMURA K, TANAKA Y. The arabidopsis COPII components, AtSEC23A and AtSEC23D, are essential for pollen wall development and exine patterning., 2018, 69(7): 1615-1633.
[26] FROMME J C, ORCI L, SCHEKMAN R. Coordination of COPII vesicle trafficking by Sec23., 2008, 18(7): 330-336.
[27] KUEHN M J, HERRMANN J M, SCHEKMAN R. COPII-cargo interactions direct protein sorting into ER-derived transport vesicles., 1998, 391(6663): 187-190.
[28] MANCIAS J D, GOLDBERG J. The transport signal on Sec22 for packaging into COPII-coated vesicles is a conformational epitope., 2007, 26(3): 403-414.
[29] BOYADJIEV S A, FROMME J C, BEN J, CHONG S S, NAUTA C, HUR D J, ZHANG G, HAMAMOTO S, SCHEKMAN R, RAVAZZOLA M, ORCI L, EYAID W. Cranio-lenticulosutural dysplasia is caused by a SEC23A mutation leading to abnormal endoplasmic-reticulum-to-Golgi trafficking., 2006, 38(10): 1192-1197.
[30] FROMME J C, RAVAZZOLA M, HAMAMOTO S, AL-BALWI M, EYAID W, BOYADJIEV S A, COSSON P, SCHEKMAN R, ORCI L. The genetic basis of a craniofacial disease provides insight into COPII coat assembly., 2007, 13(5): 623-634.
[31] LANG M R, LAPIERRE L A, FROTSCHER M, GOLDENRING J R, KNAPIK E W. Secretory COPII coat component Sec23a is essential for craniofacial chondrocyte maturation., 2006, 38(10): 1198-1203
[32] ZENG Y, CHUNG K P, LI B, LAI C M, LAM S K, WANG X, CUI Y, GAO C, LUO M, WONG K B, SCHEKMAN R, JIANG L. Unique COPII component AtSar1a/AtSec23a pair is required for the distinct function of protein ER export in., 2015, 112(46): 14360-14365.
[33] TANAKA Y, NISHIMURA K, KAWAMUKAI M, OSHIMA A, NAKAGAWA T. Redundant function of twoCOPII components, AtSec24B and AtSec24C, is essential for male and female gametogenesis., 2013, 238(3): 561-575.
[34] CONGER R, CHEN Y, FORNACIARI S, FASO C, HELD MA, RENNA L, BRANDIZZI F. Evidence for the involvement of theSEC24A in male transmission., 2011, 62(14): 4917-4926.
[35] NAKANO R T, MATSUSHIMA R, UEDA H, TAMURA K, SHIMADA T, LI L, HAYASHI Y, KONDO M, NISHIMURA M, HARA-NISHIMURA I. GNOMLIKE1/ERMO1 and SEC24a/ ERMO2 are required for maintenance of endoplasmic reticulum morphology in., 2009, 21(11): 3672-3685.
[36] QU X, CHATTY P R, ROEDER A H. Endomembrane trafficking protein SEC24A regulates cell size patterning in., 2014, 166(4): 1877-1890.
Cloning and Functional Analysis of a Root Development Related GeneL.)
Zhou Jiaqin1, Zhu Junzhao2, YangSixue2, Zhu Zhoujie2, Yao Jie2, Zheng Wenjuan2, Zhu Shihua2, Ding Wona2
(1School of Marine Science, Ningbo University, Ningbo 315211, Zhejiang;2College of Science and Technology, Ningbo University, Ningbo 315212, Zhejiang)
【Objective】The root system of rice is an important agronomic trait closely related to shoot growth and yield. Identifying new root development-related genes in rice will help further clarification of the underlying molecular mechanisms.【Method】In the present study, a mutant with significantly shorter roots was isolated from an EMS (ethyl methane sulfonate)-generated mutant library of rice and designated as(). By using solution culture and field planting, analysis of young seedling phenotype and main agronomic traits of mature plants was conducted. The F2populations from crossing ofwithKasalath andNipponbare were used for genetic analysis and map-based cloning, respectively. Candidate genes were examined by DNA sequencing. Complementation analysis of themutant with the protein-coding region ofdriven by the 35S promoter was performed using-mediated transformation. 【Result】At the seedling stage, the elongation of primary roots, adventitious roots, lateral roots and root hairs inwas severely impaired. The length of primary roots, adventitious roots and lateral roots ofwas only 33%, 38.9% and 35.3% of those of the wild type, respectively. Nevertheless, the number of adventitious roots ofwas significantly increased when compared with the wild type. At the maturation stage, the agronomic traits ofwere also significantly compromized, including the shoot height, panicle number, clum thickness, seed setting rate, 1000-grain weight and length and width of flag leaves. Among them, the panicle number and seed setting rate ofdramatically decreased to only 56.3% and 37.3% of those of the wild type, respectively. Genetic analysis showed that the growth of F1plants from the crossing ofwithKasalath was similar to the wild type and the segregation ratio of wild type and mutant phenotype plants in the corresponding F2population fitted a ratio of 3:1, indicating that the mutant trait ofwas controlled by a single recessive nuclear gene. Thelocus was further mapped between InDel markers IND1 and IND2 on chromosome 11 with a physical distance of 143 kb, where there were 25 predicted genes with annotation. Sequencing analysis found a point mutation (T73to A) in the first exon of the genewithin this region in, resulting in an amino acid substitution (Trp25to Arg). The gene encodes a putative rice homolog of the SEC23 subunit of the coat protein complex II (COPII) involved in ER-to-Golgi transport. RT-PCR analysis revealed no significant difference in the expression level ofbetween the wild type and. Transformation ofwith the coding sequence ofdriven by the 35S promoter could successfully restore its growth defects, confirming that the mutation inwas responsible for the mutant phenotype of.【Conclusion】is a rice short root mutant, and yield-related agronomic traits are significantly suppressed in.is confirmed to be within the locus, which encodes the SEC23 subunit of the coat protein complex II (COPII).is not allelic to any previously reported rice root gene and is a newly identified regulator of root development in rice.
L.; short root mutant; genetic analysis; map-based cloning; functional complementation
10.3864/j.issn.0578-1752.2019.05.001
2018-07-18;
2018-08-26
国家自然科学基金(31300246)、浙江省自然科学基金(LY17C020002)、宁波市自然科学基金(2017A610291,2017A610292)
周佳琴,E-mail:zjq668679@163.com。朱俊兆,E-mail:zhujunzhaosw@163.com。周佳琴和朱俊兆为同等贡献作者。通信作者丁沃娜,E-mail:dwn@zju.edu.cn
(责任编辑 李莉)

