香港地区汉族人群下颌前突与候选基因GHR多态性关联研究
薛 凡,陈 红
0 引 言
下颌前突是一种常见的牙颌面畸形,临床特征为向前突出的下颌和前牙的反合,在各种族人群中的发病率不同,其中以东亚裔人群中的发病率较高,约为 15%[1⁃3]。近 50% 的患者需接受包括正颌外科手术等不同的矫正治疗,以获得正常的咬合关系及面部容貌的改善。研究表明,遗传因素和各种环境因子相互作用于处于生长发育期的下颌骨髁状突软骨是造成下颌骨前突的主要原因[5]。近年来的家族和双胞胎研究也表明,遗传因素在下颌骨前突的发生中占有重要作用,下颌骨前突是一种具有多基因遗传模式的复杂疾病[4⁃6]。
目前国内外对下颌骨前突易感基因定位的研究主要集中在连锁分析研究。染色体位点1p22⁃p36和12q13⁃q24被认为是最有可能包含有下颌前突易感基因的位点[7⁃10]。关联研究发现位于1p22⁃p36位点内与下颌前突阳性关联的基因有EPB41、MATN1、SSX2IP和PLXNA[9,11⁃12]。此外也有研究发现,位于12q13⁃q24位点内的阳性关联基因有COL2A1、MYO1H、TGFB3 和LTBP2[10,13⁃15]。除了以上位点内根据生物学功能与软骨及骨生长和颅面发育等相关的候选基因,研究表明GHR基因对颅颌面骨的生长发育有重要的影响,但GHR与下颌前突的关联仍不能明确[16⁃21]。本研究对 211 例下颌前突病例和224例对照的GHR基因进行SNP基因分型,通过对SNP及其单倍型分析,鉴定与下颌前突关联的易感基因。
1 资料与方法
1.1 研究对象收集2006年9月至2009年6月期间香港大学牙医学院菲腊牙科医院登记注册患者。包括下颌前突患者211例(下颌前突组),其中男108例、女103例;对照组均为咬合关系与面形正常患者(n=224),其中男115例、女109例。纳入标准:I类错牙合的上下颌第一磨牙呈关系中性,0°<ANB角<5°;下颌前突的基本临床特征评估包括是否存在前牙反牙合(10,16)。下颌前突最终诊断标准基于头影测量关键参数ANB角 <⁃2°(17,18)。排除唇腭裂患者。所有研究对象均为现居住在香港的汉族居民。本研究经香港大学及医管局港岛西医院联网研究伦理委员会批准(批准号:UW07⁃102),所有调查取样均经受试者同意并签署知情同意书。
1.2 SNPs选择和基因分型通过HapMap和NCBI dbSNP数据库(www.ncbi.nlm.nih.gov/snp)选择SNPs。仅选择中国汉族人群中具有最小等位基因频率(mi⁃nor allele frequency,MAF)至少 10% 的 SNPs。选择7个位于GHR基因的tSNPs进行基因分型。抽取受试者外周血1~2 mL,用乙二胺四乙酸二钾(EDTA⁃K)抗凝,使用Omega Bio⁃Tek DNA提取试剂盒提法提取全基因组DNA(Doraville,GA,USA),紫外分光光度计和0.8%琼脂糖凝胶电泳检测DNA浓度和纯度,置于⁃20℃保存备用。所有DNA样本按照要求在96孔板上稀释及排列并被送到指定地点(香港大学基因组研究中心),在Sequenom MassArray平台(San Diego,CA,USA)上对每个样本进行SNPs基因分型。根据NCBI数据库中的人类基因组序列,选取每个SNP两侧的侧翼序列(250~300 bp)用于设计PCR引物。每个SNP基因分型检出率>98%,重复校验的成功率>99.5%。
1.3 统计学分析群体基因型频率经Hardy⁃Wein⁃berg平衡检验(www.oege.org/software),检验水准α=0.01,P>0.01被认为为符合Hardy⁃Weinberg遗传平衡。采用卡方检验对每个SNP基因型分布和等位基因频率进行组间比较,以P≤0.05为差异有统计学意义。等位基因关联强度以比值比(OR)表示。对每一对SNPs进行连锁不平衡评估,并通过Haplo⁃view 4.1软件进行单倍型分析(Broad Institute,Cam⁃bridge,MA,USA)。
2 结 果
2.1 SNPs关联分析 所有7个tSNPs分别检验后均符合Hardy⁃Weinberg平衡定律,每个SNP等位基因的MAF符合NCBI dbSNP数据库的信息。GHR基因中7个tSNPs的选择和基因分型见表1。SNP rs6898743与下颌前突的关联分析显示2组间基因型和等位基因频率有显著性差异(P=0.036、P=0.015);下颌前突组rs6898743等位基因G频率较对照组减少,具有显著性减低下颌前突的风险(OR=0.72、95%CI 0.55~0.94),见表2。

表1 SNPs选择和基因分型Table 1 Selection and genotyping of the SNPs of the GHR gene

表2 GHR基因SNP位点rs6898743的关联分析Table 2 Results of the association analysis on the SNP locus rs6898743 of the GHR gene
2.2 连锁不平衡与单倍型分析GHR基因中的7个tSNPs形成1个连锁不平衡区块,共构建了6个单倍型。连锁不平衡分析显示两组间比较单倍型GAA和GAG有显著性差异(P=0.014、P=0.012)。见图1。
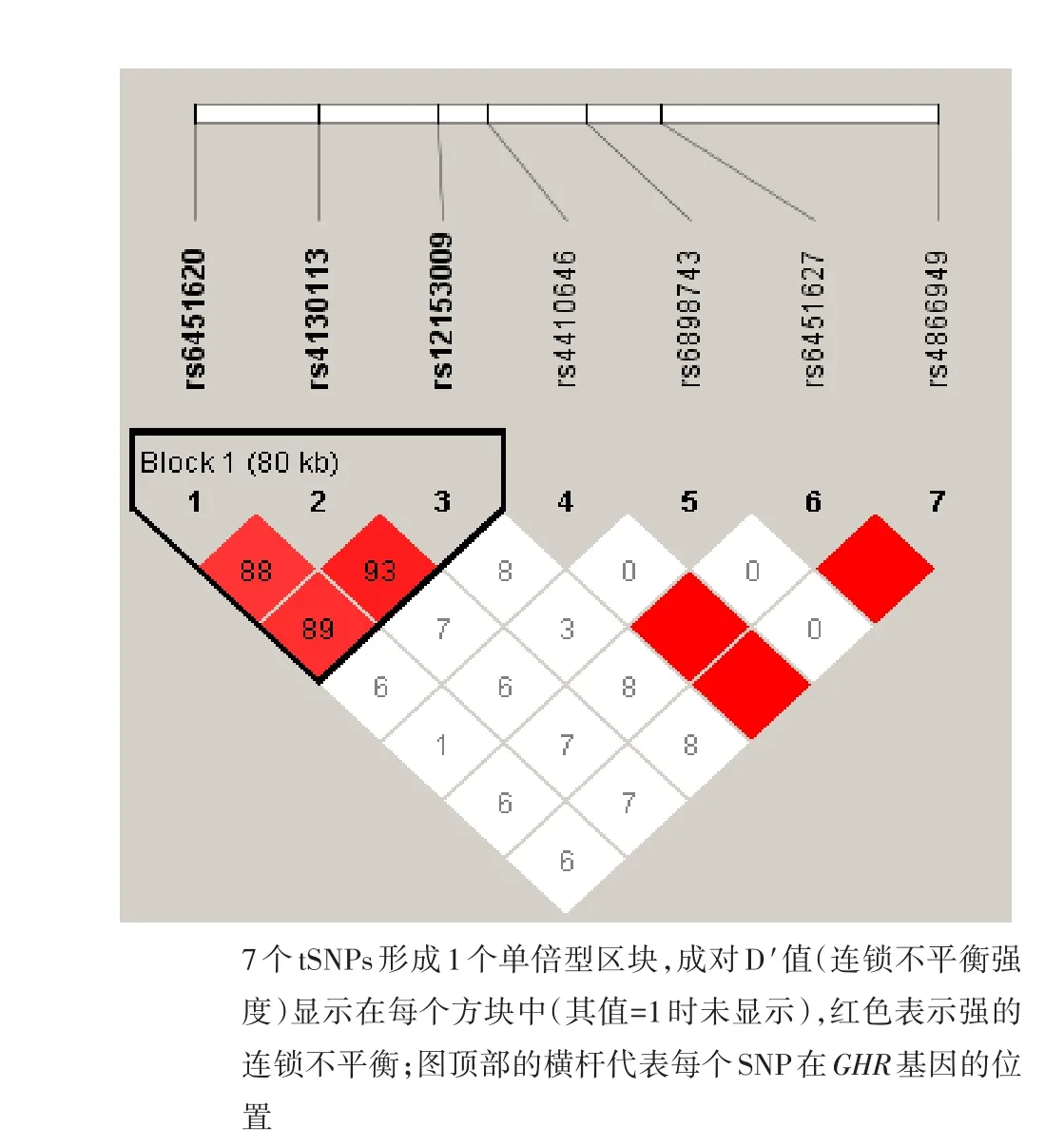
图1 GHR基因的连锁不平衡和单倍型区块Figure 1 Linkage disequilibrium and haplotype blocks of the GHR gene
3 讨 论
GHR是生长激素发挥作用的生理基础。生长激素必须与靶细胞表面的GHR结合,诱导GHR分子同源二聚化,然后激活细胞内一系列信号传导。近年来的一些研究发现,GHR基因的多态性可能导致GHR在不同个体中的表达水平或功能上的差异,进而影响生长激素生物学效应的发挥,其突变的位点多导致GHR细胞外区功能缺陷,也可引起细胞内信号转导障碍,导致IGF⁃1的分泌变化,主要影响软骨的生长,造成生长发育障碍,以及对某些疾病的易感性。GHR基因定位于第5号染色体近端短臂上p12~p13.1,是一个由单一基因编码含620个氨基酸的跨膜糖蛋白,是促乳素/生长激素/细胞因子/促红细胞生成素受体超家族成员之一。
近年来一些研究发现GHR基因多态性与下颌发育生长有显著性关联[16⁃17],分别有中国、日本和韩国研究结果表明GHR基因多态性与东亚人群下颌高度有相关性,其中日本学者的研究首先报道正常日本人口中GHR基因P56IT多态性影响下颌支长度;对中国人群的研究表明GHR基因I526L多态性与正常个体的下颌支长度有关[18];而韩国研究则并不支持GHR基因I526L多态性与正常个体的下颌支长度有关,其研究显示有效的下颌长度和下面部高度与SNP位点P561T变异有关[19]。Bayram等[20]研究显示GHR可能是下颌前突群体下颌骨形态发生的候选基因,同样显示有效的下颌长度和下面部高度与SNP位点P561T变异有关。此外,有研究显示GHR基因中SNP位点P561T的杂合错义突变在儿童时期影响其下颌骨的生长发育[21]。
然而,以上研究并没有直接证据表明GHR基因多态性与下颌前突的关联性。本研究通过香港地区435例病例对照人群发现GHR基因中SNP rs6898743与下颌前突存在统计学意义的关联。SNP关联分析显示SNP rs6898743的基因型和等位基因频率两组间差异统计学意义。分析结果表明,GHR基因多态性与中国人群下颌前突有阳性关联。连锁不平衡分析显示7个tSNPs在GHR基因中形成1个单倍型区块,单倍型GAA和GAG在下颌前突组和对照组组间比较差异有统计学意义。本研究只选择一组平均分布于GHR基因中的7个tSNPs,均位于内含子,关联分析仍需调查更多的SNPs,进一步对GHR基因内的tSNPs进行基因分型才能明确与下颌前突的风险关联。
本研究通过对下颌前突的候选基因GHR进行评估,候选基因的选择是基于其生物学功能和遗传学研究证据,提出GHR基因可能是一个新的可用于研究中国汉族人群下颌前突遗传危险因素的易感基因。鉴于各种被研究人群的数量和结果差异性较大,关于GHR基因多态性是否与下颌前突存在关联性的仍需不同种族人群的重复性研究,下颌前突的遗传病因学仍有待进一步研究。

