香菇种质资源遗传多样性研究
陈小敏,吴海冰 ,辜运富
(1.四川省绵阳市农业科学研究院食用菌研究所,四川 绵阳 621000;2.四川农业大学资源学院,成都 温江 611130)
香菇作为世界第2大食用菌,味道鲜美,营养丰富,被称为“山珍”和“植物皇后”。我国拥有丰富的香菇菌种资源,人工种植地主要集中在河南、福建、四川等地[1-2]。香菇子实体的表型特征容易受环境因素的影响而出现不稳定,生长环境不同导致同一菌种在形态特征上表现出明显的差异,所以仅靠传统形态学特征对香菇进行分类和鉴定存在一定的局限性。分子生物学的迅速发展,分子水平鉴定方法突破了传统方法的局限,为学者们从分子水平上研究食用菌差异性和亲缘关系提供了有力的手段。
DNA分子标记技术不受环境因子以及基因表达与否的限制,具有较高的多态性[3]。相较于形态学特征以及生理生化指标,分子标记技术因客观性更强、操作简便易行、特异性好、准确性高而越发受到关注。基于DNA片段的分子标记技术,能够准确反映生物体由于遗传变异所引发的核苷酸序列的差异性,在食用菌菌株遗传多样性研究、菌株鉴定、基因定位、种质资源评价等领域发挥着不可或缺的作用[4]。目前研究食用菌遗传多样性,应用较广泛的DNA分子标记技术主要包括:ITS、AFLP、SRAP、ISSR和RFLP等。
ISSR技术能较好地揭示和分析不同物种的遗传多样性和遗传结构,广泛应用于研究遗传多样性、基因定位、遗传图谱构建和种质资源鉴定等领域[5]。同样,ISSR技术也在广泛用于研究大型真菌基因组,检测基因组多个位点的差异性,例如研究香菇、黑木耳、金针菇、茯苓、蘑菇、羊肚菌等食用菌的遗传多样性、菌株鉴别[6]。利用ISSR技术研究不同遗传背景的野生毛木耳菌株的遗传差异性,有助于提升毛木耳育种水平,为培育优质的毛木耳新品种奠定理论依据[7]。利用ISSR技术研究中欧两国蜜环菌菌株的遗传多样性,研究表明地理隔离导致蜜环菌菌株表现出遗传差异,在系统发育树上位于各自的进化分支[8]。采用ISSR技术分析不同来源的桦褐孔菌菌株,成功地将21个菌株划分为6大类群[9]。以香菇为材料,采用ISSR技术确证了(TGTA)n微卫星基序的存在,在后续研究中结合ITS序列分析,有效鉴定了香菇生产菌株[10-11]。Zhang等研究17个中国香菇菌株ISSR指纹图谱,成功鉴定出全部供试菌株[12]。
目前香菇菌种混乱,品质稳定性较差且存在退化现象,严重阻碍了香菇产业的发展。因此本研究广泛收集香菇种质资源和野生资源,通过ITS和ISSR分子手段评价种质资源的遗传多样性,为后续杂交育种、原生质体融合育种提供理论依据。
1 材料与方法
1.1 供试菌株
供试香菇菌株共计75株,引自全国各地,从上海食用菌研究所、华中农大菌种实验中心、福建三明真菌研究所等地引进香菇资源53份。野生资源22份,采自四川石棉、泸定和冕宁等地,具体信息见表1所示。
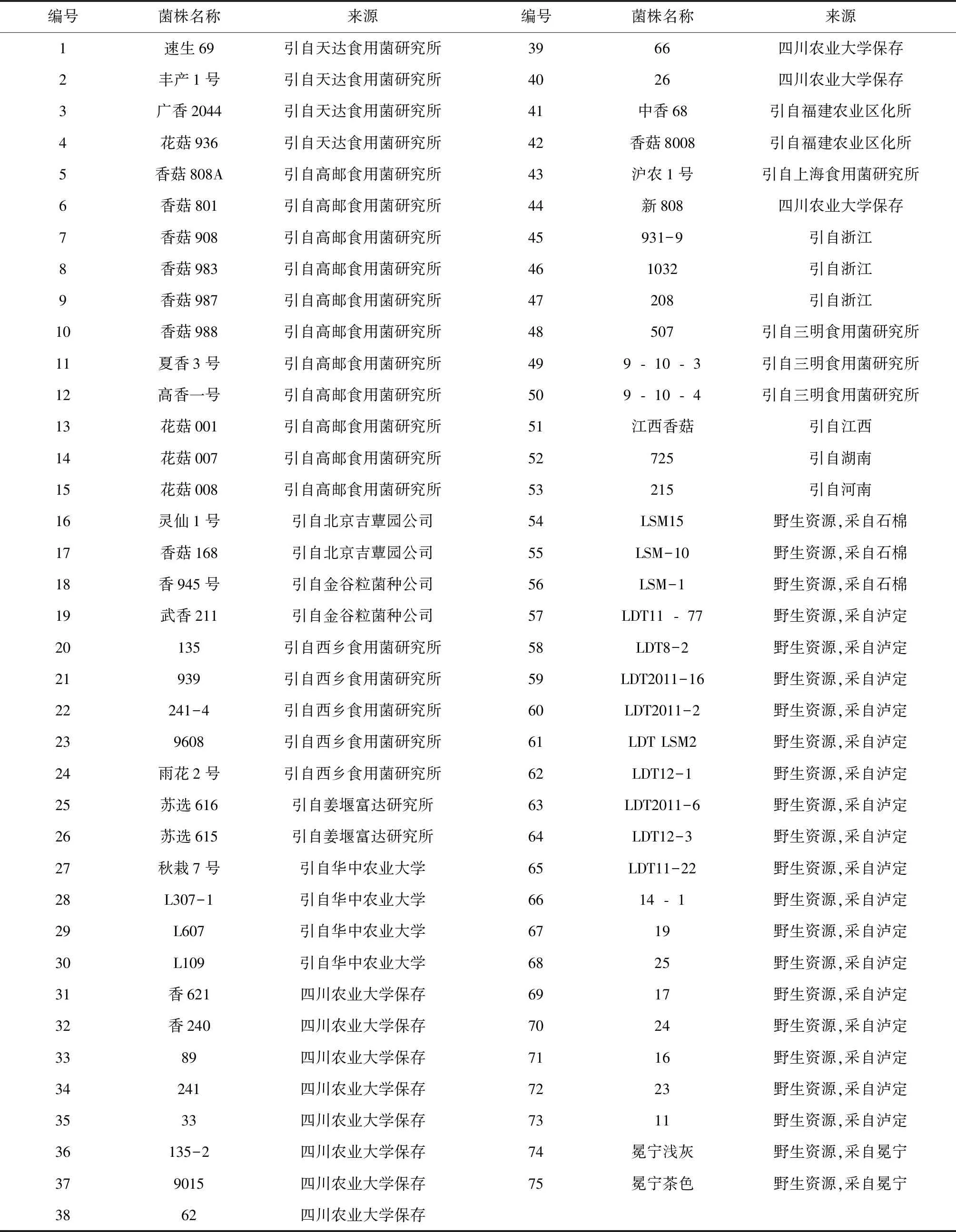
表1 供试菌株
1.2 野生资源的分离及引进资源的活化
PDA固体培养基配制:土豆去皮切成小块,称取200g,加入适量的水,沸水煮20min,经4层纱布过滤,取滤液[13]。往滤液中加无水葡萄糖20g、琼脂20g煮沸,最后加水补足到1000mL,pH自然。
野生香菇菌株分离:清理子实体表面杂质,用75%酒精对表面进行消毒。撕开子实体,用镊子取黄豆大小菌肉置于PDA斜面培养基中,25℃恒温避光培养。查看香菇菌丝的生长情况,选取未污染的菌丝进行纯化。
1.3 香菇遗传多样性研究
1.3.1 香菇菌株DNA提取 利用Ezup柱式真菌基因组DNA抽提试剂盒(上海生工Sangon Biotech)提取供试香菇样品的DNA。挑取培养基中表面的菌皮,用滤纸吸干菌丝表面水分,用液氮研磨,按照试剂盒给定的步骤依次进行操作。在含EB的1%琼脂糖凝胶上水平电泳,120 V/cm电泳10min,以DNA MarkerDL 2000作为分子量标准,用凝胶成像仪检验DNA条带,分光光度计检测DNA的质量和浓度。DNA纯度高,无降解,RNA干扰小,可进行后续实验,提取出的DNA于-20℃保存备用。
1.3.2 ITS扩增 用通用引物ITS1(5’-GGAAGTAAAAGTCGTAACAAGG-3’)和ITS4(5’-TCCTCCGCTTATTGATATGC-3’)对75株香菇菌株的ITS序列进行扩增[14]。反应体系为30μL:2×Taq PCR Master Mix 15μL(TIANGEN)、引物ITS1/ITS4各0.5μL(10μmol.L-1)、DNA模板1μL(10ng.μL-1),加ddH2O补齐至30μL。扩增程序:94℃预变性5min;94℃变性1min,55℃退火1min,72℃延伸1min,32个循环;72℃最终延伸1min;10℃终止反应[15]。取PCR产物3μL在含EB的1%琼脂糖凝胶上水平电泳检测,120V/cm电泳20min,以DNA MarkerDL2000作为分子量标准,自动凝胶成像系统拍照检测。将其产物交由擎科生物技术有限公司(成都)完成测序,向NCBI提交供试香菇菌株的ITS序列,获得基因登录号。
1.3.3 ISSR扩增 在前期的试验中,选用引物P1-P49进行ISSR扩增,结果表明引物P8(5’-GGAGGAGGAGGAGGA-3’)扩增效果最佳,谱带稳定、清晰、多态性丰富。反应体系为20μL:2×Taq PCR Master Mix 10μL(TIANGEN)、DNA模板1μL(10ng.μL-1)、引物P82μL(10μmol.L-1),ddH2O补齐至20μL。扩增程序:94℃预变性2min;94℃变性1min,52℃复性1min,65℃延伸8min,30个循环;65℃最终延伸16min;4℃终止反应[15]。取PCR产物8μL在含EB的2%琼脂糖凝胶上水平电泳检测,80V/cm电泳1.5h,以DNA MarkerDL2000作为分子量标准,自动凝胶成像系统拍照检测。根据ISSR扩增出的谱带图,将同一位置条带出现与否记为1、0,构建数字化矩阵,用软件NTSYSpc2.1进行遗传聚类分析[16]。
1.4 数据处理
实验数据使用Excel进行平均值和标准值计算,SPSS进行方差、显著性和相关性分析。利用NTSYSpc2.1构建基于ISSR分析的聚类图谱。用DNAMAN分析香菇供试菌株间的相似性,在Genbank中下载相似度较高的序列,利用MEGA5的Neighbor-Joining tree程序构建基于ITS序列的系统发育树。
2 结果与分析
2.1 ISSR聚类分析
对供试香菇菌株进行ISSR-PCR扩增,筛选得到引物P8,扩增出的条带清晰,多态性及稳定性较高,样品间重复性较好。供试香菇ISSR-PCR扩增出的条带大小在2000 bp-100 bp之间,供试菌株间扩增出的条带表现出明显的差异性,表明不同香菇菌株间存在明显的遗传差异性。根据ISSR扩增出的条带进行聚类分析,得到香菇菌株间的聚类图谱(图1)。
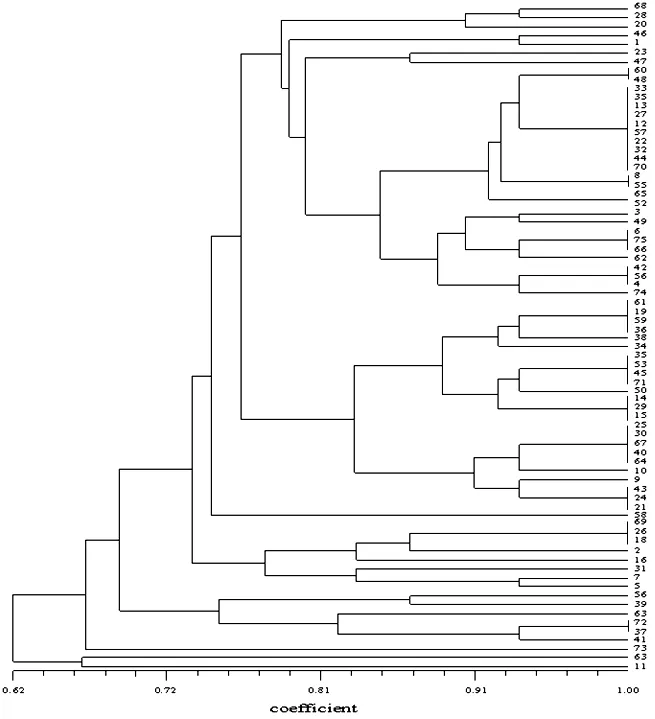
图1 供试菌株的ISSR聚类分析
从图中可以看出,供试的75株香菇菌株总体相似性为62%,具有明显的遗传多样性。遗传相似系数小于0.62时,所有的供试菌株都聚为一类。在0.74的水平上,供试菌株大致分为6类,其中菌株11、63、73单独聚为一类,菌株56、39、63、72和37聚为一类,菌株69、26、18、2、16、31、7和5聚为一类,其余的香菇菌株聚在一类。
2.2 供试菌株系统发育树
对供试75株菌株进行ITS-PCR扩增,PCR产物经电泳后检测得到单一条带(图2所示),对PCR产物进行测序。供试菌株的ITS序列片段长度大约为750bp,将测序得到的75个香菇菌株的ITS序列进行Blast(NCBI数据库)比对,比对结果显示供试菌株均与香菇菌株的相识度较高,这表明供试菌株均为香菇菌株。

图2 供试菌株ITS-PCR扩增条带
选择匹配度最高的香菇参比菌株,并提交至NCBI,并获得了基因登录号。以糙皮侧耳平菇(Pleurotusostreatus)和金针菇(Flammulinavelutiper)为外类群,对75个供试香菇菌株和5个GenBank参比序列构建系统发育树构建系统发育树(图3所示)。如图3所示,供试香菇菌株与4株香菇参比菌株聚在一起,而与外类群平菇明显分开。供试菌株大致可以分为5大类群,其中57、60、68、67、69、72、66和63聚为一类,54聚为一类,55聚为一类,61、73、62、64、65、58、71和56聚为一类,其余菌株聚为一类。其中57和60相识度为99%,说明这两个菌株的亲缘关系较近。
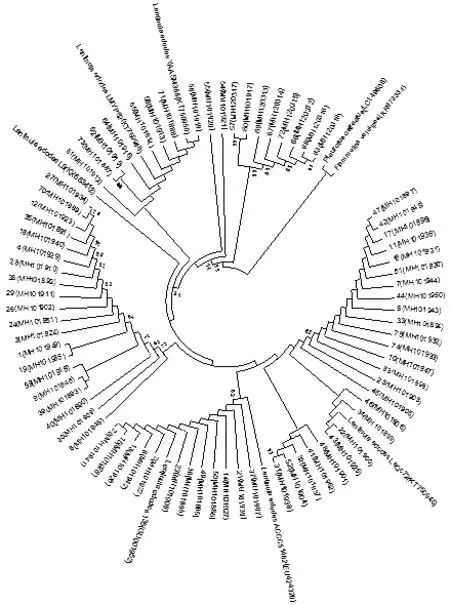
图3 供试菌株系统发育树
3 讨论
分子生物学的发展,推动了分子标记技术在食用菌遗传多样性上的研究,学者们采用DNA序列分析研究食用菌的系统发育学,自1994年ISSR分子标记技术提出以来,广泛用于评价灵芝、香菇等食用菌遗传多样性[17-18]和菌株的鉴定鉴别[19]。不同的香菇菌株的DNA序列存在差异,在ISSR-PCR扩增时不同香菇菌株DNA序列与引物P8结合,扩增出独特的ISSR条带,因此ISSR分子标记技术能反应出供试香菇菌株间的遗传多样性。在本试验中,供试香菇菌株的ISSR电泳图谱具有明显的差异性,聚类分析显示出相似系数小于0.62时,供试75株香菇菌株聚为一类,在相似系数为0.74时,供试菌株聚类6大类群,说明供试75株香菇菌株间表现出明显的遗传多样性。
基于ITS序列构建系统发育树是目前研究灵芝、香菇等食用菌种间、种内水平差异的主要分子手段。本研究中基于ITS序列构建的系统发育树将供试75株香菇菌株分为5大类群,其中菌株57(LDT11-77)和60(LDT2011.2)相似度99%,且这两个野生菌株均采集同一地区,则野生菌株LDT11-77和LDT2011.2疑是同种异名菌株。从系统发育树中可以看出,引进资源聚在一个类群,明显的与野生资源分开,这可能是四川野生资源与引进资源存在地理隔离,从而引起遗传差异。前人通过ITS测序研究中国香菇种质资源的遗传多样性,结果表明西南地区野生香菇资源遗传多样性最为丰富。从本研究系统发育树上看,采自于四川的野生野生资源分属于不同的类群,说明四川野生香菇资源具有丰富的遗传多样性。

