利用CRISPR/Cas9敲除人源细胞系中LMNA基因的研究
刘恒,李东明,朱兰玉,赖乐锦,闫婉云,陆玉双,韦伊,黄月琪,方媚,苏元港,杨芳,舒伟
利用CRISPR/Cas9敲除人源细胞系中基因的研究
刘恒1,李东明1,朱兰玉1,赖乐锦1,闫婉云1,陆玉双1,韦伊1,黄月琪1,方媚1,苏元港1,杨芳2,舒伟1
1. 广西医科大学基础医学院细胞生物学与遗传教研室,南宁 530021 2. 广西师范大学环境与资源学院,桂林 541004
基因编码A型和C型核纤层蛋白,参与细胞核核膜的组织,影响基因组稳定性并对细胞分化产生影响。人类肿瘤中表达异常普遍存在,其突变造成多种核纤层蛋白病,如Emery-Dreifuss 肌营养不良症(Emery-Dreifuss muscular dystrophy, EDMD)、扩张型心肌病(dilated cardiomyopathy, DCM)和儿童早老症(Hutchinson-Gliford progeria syndrome, HGPS)等。为进一步研究在细胞内的功能,本研究利用CRISPR/Cas9技术对体外培养的293T与HepG2细胞株的基因进行编辑,获得两株基因敲除(KO)的稳定细胞系。与野生型相比,KO细胞系增殖能力相对减弱,凋亡增加。同时,细胞形态上也发生显著改变,核膜凹凸不平。本研究首次报道了KO永生细胞系构建和形态研究结果,为后续基因功能研究和致病突变体研究奠定基础。
基因;CRISPR/Cas9;293T;HepG2;细胞形态
细胞核内核膜的核纤层在维持核结构和核稳定方面起到关键作用,也影响到基因的组织和表达[1~3]。核纤层由5种中间纤维蛋白组成,包括A型核纤层蛋白(Lamin A)、C型核纤层蛋白(Lamin C)、B1型核纤层蛋白(Lamin B1)、B2型核纤层蛋白(Lamin B2),以及核纤层相关蛋白。其中A型与C型核纤层蛋白由基因编码表达,基因的功能包括支撑细胞核膜并调控核膜的分解与重构,稳固染色质和核周转录因子,提供基因组需要的活动场所,还参与细胞骨架到细胞核之间的信号传导。此外,蛋白直接与核稳定性和细胞可塑性相关[4]。最近的研究表明,核纤层蛋白在人类恶性肿瘤中异常表达[5,6]。在前列腺癌、乳腺癌、结肠癌、卵巢癌和胃癌中,研究人员发现核纤层蛋白表达下调,而且常常与预后不良有关[7]。在结直肠癌、前列腺癌和乳腺癌的研究报告中,Lamin A/C的表达变化与预后存在关联[8]。基因突变与十几种退行性疾病有关,包括肌肉萎缩症(如Emery-Dreyfus肌肉萎缩症,EDMD)、外周神经病变(如 Charcot-Marie-牙病2B1型, CMT2B1)、脂肪代谢障碍、儿童早老综合征(如Hutchinson–Gilford早老综合征,HGPS),此外还有非典型沃纳综合征(Atypical Werner syndrome, AWS)、硬皮病(restrictive dermopathy, RD)[9~12]等。基因的不同突变能导致同种类型的疾病,而且同一碱基的不同替换也会导致不同的疾病,同样的突变在一些个体会导致疾病,一些个体却不会有临床症状,这些都表明了功能复杂而多样[13,14]。人们对于核纤层蛋白病中的基因型和表型之间的联系不甚了解,对基因的生物学功能也有待进一步研究。
Lamin A和C是由基因选择性剪切产生,位于细胞核内膜内侧,主要在已分化细胞内表达[15]。Lamin C是由基因在第10号外显子选择性剪切产生,与Lamin A功能类似,共同在细胞核膜结构和功能中发挥重要作用[16]。为研究基因在细胞内的功能,本研究利用CRISPR/Cas9技术对不同来源的体外培养细胞(293T和HepG2)中进行基因编辑,通过敲除基因使Lamin A/C都不表达,并对敲除后细胞系的形态和增殖情况进行了观察。
1 材料与方法
1.1 材料
293T与HepG2细胞由本实验室保存;PX459质粒购自美国Addgene公司。
1.2 gRNA设计与合成
利用美国麻省理工学院 CRISPR Design 软件(http://crispr.mit.edu/)进行gRNA的设计,然后用NCBI的Primer-Blast对所设计的引物进行验证。一共设计4条gRNA,如表1所示。gRNA合成由上海生工生物工程技术服务有限公司负责完成。
1.3 载体构建
将合成的gRNA正义链和反义链进行复性处理形成双链,反应体系为正、反义链各1 μL,T4连接酶缓冲液(10×) 1 μL,T4多核苷酸激酶(PNK) 0.5 μL,双蒸水6.5 μL,95℃ 5 min,自然冷却至16℃保存10 min。然后利用Ⅰ内切酶对PX4594℃条件下剪切过夜,电泳切胶回收剪切后的质粒。将寡核苷酸双链与剪切后质粒用T4连接酶进行重组。反应体系为:PX459质粒 50 ng,寡核苷酸双链2 μL,T4连接酶1.5 μL,T4连接酶缓冲液1.5 μL,双蒸水补至15 μL。反应程序:16℃连接10 min后,4℃静置过夜。将重组后的载体悬液产物5~10 μL加入到50 μL的DH5α感受态细胞中,轻弹混匀后冰浴30 min,42℃热激90 s,冰上静置2 min,然后直接涂于氨苄抗性的LB平板。37℃培养箱过夜后挑3~5个白色菌落摇菌培养,提取质粒DNA进行测序验证。测序引物为:上游引物5′-GAGGGCCTATTTCCCATGAT- 3′,下游引物5′-GGGCGTACTTGGCATATGAT-3′。引物由上海生工生物工程技术服务有限公司负责合成。

表1 靶向人源LMNA基因敲除的gRNA序列
FO与RO分别为gRNA的正义链与反义链。
1.4 细胞转染
在6孔板中每孔加入2×106个细胞,12 h后PBS清洗两次;将脂质体(Lipofectamine 3000 Transfection reagent,美国Invitrogen) 10 μL与250 μL无血清培养基混匀,室温放置5 min,同时把5 μg重组质粒与250 μL无血清培养基混匀,室温静置5 min;然后将两种液体混匀,室温静置20 min,最后加入培养孔。在转染6~8 h后换完全培养基,24 h后更换新鲜完全培养基,并加入嘌呤霉素进行筛选(293T的工作浓度为2.0 ug/mL,HepG2的工作浓度为1.5 μg/mL)。
1.5 单克隆测序分析
在嘌呤霉素筛选3天后,用胰酶把细胞消化下来,利用有限稀释法稀释细胞至每1个/100 μL,然后在96孔板的每孔中加入100 μL细胞悬液,显微镜观察之后将只有单个细胞的培养孔做好标记;培养五天后更换新鲜培养基,细胞铺板80%以上消化细胞,一半提取DNA测序验证,一半细胞换六孔板扩大培养。
收集单克隆细胞,在提取DNA后进行PCR扩增。PCR体系为:DNA 100 ng,上下游引物各0.2 μL,2× Taq mastermix 25 μL,双蒸水补至50 μL;取PCR扩增产物进行1%琼脂糖凝胶,100 V、25 min电泳,得到目的条带后,剩余PCR扩增产物送测序。由于HepG2 gRNA的设计位点(PX-459-gRNA1和PX- 459-gRNA2)与293T gRNA的设计位点(PX-459- gRNA3和PX-459-gRNA4)不同,所以设计了2对不同的的引物。其中Primer1用于293T细胞,Primer2用于HepG2细胞。基因组测序引物如表2所示。由上海生工生物工程技术服务有限公司负责合成。PCR产物电泳切胶回收,在微量离心管中加入T载体1 μL (50 ng),加入等摩尔数PCR产物;加入含ATP的10× Buffer 1 μL,T4 DNA连接酶合适单位,用双蒸水补足至10 μL;低速离心,4℃连接过夜;转化,蓝白斑筛选并菌落PCR测序。
1.6 免疫荧光检测
将细胞爬片用PBS浸洗;用4%的多聚甲醛固定爬片15 min,然后用PBS浸洗玻片3次,每次3 min;再使用0.5% Triton X-100 (PBS配制)室温通透20 min;PBS浸洗玻片后用吸水纸吸干PBS,用5%脱脂奶粉(PBST)室温封闭30 min;吸水纸吸掉封闭液之后用稀释一抗(Lamin B, AF0219购自中国上海碧云天生物技术有限公司;β-actin, ab8226购自美国Abcam公司) 4℃孵育过夜;然后用PBST浸洗爬片,吸水纸吸干后滴加荧光二抗,室温孵育孵育1 h后用PBST浸洗爬片3次,每次3 min;最后用荧光显微镜观察。

表2 LMNA基因敲除位点的测序引物序列
Primer1用于293T细胞;Primer2用于HepG2细胞。F为正义链,R为反义链。
1.7 蛋白免疫印记检测
293T与HepG2的野生型和KO型细胞六孔板铺板24 h后,RIPA裂解液(加pmsf)裂解细胞收集蛋白,聚丙烯酰胺凝胶电泳后转PVDF膜。封闭后,相应一抗(Lamin A,ab8089和Lamin C,ab106682均购自美国Abcam公司) 4℃孵育过夜,二抗室温1 h。凝胶成像系统成像。
1.8 细胞增殖实验
把293T与HepG2的野生型和KO型细胞胰酶消化成单个细胞,在96孔板种板中每孔加入1500个细胞,37℃培养箱中培养;18 h后加入Cell Counting Kit-8,1 h后酶标仪450 nm测吸光度,每种细胞测3个孔,取平均值。每隔一天测一次吸光度,直到其中一种细胞铺板达70%以上就停止细胞增殖实验。
1.9 细胞凋亡实验
用无EDTA胰酶消化细胞长满50%~75%状态良好的293T与HepG2的野生型和KO型细胞,PBS清洗之后1000 r/min 3 min收集细胞。利用Annexin V-FITC/PI双染细胞凋亡试剂盒染色,避光孵育10 min后用流式细胞仪检测。
1.10 扫描电镜观察
收集离心细胞后,用3%戊二醛在4℃固定2 h后0.1 mmol/L PPS缓冲液4℃下浸泡清洗3次,每次10 min;然后用四氧化锇在4℃固定1 h,再用0.1 mmol/L PPS缓冲液4℃下浸泡清洗3次,每次15 min;依次从50%、70%、80%、90%、100% (3次)的乙醇浓度浸泡脱水,每次15 min;再使用100%六甲基二硅胺烷浸泡3次,每次10 min,然后放入真空干燥器抽真空干燥;粘样本到样本座,IB3 (IB5)离子溅射仪喷镀后放入电镜(VEGA 3 LMU型,捷克TESCAN公司)观察。
1.11 透射电镜观察
收集离心细胞,用3%戊二醛在4℃固定2 h;0.1 mmol/LPPS缓冲液4℃下浸泡清洗3次,每次10 min;用四氧化锇在4℃固定1 h;0.1 mmol/LPPS 缓冲液4℃下浸泡清洗3次,每次15 min;乙醇浓度依次从50%、70%、80%、90%、1∶1 (90%乙醇∶90%丙酮)、90%丙酮、100%丙酮(3次)浸泡脱水,每次15 min;1∶1(丙酮∶包埋剂)室温渗透1 h,纯包埋剂室温浸透2 h;环氧树脂包埋;依次从35℃ 15 h,45℃ 12 h,60℃ 24 h聚合;在修块、超薄切片与染色之后,利用电镜(H-7650型,日本HTACHI公司)观察。
2 结果与分析
2.1 PX-459-gRNA(LMNA)重组质粒构建和单克隆测序分析
设计并合成gRNA后,把gRNA与PX459载体连接。PX459具有嘌呤霉素抗性,方便于之后的的细胞单克隆筛选。在转染重组质粒与挑纯之后,提取单克隆细胞的DNA,扩增gRNA两侧400 bp左右序列,测序结果显示在gRNA附近位点开始出现杂峰,经人工比对后,杂峰中不存在原始序列,说明成功的剪切了293T和HepG2的两个等位基因。然后我们利用TA克隆测序法验证得出两株细胞的等位都缺失若干碱基(图1A)。将测序结果与全基因序列进行比对后,发现293T单克隆细胞株在等位基因分别缺失40和42个碱基,HepG2单克隆细胞株在等位基因分别缺失35和39个碱基(图1B)。
2.2 敲除细胞内LMNA基因表达分析
LMNA基因共有12个外显子,选择性剪切编码Lamin A和Lamin C蛋白。Lamin A 由1-12号外显子编码,Lamin C由1~10号外显子编码(图2A)。PX-459-gRNA1和PX-459-gRNA2(用于HepG2)的识别位点位于第9号外显子;PX-459-gRNA3和PX- 459-gRNA4 (用于293T)的识别位点在第7号外显子。Lamin A蛋白免疫印迹结果显示,293T KO和HepG2 KO不表达Lamin A (图2B)。免疫荧光技术分析结果显示,低倍镜下293T KO和HepG2 KO细胞 Lamin A (绿色)未见荧光信号(图2C)。上述两个结果显示,利用CRISPR/Cas9技术成功获得了敲除基因293T与HepG2细胞株。同时,Lamin C蛋白免疫印迹结果显示两株KO细胞也不能表达Lamin C (图2D)。
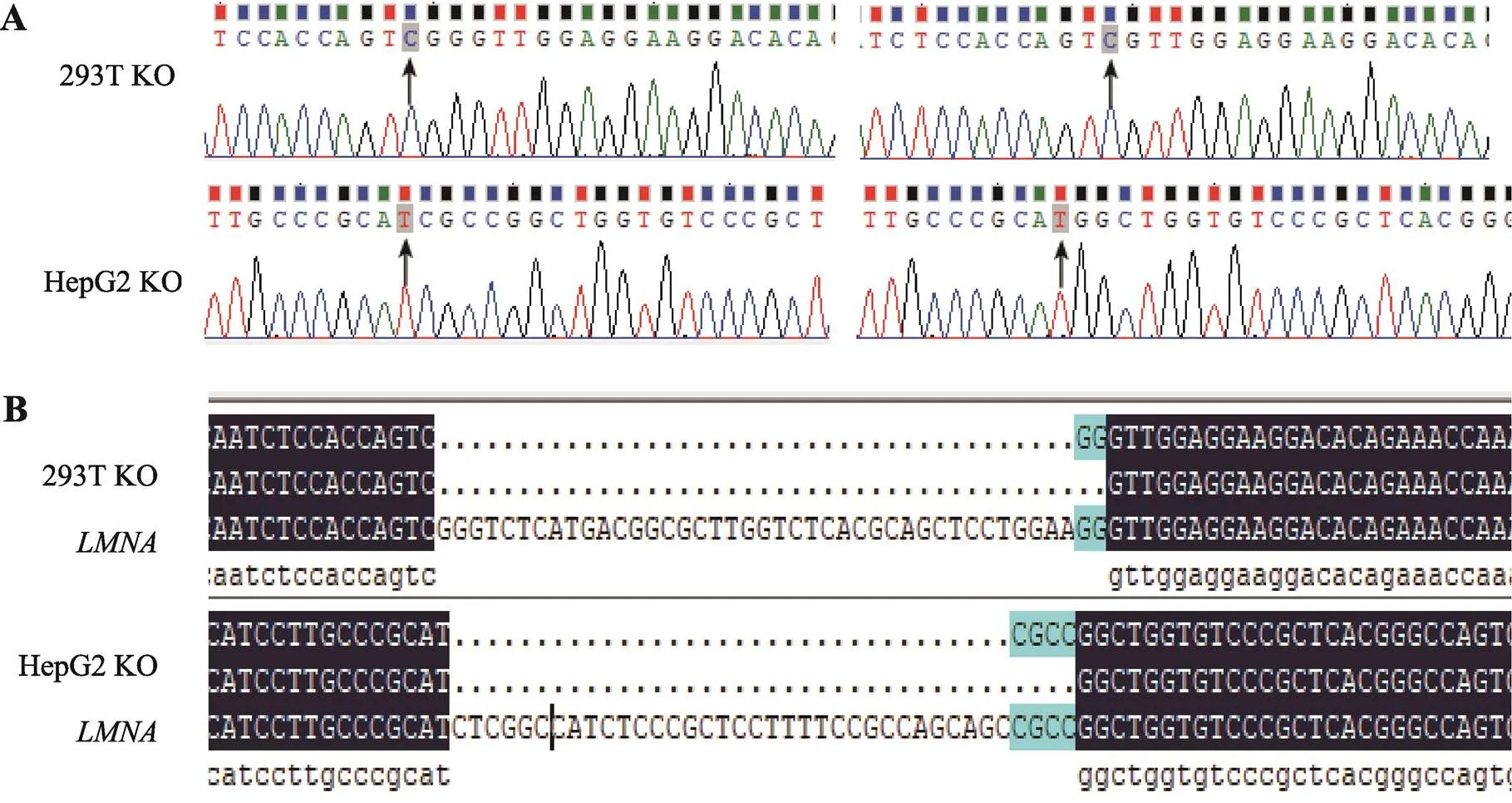
图1 LMNA基因敲除单克隆细胞株的TA克隆测序鉴定
A:阳性单克隆细胞的鉴定。黑色箭头指示为CRISPR/Cas9系统开始剪切的位点。B:阳性克隆序列与全基因序列对比。序列比对显示在gRNA处剪切,293T的等位基因分别缺失40和42个碱基,HepG2的等位基因分别缺失35和39个碱基。KO为敲除细胞。

图2 LMNA基因敲除细胞内Lamin A和Lamin C表达分析
A:与敲除细胞株剪切位点示意图。两株敲除细胞系的剪切位点都位于LMNC选择性剪切位点之前。B:Lamin A的蛋白免疫印迹。C:敲除后细胞的免疫荧光比较(400×)。蓝色为DAPI,绿色为Lamin A,红色为Lamin B。D:Lamin C的蛋白免疫印迹。WT为野生型细胞,KO为敲除细胞。
2.3 LMNA基因敲除后对细胞增殖和凋亡的影响
CCK-8分析结果显示,基因敲除之后293T和HepG2两株细胞的增殖受到明显的抑制(图3A),其中对肝癌细胞HepG2的增殖抑制更为明显。同时,利用流式细胞仪对细胞凋亡情况进行检测,Lamin A敲除后,两株细胞的凋亡率都显著上升(图3:B, C)。
2.4 LMNA基因敲除后细胞核和细胞整体形态的变化
基因敲除后,与野生型对比,光镜下观察这两株细胞形态上发生了明显改变(图4A)。293T野生型细胞有3~4个突出,在敲除后,293T细胞胞体变小变圆,突出连接减少变细;HepG2野生型为长梭型,细胞核在正中突出,胞质主要分布在细胞的梭型两端,Lamin A敲除后,细胞变成多边形,胞质较为均匀的分布在核周。DAPI染色后,未发现细胞核发生核凹陷、核畸形等改变(图4B)。
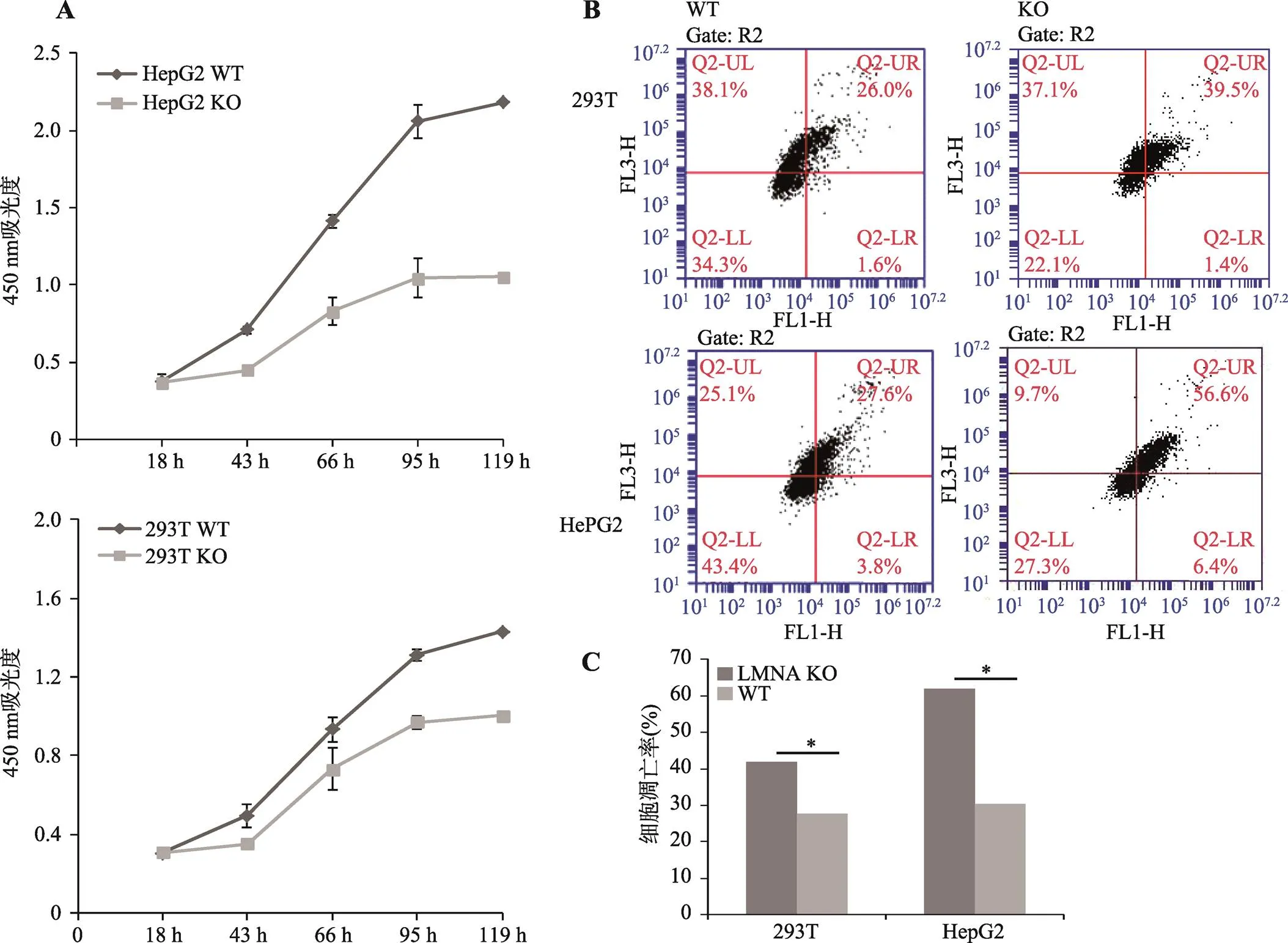
图3 LMNA敲除细胞株和野生细胞株的细胞增殖能力和凋亡比较
A:细胞增殖曲线。敲除后细胞增殖能力都明显下降(=3)。B:流式细胞仪检测细胞凋亡结果。敲除后细胞凋亡率都显著上升,FL1-H为绿光通道(Annexin V-FITC),FL3-H为红光通道(PI)。C:细胞凋亡的统计学分析(<0.05,=3)。293T WT为293T野生型细胞,293T KO为293T基因敲除细胞,HepG2 WT为HepG2野生型细胞,HepG2 KO为HepG2基因敲除细胞。

图4 LMNA基因敲除后细胞核和细胞整体形态的变化
A:普通光学显微镜下的细胞形态。敲除后细胞形态发生明显改变。B:DAPI染色后的细胞核形态未观察到显著差异。C:扫描电镜下的细胞形态。D:透射电镜下的细胞核形态。与野生型对比,KO细胞核膜凹凸不平WT为野生型细胞,KO为敲除细胞。
为了更清楚的观察细胞形态的改变,利用扫描电镜把细胞放大3000倍(图4C),形态差异更为显著;利用透射电镜将细胞膜放大15 000倍,发现KO细胞的核膜变得凹凸不平(图4D)。通过免疫荧光技术对细胞骨架(β-actin)进行染色,油镜下也观察到相同的形态变化(图5)。
3 讨论
本文首次利用CRISPR/Cas9系统成功敲除293T和HepG2的基因。虽然敲除小鼠早有报道[17],研究人员也获得了敲除小鼠来源的体外培养成纤维细胞系[18],但人源基因敲除的细胞系未见报道。本研究构建的人源基因敲除细胞系为进一步基因功能研究奠定基础。
基因不同突变会导致多种疾病,这与核纤层蛋白所发挥的多种细胞功能相一致[9,19,20]。核纤层蛋白病是人类疾病中最耐人寻味的退行性疾病之一,基因型与表型之间的关系仍然不甚清楚[21,22]。有趣的是,尽管核纤层蛋白普遍表达,但在每一种核纤层蛋白病中,只有一种或几种组织受到损害,为了解释这个复杂的谜题,研究人员常常构建不同的基因突变体(G608G,R527C和H222P突变等),然后在不同体外培养细胞系中研究突变体的功能[23]。然而,较多的突变体,尤其是隐性遗传的致病突变体,往往在有内源性Lamin A和Lamin C存在的情况下,很难观察到突变体对细胞的影响。本研究构建的敲除细胞系可以避免内源性Lamin A和Lamin C对突变体功能研究的影响,是很好的研究隐性遗传的致病突变体的材料。

图5 免疫荧光技术染色微丝观察LMNA敲除后细胞形态的改变
敲除后细胞骨架微丝的改变更为明显,蓝色为DAPI染色的细胞核,绿色为β-actin 293T WT为293T野生型细胞,293T KO为293T基因敲除细胞,HepG2 WT为HepG2野生型细胞,HepG2 KO为HepG2基因敲除细胞。
CRISPR/Cas9基因编辑技术作为新一代基因编辑技术,尽管具有设计简单,操作方便,效率高,成本低并且可同时进行多点编辑等优势,但也存在敲除效率不高的问题[24,25]。有研究显示,CRISPR/ Cas9系统导致的DNA双链断裂通过同源重组介导的DNA修复后剪切成功比例常不足10%[26~28]。前期实验中发现设计两条位点相近的gRNA共同转染能显著提高CRISPR/Cas9系统的双等位基因敲除效率。
在本研究中,构建的两株KO细胞株与野生型细胞相比增殖能力都明显下降,其中肝癌细胞HepG2的增殖能力下降更为显著,这说明对肿瘤增殖十分重要。通过流式细胞术检测细胞凋亡情况,我们发现,KO之后两株细胞的凋亡情况也大幅上升。虽然使用两株细胞特性差异较大的细胞系,293T为胚肾来源,HepG2为肝肿瘤来源。但是在增殖,凋亡与形态上的改变都类似效应(图3)。值得一提的是,高表达的肿瘤细胞系HepG2,敲除后其增殖和凋亡变化较为显著。较多的临床样本研究中已经发现的基因在多种肿瘤中异常表达[29,30],但异常表达在肿瘤发生发展中的意义未知,本研究观察到KO对肿瘤细胞增殖与凋亡都有显著影响,这是否与肿瘤的致瘤性相关有待进一步研究。
文献报道在Lamin A缺失的小鼠成纤维细胞中,微管组织中心(MTOC)就会压迫细胞核,形成较多变形的凹型/新月形细胞核[31]。该文献提出假说,细胞核与MTOC相互有一个作用力,只有当这个力度平衡时,细胞核才能维持正常形态[32]。而核纤层蛋白正是细胞核的这种作用力的提供者之一[33]。有趣的是,本研究获得的两株基因敲除细胞系,却并未发现明显的核凹陷变形(图4和图5)。而只有通过透射电镜才发现核膜由平整光滑变得凹凸不平(图5)。本研究中使用的体外培养的永生化细胞系与文献报道中使用的原代培养的小鼠成纤维细胞不同,由此推测,可能是由于在Lamin A减少或者缺失时,Lamin B的表达相对会增加,而体外培养的永生化细胞内Lamin B的表达大大增加[34]正好调整了这种失衡。
本研究发现两株KO细胞与野生型相比形态发生明显改变(图4)。编码核纤层蛋白Lamin A/C,为细胞核提供结构支持。表达水平与核刚度、组织的硬度和可塑性直接相关[5, 35,36]。核刚度对于细胞的生存能力和维持分化细胞的功能都很重要,尤其是在肌肉和皮肤等承受强烈机械张力的组织中[36,37]。
综上所述,基因功能与细胞增殖、凋亡相关,也影响细胞骨架的分布。本研究成功获得两株体外培养的KO细胞,为将来研究基因在肝肿瘤中的作用奠定基础;也为研究中隐性致病突变是如何对不同细胞产生影响,以及这些突变体如何导致细胞功能的改变提供实验材料。
[1] Burke B, Stewart CL. Functional architecture of the cell's nucleus in development, aging, and disease., 2014, 109: 1–52.
[2] Gruenbaum Y, Foisner R. Lamins: Nuclear intermediate filament proteins with fundamental functions in nuclear mechanics and genome regulation., 2015, 84: 131–164.
[3] Gruenbaum Y, Medalia O. Lamins: The structure and protein complexes., 2015, 32: 7–12.
[4] Swift J, Ivanovska IL, Buxboim A, Harada T, Dingal PCDP, Pinter J, Pajerowski JD, Spinler KR, Shin JW, Tewari M, Rehfeldt F, Speicher DW, Discher DE. Nuclear lamin-A scales with tissue stiffness and enhances matrix-directed differentiation., 2013, 341(6149): 1240104.
[5] Davidson PM, Denais C, Bakshi MC, Lammerding J. Nuclear deformability constitutes a rate-limiting step during cell migration in 3-D environments., 2014, 7(3): 293–306.
[6] Kohsaka S, Saito T, Akaike K, Suehara Y, Hayashi T, Takagi T, Kaneko K, Ueno T, Kojima S, Kohashi KI, Mano H, Oda Y, Yao T. Pediatric soft tissue tumor of the upper arm with LMNA-NTRK1 fusion., 2018, 72: 167–173.
[7] Sakthivel KM, Sehgal P. A novel role of lamins from genetic disease to cancer biomarkers., 2016, 10(2): 309.
[8] Butin-Israeli V, Adam SA, Goldman AE, Goldman RD. Nuclear lamin functions and disease., 2012, 28(9): 464–471.
[9] Worman HJ, Fong LG, Muchir A, Young SG. Laminopathies and the long strange trip from basic cell biology to therapy., 2009, 119(7): 1825–1836.
[10] Gordon LB, Rothman FG, López-Otín C, MisteliT. Progeria: A paradigm for translational medicine., 2014, 156(3): 400–407.
[11] Gonzalo S, Kreienkamp R. DNA repair defects and genome instability in Hutchinson-Gilford Progeria Syndrome.,2015, 34: 75–83.
[12] Vidak S, Foisner R. Molecular insights into the premature aging disease progeria., 2016, 145(4): 401–417.
[13] Rodríguez S, Eríksson M. Low and high expressing alleles of the LMNA gene: Implications for laminopathy disease development., 2011, 6(9): e25472.
[14] Vigouroux C, Guénantin AC, Vatier C, Capel E, Le Dour C, Afonso P, Bidault G, Béréziat V, Lascols O, Capeau J, Briand N, Jéru I. Lipodystrophic syndromes due to LMNA mutations: Recent developments on biomolecular aspects, pathophysiological hypotheses and therapeutic perspectives., 2018, 9(1): 251–264.
[15] Cenni V, D'Apice MR, Garagnani P, Columbaro M, Novelli G, Franceschi C, Lattanzi G. Mandibuloacral dysplasia: A premature ageing disease with aspects of physiological ageing., 2018, 42: 1–13.
[16] Al-Saaidi R, Bross P. Do lamin A and lamin C have unique roles?,2015, 124(1): 1–12.
[17] Young SG, Jung HJ, Lee JM, Fong LG. Nuclear lamins and neurobiology., 2014, 34(15): 2776– 2785.
[18] Jahn D, Schramm S, Schnölzer M, Heilmann CJ, de Koster CG, Schutz W, Benavente R, Alsheimer M. A truncated lamin A in the Lmna–/–mouse line: Implications for the understanding of laminopathies., 2012, 3(5): 463–474.
[19] Capell BC, Collins FS. Human laminopathies: Nuclei gone genetically awry., 2006, 7(12): 940–952.
[20] Camozzi D, Capanni C, Cenni V, Mattioli W, Columbaro M, Squarzoni S, Lattanzi G. Diverse lamin-dependent mechanisms interact to control chromatin dynamics. Focus on laminopathies., 2014, 5(5): 427–440.
[21] Bertrand AT, Chikhaoui K, Yaou RB, Bonne G. Clinical and genetic heterogeneity in laminopathies., 2011, 39(6): 1687–1692.
[22] Smith ED, Kudlow BA, Frock RL, Kennedy BK. A-type nuclear lamins, progerias and other degenerative disorders., 2005, 126(4): 447–460.
[23] Manju K, Muralikrishna B, Parnaik VK. Expression of disease-causing lamin A mutants impairs the formation of DNA repair foci., 2006, 119(Pt 13): 2704–2714.
[24] Ul Ain Q, Chung JY, Kim YH. Current and future delivery systems for engineered nucleases: ZFN, TALEN and RGEN., 2015, 205: 120–127.
[25] Zheng W, Gu F. Progress of application and off-target effects of CRISPR/Cas9., 2015, 37(10): 1003–1010.郑武, 谷峰. CRISPR/Cas9 的应用及脱靶效应研究进展. 遗传, 2015, 37(10): 1003–1010.
[26] Paquet D, Kwart D, Chen A, Sproul A, Jacob S, Teo S, Olsen KM, Gregg A, Noggle S, Tessier-Lavigne M. Efficient introduction of specific homozygous and heterozygous mutations using CRISPR/Cas9., 2016, 533(7601): 125–129.
[27] Miyaoka Y, Berman JR, Cooper SB, Mayerl SJ, Chan AH, Zhang B, Karlin-Neumann GA, Conklin BR. Systematic quantification of HDR and NHEJ reveals effects of locus, nuclease, and cell type on genome-editing., 2016, 6: 23549.
[28] Liu DY, Qiu T, Ding XH, Li M, Zhu MY, Wang JH. Rapid construction of multiple sgRNA vectors and knockout of the Arabidopsis IAA2 gene using the CRISPR/Cas9 genomic editing technology., 2016, 38(8): 756–764.刘丁源, 邱婷, 丁晓辉, 李苗苗, 朱睦元, 王君晖.快速构建多重sgRNA载体利用CRISPR/Cas9技术敲除拟南芥IAA2基因. 遗传, 2016, 38(8): 756–764.
[29] Graziano S, Kreienkamp R, Coll-Bonfill N, Gonzalo S. Causes and consequences of genomic instability in laminopathies: Replication stress and interferon response., 2018, 9(1): 289–306.
[30] Rauschert I, Aldunate F, Preussner J, Arocena-Sutz M, Peraza V, Looso M, Benech JC, Agrelo R. Promoter hypermethylation as a mechanism for Lamin A/C silencing in a subset of neuroblastoma cells., 2017, 12(4): e0175953.
[31] Tariq Z, Zhang H, Chia-Liu A, Shen Y, Gete Y, Xiong ZM, Tocheny C, Campanello L, Wu D, Losert W, Cao K. Lamin A and microtubules collaborate to maintain nuclear morphology., 2017, 8(4): 433–446.
[32] Dahl KN, Kahn SM, Wilson KL, Discher DE. The nuclear envelope lamina network has elasticity and a compressibility limit suggestive of a molecular shock absorber., 2004, 117(Pt 20): 4779–4786.
[33] Reddy S, Comai L. Recent advances in understanding the role of lamins in health and disease., 2016, 5: 2536.
[34] Liu GH, Barkho BZ, Ruiz S, Diep D, Qu J, Yang SL, Panopoulos AD, Suzuki K, Kurian L, Walsh C, Thompson J, Boue S, Fung HL, Sancho-Martinez I, Zhang K, Yates J 3rd, Izpisua Belmonte JC. Recapitulation of premature ageing with iPSCs from Hutchinson-Gilford progeria syndrome., 2011, 472(7342): 221–225.
[35] Denais CM, Gilbert RM, Isermann P, McGregor AL, te Lindert M, Weigelin B, Davidson PM, Friedl P, Wolf K, Lammerding J. Nuclear envelope rupture and repair during cancer cell migration.,2016, 352(6283): 353–358.
[36] Harada T, Swift J, Irianto J, Shin JW, Spinler KR, Athirasala A, Diegmiller R, Dingal PCDP, Ivanovska IL, DischerDE. Nuclear lamin stiffness is a barrier to 3D migration, but softness can limit survival., 2014, 204(5): 669–682.
[37] Bell ES, Lammerding J. Causes and consequences of nuclear envelope alterations in tumour progression., 2016, 95(11): 449–464.
Research on the knockout ofgene by CRISPR/Cas9 system in human cell lines
Heng Liu1, Dongming Li1, Lanyu Zhu1, Lejin Lai1, Wanyun Yan1, Yushuang Lu1, Yi Wei1, Yueqi Huang1, Mei Fang1, Yuangang Su1, Fang Yang2, Wei Shu1
Thegene encodes the nuclear Lamin A and Lamin C proteins, and is related to nuclear membrane organization, genome stability and cell differentiation. Abnormal expression ofis ubiquitous in human tumors, and its mutation leads to various forms of laminopathies, including Emery-Dreifuss muscular dystrophy (EDMD), dilated cardiomyopathy (DCM), and Hutchinson-Gliford progeria syndrome (HGPS). To further determine the functions of thegene in cellular physiology, the present study used the CRISPR/Cas9 technique to edit thegene of 293T and HepG2 cells, which resulted in two stablegene knockout (KO) cell lines. Compared to the respective wild type cells, theKO cell lines showed decrease in proliferation ability, increase in apoptosis, alteration in cellular morphology and uneven structures in the nucleus membrane. In this study, we report for the first time the results on the construction ofKO immortalized cell lines and characterization of their morphological changes, thereby laying the foundation for the further studies of thegene functions and pathogenic mutations.
gene; CRISPR/Cas9; 293T; HepG2; cell morphology
2018-09-13;
2018-11-21
国家自然科学基金项目(编号:31660311)和广西医科大学创新创业项目(编号:201710598055)资助[Supported by the National Natural Science Foundation of China (No. 31660311) and Guangxi Medical University Innovation and Entrepreneurship Project (No. 201710598055)]
刘恒,硕士研究生,专业方向:分子生物学与生物化学。E-mail: 240627592@QQ.com
李东明,硕士研究生,专业方向:分子生物与生物化学。E-mail: abcd.1456@163.com
刘恒和李东明并列第一作者。
舒伟,博士,副教授,研究方向:分子遗传。E-mail: shuwei7866@126.com
10.16288/j.yczz.18-146
2018/12/6 11:18:49
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20181206.1118.004.html
(责任编委: 谷峰)

