基因组选择在羊育种中的应用研究进展
张统雨,魏 霞,张 勤,杜立新,王立贤*,赵福平*
(1. 中国农业科学院北京畜牧兽医研究所,农业部动物遗传育种与繁殖(家禽)重点实验室,北京 100193;2中国农业大学动物科技学院,北京 100193)
基因组选择(genomic selection,GS)作为当前畜禽分子选育的前沿技术,其理论源于2001年Meuwissen等[1]的研究报道。其核心思想是基于畜禽全基因组高密度SNP 遗传标记的芯片技术,通过构建相应统计方法估计畜禽个体全基因组估计育种值(genome-wide estimated breading value,GEBV)。基因组选择理论和方法已在畜禽育种实践中成功实施[2],特别是给奶牛育种带来了革命性的变化[3],已经成为了奶牛育种的常规技术。彻底改变了常规育种的思路,极大提高了遗传进展。
随着生物信息技术的发展,绵羊和山羊的基因组信息不断完善,推动了不同密度SNP芯片的生产和应用。2009年,首张绵羊芯片——OvineSNP50 BeadChip array[4]得以问世。2010年,推出了山羊第一张芯片GoatSNP50 BeadChip array[5]。2014年,推出了绵羊600K高密度SNP芯片——Ovine HD SNP BeadChip[6]。2015年新西兰羊公司内部推出了5K绵羊芯片[7]。同年又推出了12K SNPs的低密度绵羊芯片[8]。2017年8月,绒山羊的66K SNP芯片也被研发出来[9]。这些不同密度SNP芯片的推出,为基因组选择在羊育种中的应用起到了技术支撑作用。最先报道的是2009年基于肉用绵羊和毛用绵羊的模拟研究结果[10]。目前,新西兰、澳大利亚、法国等养羊大国已经启动了羊的基因组选择,并且取得了显著成效,开启了羊育种的新篇章。Mrode等[11]也对非洲各国和印度开展羊基因组选择进行了可行性分析。中国也在2015年提出了《全国肉羊遗传改良计划(2015-2025)》,并在2016年确定了天津奥群牧业有限公司等6家企业作为国家核心育种场。随着环保意识的不断增强,在农区大力推行设施养羊,这为适合设施养殖的湖羊品种的推广提供了很好的机会,扩大了其遗传影响力。2018年5月,江苏乾宝湖羊产业公司、润林木业和湖州咩咩羊3家公司签订了湖羊产业联合发展协议。这为在中国开展第一个中国地方绵羊品种的基因组选择打下了基础,也推动了基因组选择在中国羊育种中的实施,将加快其遗传进展,促使中国羊的育种进入基因组选择时代。本文总结了基因组选择在羊中的应用现状、影响因素、优势以及存在问题和展望,为基因组选择在羊育种中的应用提供参考依据。
1 羊重要生产性状基因组选择的应用现状
1.1 毛用性状
羊毛是人在纺织史上开发利用较早的天然纤维之一,羊毛加工后的毛制品手感丰满、吸湿性强,保暖性好,是纺织工业的重要原材料。Daetwyler等[12]对Merino羊毛重量、纤维直径、纤维强度等毛用性状进行GEBV估计时,发现其估计准确性范围为0.15~0.79。Moghaddar等[13]研究发现,Merino羊的一岁羔羊和成年羔羊的羊毛性状GEBV估计的准确率为0.33~0.75。Bolormaa等[14]利用BayesR和GBLUP对3个羊毛质量性状进行GEBV估计,结果发现,BayesR 和 GBLUP的平均GEBV精确度比较相似,约为0.22,BayesR在羊毛产量和纤维直径上的精确度最高,均大于0.40,而在羊皮质量和沾粪便结块污毛得分最低。
1.2 肉用性状
肉用绵羊具备的优良性状包括适应性好、繁殖力强、生长速度快、胴体品质优良 4 个方面的特征。澳大利亚肉用绵羊上开展的GS效果显著,其胴体性状和肉质性状的GEBV准确性估计提高到0.05~0.10[12, 15-16]。Van Der Werf[10]采用选择指数理论对肉羊和细毛羊群体进行模拟研究,发现GS的应用可以使肉羊的产肉指数在25年中增长32%,大大加快了绵羊的育种进展。Slack-Smith等[17]研究发现,澳大利亚绵羊胴体和生长性状、冻后眼肌面积和肌内脂肪含量GEBV的基因组预测准确性分别为0.45、0.72 和 0.32。Brito等[18]对新西兰绵羊活体测量性状、胴体性状和肉质性状的GEBV估计准确性范围分别为(0.18±0.07)~(0.33±0.10)、(0.28±0.09)~(0.55±0.05)和(0.21±0.07)~(0.36±0.08)。Moghaddar等[13]研究发现,Poll Dorset羊和White Suffolk羊体重的GEBV估计准确性在0.11~0.27之间,而Border Leicester羊和Merino羊在0.25~0.63之间。Shumbusho等[19]计算出绵羊产肉性状年度遗传进展在传统育种方案下的标准方差σa为0.095,在肉用绵羊品种中,母系性状σa为0.061,而奶山羊的σa为0.147。而优化决策变量后,传统选择方法下的产肉性状年度遗传进展提高了0.139σa,奶绵羊σa为0.174。因此,当优化育种方案或者使用全基因组信息均可提高年度遗传进展。
1.3 产奶性状
产奶性状主要包括产奶量、乳蛋白率、乳蛋白量、乳脂率和乳脂量等。山羊奶与绵羊奶总体组成很相似,但是山羊奶的各个营养成分和理化性质都比绵羊奶低。Saanen奶山羊是世界上最优良的奶山羊品种,产奶量高(最高年产奶量可达3 430 kg),营养价值丰富,已成为乳制品行业重要的原料[20]。目前法国是Saanen奶山羊开展GS最成熟的国家。Carillier等[21]针对法国Saanen奶山羊和Alpine羊基因组评估进行交叉验证时得到,GEBV的准确性为36%~53%。Larroque等[22]研究发现,与荷斯坦奶牛相比,使用GBLUP方法对奶山羊和奶绵羊进行基因组预测准确性较低,而ssGBLUP方法可以提高其准确性。通过交叉验证发现,法国Lacaune奶绵羊基因组预测准确性增加了0.47,而Lacaune奶山羊基因组预测准确性增加了0.43。Duchemin等[23]针对法国Lacaune奶山羊3个泌乳性状(羊奶产量、脂肪含量和体细胞分数),比较使用不同的模型(只有无穷小、只有标记、联合估计无穷小、标记效应)和方法(BLUP、Bayes Cπ、偏最小二乘法(PLS)和稀疏矩阵PLS),进行全基因组育种值估计,结果表明,基因组方法的准确性为0.4~0.6;联合估计无穷小模型可改变预测回归方程的斜率;Bayes Cπ和偏最小二乘法对一些模型和性状存在有利影响。Baloche等[24]比较了利用平均父母代和基因组选择两种方法对法国Lacaune奶山羊后代测定公羊结果准确性的差异,结果发现,基因组选择的准确性要比传统依靠父母代测定后代公羊的准确性更高,达到了52%。而使用ssGBLUP方法准确性要比BLUP方法准确性高58%。Baloche等[25]利用2 892只法国Lacaune奶山羊构建参考群,使用3种方法(1)pseudo-BLUP:使用所有的公羊和后代女儿方差;2)pseudo-ssGBLUP:使用所有的公羊和后代女儿方差;3)常规ssGBLUP:使用所有动物的表型和系谱信息)进行基因组预测,结果表明,基因组选择的准确性在双亲平均水平上的增加幅度为0.10~0.20。使用常规ssGBLUP可以发现最高的准确性和最低的偏差。另外美国、意大利和西班牙也在奶山羊上开展过GS研究。Mucha等[26]对美国Alpine羊、Saanen羊和Toggenburg羊3个奶山羊品种的产奶量性状进行GEBV估计,研究表明,采用一步法(ssBLUP)的准确性最高,达到了0.61。Molina等[27]基于55K奶山羊芯片,利用ssGBLUP方法进行基因组评估发现,西班牙Florida奶山羊GEBVs的平均可靠性只增加了5.86%。
1.4 疾病性状
头痛性中毒,俗称面部湿疹(facial eczema, FE)是由致毒性的嗜铁杆菌菌株引起的一类肝性疾病,常见于夏末初秋的季节。腐生真菌产生的毒素寄生在温暖潮湿的牧草上,逐渐形成真菌孢子,当这些孢子随着牧草被牛羊吃掉后,会引起肝的炎症反应[28]。该疾病在新西兰已爆发100多年,对当地的畜牧业产生了重大负面影响。为了筛选出FE耐受性绵羊,Phua等[29]对新西兰Romney羊进行基因组选择,得到FE最佳基因组预测的准确度是0.38。虽然该结果不是很理想,但依然可以作为其他经济性状在最小范围内的基因组预测值。
1.5 繁殖性状
Newton等[30]随机选择不同年龄段的公羊和母羊,基于基因组信息针对澳大利亚青年绵羊的生育能力,评估其遗传进展,研究发现,不同年龄段的基因组信息均会影响到遗传增益,青年母羊的遗传增益相对较高。一岁公羊基因组信息的遗传增益显著高于2岁公羊。如果生育水平达到10%以上,则遗传进展就可能会有不利影响。Pickering等[31]构建了4 237只群体规模的参考群和候选群,利用基因组选择预测Romney羊繁殖性状和存活率GEBV的准确性,结果表明,罗姆尼羊GEBVs的估计准确性范围是0.16~0.52。总之,在新西兰双重用途绵羊中应用GEBVs可获得的遗传进展有望提高到84%。Bolormaa等[32]对Merino羊的出生羔羊数(number of lambs born,NLB)和产仔数(litter size,LS)进行基因组预测值评估时,基于混合记录的NLB和LS的GEBV估计准确性达到了0.41~0.54。
1.6 其他性状
目前,环境问题成为限制畜牧业发展的一个重要因素。Rowe等[33]利用GBLUP对1 872只新西兰绵羊肠道排放CH4性状和在摄入饲料后CH4排放比例(gCH4/kgDMI)进行基因组选择,结果发现,CH4排放量的基因组预测值为0.37,gCH4/kg DMI为0.43,与之相关性状的标记位点分别在9号和25号染色体上。这些位点从生理学上看接近候选位点。该研究旨在培育出肠道排放CH4气体含量较少的绵羊,从而为保护环境提供一定的参考。
2 羊基因组选择效果的影响因素
目前基因组选择在羊育种中的应用受到了诸多因素的影响,其中包括表型数据收集的准确性、参考群规模和结构、芯片密度、GEBV估计方法和统计模型等。
2.1 表型数据收集准确性
表型数据收集是开展基因组选择的第一步,是进行基因组选择的先决条件。只有收集到准确的表型数据,才能确保基因组选择的准确性。目前,除了自动化测量体重和体尺数据外,超声波胴体测量、抗病性状的测量、个体采食量测定等技术也在不断更新。计算机断层扫描(computed tomography, CT)技术也被应用于胴体组成、肉质、器官病变等表型数据的测定,这些测定技术的应用可有效提高表型值测定的准确性。CT技术的应用不仅提高了表型测定的准确性,而且还提高了GEBV估计的准确性。研究发现,公羊的参考群使用CT表型数据的遗传增益要比没有使用CT表型数据的遗传增益提高55%,如果引入到GEBV预测,则GS选择的准确性可能更高[34]。
2.2 参考群有效群体大小
基因组选择首先需要建立参考群,通过参考群计算标记的效应大小。参考群的结构[35]、遗传多样性[36]和有效群体大小对基因组预测准确性具有重要影响。Carillier-Jacquin[37]构建了不同水平的Saanen奶山羊和Alpine羊参考群(20~705只公羊和1 946只母羊),发现随着群体规模的增大,相关性状GEBV的准确性从+2%增加到+31%。而另一方面,Carillier-Jacquin等[38]构建了多品种参考群,由于参考群规模较小,基因组预测准确性有所降低,范围是-5%~38%。因此在构建参考群时,应尽量选择没有亲缘关系的公羊,让参考群体拥有丰富的遗传信息,确保参考群有效群体足够大。表1总结了新西兰、澳大利亚、法国等养羊大国参考群的规模和结构。新西兰建立了由13 420只Romney纯种羊和杂种绵羊(Coopworth羊)组成的参考群,这也是目前世界上规模最大的羊参考群[39]。英国和法国分别建立了2 400和2 700只山羊参考群,法国建立了4 800只Lacaune奶绵羊参考群[22],澳大利亚建立了8 000只杂交肉用绵羊参考群,但是相比奶牛的参考群,羊的参考群规模还相对较小[12]。参考群规模和目标性状的遗传力直接影响着遗传进展,因此扩大参考群规模可以有效提高遗传进展[15]。
表1世界各国建立的羊参考群结果汇总
Table1Summaryofreferencepopulationresultsofsheepandgoatsindifferentcountriesaroundtheworld
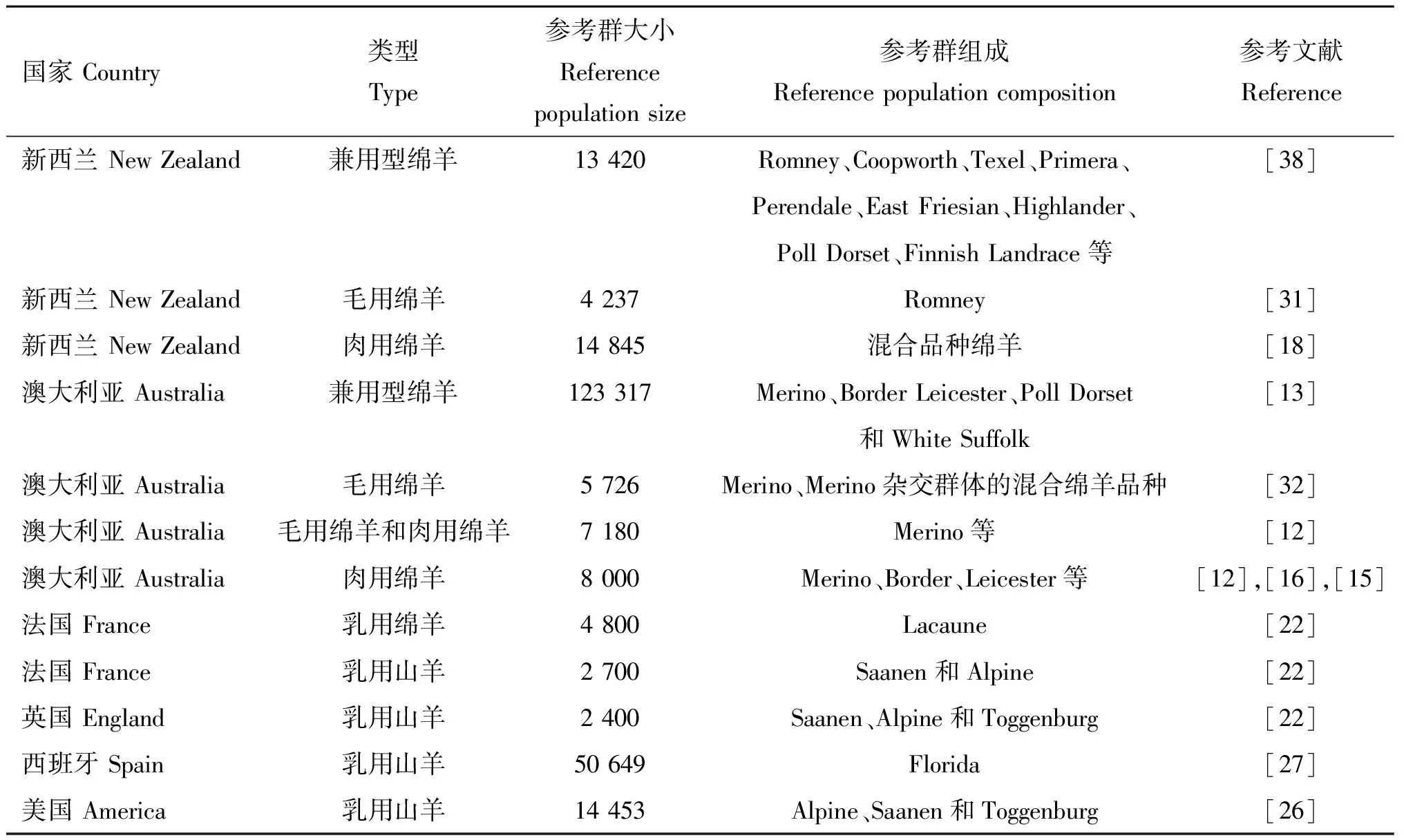
国家 Country类型Type参考群大小Reference population size参考群组成Reference population composition参考文献Reference新西兰 New Zealand兼用型绵羊13 420Romney、Coopworth、Texel、Primera、[38]Perendale、East Friesian、Highlander、Poll Dorset、Finnish Landrace等新西兰 New Zealand毛用绵羊4 237Romney[31]新西兰 New Zealand肉用绵羊14 845混合品种绵羊[18]澳大利亚 Australia兼用型绵羊123 317Merino、Border Leicester、Poll Dorset[13]和White Suffolk澳大利亚 Australia毛用绵羊5 726Merino、Merino杂交群体的混合绵羊品种[32]澳大利亚 Australia毛用绵羊和肉用绵羊7 180Merino等[12]澳大利亚 Australia肉用绵羊8 000Merino、Border、Leicester等[12],[16],[15]法国 France乳用绵羊4 800Lacaune[22]法国 France 乳用山羊2 700Saanen和Alpine[22]英国 England乳用山羊2 400Saanen、Alpine和Toggenburg[22]西班牙 Spain乳用山羊50 649Florida[27]美国 America乳用山羊14 453Alpine、Saanen和Toggenburg[26]
2.3 所用SNP芯片的密度
SNP芯片密度越高,标记间的连锁不平衡就越高,期望获得的准确性也越高。Bolormaa等[8]发现,12K芯片数据的品种和性状之间的GEBVs为0.08,虽然其准确性范围变异较大,但12K芯片在基因组选择上仍有应用价值。Raoul等[40]探究了参考群的45K中密度芯片和1K极低密度芯片之间的遗传进展关系。结果发现,参考群中只有中密度芯片数据的公羊时,其遗传进展增加了26%,而参考群中包含中密度芯片的公羊和母羊时,其遗传进展增加了54%。当候选群中使用1K的极低密度芯片时,若参考群中只有公羊,则其遗传进展增加22%,若参考群中既有公羊又有母羊,则遗传进展增加42%。Clarke等[41]针对新西兰奶山羊设计了一种新的基因分型技术,该方法包括大约80 000个SNPs,价格低于20美元/样品。若基因分型技术采用高密度基因分型(>200 kb SNPs),则基因分型技术在奶山羊的应用前景就非常可观。
2.4 参考群体与目标群体个体间的相关性
参考群与选择个体间的相关性越大,则参考群的系谱信息以及基因组信息的代表性越明显,那么基因组选择准确性越高。基因组选择的基本过程是通过参考群每个个体的SNP基因型和表型计算每个SNP的效应,然后结合目标群体的SNP基因型计算个体的GEBV。由于羊品种繁多,每个品种规模有限,在进行基因组选择时可以通过组建一个含有多个品种的混合参考群,以便增加参考群规模,以此增加参考群与目标群体个体间的相关性,提高参考群的利用效率。
2.5 GEBV估计方法和统计模型
自从基因组选择方法提出之后,GEBV估计方法层出不穷,大致可以分为3大类[42-43]:1) 基于BLUP理论的方法,有RRBLUP、GBLUP、ssGBLUP等;2)基于贝叶斯理论的方法,有BayesA、BayesB、BayesCπ、anteBayes、BayesR等;3)其他方法,有半参数方法、RKHS、机器学习、神经网络等。这些方法中没有哪一个方法能在所有情况下都具有绝对优势。由于第三类方法中考虑互作效应以及其他非线性效应使得模型非常复杂或者计算难度比较大。因此,目前研究和应用最多的还是第一类和第二类方法。
随着检测个体数据量和遗传信息量的增加,各方法都在不断的改进。对BLUP理论的方法而言,随着G矩阵或者H矩阵的增大,其求逆就是个很大的问题[44]。贝叶斯的计算速度一直是限制其应用的一个重要因素,特别是在丰富遗传信息的测序数据分析下如何加快其运算速度,提高运算效率。因为贝叶斯方法在对参数求解时是基于参数的先验信息结合似然函数推断出后验分布,从后验分布中得到参数的估计值[45-46]。常用的算法有MCMC和EM算法。MCMC算法是一种基于Markov链模型的随机抽样技术,需要大量的抽样时间,但EM算法是通过迭代进行求解,虽然其耗时短但是准确性较MCMC算法差[47-48]。为了避免各自的缺点,Wang等[49]提出了去掉MCMC的burnin阶段,直接用EM算法估计值进行MCMC抽样的杂交方法,该方法节省了burnin的抽样时间。随着计算机设备的升级,多核的并行计算也提高了MCMC的计算速度[50-51]。随着生物信息学知识和不同组学数据不断涌现,GEBV估计时也结合其生物学信息以期提高其估计准确性,有结合GWAS研究结果的BLUP|GA[52]、结合组学分析中富集分析结果的基因组特征GFBLUP模型[53]、结合生物信息的sgBLUP[54]和BayesRC[55],以及结合生物信息功能和上位效应构建单倍型区段进行基因组信息预测等[56]。这些方法在不同程度上都提高了估计的准确性。
3 在羊上开展基因组选择的优势
绵山羊以传统的粗放饲养为主,集约化饲养仍处于起步阶段,在实际生产中很难得到准确的系谱信息和交配记录,SNP芯片数据能准确计算个体间的遗传相关,提高基因组育种值估计的准确性,同时还可以避免个体间的近交,保持群体内的遗传变异。其次,SNPs标记分布在整个基因组中能捕获全部遗传变异,提高选择准确性。再次,基因组选择典型的优势是可以不依赖表型信息直接根据标记信息进行个体选择,不仅能开展早期选择,缩短世代间隔,还能提高选择强度,降低育种成本。这些优势使得基因组选择,尤其是对一些低遗传力性状、难以测量的性状、限性性状、生长后期测定性状和免疫力性状等,在羔羊出生或者胚胎期就可以进行。根据用途,羊可以分为肉用、奶用、毛用、皮用以及肉毛、肉奶兼用等品种,由于不同品种和用途的羊其育种规划不尽相同,所以基因组选择在不同育种规划中的优势也表现不同。在羊的育种中,基因组选择为制定科学的育种规划、提高遗传进展提供了技术飞跃[57]。
4 存在问题
虽然GS在羊育种的应用前景广阔,但目前仍然面临以下几方面的问题。第一,羊的SNP芯片成本比猪和奶牛要高,在羊上开展基因组选择,大半的经费花在了SNP芯片上,极大的限制了基因组选择在羊上的应用。第二,羊的品种资源丰富,种类繁多,没有一个品种能主导一个产业,而且各个羊场间联系较少,如何建立有效的参考群是个难题。第三,羊每胎产羔数较低,公羊的影响也没有种公牛效应那么大,优秀基因传递和扩散速度比较慢。第四,放牧的羊需要经受寒冷冬季、炎热的夏季,还需要适应干旱戈壁和盐碱之地等恶劣的自然环境,要提高其生产性状的遗传进展还需要提高其自然的适应性。如何在选择目标性状的同时兼顾适应性状,使其在当地的气候条件下,最大程度的提高其遗传进展也是当前需要解决的问题之一。第五,随着基因组数据、表型组数据、代谢组数据、表达组数据等各个组学数据的出现,该如何整合多种组学数据信息,提高GEBV估计的准确性也需要研究者们认真思考。
5 展 望
目前,羊基因组选择已经在新西兰、澳大利亚、法国等养羊大国开始实施,并取得显著成效。但上述问题对羊基因组选择提出了新的方向和挑战。首先,随着测序价格的下降,基于测序数据的GS优势日趋明显。它不仅可以降低基因分型的成本,而且还克服了已有SNP芯片的偏倚(acertainment bias, AB)问题[58],有望在5~10年内替代高密度SNP芯片,成为GS应用的主要分型手段。其次,海量的组学数据收集,对计算机速度和统计方法提出了更高的要求和挑战,需要更快的计算速度和准确的估计方法。再者,对多个群体和品种构建的参考群,需要建立群体之间遗传联系性,扩大群体的有效含量,提高选择群体估计的准确性。从次,制定最优育种目标需结合多性状信息选择最佳的个体。除此之外,扩大基因组选择的种羊基因的影响,需结合人工授精、MOET、JIVET和基因编辑等先进的生物繁殖技术加快优秀基因的扩散和遗传进展。基因组选择是一个系统工程,需要各个影响因素配置成最佳,才能选择最优的个体,它必将加快羊的遗传进展,揭开羊育种的新篇章。

