中国莲P1B -ATPase亚家族成员的生物信息学分析
毛立彦, 龙凌云, 谢振兴, 於艳萍, 丁丽琼, 黄秋伟, 宾振钧, 金 刚
(广西壮族自治区亚热带作物研究所,广西南宁 530001)
近年来,随着工农业的飞速发展,农药化肥的过度施用、矿区的不合理开采以及工厂产生的重金属废渣、废液的不合理排放,致使Cd、Zn、Cu、Pb、Co等重金属及其有毒化合物大量渗入水体,水体重金属污染问题日益严重,已引起世界范围广泛关注[1-5]。水体中重金属的超标不仅会污染自然环境,同时还会诱发各种疾病,对人体健康造成直接或间接危害。重金属污染水体的修复工程任务艰巨,以往采用的物理或化学修复手段投资巨大且效果不佳,而大型水生植物生长速度快,生物量大,易于人工栽培管理,且可作为较好的景观观赏材料,将其用于治理水体重金属污染可降低投资成本,提高治理效率,并能获得良好的环境生态效益[6],因此,探明大型水生植物对重金属的耐受性和富集机制,提高它们对重金属的耐受和富集能力,实现大型水生植物修复水体重金属污染的综合开发利用已成为当今环境科学领域的研究热点。
P1B-ATPase蛋白(P1B型ATPase蛋白,别称heavy metal transporting ATPase,简称HMA)属于P型ATPase蛋白家族中的一个亚家族,也是该家族中唯一参与重金属阳离子稳态的转运蛋白,可通过水解ATP跨膜运输金属阳离子,选择性吸收和运输植物生长发育必需的金属离子(Cu+、Cu2+、Zn2+、Co2+)以及一些非必需重金属离子(Cd2+、Pb2+),在重金属的抗性、吸收、转运过程中起着重要作用,是植物修复重金属污染水体和土壤不可或缺的组成部分[7-8]。低等、高等植物中存在多个P1B-ATPase蛋白,广泛分布于植物细胞的质膜、叶绿体、液泡膜、高尔基体等细胞结构上[9-11]。关于植物P1B-ATPase蛋白结构和功能的研究,现主要集中在拟南芥、水稻、大麦、大豆等模式植物上,已有研究证实拟南芥(Arabidopsisthaliana)含有8个P1B-ATPase蛋白(AtHMA1-8)[12-13],水稻(Oryzasativa)中有9个(OsHMA1-9)[14],大麦(Hordeivulgaris)中有10个(HvHMA1-10)[15],大豆(Glycinemax)中发现9个(GmHMA1-9)[16]。此外,有研究者还从甘蓝型油菜(Brassicanapus)、鼠耳芥(Arabidopsishalleri)、遏蓝菜(Thlaspicaerulescens)等植物中分别克隆获得P1B-ATPase蛋白成员BnHMA1、AhHMA4、TcHMA4的编码基因[17-20]。目前已报道的高等植物P1B-ATPase蛋白多存在于陆生植物中,该类蛋白在水生植物中的相关研究较少,在睡莲科(Nymphaeaceae)水生植物中尚未见相关的研究报道。睡莲科植物是多年生水生植物,共有莲属(NelumboGaertn.)、睡莲属、芡实属、王莲属等9个属,不同属内均具有较多种类的大型水生植物物种,如睡莲(Nymphaeatetragona)、中国莲(Nelumbonucifera)、荷花等,它们均具有抗性强、分布广、生长量大和繁殖能力强等特点,同时还具有较高的观赏价值,可作为修复水体重金属污染的植物材料,能起到兼顾水体污染修复和景观改造的功能,具有较广泛的应用前景。目前,睡莲科莲属的中国莲基因组测序工作已完成[21],全基因组测序结果已公开,大量遗传信息的获得为系统解析睡莲科植物的水体重金属修复机制、促进睡莲科植物在水体修复中的应用奠定了基础。本研究采用生物信息学方法,对中国莲(N.nucifera)可能存在的P1B-ATPase蛋白亚家族成员进行预测并利用生物信息学软件分析其系统进化地位、蛋白质的理化性质、结构特征和保守结构域等特点,以期为进一步揭示中国莲等大型水生植物P1B-ATPase蛋白的结构和功能奠定基础。
1 材料与方法
1.1 中国莲P1B-ATPase蛋白亚家族序列的获取和确定
试验地点位于广西壮族自治区亚热带作物研究所实验室,于2017年9月从中国莲全基因组数据库(http://lotus-db.wbgcas.cn/)下载所有已注释的蛋白序列,以FASTA格式保存,根据拟南芥、水稻基因组中已鉴定的P1B-ATPase蛋白成员的保守基序和结构特征[7],从下载的中国莲数据库已翻译的蛋白序列中删除不含P1B-ATPase蛋白成员保守基序和结构特征的冗余序列,最终筛选出中国莲中符合P1B-ATPase蛋白特征的目的序列。
1.2 中国莲P1B-ATPase蛋白的系统发育树构建和模体识别
利用多序列比对工具Clustal X对中国莲、拟南芥、水稻的P1B-ATPase蛋白序列进行比对,并用GeneDoc软件查看比对序列的保守基序。采用MEGA 5.0软件中的最大似然法构建系统发育树。通过随机逐步比较的方法搜索最佳系统进化树,对生成的系统树进行Bootstrap校正。同时,利用MEME program3(http://meme-suite.org/tools/meme)模体检索工具识别中国莲、拟南芥、水稻的P1B-ATPase蛋白所共有的模体,并对相关参数进行修改,将模体数最大值调整为5个,其他参数均为默认值。
1.3 中国莲P1B-ATPase蛋白序列分析
利用瑞士生物信息学研究所提供的ProtParam(http://web.expasy.org/protparam/)程序,对上述4种蛋白质的氨基酸残基数目、组成、相对分子量、理论等电点及稳定性等理化性质进行在线分析。利用Plant-mPLoc(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)分析蛋白亚细胞定位。利用ProtScale(http://web.expasy.org/protscale/)分析蛋白亲/疏水性。利用TMHMM在线程序(http://www.sacs.ucsf.edu/cgi-bin/tmhmm.py)预测蛋白的跨膜结构,并用TOPO2在线程序(http://www.sacs.ucsf.edu/cgi-bin/open-topo2.py)显示跨膜拓扑结构图。利用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)分析蛋白二级结构。利用Swiss-Model(https://www.swissmodel.expasy.org/)分析蛋白的三级结构。
2 结果与分析
2.1 中国莲P1B-ATPase蛋白成员的筛选和命名
通过对拟南芥、水稻等已知的P1B-ATPase蛋白亚家族成员序列和结构进行分析,依据已知P1B-ATPase蛋白成员的序列长度范围,剔除获取的中国莲蛋白模型中氨基酸数目<400个或>1 500个的冗余序列,并将获得的非冗余序列与拟南芥、水稻已知P1B-ATPase蛋白进行比对,依据该亚家族成员序列中高度保守的DKTGT、GDGxNDxP、TGE、C/SPx、HP等保守基序进行筛选,最终获得4个中国莲P1B-ATPase蛋白,分别命名为NnHMA1、NnHMA2、NnHMA3、NnHMA4(表1)。

表1 中国莲P1B-ATPase蛋白成员信息
2.2 中国莲P1B-ATPase蛋白系统进化和保守序列分析
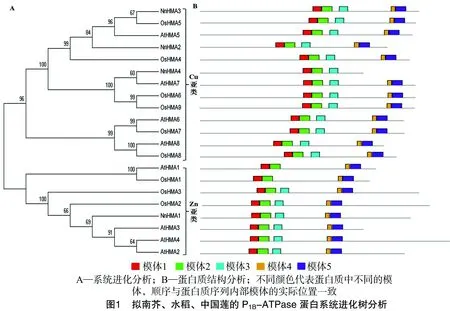
对拟南芥、水稻、中国莲中21个P1B-ATPase蛋白进行氨基酸多序列比对,并构建系统进化树(图1-A),分析发现,这21个蛋白可聚类为2个独立的进化分支。植物中存在多种P1B-ATPase蛋白,目前已鉴定的这类蛋白可根据金属底物特异性划分为2个亚类,分别为Zn2+/Co2+/Cd2+/Pb2+P1B-ATPase(Zn亚类)、Cu+/Ag+P1B-ATPase(Cu亚类)[7,15],其中AtHMA1、AtHMA2、AtHMA3、AtHMA4属于Zn亚类,而AtHMA5、AtHMA6、AtHMA7、AtHMA8属于Cu亚类[9];水稻OsHMA1、OsHMA2、OsHMA3属于Zn亚类,OsHMA4、OsHMA5、OsHMA6、OsHMA7、OsHMA8、OsHMA9属于Cu亚类[1,14]。从图1-A可以看出,中国莲的NnHMA1属于Zn亚类,NnHMA2、NnHMA3、NnHMA4属于Cu亚类。模体分析结果(图1-B)显示,供试的21个蛋白均含有模体1和模体2,预测NnHMA1、NnHMA2、NnHMA3含有的模体与其所属进化分支中拟南芥、水稻的P1B-ATPase蛋白成员含有相似模体,而NnHMA4与其所属进化分支上的拟南芥、水稻P1B-ATPase蛋白相比,缺少模体4和模体5,推测这可能是由于在进化过程中,NnHMA4蛋白编码基因出现部分序列缺失所致。此外,利用GeneDoc软件对拟南芥、水稻和中国莲3个物种中的P1B-ATPase蛋白进行序列比对发掘保守基序,从图2可以看出,获得的中国莲4个蛋白均含有植物P1B-ATPase蛋白所特有的DKTGT、TGE、C/SPx、HP等保守基序,进一步验证本研究预测的NnHMA1、NnHMA2、NnHMA3、NnHMA4等4个蛋白属于植物P1B-ATPase蛋白亚家族成员。与其他序列相比,NnHMA4不含有GDGxNDxP保守序列,推测该蛋白在进化过程中,该部分序列出现缺失或插入的现象。
2.3 中国莲P1B-ATPase蛋白理化性质分析和亚细胞定位预测
对筛选出的4个中国莲P1B-ATPase蛋白进行理化性质分析,结果(表2)显示,中国莲的P1B-ATPase蛋白序列长度为762~1 022个氨基酸,分子量为82 672.23~111 589.13 u,理论等电点为5.17~7.17。4个P1B-ATPase蛋白的总平均疏水性均为正值,说明它们均为疏水性蛋白,这可能与P1B-ATPase蛋白具有跨膜转运金属离子的功能有关,其中NnHMA1疏水性最弱,NnHMA4疏水性最强。此外,4个蛋白的不稳定指数为30.60~38.75,其中NnHMA2最小,表明预测的蛋白都相对稳定,可供体外试验进行选择。亚细胞定位预测结果显示,中国莲的4个P1B-ATPase蛋白均位于质膜上,进一步证明中国莲4个P1B-ATPase蛋白可能为跨膜蛋白,推测它们具有参与金属离子跨膜转运的功能。
2.4 中国莲P1B-ATPase蛋白亲/疏水性和跨膜结构分析
氨基酸的亲/疏水性是蛋白质空间折叠的主要驱动力来源之一,不同氨基酸的亲/疏水性差异决定了蛋白质在折叠过程中可形成亲水性表面或疏水性内核结构,通常在跨膜区域内会形成类似的疏水性区域。通过ProtScal在线程序对中国莲的4个P1B-ATPase蛋白进行亲/疏水性预测。如图3所示,以0为分界线,正值越大表示疏水性越强,负值越大表示亲水性越强,分值的绝对值在0.5之内的氨基酸可定性为两性氨基酸[22-23]。由图3中可知,中国莲P1B-ATPase蛋白的N端均存在20~60个数量不等、亲/疏水性得分值<0.5的氨基酸残基,且NnHMA1和NnHMA4的C端亲/疏水性得分值<0.5的氨基酸残基数目明显多于NnHMA2和NnHMA3。此外,蛋白中氨基酸残基亲/疏水性得分值连续高于1的区段数量,NnHMA1有11个,NnHMA2有8个,NnHMA3有9个,NnHMA4有5个。通常在蛋白质亲/疏水性预测分析过程中,亲/疏水性>1.0可判定为跨膜区,介于0.6~1.0的片段为疑似跨膜区[22-23],以此可预测蛋白的跨膜区数目。
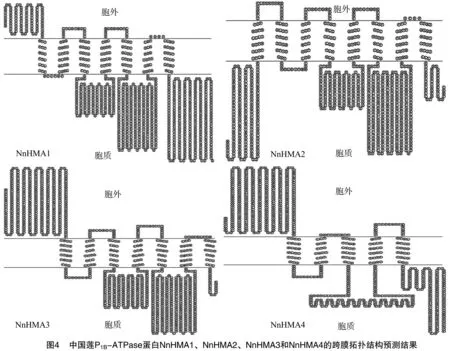
为论证亲/疏水性分析的预测结果,进一步利用TMHMM和TOPO2在线程序对获得的4个中国莲P1B-ATPase蛋白进行跨膜区域和拓扑结构预测。由图4可知,NnHMA1蛋白存在7个跨膜结构,NnHMA2有8个,NnHMA3有7个,而NnHMA4仅有5个,这与图3亲/疏水性预测分析的推断相类似,进一步说明经本研究预测得到的中国莲P1B-ATPase蛋白均为跨膜蛋白。中国莲P1B-ATPase蛋白均含有2个大小不一的细胞质内环,有研究表明,这2个胞质环是P1B-ATPase蛋白亚家族成员的特有结构,在环上含有3个重要的保守结构域,分别为磷酸化位点(P-domain)、脱磷酸化位点(A-domain)、ATP结合区域(N-domain)以及多个保守基序,如DKTGT、GDGxNDxP、S/TGE、HP等[7,10,15,24-28],这些保守基序在植物P1B-ATPase蛋白跨膜转运离子和水解ATP提供能量过程中起着重要作用[10,27-28],结合图2中保守基序分析结果进一步证明,本研究从中国莲基因组数据库中筛选确定的NnHMA1、NnHMA2、NnHMA3和NnHMA4属于P1B-ATPase蛋白。


表2 中国莲P1B-ATPase蛋白的理化性质参数和亚细胞定位
2.5 中国莲P1B-ATPase蛋白的二级结构与三级结构分析
蛋白质的结构、功能与生物体的各类生命活动密切相关,蛋白质的二级结构是构成三级结构的基本单元,探明蛋白质的二、三级结构,对深入探究蛋白质的折叠构造和生物学功能有重要的研究价值[29-31]。本研究采用SOPMA在线程序预测中国莲P1B-ATPase蛋白(NnHMA1、NnHMA2、NnHMA3和NnHMA4)的二级结构。由表3可知,4个中国莲P1B-ATPase蛋白的α-螺旋结构含量均在30%以上,除NnHMA3之外,其他3个中国莲P1B-ATPase蛋白的无规卷曲结构含量均高于25%;4个蛋白的β-转角结构所占比例均较低,最高含量不超过11%。二级结构元件分布情况显示,4个蛋白以α-螺旋和无规卷曲为主要的二级结构元件,无规卷曲散乱分布于蛋白结构中。

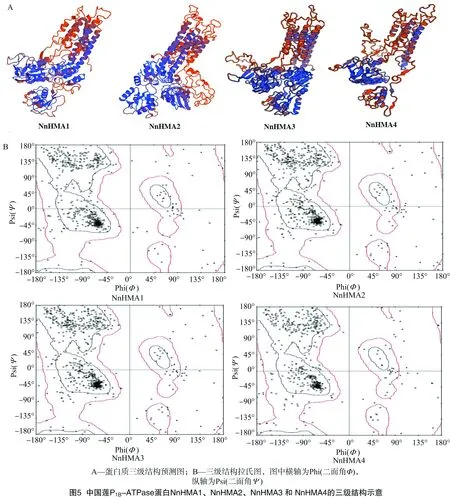
进一步利用同源建模的方法预测这4个蛋白的三级结构。由图5-A可知,中国莲的4个P1B-ATPase蛋白都主要由α-螺旋、无规卷曲元件组成,这与二级结构预测的4个蛋白中α-螺旋、无规卷曲所占比例高的结果相符合,它们与二级结构元件在种类及比例上存在较大的相似性,推测这可能与中国莲P1B-ATPase蛋白在进化过程中存在较多的保守序列有关。此外,NnHMA2和NnHMA3蛋白的三级结构较相似,说明这2种蛋白在亲缘关系上较近,这与图1中系统进化树上显示的结果一致。NnHMA4的三级结构与其他3个蛋白的差异较明显,结合图1中4个蛋白所含模体的类型和数量比对情况,推测这可能与筛选得到的NnHMA4蛋白的编码基因在进化过程中出现部分序列缺失,导致蛋白质序列长度较短有关。
注:蓝色区域表示α-螺旋,绿色区域表示β-转角,红色区域表示延伸链,紫色区域表示无规卷曲。
在蛋白质三级结构预测模型的检验拉式图(Ramachandran)中,蓝线内是最合理的主链二面角Phi(Φ)、Psi(Ψ)分布区域,红色线内则为可接受区域,而在红线之外的区域则为不合理区域。如果预测蛋白质残基的二面角Phi、Psi有90%以上落于蓝线内,则表示预测的蛋白质三维结构合理,具有较稳定的空间结构。由图5-B可见,同源建模构建的4个中国莲P1B-ATPase蛋白空间结构主链二面角Phi、Psi,落于蓝线内中心区域的氨基酸残基占90%以上,落于红线内的主链二面角Phi、Psi亦有9%左右,绝大多数主链二面角Phi、Psi均在正常范围内,仅有少量氨基酸残基的二面角Phi、Psi落于不合理区域。从理论上表明,本研究预测的4个中国莲P1B-ATPase蛋白的三维空间结构是合理可靠的。
3 讨论与结论
P1B-ATPase蛋白是植物吸收、转运重金属离子过程中的一类重要转运蛋白,已有研究结果显示,不同P1B-ATPase蛋白对重金属离子的种类具有一定的选择性,可依据转运重金属的特异性将它们划分为Zn亚类(转运重金属离子Zn2+/Co2+/Cd2+/Pb2+)和Cu亚类(Cu+/Ag+)[28]。本研究通过对拟南芥、水稻、中国莲P1B-ATPase蛋白的保守基序预测,同样将P1B-ATPase蛋白划分为Zn亚类和Cu亚类,拟南芥的AtHMA1、AtHMA2、AtHMA3、AtHMA4与水稻的OsHMA1、OsHMA2、OsHMA3及中国莲的NnHMA1属于Zn亚类,而拟南芥的AtHMA5、AtHMA6、AtHMA7、AtHMA8与水稻的OsHMA4、OsHMA5、OsHMA6、OsHMA7、OsHMA8、OsHMA9以及中国莲的NnHMA2、NnHMA3、NnHMA4属于Cu亚类。这2个亚类的P1B-ATPase蛋白均含有一些保守基序,如DKTGT、GDGxNDxP、TGE、C/SPx、HP。有研究表明,P1B-ATPase蛋白的若干保守基序通常位于蛋白的不同结构域中,直接或间接参与该类蛋白的金属离子跨膜转运,它们的变异可能会导致相应基因及其编码蛋白的功能发生改变[7,10,15,24,26-27],如DKTGT、GDGxNDxP基序通常位于P1B-ATPase蛋白的大胞质环上的磷酸化位点(P-domain)结构域,与该类蛋白的金属离子转运能力有关[32-33],GDGxNDxP基序中的D(天冬氨酸,Asp)残基影响着蛋白与Mg2+的结合[34],TGE基序通常位于小胞质环上的脱磷酸化位点(A-domain),参与金属离子转运过程[25],而HP基序一般较为保守,位于ATP结合区域(N-domain),有研究表明,该区域参与了蛋白与ATP相互结合的过程,HP基序在此过程中扮演着重要角色[35]。此外,Mills等的研究结果显示,C/SPx基序中的Cys(半胱氨酸)对蛋白的活性起到重要作用,它的突变可以导致P1B-ATPase蛋白转运金属离子活性缺失[36]。本研究发现,中国莲NnHMA4不含有GDGxNDxP基序,但含有上述的其他保守基序,由此推测,NnHMA4可能不参与Mg2+的转运过程。本研究通过采用中国莲、拟南芥、水稻的 P1B-ATPase蛋白的氨基酸序列构建进化树发现,3个物种的P1B-ATPase蛋白明显聚类为2个亚类,其中中国莲的NnHMA1属于Zn亚类,中国莲的NnHMA2、NnHMA3、NnHMA4属于Cu亚类,表明依据序列的相似性程度推断蛋白的功能具有一定的准确性。
探明蛋白质的二、三级结构是解析蛋白空间结构与功能相关性的关键,利用生物信息学软件预测蛋白的二、三级结构是一种简捷、有效的方法[37]。本研究对中国莲的P1B-ATPase蛋白结构进行生物信息学预测和分析,二级结构预测结果显示,4个中国莲P1B-ATPase蛋白由α-螺旋、无规卷曲、延伸链和β-转角4个元件构成,其中无规卷曲和α-螺旋所占比例之和高于50%,在NnHMA1中无规卷曲在该蛋白所有二级结构单元中所占比例最大,NnHMA2、NnHMA3和NnHMA4中均以α-螺旋为主。目前,同源建模、折叠识别和从头预测法是蛋白质三级结构预测最常用的3种方法[37]。本研究利用同源建模方法对中国莲P1B-ATPase蛋白进行三级结构模拟,由三级结构图可知,4个蛋白以α-螺旋、无规卷曲为主要结构单元,这与二级结构预测的结果相符合。此外,还发现不同中国莲P1B-ATPase蛋白的序列中虽然存在众多保守基序如DKTGT、HP等,部分空间结构也存在一定的相似性,但它们的空间整体构型存在较大差异,特别是进化关系相对较远的蛋白之间差异更加明显,这可能与它们所具有的转运不同重金属离子的功能有关。
已有的关于植物P1B-ATPase蛋白及其编码基因的结构和功能等方面研究,目前主要集中在拟南芥、水稻、大麦、大豆等陆生草本植物上,在大型水生植物上的研究投入相对较少。而大型水生植物在生长速度、生物量和潜在的景观效应方面具有一定优势,使其在植物修复水体重金属污染方面具有较大的应用价值。本研究应用生物信息学手段系统预测和分析了中国莲中可能存在的P1B-ATPase蛋白亚家族成员的基本信息,探讨了其结构与重金属转运、吸收等功能的关系,这些信息为进一步探究大型水生植物的重金属修复分子机制提供了理论参考,对今后开展基因工程育种研究具有重要的指导意义。

