花生AhFAD2基因抑制表达的转基因后代分析
徐平丽,唐桂英,毕玉平,柳展基,单雷
花生基因抑制表达的转基因后代分析
徐平丽1,2,唐桂英1,2,毕玉平1,2,柳展基3,单雷1,2
1 山东省农业科学院 生物技术研究中心,山东 济南 250100 2 山东省作物遗传改良与生理生态重点实验室,山东 济南 250100 3 山东省农业科学院 棉花研究中心,山东 济南 250100

徐平丽, 唐桂英, 毕玉平, 等. 花生AhFAD2基因抑制表达的转基因后代分析.生物工程学报, 2018, 34(9): 1469–1477.Xu PL, Tang GY, Bi YP, et al. Analysis of the peanut transgenic offspring with depressing AhFAD2 gene. Chin J Biotech, 2018, 34(9): 1469–1477.
FAD2 (Δ12fatty acid desaturase,Δ12FAD或FAD2) 是催化油酸在脂肪酸碳链Δ12位脱氢生成亚油酸的关键酶。在花生中,FAD2酶活性下降或失活可提高籽粒中油酸的相对含量,改善花生籽粒及制品的品质和氧化稳定性。通过将种子特异性表达Lectin启动子和CaMV35S启动子驱动的倒位重复基因RNAi干扰结构转入花生,获得了以丰花1号(FH1) 和花育23 (HY23) 为受体、携带上述2种转化结构、稳定遗传的花生转基因纯合体株系,转基因花生主要农艺性状与非转基因对照基本一致。实时荧光定量分析发现,各转基因株系发育种子中基因的转录水平普遍下调。气相色谱法进一步测定了部分转基因后代株系种子的脂肪酸含量及组成,籽粒中油酸含量分别平均提高了15.09% (HY23为受体)、36.40% (FH1为受体),相应地,亚油酸含量平均下降了16.19%、29.81%,油亚比平均增加了38.02%、98.10%。各转基因株系的油酸含量显著提高;且在以FH1为受体的转基因株系后代籽粒以及种子特异性启动子驱动的转化结构中,RNAi的抑制效果更明显。通过RNAi技术抑制花生FAD2基因的表达,可以有效提高花生籽粒油酸含量。该技术体系可以为花生品质育种提供借鉴。
花生,Δ12脂肪酸脱氢酶,RNA干扰,转基因,油酸与亚油酸比值
花生是我国重要的经济作物和油料作物,花生籽粒含油量达50%左右,油酸、亚油酸分别占其总脂肪酸含量的36%–67%和15%–43%[1]。油酸为18碳单不饱和脂肪酸,化学性质相比亚油酸、亚麻酸等多不饱和脂肪酸要稳定得多。提高油酸含量可以改善花生及花生制品的口味和氧化稳定性。Mozingo等报道称,烘焙的高油酸花生比普通含量油酸花生贮存期延长8倍[2-4]。因此,提高花生籽粒中油酸的相对含量已成为目前花生品质育种的重要目标之一[5-7]。
微粒体Δ12脂肪酸脱氢酶 (Δ12fatty acid desaturase,Δ12FAD或FAD2),是催化脂肪酸碳链上油酸在C12处脱氢生成亚油酸的关键酶,对于植物贮藏脂和膜脂中的不饱和脂肪酸与饱和脂肪酸的比例有着重要调控作用[8-9]。花生基因组中存在两对能编码完整蛋白的基因 (和)。和基因编码区序列变化所引起编码蛋白活性的差异与花生籽粒油酸、亚油酸含量密切相关[10-11]。
近几年,反义、共抑制或RNAi技术在改良脂肪酸组成方面的研究,已在油菜、大豆、棉花、亚麻等作物取得了突破[12-17]。Peng等构建独特内含子间隔的脂肪酸延长酶1 (Fatty acid elongase1,) 基因融合片段的发卡RNA干扰载体,同时抑制基因的表达,使得转基因油菜种子中的油酸含量从15%提高到85%;多不饱和脂肪酸含量下降到10%,其中芥酸含量为0[12]。Lee等通过反义RNA特异抑制种子基因,获得了高油酸、低多不饱和脂肪酸的油菜[13]。Kinney等1996年通过正义-共抑制途径,降低FAD2的酶活性,使得转基因大豆的油酸含量从67%提高到超过80%[14]。Buhr等则利用RNAi同时抑制酰基转移酶基因 () 和基因的表达,获得了低棕榈酸和高油酸的转基因大豆[15]。Liu等通过构建种子特异性启动子驱动的hpRNA (Hairpin RNA) 载体,抑制基因的表达,使得棉籽油中油酸的含量从15%提高到77%,亚油酸含量从60%下降到4%[16]。Chen等通过RNAi干扰基因的表达,使得转基因T3代亚麻中油酸含量达到77%[17]。
我们实验室前期构建了基因RNAi抑制表达载体,通过农杆菌介导法,分别将花椰菜花叶病毒 (Cauliflower mosaic virus,CaMV) 35S启动子和种子特异性表达的大豆凝集素 (Lectin) 启动子驱动的倒位重复片段转入花生,获得了一批PCR检测的阳性转基因植株[18]。本研究在此基础上,经遗传转化、温室种植、分子水平检测筛选等,获得10个稳定遗传的转基因株系。利用qRT-PCR分析了基因在部分转基因花生后代种子中的转录水平表达情况;调查了转基因株系的主要农艺性状;利用气相色谱测定了部分转基因花生后代种子的脂肪酸含量和组成。
1 材料与方法
1.1 植物材料
花生L.品种:花育23号 (HY23,CK1即非转基因对照1),由山东省花生研究所提供;丰花1号 (FH1,CK2即非转基因对照2),由山东农业大学农学院万勇善教授提供。
花生转基因株系由本实验室通过RNAi策略,采用农杆菌介导法将基因倒位重复结构转入受体花生FH1[18]和HY23获得。文中分析的以HY23为受体的株系包括:201301021903、201301061108、201201030109 (带有种子特异启动子Lectin,下文分别简写为H1903、H1108、H0109) 和201302011308、201202100319、201202120104 (带有组成型表达启动子CaMV35S,下文分别简写为H1308、H0319、H0104)。FH1为受体的株系:201001010104、201001040101 (带有种子特异启动子Lectin,下文分别简写为F0104、F0101) 和 201002123501、201202060904 (带有组成型表达启动子CaMV35S,下文分别简写为F3501、F0904)。花生转基因后代及非转基因对照均种植在山东省农业科学院生物技术研究中心饮马泉实验基地温室。
1.2 试剂
DNA聚合酶、PrimeScript Ⅱ1st Strand cDNA Synthesis Kit、DNA分子量标准DL2000购自大连TaKaRa生物工程有限公司。TransStart Green qPCR SuperMix UDG购自北京全式金生物技术有限公司。新型植物RNA快速提取试剂盒购自北京华越洋生物科技有限公司。PCR引物由上海生工生物工程有限公司合成。其余均为国产或进口分析纯试剂。
1.3 实时荧光定量PCR引物设计
遵循荧光定量引物设计原则,使用Primer5.0引物设计软件设计荧光定量PCR引物。引物由上海生工生物工程技术服务有限公司合成。
qRT-PCR检测花生内源基因转录水平的引物序列,2F:5′-CGACCGCAACGAAGT GTT-3′;2-R:5′-CCCTCCCTGGTGGATTGT-3′;花生内参Actin基因转录水平的检测引物序列,Actin-F:5′-ATGTATGTAGCCATCCAAG-3′;Actin-R:5′-ACCAGAGTCCAGAACAATA-3′。
1.4 方法
1.4.1 稳定遗传的转基因株系获得
实验室前期已获得以FH1为受体的RNAi抑制基因表达的2种转化结构的阳性转基因花生植株[18]。在此基础上,以HY23为受体进行了同样的转化,最终,共获得2个转化结构的PCR检测阳性的转基因T0代植株20株,部分植株PCR产物经测序验证。来源于受体FH1和HY23的转基因T0代植株经3–4代温室种植、田间取样PCR检测、DNA序列测定筛选等过程,最终获得单拷贝插入稳定遗传的纯合体株系10个,用于后续实验。
1.4.2基因在转基因后代的转录水平分析
按照北京华越洋生物科技有限公司新型植物RNA快速提取试剂盒提供的方法,分别提取转基因株系及非转基因对照开花后30 d和60 d未成熟种子的总RNA。紫外分光光度计测定其纯度、浓度,取0.2–0.5 μg 总RNA在1.2%琼脂糖凝胶电泳,检测总RNA的完整性。取0.5 μg总RNA,在10 μL体系中参照TaKaRa PrimeScript Ⅱ 1st Strand cDNA Synthesis Kit提供步骤和条件合成cDNA第一链,储存于–20 ℃备用。
选择受体为HY23的转基因后代4个转化事件 (包括Lectin启动子的2个,CaMV35S启动子的2个),受体为FH1的转基因后代4个转化事件 (包括Lectin启动子的2个,CaMV35S启动子的2个),以花生Actin基因作为参比,实时荧光定量法对转基因种子中基因转录水平表达情况进行分析。每一株系的检测均设3个生物学重复,相对表达量用2–ΔΔCT的表示。
荧光实时定量PCR反应体系包括:10 μL 2×TransStart Green qPCR SuperMix UDG,10 μmol/LPCR正向引物和反向引物各0.4 μL,cDNA模板2 μL (约50 ng),Passive Reference Dye (50×) 0.4 μL,ddH2O补充体积至20 μL。反应条件:95 ℃预变性2 min;然后95 ℃变性10 s,60 ℃退火延伸30 s,共40个循环。反应由ABI公司的7500型荧光定量PCR仪完成。
1.4.3 转基因花生株系主要农艺性状调查
主要通过观察比较方法,对种植在温室的转基因花生及对照的株高、花期、生育期及感病情况等进行调查。收获后的花生,则待自然晒干,分别选取不同转基因株系的10个单株,每单株随机选10个饱果,计算百果重;之后,随机选取100粒饱满种子,计算百仁重;统计记录单株饱果数。
1.4.4 气相色谱法 (Gas chromatography,GC) 检测转基因花生籽粒脂肪酸组成及含量
近红外反射光谱法首先从T2代开始,对转基因后代成熟籽粒的油酸含量进行初步筛选,选取油酸含量升高的后代株系进行后续实验。之后,综合主要农艺性状调查和近红外反射光谱测定结果,在每个表达结构的转基因株系中选取2–3个转化事件,每个转化事件选取3个所测结果接近平均值的单株籽粒为气相色谱测定对象,每个样品10 g。参考Sukhija等[19]改进的一步法抽提脂肪酸并进行甲酯化,利用配置色谱柱DB-23 (60.0 m×250 μm×0.25 μm)、检测器FID 270 ℃的气相色谱仪Agilent 6890,参考国标GB/T 21514-2008[20]分析种子脂肪酸组成及其含量,测定由中国农业大学饲料分析测试中心完成。
2 结果与分析
2.1 AhFAD2基因在转基因花生种子中的转录水平分析
qRT-PCR结果显示 (图1),与非转基因对照相比,花生基因的相对表达量在各转基因株系后代发育种子中均有不同程度的降低,但降低幅度在不同启动子驱动的转化结构、种子不同的发育时期及不同的转基因株系间不同。以HY23为受体的4个转基因株系,在种子发育早期 (花后30 d),与非转基因对照相比,所测基因的相对表达量下降幅度为8.45%–51.30%,平均下降23.70%,种子特异性启动子、CaMV35S启动子驱动的转化结构平均下降幅度分别为34.95%、12.46%;而在种子发育后期 (花后60 d),基因表达受到更明显抑制,基因相对表达量下降幅度为20.14%–88.98%,平均下降68.76%,种子特异性启动子、CaMV35S启动子驱动的转化结构平均下降幅度分别为82.96%、54.56%。以FH1为受体的4个转基因株系中,与非转基因对照相比,花后30 d发育种子中基因的相对表达量下降幅度为16.38%–80.47%,平均下降48.88%,种子特异性启动子、CaMV35S启动子驱动的转化结构平均下降幅度分别为74.06%、23.70%;而花后60 d种子中,基因表达受到的抑制更明显,相对表达量下降幅度为31.16%–94.4%,平均下降71.04%,种子特异性启动子、CaMV35S启动子驱动的平均下降幅度分别为86.00%、56.09%。因此,种子特异性启动子驱动下,抑制基因表达的效果更明显;在以FH1为受体的转基因株系籽粒中,基因的相对表达量较低,RNAi的抑制效果更理想。
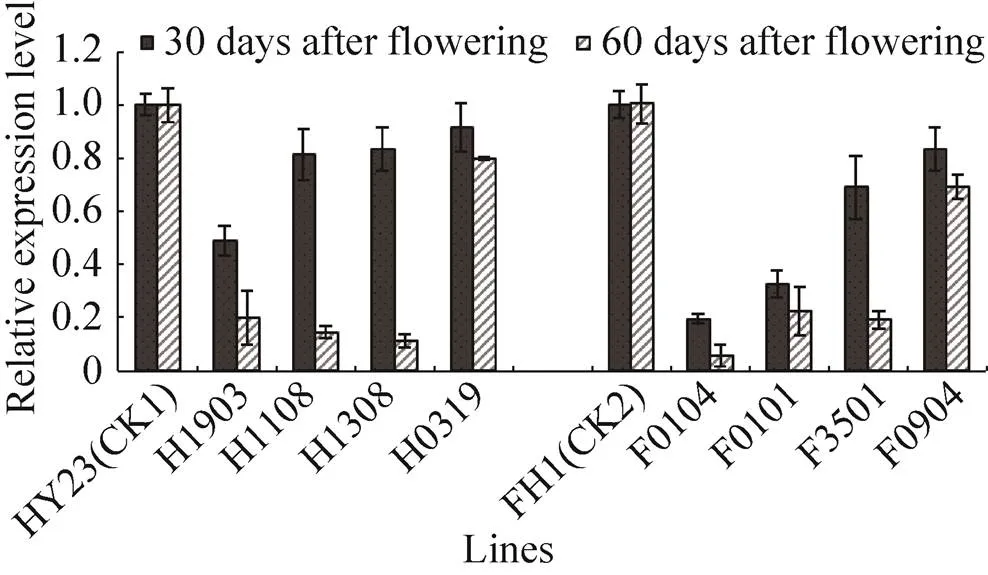
图1 AhFAD2基因在不同转基因株系发育种子中的转录水平
2.2 转基因花生主要农艺性状调查
对2个转化结构的转基因花生主要农艺性状分析表明,转基因花生生育期分别为130 d和135 d左右 (以HY23、FH1为受体),主茎平均高度分别为37.0 cm和45.0 cm,均属连续开花型,整个生命周期无明显感病症状,分别与非转基因HY23、FH1的表现基本一致。收获后,调查单株花生荚果及种子的部分性状,转基因花生果形、粒形、种皮颜色与非转基因对照基本一致;百仁重、百果重等也无明显变化 (图2,表1)。调查结果可见,无论是组成型表达35S启动子还是种子特异性Lectin启动子驱动的基因抑制表达都没有引起转基因花生主要农艺性状的明显改变。
2.3 气相色谱法 (GC) 分析转基因花生株系籽粒脂肪酸组成
综合考虑近红外反射光谱检测情况 (数据未展示) 和农艺性状调查结果,我们共选取2个转化结构的10个不同转基因代表单株 (6个HY23为受体,4个FH1为受体),利用气相色谱法测定了籽粒中的主要脂肪酸含量及组成 (表2)。分析发现,花生转基因株系种子中的8种主要脂肪酸含量发生了变化,包括棕榈酸 (C16:0)、硬脂酸 (C18:0)、油酸 (C18:1n9c)、亚油酸 (C18:2n6c)、花生酸 (C20:0)、山嵛酸 (C22:0)、木蜡酸 (C24:0) 等 (表3,含量低于2%的数据未展示)。以HY23为受体的转基因株系种子中油酸含量从44.19%提高到48.15%–53.69%,平均提高了15.09%,最高可达21.5% (H1308);亚油酸含量从34.48%下降到26.19%–31.04%,平均下降16.19%;油亚比从1.28提高到1.55–2.05,平均提高了38.02%。以FH1为受体的转基因株系种子中油酸含量从37.20%提高到42.01%–55.22%,平均提高了36.40%,最高提高幅度为48.44% (F0104);亚油酸含量从40.51%下降到24.87%–34.36%,平均下降29.81%;油亚比从0.92提高到1.22–2.22,平均提高了98.10%。转基因株系种子中油酸含量、油亚比普遍提高,转基因株系与非转基因对照之间种子油酸、亚油酸含量差异显著,说明倒位重复序列片段串联组成的RNAi结构,可以有效抑制基因的表达;不同的转基因株系之间,油酸含量差异显著,不同启动子驱动下的油亚比提高率差异明显,说明不同的启动子驱动、不同的转化受体、不同株系间基因表达的RNAi抑制效果明显不同 (表2,图3)。

图2 收获期转基因花生和非转基因对照的表型
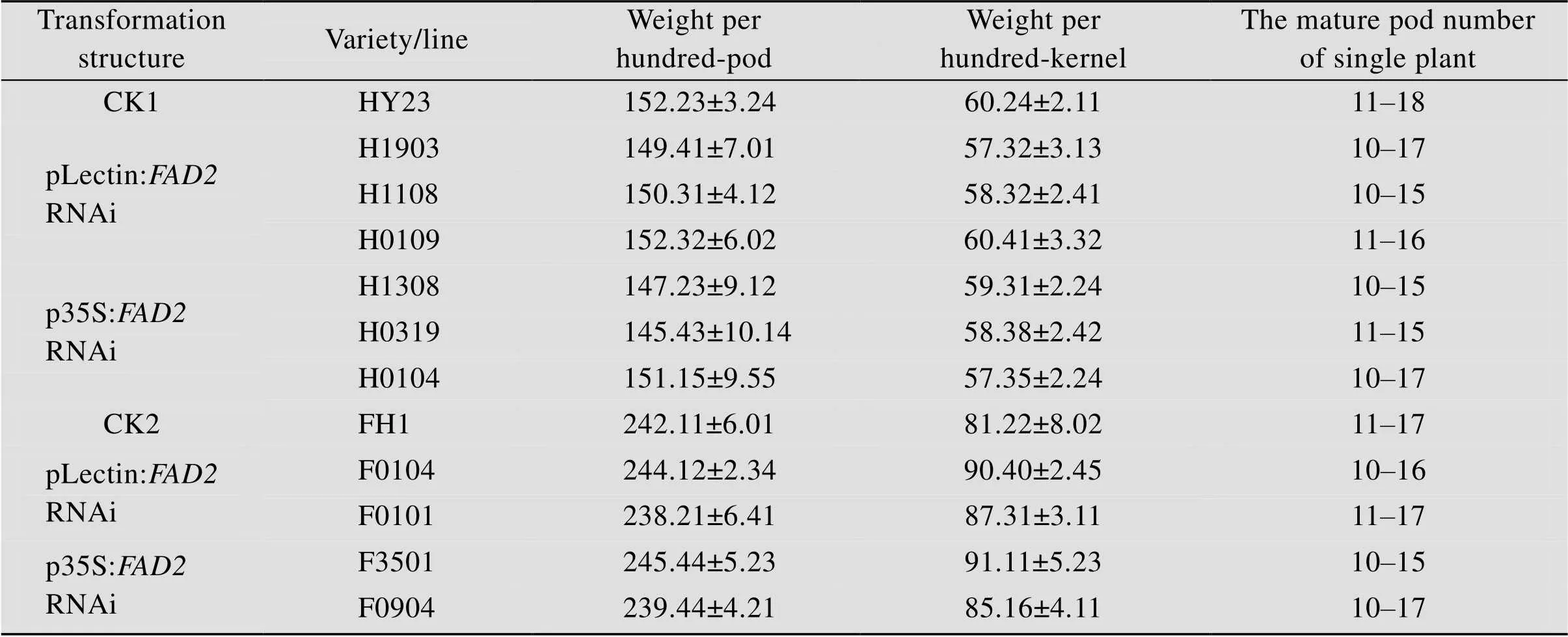
表1 部分转基因花生荚果产量性状统计
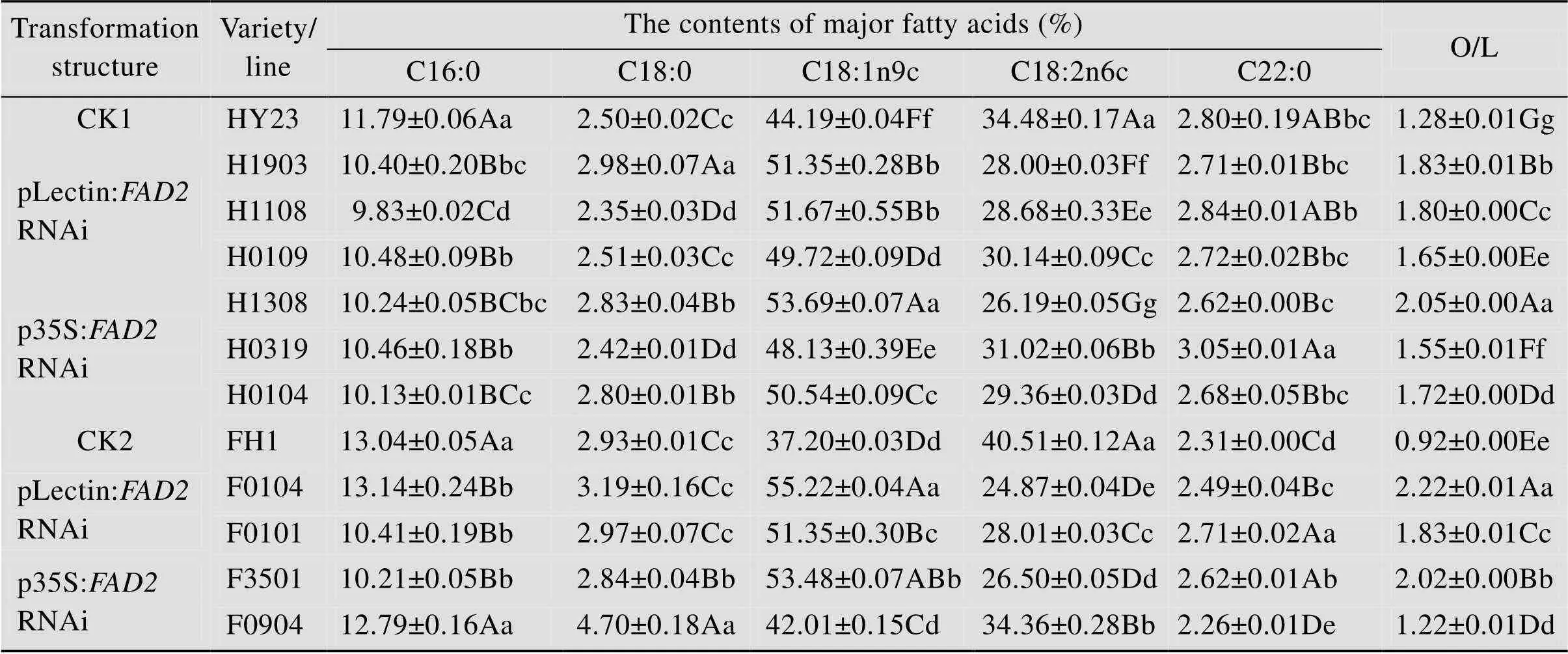
表2 转基因花生籽粒中主要脂肪酸组分含量
CK1 is untransformed control HY23; CK2 is untransformed control FH1; capital letter represents significant variation at the 0.01 probability level; lowercase letter represents significant difference at the 0.05 probability level.
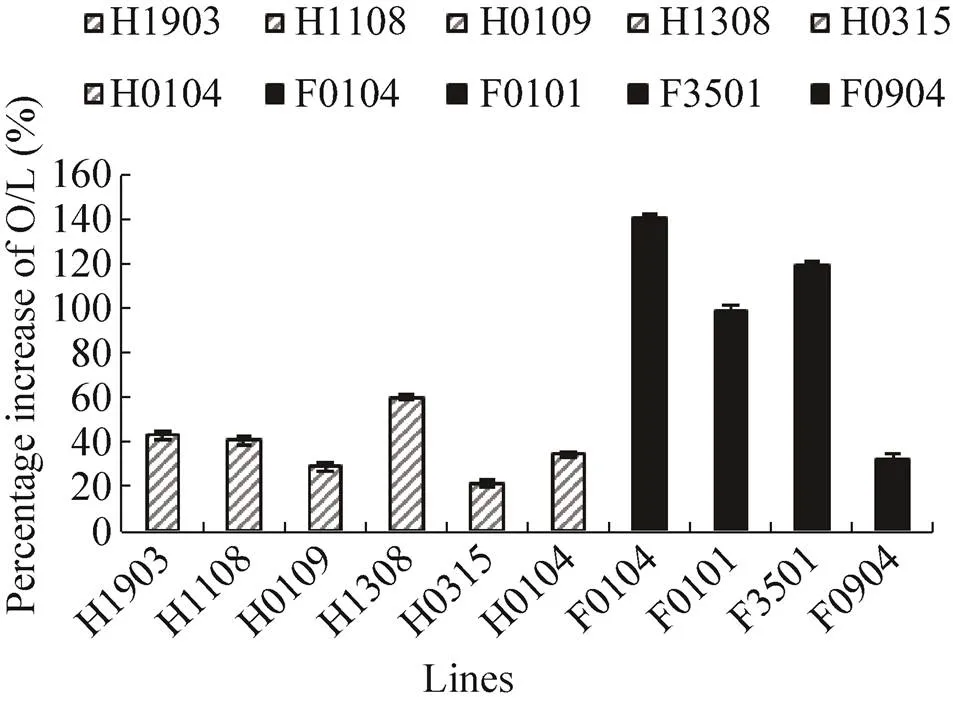
图3 花生不同转基因株系籽粒的油亚比增加率
3 讨论
3.1 RNAi有效抑制转基因花生种子中FAD2基因的表达
花生中含有两个基因 (、),它们在ORF区具有99%的序列一致性,因此,我们选择位于编码区5′端9–235 bp的保守区域设计RNAi表达结构可以同时抑制和的表达[18]。本研究获得的转基因株系种子中基因的表达受到不同程度抑制,但其他表型没有发生明显变化。一方面,干扰片段与其他基因的编码序列或EST序列没有同源性,不会意外沉默其他基因;另一方面,花生高油酸突变体的研究表明,正常生长条件下,这两个基因在根、叶等营养体中的抑制表达或编码酶活性降低,并不会造成明显的表型变化。
Stoujesijk等在拟南芥中的研究表明,FAD2的RNA干扰效果在拟南芥子代稳定遗传[21]。我们的研究结果也基本印证了这一结论。本研究qRT-PCR分析的是T3或T4代纯合株系种子中的转录水平,发现大多数转基因株系中RNAi抑制基因表达的抑制效果明显,说明倒位重复序列片段串联组成的RNAi结构,可以有效抑制基因的表达。
对基因在不同转基因株系发育种子中的转录水平分析发现,种子特异性启动子驱动的转化结构,RNAi抑制基因表达的效果更为明显。这或许是由于CaMV35S启动子属于组成型的表达启动子,在整株水平上抑制基因的表达容易影响植株的膜脂结构、植株的生长速率及组培再生苗的生根等[18]。Lectin启动子为种子特异表达启动子,仅在种子发育阶段驱动RNAi抑制基因的表达,一般不会对植株整体造成影响。脂肪酸代谢是植株生命体的基本生命活动之一,基因是脂肪酸代谢途径的关键酶,因此,从籽仁油酸含量改良角度出发,种子特异启动子调控的RNAi转化结构,在改善花生油品质等方面会有更大的应用空间。
3.2 转基因花生籽粒的O/L比不同程度提高
FAD2是催化油酸在脂肪酸碳链C12位脱氢生成亚油酸的关键酶,与植物贮藏脂中油酸、亚油酸含量密切相关。花生中和两基因编码的酶均具有催化活性,但它们对油酸含量的贡献不同,基因突变的贡献性更大一些。编码区448位一个点突变致使编码的酶活性几乎丧失,然而仅这一基因突变,花生籽粒只表现出普通油酸性状;而突变导致的表达降低或酶活性丧失,则花生籽粒会表现出中等油酸性状;两基因同时突变,突变体籽粒表现高油酸性状,油酸含量可达75%以上[10,22]。
本研究以HY23为受体的转基因株系种子中油亚比从1.28提高到1.55–2.05,平均提高了38.02%;而FH1为受体的转化事件种子中油亚比从0.92提高到1.22–2.22,平均提高了98.10%。不同受体花生获得的转基因材料种子油亚比提高幅度明显不同。以往的研究发现,转化受体HY23基因发生突变,编码的酶本身已不具备催化功能,因此其籽粒的O/L与其他珍珠豆型品种相比有所提高;而受体FH1携带的是野生型和基因,籽粒O/L处于较低水平[10]。按设计我们转入的干扰片段应对两基因具有同等的抑制效果,本研究发现,转录水平上,开花60 d的HY23和FH1转基因株系间的抑制效果确实没有明显差别,但由于HY23受体突变的AhFAD2A本身的酶活性几近丧失,因此转基因种子中基因的抑制效果仅为基因抑制表达的结果,O/L提高幅度就相对偏低。FH1为受体的转基因株系和两基因表达均受到抑制,尽管它们的油酸含量和O/L总体上与HY23为受体的转基因株系持平,但油酸含量和O/L的提高幅度相对更大。
以往在油菜、棉花、大豆等作物高油酸转基因研究中发现,介导的转录后基因沉默策略比反义RNA、RNA共抑制等技术更为有效[12-17]。以油菜基因的3′UTR (Untranslated region,UTR) 219 bp片段为干扰片段的结构,使得转基因油菜油酸含量从67%提高到85%[13]。油菜、两个基因的融合片段形成的结构,可同时抑制转基因油菜种子中与两个基因的表达,使油酸含量高达85%且不含芥子酸[12]。Liu等在棉花中抑制ghFAD2-1基因表达,也使转基因棉花籽粒中油酸从15%提高到了77%[16]。然而,本研究两种受体材料获得的转基因株系中油酸含量最高的分别为53.69%、55.22%,O/L分别为1.55–2.05、1.22–2.22。此结果与黄冰艳等获得的RNAi转基因花生T1代籽粒O/L接近[23]。黄冰艳[23]等在研究中采用的也是具有内含子间隔的具发卡环的RNAi结构,但文中没有介绍所选基因干扰片段的位置。同一转化结构产生的不同转基因株系目标性状之间存在差异,这在其他植物转基因研究中也常发生,推测是由于外源片段插入受体基因组的不同位置所造成[17,21]。但是,同样运用hpRNA介导的策略抑制基因的表达,我们和黄冰艳等[23]在花生中的研究结果与他人在油菜、棉花等作物中的结果相比差距很大,可能是由于所选靶基因片段的位置不同造成了不同的抑制效果。
[1] Moore KM, Knauft DA. The inheritance of high oleic acid in peanut. J Hered, 1989, 80(3): 252–253.
[2] Braddock JC, Sims CA, O′Keefe SF. Flavor and oxidative stability of roasted high oleic acid peanut. J Food Sci, 1995, 60(3): 489–493.
[3] Mugendi JB, Sims CA, Gorbet DW, et al. Flavor stability of high-oleic peanuts stored at low humidity. J Am Oil Chem Soc, 1998, 75(1): 21–25.
[4] Mozingo RW, O’Keefe SF, Sanders TH, et al. Improving shelf life of roasted and salted inshell peanuts using high oleic fatty acid chemistry. Peanut Sci, 2004, 31(1): 40–45.
[5] Rogalski M, Carrer H. Engineering plastid fatty acid biosynthesis to improve food quality and biofuel production in higher plants. Plant Biotech J, 2011, 9(5): 554–564.
[6] Napier JA. The production of unusual fatty acids in transgenic plants. Annu Rev Plant Biol, 2007, 58: 295–319.
[7] Takeno S, Sakuradani E, Tomi A, et al. Improvement of the fatty acid composition of an oil-producing filamentous fungus,1S-4, through RNA interference with Δ12-desaturase gene expression. Appl Environ Microbiol, 2005, 71(9): 5124–5128.
[8] Jung S, Swift D, Sengoku E, et al. The high oleate trait in the cultivated peanut [L.]: I. Isolation and characterization of two genes encoding microsomal oleoyl-PC desaturases. Mol Gen Genet, 2000, 263(5): 796–805.
[9] Jung S, Powell G, Moore K, et al. The high oleate trait in the cultivated peanut [L.]: II. Molecular basis and genetics of the trait. Mol Gen Genet, 2000, 263(5): 806–811.
[10] Zhou LX, Tang GY, Chen G, et al. Correlation betweenpolymorphism and oleic acid/linoleic acid ratio in peanut seeds. Acta Agron Sin, 2011, 37(3): 415–423 (in Chinese). 周丽侠, 唐桂英, 陈高, 等. 花生基因的多态性及其与籽粒油酸/亚油酸比值间的相关性. 作物学报, 2011, 37(3): 415–423.
[11] Nayeri FD, Yarizade K. Bioinformatics study of delta-12 fatty acid desaturase 2 (FAD2) gene in oilseeds. Mol Biol Rep, 2014, 41(8): 5077–5087.
[12] Peng Q, Hu Y, Wei R, et al. Simultaneous silencing ofandgenes affects both oleic acid and erucic acid contents inseeds. Plant Cell Rep, 2010, 29: 317–325.
[13] Lee KR, Kim EH, Roh KH, et al. High-oleic oilseed rapes developed with seed-specific suppression ofgene expression. Appl Biol Chem, 2016, 59(4): 669–676.
[14] Kinney AJ. Development of genetically engineered soybean oils for food applications. J Food Lipids, 1996, 3(4): 273–292.
[15] Buhr T, Sato S, Ebrahim F, et al. Ribozyme termination of RNA transcripts down-regulate seed fatty acid genes in transgenic soybean. Plant J, 2002, 30(2): 155–163.
[16] Liu Q, Singh SP, Green AG. High-stearic and high-oleic cottonseed oils produced by hairpin RNA-mediated post-transcriptional gene silencing. Plant Physiol, 2002, 129(4): 1732–1743.
[17] Chen YR, Zhou XR, Zhang ZJ, et al. Development of high oleic oil crop platform in flax through RNAi-mediated multiplegene silencing. Plant Cell Rep, 2015, 34(4): 643–653.
[18] Zhang XQ, Shan L, Tang GY, et al. Transformation of RNAi suppressed expression vector containing of Δ12fatty acid desaturase gene viainfection inL. Chin J Oil Crop Sci, 2007, 29(4): 409–415, 419 (in Chinese). 张小茜, 单雷, 唐桂英, 等. 农杆菌介导的花生Δ12脂肪酸脱氢酶基因RNAi抑制表达遗传转化研究. 中国油料作物学报, 2007, 29(4): 409–415, 419.
[19] Sukhija PS, Palmquist DL. Rapid method for determination of total fatty acid content and composition of feedstuffs and feces. J Agric Food Chem, 1988, 36(6): 1202–1206.
[20] 中华人民共和国国家质量监督检验检疫总局. GB/T 21514−2008/ISO/TS 17764:2002, 饲料中脂肪酸含量的测定. 北京: 中国标准出版社, 2008.
[21] Stoutjesdijk PA, Singh SP, Liu Q, et al. HpRNA-mediated targeting of the Arabidopsisgene gives highly efficient and stable silencing. Plant Physiol, 2002, 129(4): 1723–1731.
[22] Wang ML, Barkley NA, Chen ZB, et al.gene mutations significantly alter fatty acid profiles in cultivated peanutsBiochem Genet, 2011, 49(11/12): 748–759.
[23] Huang BY, Zhang XY, Miao LJ, et al. RNAi transformation ofgene and fatty acid analysis of transgenic seeds. Chin J Oil Crop Sci, 2008, 30(3): 290–293 (in Chinese). 黄冰艳, 张新友, 苗利娟, 等. 花生基因RNAi载体转化及转基因籽粒脂肪酸分析. 中国油料作物学报, 2008, 30(3): 290–293.
(本文责编 郝丽芳)
Analysis of the peanut transgenic offspring with depressinggene
Pingli Xu1,2, Guiying Tang1,2, Yuping Bi1,2, Zhanji Liu3, and Lei Shan1,2
1 Bio-Tech Research Center, Shandong Academy of Agricultural Sciences, Jinan 250100, Shandong, China 2 Shandong Provincial Key Laboratory of Crop Genetic Improvement, Ecology and Physiology, Jinan 250100, Shandong, China 3 Shandong Cotton Research Center, Shandong Academy of Agricultural Sciences, Jinan 250100, Shandong, China
The delta-12 fatty acid desaturase (Δ12FAD or FAD2) is a key enzyme that catalyzes oleic acid to linoleic acid by dehydrogenation at Δ12position offatty acid carbon chain. In peanut, reduction or loss of FAD2 activity could enhance the relative content of oleic acid in kernels, and improve the quality and oxidation stability of peanut kernels and products. RNA interference (RNAi) technology could lead to non-expression or down-regulated expression ofgene. We constructed two RNA interference expression vectors with the inverted repeat sequence of partialgene, which were driven separately by cauliflower mosaic virus (CaMV) 35S promoter or soybean agglutinin lectin seed-specific promoter. Homozygous transgenic lines carrying the two constructs stably in genetics were developed by peanut genetic transformation. There were no significant differences between the transgenic lines and the control through investigating the main agronomic traits. We analyzed the transcriptional level expression ofgene in transgenic lines and the control by real-time fluorescence quantitative PCR (qRT-PCR). The results suggested that the target genes of transgenic lines were likely suppressed by RNA interference, but showed different transcriptional levels in different peanut transgenic lines. Compared with untransformed lines, the resulting down-regulation ofgene resulted in a 15.09% or 36.40% increase in oleic acid content in the seeds of transformed HY23 and FH1 lines respectively, and the content of linoleic acid decreased by 16.19% or 29.81%, correspondingly, the ratio of oleic acid and linoleic acid (O/L) improved by 38.02%, 98.10%. The oleic acid content had significant differences between the two transformation constructs, and also among different transgenic lines. Moreover, the inhibition effect of RNAi was more obvious in the transgenic lines with FH1 as the receptor, and with transformation structure driven by seed specific promoter. The suppressed expression ofgene enabled the development of peanut fatty acid, which indicated that RNA interference would be a reliable technique for the genetic modification of peanut seed quality and the potential for improvement of other traits as well.
peanut(L.), Δ12fatty acid desaturase (FAD2), RNA interference (RNAi), transgenic, the ratio of oleic acid and linoleic acid (O/L)
December 23, 2017;
June 8, 2018
National Natural Science Foundation of China (Nos. 31201272, 31470349), Agricultural Improved Seed Project of Shandong Province (2014-2017).
Lei Shan. Tel: +86-531-66659435; E-mail: shlei1025@sina.com
国家自然科学基金(Nos. 31201272,31470349),山东省农业良种工程(2014-2017) 资助。
2018-07-06
10.13345/j.cjb.170508
http://kns.cnki.net/kcms/detail/11.1998.Q.20180709.1036.001.html

