耳聋基因检测技术应用和研究现状
查树伟 查佶 傅雅丽 邹文霓 许豪勤
耳聋基因筛查研究主要集中在耳聋患者、正常孕期人群、新生儿以及孕(婚)前耳聋基因筛查人群,筛查方法多采用耳聋基因芯片检测常见遗传性耳聋的4个基因9个位点。目前临床耳聋基因筛查和诊断研究还涉及基质辅助激光解吸电离飞行时间质谱、全外显子组测序、多重置换扩增和单核苷酸多态分型等技术。本文简要阐述有关耳聋基因检测技术应用和研究现状。
一、6个通过国家食品药品监督局(SFDA)批准的耳聋基因筛查试剂盒检测特点
目前耳聋基因筛查检测方法主要是2009年—2014年6个通过SFDA批准的耳聋基因筛查试剂盒[1],孕前耳聋基因筛查研究采用9项遗传性耳聋基因检测试剂盒,检测4个常见耳聋基因中的9个位点,包括GJB2基因c.35 del G、c.176 del 16、c.235 del C、c.299 del AT,GJB3基因c.538 C>T,SLC26A4基因c.2168 A>G、IVS7-2 A>G和线粒体12S rRNA基因1494 C>T、1555 A>G。6个试剂盒检测特点见表1。
二、耳聋基因筛查(检测)获得专利的检测方法
2014年—2016年(万方数据-专利:“耳聋基因”,检索日期2017年2月12日)[2]中国已有16个申请获得的耳聋基因筛查(检测)专利(见表2)。在这些专利中,中南大学湘雅医院(冯永等)的专利“耳聋基因筛查芯片”具有高通量和覆盖面广的优势:芯片共包含384个检测位点,涵盖了线粒体及46个位于常染色体上的不同耳聋基因,包含240个耳聋突变位点和144个单核苷酸多态性(SNP)位点,其中耳聋突变位点中包括200个非综合症耳聋位点和40个常见综合征耳聋位点。济南英盛生物技术有限公司(冯振等)分别申请并获得3项有专门针对性基因的检测专利:①“一种先天性耳聋基因多通道荧光PCR检测试剂盒”,首次实现了应用多通道荧光PCR方法在同一个反应管中同时检测先天性耳聋的8个突变位点,包括GJB2基因的6个位点和GJB3基因的2个位点;②“一种药物性耳聋基因多通道荧光PCR检测试剂盒”,同时检测药物性耳聋的4个突变位点,即mtDNA 12S rRNA基因的4个位点,以有效预防药物性耳聋发生;③“一种迟发性耳聋基因多通道荧光PCR检测试剂盒”,同时检测迟发性耳聋的7个突变位点,即SLC26A4基因的7个位点,以有效预防迟发性耳聋发生。

表1 6个通过SFDA批准的耳聋基因筛查试剂盒检测特点
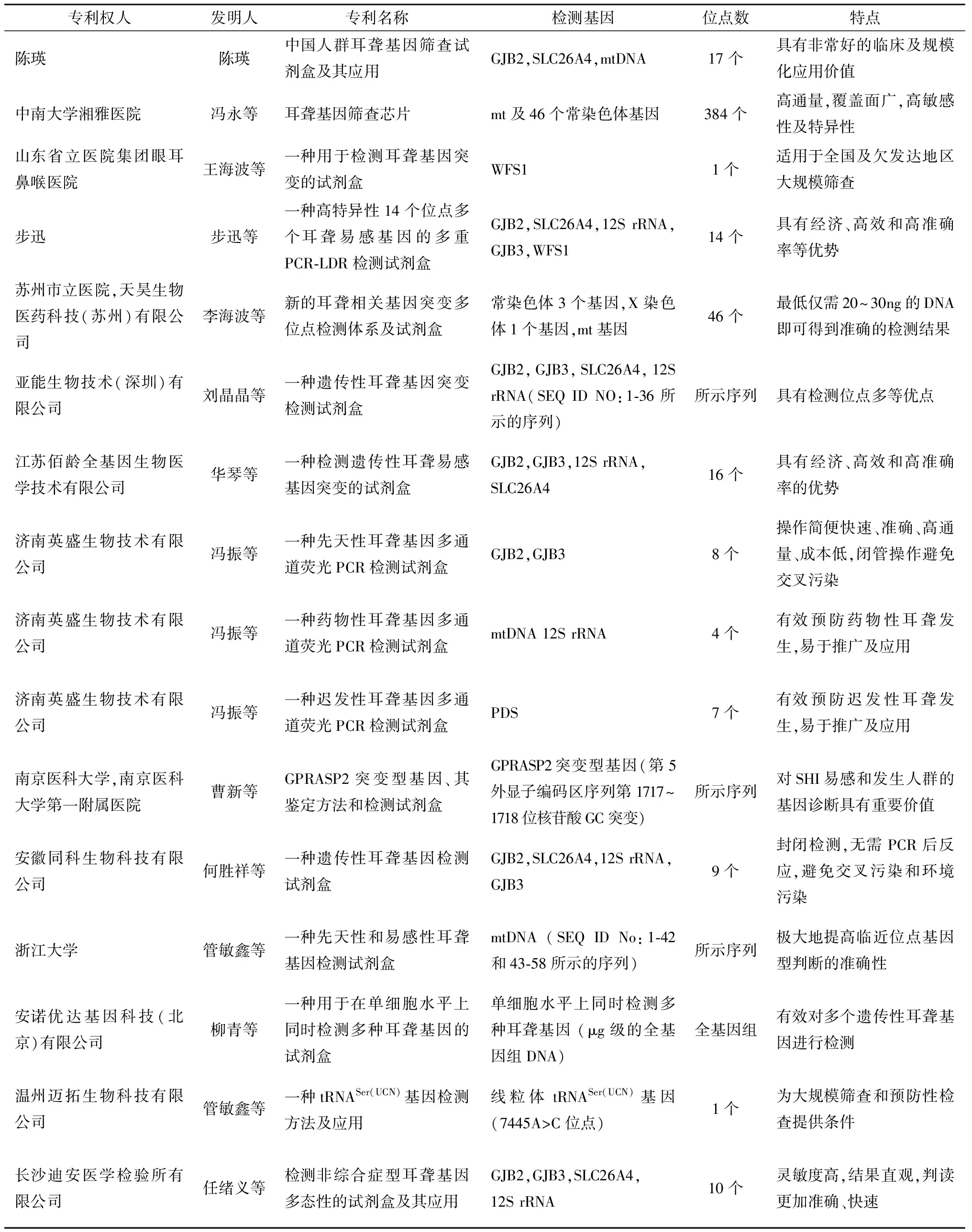
表2 2014年—2016年申请获得的耳聋基因筛查(检测)专利
三、基质辅助激光解吸电离飞行时间质谱(MALDI-TOF-MS)技术:
MALDI-TOF-MS技术是在化学、生物、医疗卫生等众多领域得到广泛研究应用的一种新型“软电离”质谱技术,它可以对样品进行定性或定量分析,从质谱图上可获得样品中每一分子的分子量,以及通过谱峰强度计算样品的平均分子量与分子量分布宽度[3]。用针对耳聋基因的20个位点,较9项试剂盒增加11个位点,即GJB2:176-191del16,GJB3:547G>A,SLC26A4:281C>T、589G>A、1174A>T、1226G>A、1229C>T、IVS15+5G>A、1975G>C、2027T>A、2162C>T。因此具有检测位点多、覆盖率高、准确、高通量、低成本和周期短等特点,能够满足临床对耳聋基因检测的要求,适合大范围、大规模的检测项目[4],并已在许多新生儿耳聋基因筛查大规模检测项目中应用和研究。
四、测序技术发展与耳聋基因诊断研究
DNA测序技术是现代分子生物学和生物医学研究中常用的一种技术手段。1954年Whitfeld等报道用化学降解进行多聚核糖核苷酸测序方法,1977年第一代测序技术(Sanger测序法)出现,由于高昂的测序成本限制了第一代测序技术更常规的使用,2005年以后出现的新(下)一代测序技术(nextgeneration sequencing technology,NGS),又称第二代测序技术,或高通量测序(high-throughput sequencing),或深度测序(deep sequencing)技术,由于测序成本的大幅度降低,使之成为解决一般性的基因分子生物学问题的有效工具。高通量测序技术能一次并行对几十万到几百万条DNA分子进行序列测定,就耳聋基因研究和基因诊断而言,基于靶向捕获和高通量测序,可针对绝大多数已知耳聋基因进行一次性全序列突变检测。第三代测序技术是指单分子测序技术,DNA测序时,不需要经过PCR扩增,实现了对每一条DNA分子的单独测序。虽然测序技术越来越成熟,成本也越来越低,但在遗传性耳聋方面一个很大的挑战是如何认识基因变异对耳聋发生和发展的影响。随着测序技术日趋成熟,测序研究提示更多可能导致遗传性耳聋发生的基因,同时通过相应的功能研究来证实,遗传性耳聋的本质会逐渐被揭示出来[5]。
五、全外显子组测序(whole exome sequencing,WES)技术
WES是一种只针对外显子区域DNA的新型基因组分析技术。该技术首先将全基因组外显子区域DNA捕捉并富集,再进行高通量测序,然后结合生物信息学分析鉴定罕见及常见疾病相关的致病基因。有专家学者分别阐述了目标序列捕获及平行测序在临床耳聋基因诊断中的应用,全外显子组测序技术及其在遗传性耳聋研究中的应用,运用全外显子组测序技术诊断非综合征性耳聋基因变异等[6-8],表明这一技术具有耳聋基因诊断的应用前景。
六、多重置换扩增(multiple displacement amplification,MDA)技术
MDA技术是全基因组扩增(whole genome amplification,WGA)技术的一种,能够提供高度均一完整的全基因组序列,使产物与模板的遗传序列信息保持一致,是一种真正意义的全基因组扩增方法[9]。MDA技术是1998年Lizardi等[10]基于环状滚动扩增方法创建的一种链置换扩增技术,利用phi29 DNA聚合酶和六聚体随机引物对基因组进行扩增,不需要经过聚合酶链反应(PCR)热循环过程,只需在30℃条件下恒温即可反应。扩增时,phi29 DNA聚合酶在多个位点同时起始复制,合成新的DNA链取代模板互补链,被置换出新的DNA链又成为模板来进行扩增,形成级联分支的放大系统。最终生成大量的高保真基因组DNA,为单细胞多次分析提供了可能[11]。“一种用于在单细胞水平上同时检测多种耳聋基因的试剂盒”专利就是通过MDA技术,从单细胞样本等DNA含量极其微少的样本中有效扩增得到总量在μg级的全基因组DNA。
七、单核苷酸多态分型(single nucleotide polymorphisms scan,SNPscan)技术
SNPscan技术是一个快速多基因检测系统,能在一个检测流程中同时实现对48/96/144/192个位点进行分型。SNPscan技术基本原理是利用连接酶连接反应的高特异性实现对SNP位点等位基因的识别,然后通过在连接探针末段引入不同长度的非特异序列以及通过连接酶加接反应获得位点对应的不同长度连接产物,利用荧光标记的通用引物对连接产物进行PCR扩增,通过荧光毛细管电泳对扩增产物进行电泳分离,最后通过对电泳图谱的分析获取各个SNP位点的基因型。SNPscan是一项高通量和低成本的检测技术,在对225例非综合征型耳聋患者(即225个样本3个基因115个位点)的SNPscan基因检测中,初筛中找到有明确致聋基因的患者111例(49.3%),接近60%的理想目标,另外耳聋基因携带者30例(13.3%),筛查结果均为阴性者84例(37.3%)。SNPscan检测结果经抽样Sanger测序验证,符合率达100%。应该指出,到目前为止直接测序法仍然是基因突变检测的金标准,其在结果验证和新基因突变位点的检测等领域还是不可替代的,但直接测序对于大规模基因筛查来说太过昂贵和耗时。因此有必要研究和探索适宜于耳聋基因筛查和检测的新技术和好方法[12]。
八、胚胎植入前遗传学诊断技术(PGD)
Handyside等[13]于1990年报道运用胚胎移植前遗传学诊断技术(PGD)成功妊娠,使得PGD开始应用于临床。PGD是采用辅助生殖方法,通过遗传诊断技术,挑选健康的胚胎进行子宫移植,获得表型正常的后代。PGD已广泛应用于单基因遗传性疾病和染色体异常性疾病以及线粒体疾病,遗传性耳聋绝大部分为单基因变异,是PGD适应症[14]。Altarescu等[15]于2009年报道利用极体和卵裂球活检对非综合征性遗传性耳聋患者采用PGD获得成功妊娠。2010年国立台湾大学医学院多专科联合对已生育一名因SLC26A4耳聋基因突变导致大前庭水管综合征(EVAS)患儿的夫妇进行了PGD辅助生殖干预,成功妊娠并生育基因为野生型的单胎婴儿,出生后听力筛查正常[16]。2015年中国大陆首例先天性耳聋胚胎植入前遗传学诊断健康试管婴儿诞生[17]。
另外,还有学者对Wolfram综合征的致病基因wolframin基因(WFS1)[18-20],与X连锁隐性遗传综合征型耳聋相关的G蛋白偶联受体相关分选蛋白2(GPRASP2)基因[21],线粒体导致非综合征型耳聋两个突变热点区域之一的线粒体tRNASer(UCN)基因[22,23]进行了检测技术研究。

