绵羊Y染色体特异性引物及SNPs的筛选
曹学涛,裴生伟,张晋,李发弟,2,李刚,李万宏,乐祥鹏
绵羊Y染色体特异性引物及SNPs的筛选
曹学涛1,裴生伟1,张晋3,李发弟1,2,李刚3,李万宏1,乐祥鹏1
(1兰州大学草地农业生态系统国家重点实验室/兰州大学农业农村部草牧业创新重点实验室/兰州大学草地农业科技学院,兰州 730020;2甘肃省肉羊繁育生物技术工程实验室,甘肃民勤 733300;3甘肃省家畜繁殖中心,甘肃武威 733000)
【目的】哺乳动物Y染色体雄性特异区在减数分裂过程中不与X染色体发生重组,遵循严格的父系遗传,是研究父系遗传多样性的重要遗传资源;此外绝大多数Y染色体基因主要或者特异性的在睾丸组织中表达,并在精子发生和雄性繁殖力方面扮演着至关重要的作用。由于Y染色体测序极其困难,造成很多物种Y染色体序列很少。因此本文基于目前现有牛科动物Y染色体引物信息,旨在鉴定绵羊Y染色体特异性引物,比较绵羊Y染色体基因片段与岩羊、牛、山羊和牦牛Y染色体的差异,同时筛选不同绵羊品种在Y染色体基因片段内的SNPs,将找到的Y-SNPs和绵羊的睾丸大小进行相关性分析,为鉴定绵羊Y染色体基因片段奠定基础,并为今后绵羊Y染色体单倍型的构建、绵羊胚胎性别早期鉴定和公绵羊繁殖力相关分子标记的筛选提供科学依据。【方法】根据目前文献公布的29对牛科动物Y染色体特异性引物序列,以母绵羊和水分别作阴性和空白对照,以公绵羊DNA为模板验证引物的雄性特异性;确定绵羊Y染色体特异引物后,利用DNA混合池测序结合限制性长度片段多态性等技术在萨福克羊(n=146)、白萨福克羊(n=91)、东弗里升羊(n=6)、特克塞尔羊(n=72)、南非肉用美利奴羊(n=17)、杜泊羊(n=32)、湖羊(n=55)、藏羊(n=34)、滩羊(n=43)和岩羊(n=14)公羊群体中进行Y-SNPs扫描。用Chromas和DNASTAR等软件分析混合池测序的结果,利用DNAman软件将绵羊Y染色体基因片段与牦牛、山羊、黄牛和岩羊进行同源性分析。同时,测量萨福克羊、白萨福克羊、东弗里升羊、特克塞尔羊、南非肉用美利奴羊、杜泊羊周岁的阴囊围,利用SPSS 19.0软件分析SNPs位点与公羊阴囊围的相关性。【结果】在分析的29对引物中,6对引物为绵羊Y染色体特异性引物,分别能扩增出3、6、、、11和6片段。17对引物未能出现扩增条带,6对引物在母羊DNA中出现扩增条带。通过比对发现绵羊6个基因片段与岩羊、牛、山羊、牦牛的同源性在81.51%—98.84%。此外,首次在萨福克和白萨福克羊群体的11片段中鉴定得到一个G>A的突变,通过I酶切分析发现在萨福克羊、白萨福克羊中有2种基因型(AA、GG),在其他7个绵羊品种中只有GG基因型。在白萨福克羊群体中,GG基因型的频率为0.747,AA基因型的频率为0.253;在萨福克羊群体中GG基因型的频率为0.986,AA基因型的频率为0.014;说明在萨福克羊和白萨福克羊群体中,GG基因型为优势基因型。关联分析显示在白萨福克羊群体中,GG基因型个体周岁的阴囊围显著的高于AA基因型(=0.029)。【结论】鉴定得到6个绵羊Y染色体基因片段,它们与牛、山羊、牦牛和岩羊具有较高的同源性,表明Y染色体基因在进化过程中具有一定的保守性。首次在萨福克和白萨福克羊群体11片段中找到一个Y-SNP(G>A),其与白萨福克羊周岁的睾丸大小紧密相关。
Y染色体;绵羊;单核苷酸多态性
0 引言
【研究意义】Y染色体基因在研究父系遗传多样性、维持雄性特性,精子发生和雄性繁殖力起着关键作用,Y染色体分子标记已经运用于绵羊父系起源进化研究及父系单倍型构建[1-3]。然而Y染色体测序困难,造成绵羊Y染色体基因序列极度贫乏。因此本研究基于前期牛科动物Y染色体研究成果,进行绵羊Y染色体基因片段鉴定和Y-SNPs的筛选,以期为优秀种公羊的早期选择、胚胎性别的早期鉴定和绵羊品种的父系遗传多样性的保护提供理论依据。【前人研究进展】哺乳动物的性染色体,X和Y染色体,是由一对原始的常染色体进化而来[4]。Y染色体是雄性动物特有的性染色体,由两部分组成:拟常染色体区域和Y染色体雄性特异区(male specific region of Y chromosome, MSY)组成。MSY区在减数分裂过程中不与X染色体发生重组交换,遵循严格的父系遗传。Y染色体结构非常复杂,包含有大量的重复序列和回文结构,使得其序列的组装极为困难[5]。到目前为止,绵羊Y染色体只有、等少数基因的部分序列已公布[6-8]。MEADOWS等利用23对人、牛、绵羊Y染色体特异性引物扩得到绵羊、、、、等Y染色体基因的部分片段[9]。FENG等扩增得到岩羊ZFY基因639 bp的片段[7]。刘帅兵克隆得到ZFY基因整个mRNA全长[10]。熊勇等克隆得到藏系绵羊ZFY基因447 bp的片段[11]。蒋利等利用山羊SRY基因序列设计引物,扩增得到阿勒泰羊、藏绵羊SRY基因整个编码区的雄性特异性片段,研究表明SRY基因在不同绵羊品种间具有高度的同源性[12]。【本研究切入点】目前只有人类、猩猩、猕猴、小鼠和牛等少数物种的Y染色体完成了测序[3,5,13-15]。关于绵羊Y染色体,少数研究者只扩增出基因的部分片段,未见克隆得到完整的Y染色体基因序列。【拟解决的关键问题】本研究基于牛科动物现有Y染色体特异引物鉴定绵羊Y染色体单拷贝基因,扫描Y染色体基因在不同绵羊品种中的SNPs,以期找到Y染色体分子标记,以期为筛选不同羊品种Y染色体SNPs提供基础,也为绵羊早期胚胎性别的鉴定提供了分子标记,为进一步研究绵羊Y染色体基因序列奠定基础。
1 材料与方法
试验于2016年10月至2017年5月在兰州大学草地农业生态系统国家重点实验室完成。
1.1 试验材料
496份公绵羊和14份公岩羊的血液样本,包括采自北京澳鑫牧业有限公司的萨福克羊(n=146)、白萨福克羊(n=91)、东弗里升羊(n=6)、特克塞尔羊(n=72)、南非肉用美利奴羊(n=17)、杜泊羊(n=32),甘肃金昌中天羊业有限公司的湖羊(n=55),甘肃甘南藏族自治州的藏羊(n=34),宁夏盐池县的滩羊(n=43),岩羊采自新疆动物园野生动物保护中心。采集3只湖羊母羊血液(甘肃金昌中天羊业有限公司)及3只岩羊母羊血液,作为阴性对照。同时在收集血样时测定萨福克羊、白萨福克羊、东弗里升羊、特克塞尔羊、南非肉用美利奴羊、杜泊羊周岁(周岁±15—20 d)的阴囊围直径。
1.2 DNA的提取和DNA池的构建
采用常规的酚-氯仿抽提法提取绵羊血液基因组DNA[16]。用Nano drop 2000(Thermo,美国)检测提取的DNA浓度和纯度,并用1%的琼脂糖凝胶电泳检测DNA的质量。将质量检测合格绵羊和岩羊DNA统一稀释为50 ng·µL-1,每个个体DNA取10 µL,以同一个品种尽量在一个DNA池的原则,每10个个体混成一个DNA池,共计51个DNA池,混合均匀后-20℃保存备用。
1.3 绵羊Y染色体特异性引物的筛选
查阅相关文献可知,目前在牛、牦牛、山羊等牛科动物Y染色体研究有29对引物(表1),分别能扩增出MSY区的、、、、、等基因。本研究的29对引物均由北京六合华大基因科技有限公司合成。实验以公羊DNA混合池为模板,以母羊DNA为阴性对照,灭菌水为空白对照,通过PCR扩增确定绵羊Y染色体特异性引物。PCR扩增体系为50 µL:模板2 µL,上、下游引物各1 µL,2×EasyTaq酶25 µL和ddH2O 21µL。扩增反应条件:94℃预变性30 s;94℃变性30 s,退火30 s,72℃延伸1 min/1kb,34个循环;72℃延伸5 min;4℃保存。PCR产物用1%琼脂糖凝胶电泳检测。
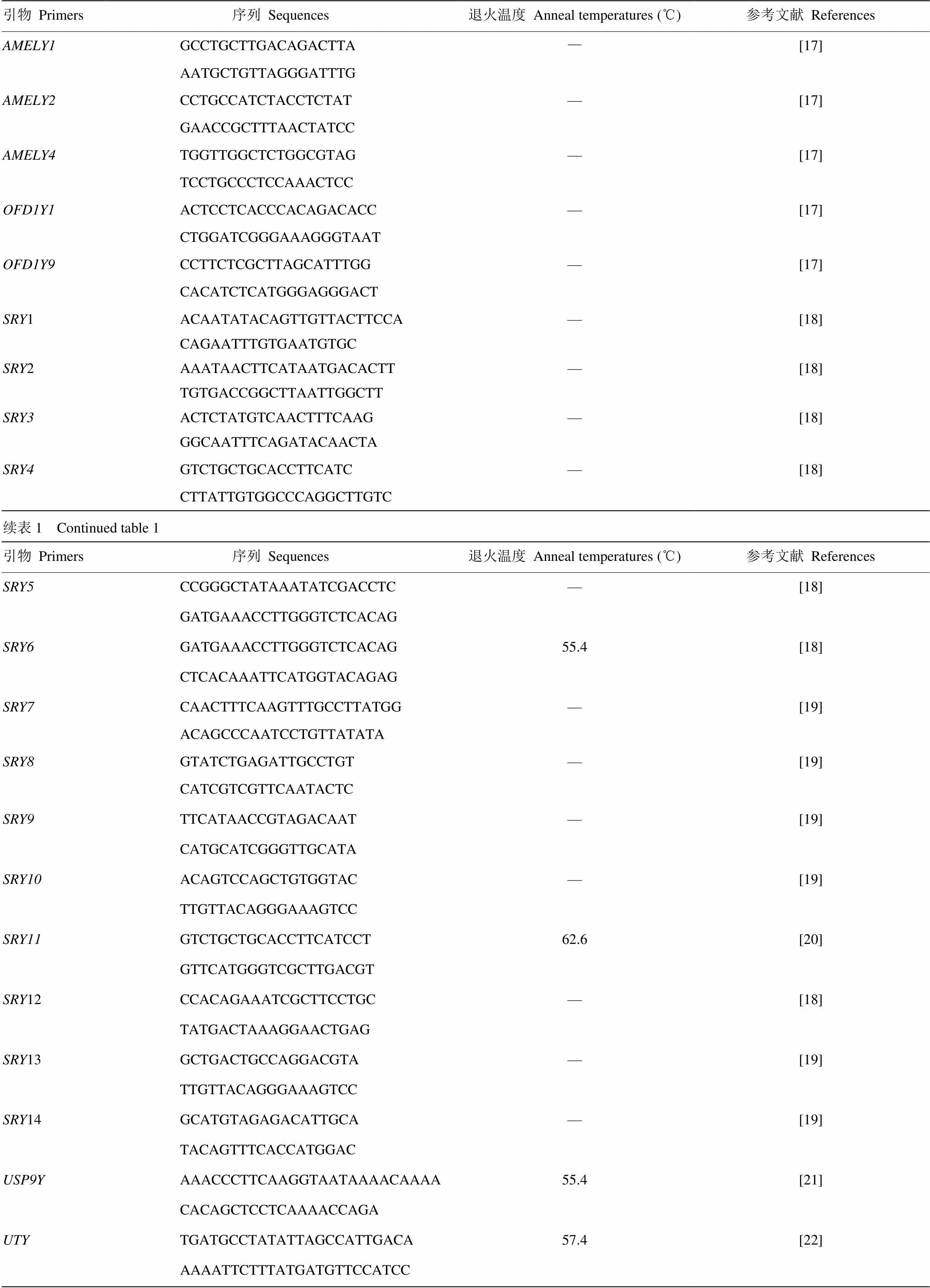
表1 引物序信息
表中仅列出绵羊Y染色体特异性引物的退火温度The annealing temperatures were listed for those ovine Y-specific primers
1.4 绵羊Y染色体特异性基因片段SNPs筛选
将上述鉴定为绵羊Y染色体特异性引物,以DNA混合池为模板的PCR产物送北京六合华大基因科技有限公司进行测序。DNAstar和Chromas软件用于测序结果分析,DNAMAN用于多序列比对及同源性分析。经过混合池测序,在11片段内鉴定得到一个G>A突变,利用Watcut软件分析(http://watcut. uwaterloo.ca/template.php?act=snp_new)发现该突变位点可被限制性内切酶188I识别。酶切反应体系:10×NEB Buffer 3 µL,PCR产物4 µL和5U188I内切酶,加灭菌水至25 µL。酶切反应条件为37℃温育3 h。取酶切消化液10 µL加样于2.5%琼脂糖凝胶上进行电泳。
1.5 数据处理与统计分析
利用SPSS 19.0软件分析基因位点与绵羊睾丸大小的相关性。先对数据进行描述性统计分析,确定是否存在离群值,根据数据特征,利用单因素方差分析(ANOVA)探究基因型效应。
2 结果
2.1 绵羊Y染色体特异性引物
通过PCR扩增,本研究发现29对引物中6对引物,包括3、6、、、11、6确定为Y染色体特异性引物(图1),只在公羊DNA上出现扩增条带,在母羊DNA及水中没有扩增产物。此外17对引物未能出现扩增条带或非特异性扩增,6对引物母羊DNA能扩增出条带(电子版附图S1)。
2.2 绵羊Y染色体特异性DNA片段与其他物种的同源性分析
通过对上述6对特异性引物的PCR产物进行测序,分析发现3片段大小为614 bp,该片段与山羊第11外显子和部分CDS区相似度较高,相似度为98.84%。6片段大小为461 bp,该片段与山羊部分CDS区相似度较高,相似度为98.84%。片段大小为444 bp,该片段与牛第26外显子相似度较高,相似度为84.96%。片段大小为256 bp,该片段与牛第19外显子相似度较高,相似度为81.51%。片段大小为440 bp,该片段与山羊SRY基因序列相似度较高,相似度为90.41%,该基因片段的部分片段(237 bp)与NCBI公布的绵羊的SRY基因CDS区(AY604735.1)相似度达100%。3片段大小为621 bp,该片段与牛ZFY基因第11外显子和部分CDS区相似度较高,相似度为97.67%。
用DNAMAN生物软件,将绵羊3、6、、、11、6基因序列与山羊、牛、牦牛和岩羊相应基因序列进行序列比对(电子版附图S2—S7),计算不同物种基因序列间的同源性大小(表2)。结果表明,绵羊3基因片段与岩羊、山羊、牛、牦牛各物种间同源性大小分别为98.84%、97.89%、97.08%和95.79%;绵羊SRY6基因片段与岩羊、山羊、牛、牦牛各物种间同源性大小分别为95.25%、93.93%、90.54%和90.32%;绵羊USP9Y基因片段与牛、牦牛各物种间同源性大小分别为84.96%、84.67%;绵羊UTY基因片段与岩羊、黄牛、牦牛各物种间同源性同为96.77%、81.51%和81.51%;绵羊SRY11基因片段与岩羊、山羊、牛、牦牛各物种间同源性大小分别为92.63%、90.41%、84.97%和84.75%;绵羊ZFY6基因片段与岩羊、山羊、牛、牦牛各物种间同源性大小分别为99.06%、89.11%、97.67%和86.76%。由于USP9Y基因是从岩羊母羊DNA中扩增出,表明该片段并不是岩羊Y染色体片段。
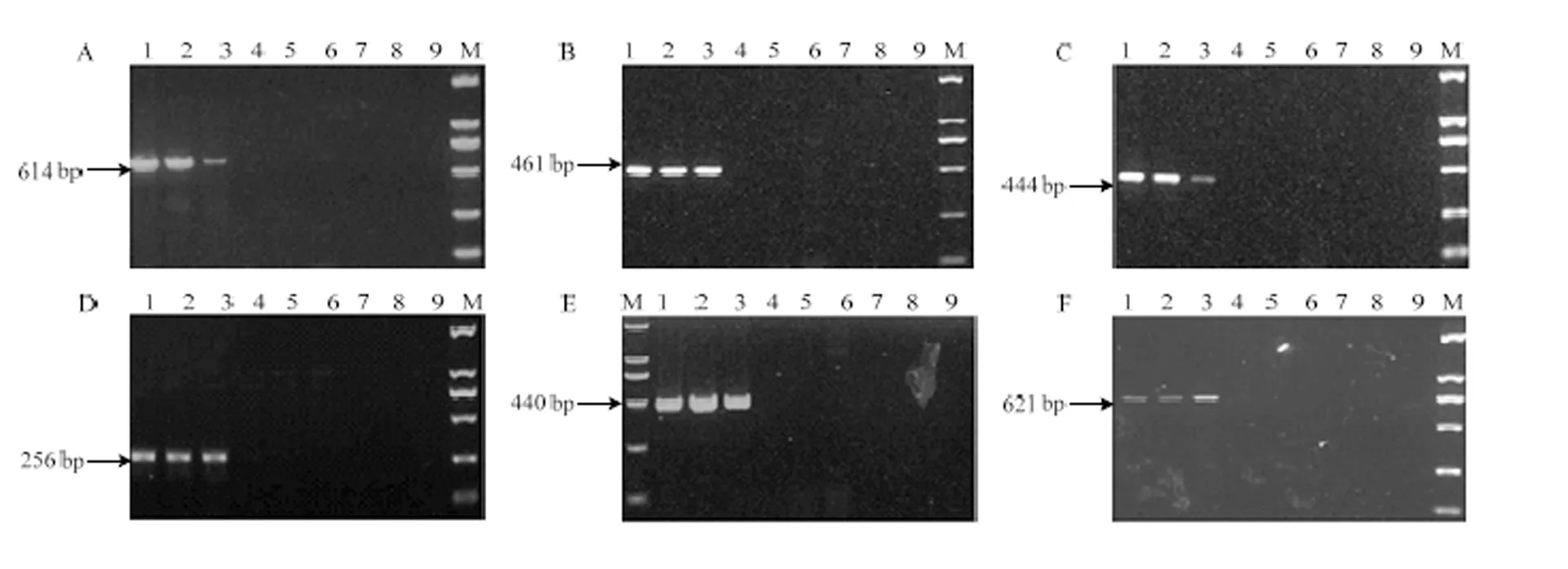
A:ZFY3基因PCR扩增结果;B:SRY6基因PCR扩增结果;C:USP9Y基因PCR扩增结果;D:UTY基因PCR扩增结果;E:SRY11基因PCR扩增结果;F:ZFY6基因PCR扩增结果;1-3公羊DNA,4-6母羊DNA,7-9 ddH2O,M.DNA相对分子质量标准DL2000
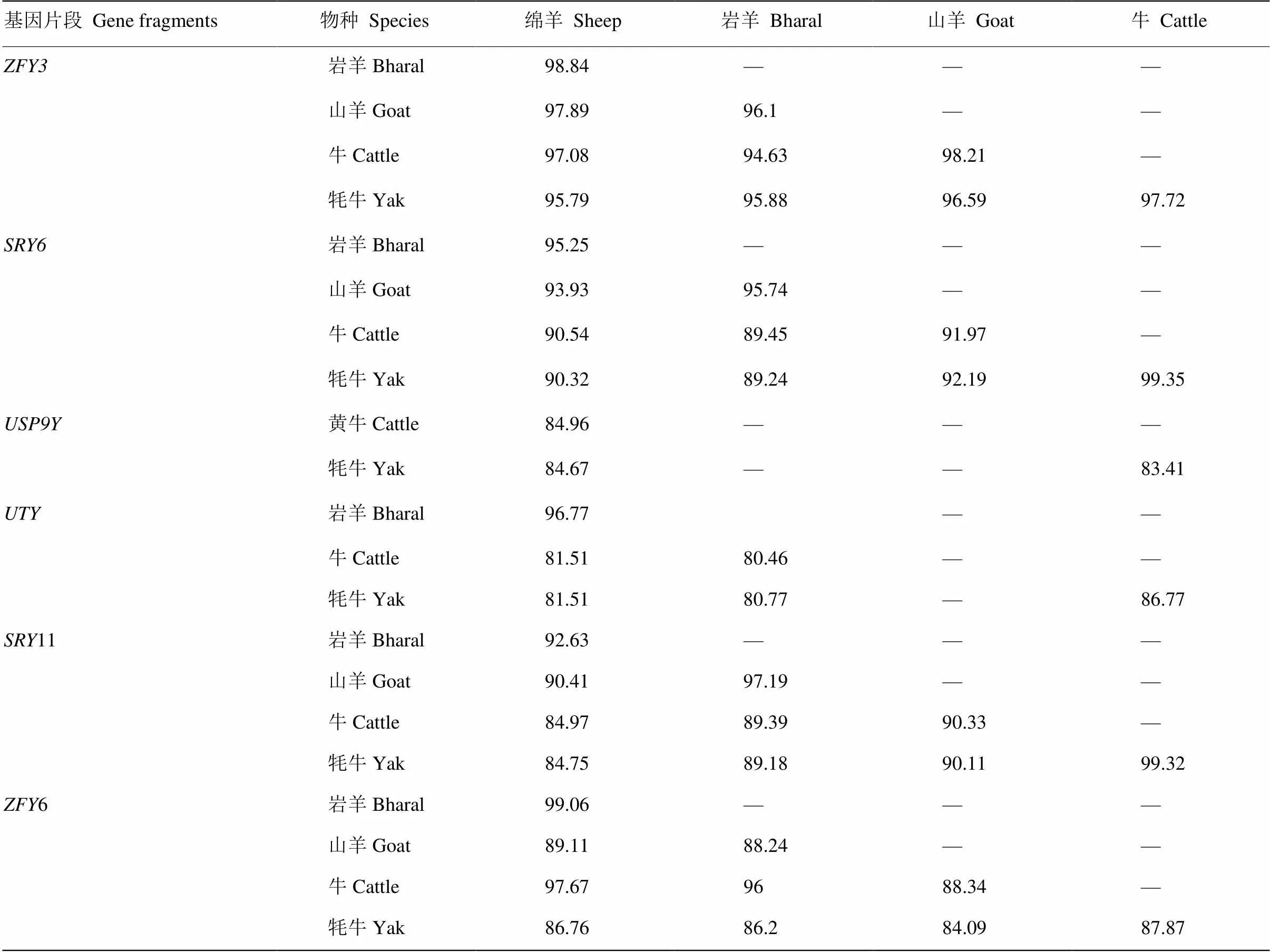
表2 绵羊Y染色体基因与不同物种同源基因的比较
2.3 绵羊Y染色体SNPs的筛选及基因效应分析
通过测序,3、6、、、6基因序列均未发现突变位点。在萨福克和白萨福克羊群体中,11片段混合池测序出现双峰,进一步对其中一个混池的每个个体分别进行测序,验证为G>A的突变(图2)。对萨福克及白萨福克的个体进行PCR-RFLP检测,PCR产物经188I酶切后产生AA和GG 2种基因型(图3)。AA基因型个体的扩增产物能被188I酶切后将产生4个片段,分别为54、63、151和172 bp;GG基因型个体的扩增产物能被188I酶切后将产生3个片段,分别为63、172和205bp。11片段G>A突变位点在萨福克羊、白萨福克羊、东弗里升羊、特克塞尔羊、南非肉用美利奴羊、杜泊羊、湖羊、藏羊、滩羊公羊群体中的基因型频率、等位基因频率见表3。结合混合池测序及酶切结果发现,在萨福克羊、白萨福克羊中存在AA和GG基因型,其中在白萨福克羊群体中AA基因型频率为0.253,GG基因型频率为0.747;在萨福克羊群体中AA基因型频率为0.014,GG基因型频率为0.986。结果显示GG基因型在萨福克羊和白萨福克羊群体中为优势基因型;而东弗里升羊、特克塞尔羊、南非肉用美利奴羊、杜泊羊、湖羊、藏羊、滩羊公羊群体中只有GG基因型。通过对岩羊个体的混合池测序,在11片段也未发现突变位点。性状关联分析表明,在白萨福克羊群体中,GG基因型个体的睾丸大小显著的高于AA基因型(表4)。
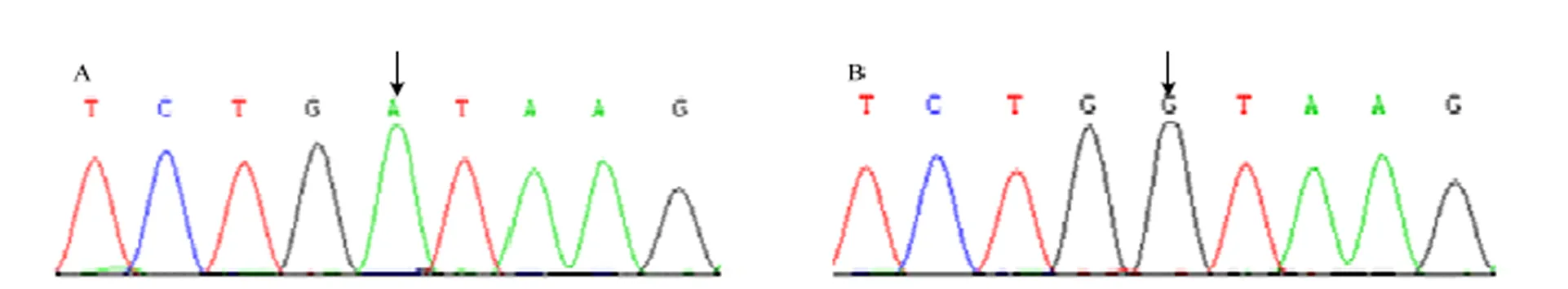
A. AA基因型; B. GG基因型
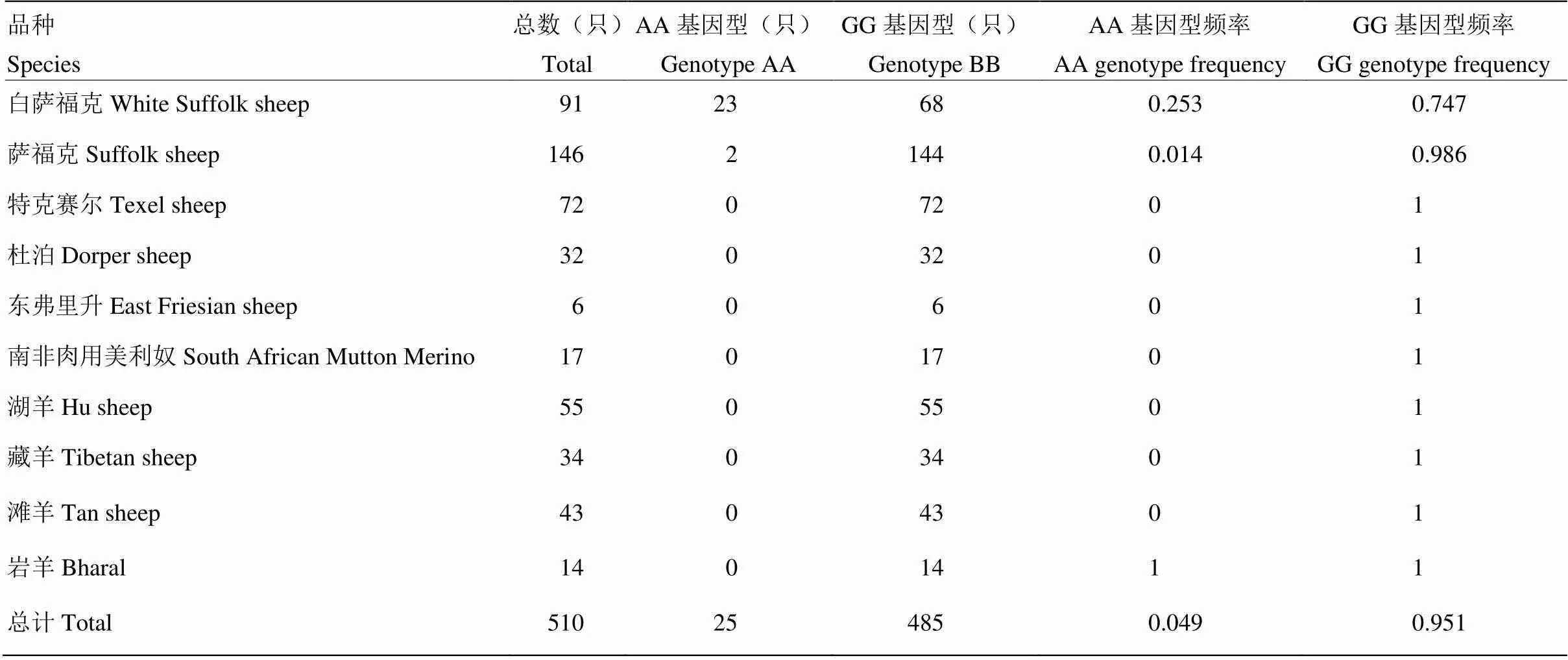
表3 SRY11片段G>A 位点在不同绵羊品种中的基因型和等位基因频率
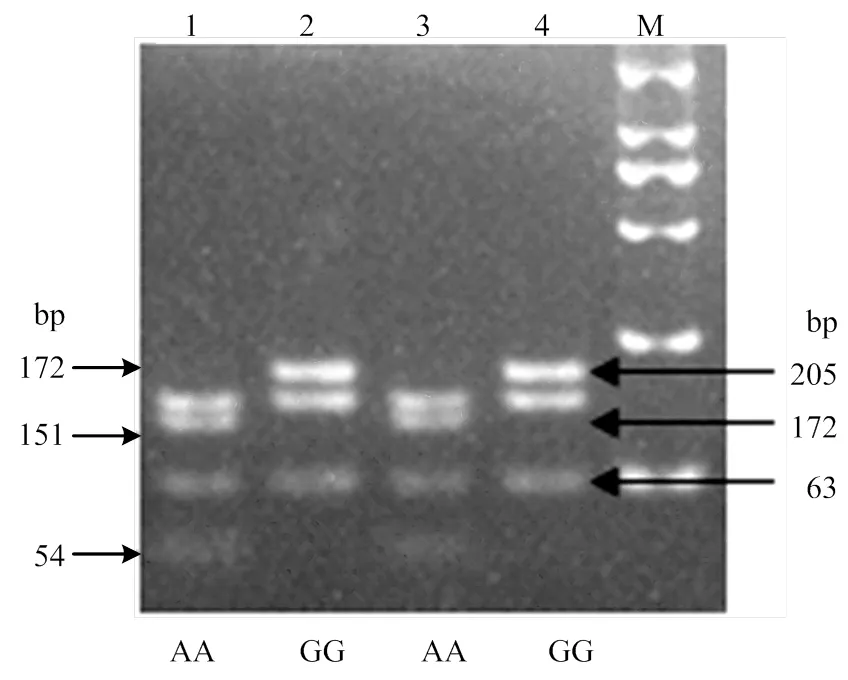
1,3:AA基因型;2,4:GG基因型;M为DNA相对分子质量标准DL2000
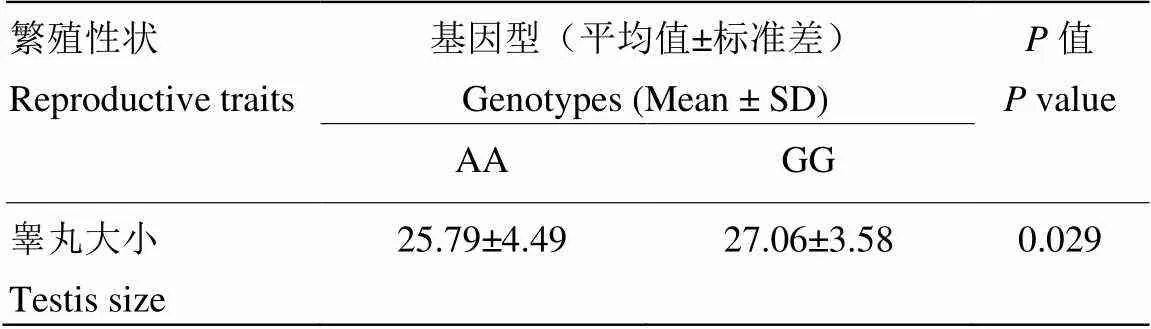
表4 SRY11片段G>A位点与白萨福克羊睾丸大小之间的关联分析
3 讨论
在目前研究绵羊起源进化及单倍型分析中,更多的是以线粒体基因遗传多样性为基础的母系遗传研究,而利用以Y-SNPs为基础的父系遗传的研究较少。绵羊的父系及母系遗传相结合才能更加全面、系统的分析绵羊起源进化、单倍型及分类。本研究筛选得到了6对绵羊Y染色体特异性引物,均为首次扩增得到,与MEADOWS等筛选得到9对绵羊Y染色体特异性引物无重复[9]。(sex-determining region on Y chromosome,)位于哺乳动物Y染色体,是性别决定直接相关的基因。该基因从人的Y染色体上首次分离得到,并被确定为睾丸决定因子[24]。MEADOWS等通过测序在SRY基因5′启动区发现8个SNPs,结合Y染色微卫星鉴定绵羊及野生绵羊单倍型[25]。MEADOWS等发现得到绵羊第一外显子与牛的同源性为94.7%,并在5′启动区鉴定得到了一个A>G的突变[9]。在本次研究扩增得到了两个SRY基因片段,其与牛的同源性分别为90.54%和84.97%,这与前人的研究相似,说明绵羊和牛SRY基因具有较高的同源性。本研究在11片段鉴定到1个G>A的同义突变,通过188I酶切分析,发现只有在萨福克羊、白萨福克羊中有AA和GG 2种基因型,而在其他7个绵羊品种和岩羊中只有GG基因型,暗示该位点可能只存在于萨福克羊和白萨福克羊的血统中,但还需要在更多的绵羊品种群体中进行验证。此外该突变位点与白萨福克羊周岁的睾丸大小紧密相关,即GG基因型个体的睾丸大小显著大于AA型个体。本研究为首次发现Y染色体突变与绵羊繁殖性状相关。该位点在更大的白萨福克羊群体进行验证后可用于白萨福克种公羊的选育。
ZFY基因即Y连锁的锌指蛋白因子(zinc finger protein, Y-linked),位于Y染色体短臂(Yp11.3)上,由11个外显子和1个随机重复区域的13个“锌-指”结构组成,在雄性动物睾丸的生长发育过程中发挥作用[26-27]。毛德才研究发现牦牛、猩猩、人、牛等9个物种间的核苷酸序列有较高同源性[28]。熊勇等研究表明藏系绵羊ZFY基因与猪、摩弗伦羊、狼、人类等其他物种之间具有很高的同源性[11]。LAWSON等通过进化树分析发现:绵羊与岩羊、山羊、牛的亲缘关系较近[8]。ARKADI克隆得到了整个牛ZFY基因整个编码区及马和猪ZFY基因编码区部分序列,通过比对发现ZFY基因编码区在3个物种的同源性较高[29]。本研究发现绵羊3和6片段与岩羊、山羊、牛、牦牛等物种间同源性大小在89.11%—99.6%,表明该基因在不同物种间也具有较高的同源性,与前人的研究基本一致。AASEN等[30]利用片段的扩增及酶切鉴别人、牛、绵羊和山羊的胚胎性别,该研究的结果也可用于绵羊胚胎性别的早期鉴定。
SATOU等[31]研究发现USP9Y基因的突变影响小鼠睾丸重量。有学者发现在牛USP9Y基因第26内含子发现了一个81 bp的插入缺失,并用于构建牛Y染色体单倍型及区分瘤牛和普通牛[21,32-33]。本研究在USP9Y基因片段中没有检测到遗传变异,今后需要在更多的绵羊品种进行Y-SNPs扫描。先前的研究利用BAC的方法在绒山羊上鉴定到USP9Y基因348 bp的片段,其与家猫、人、恒河猴、黑猩猩等的同源性在89%以上[34]。本研究扩增得到的绵羊USP9Y基因与牛、牦牛各物种间同源性大小分别为84.96%、84.67%,表明该基因在进化过程中具有一定的保守性。USP9Y基因在岩羊母羊DNA中扩增出,表明该片段并不是岩羊Y染色体片段,因此本研究的结果可作为区分岩羊和绵羊的分子标记。
UTY基因即Y连锁的泛转录三角形四肽重复因子,在人上的研究发现UTY基因有6个不同的mRNA转录本,UTY蛋白被认为是未成熟的组织适合性抗原,可启动男性特异性细胞毒性T淋巴细胞反应[35-36]。MEADOWS等扩增得到了UTY基因外显子1,通过比对发现与牛的同源性为98.1%[9]。本研究扩增得到的绵羊UTY基因与岩羊、牛、牦牛各物种间同源性达81.51%—96.77%,表明该基因在进化过程中相对保守。GINJA等扩增牛UTY基因19外显子,发现有一个C>A的突变[23]。然而在本研究中在UTY基因上并未检测到突变位点。
本研究首次在发现一个G>A的突变,在3、6、、、6片段均未发现突变,其原因可能与样本量大小及品种差异有关。但是本研究的结果为今后我国绵羊品种父系遗传多样性的研究奠定了基础。当然,为了系统和全面研究绵羊Y染色体,必须依赖绵羊Y染色体完整的序列,完成绵羊Y染色体测序是研究绵羊父系遗传多样性和绵羊雄性繁殖力育种亟待解决的难题。
4 结论
本研究首次扩增得到绵羊、、和4个基因的6个片段。通过比对发现绵羊3、6、、、6片段与岩羊、牛、山羊、牦牛的具有较高的同源性。首次在11片段发现G>A的突变,该突变位点只存在于萨福克和白萨福克公羊群体中,并与白萨福克羊睾丸大小紧密相关。

A:2 PCR扩增结果;B:4 PCR扩增结果;C:5 PCR扩增结果;D:9 PCR扩增结果;E:1 PCR扩增结果;F:9 PCR扩增结果;1-3公羊DNA,4-6母羊DNA,7-9ddH2O,M.DNA相对分子质量标准DL2000
A.PCR products of3 fragment; B.PCR products of6 fragment; C.PCR products offragment; D.PCR products offragment; E.PCR products of11 fragment; F.PCR products of3 fragment;1-3 rams DNA;4-6 ewes DNA;7-9 ddH2O;M.DL2000 marker
图S1 绵羊Y染色体非特异性PCR扩增结果
Fig. S1 PCR products of Y nonspecific fragment in sheep


图S2 不同物种3基因序列比对结果
Fig. S2 Comparison of ovine3 gene sequence with other species

图S3 不同物种6基因序列比对结果
Fig. S3 Comparison of ovine6 gene sequence with other species
图S4 不同物种基因序列比对结果
Fig. S4 Comparison of ovinegene sequence with other specie

图S5 不同个物种基因序列比对结果
Fig. S5 Comparison of ovinegene sequence with other specie

图S6 不同物种11基因序列比对结果
Fig. S6 Comparison of ovine11 gene sequence with other specie
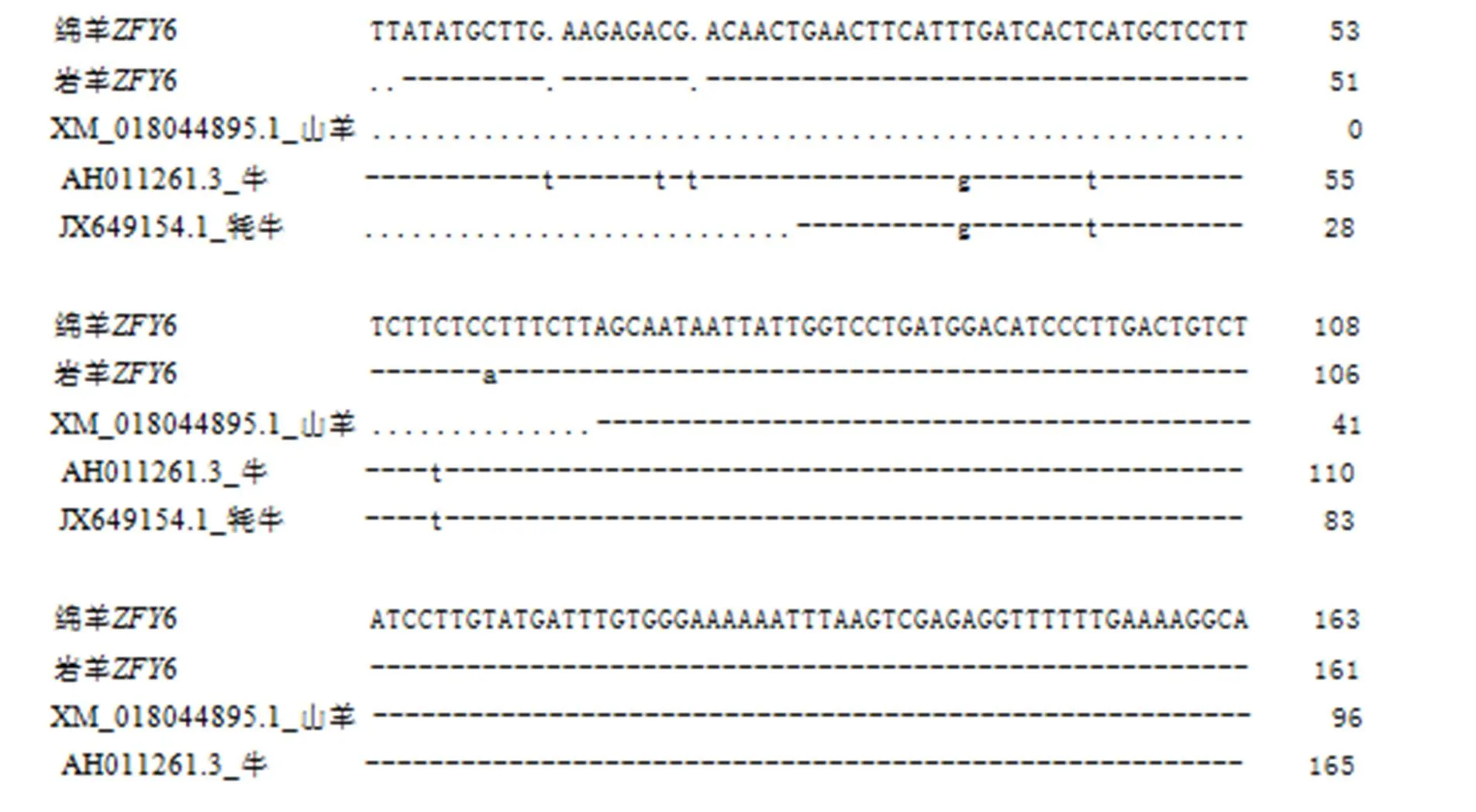
图S7 不同物种6基因序列比对结果
Fig. S7 Comparison of ovine6 gene sequence with other specie
[1] MIN Z, PENG W F, YANG G L, LV F H, LIU M J, LI W R, LIU Y G, LI J Q, FENG W, SHEN Z Q. Y chromosome haplotype diversity of domestic sheep () in northern Eurasia.2014, 45(6): 903-907.
[2] FERENCAKOVIC M, CURIK I, PÉREZ-PARDAL L, ROYO L J, CUBRIC-CURIK V, FERNÁNDEZ I, ÁLVAREZ I, KOSTELIC A, SPREM N, KRAPINEC K. Mitochondrial DNA and Y-chromosome diversity in East Adriatic sheep.2013, 44(2): 184-192.
[3] CHANG T C, YANG Y, RETZEL E F, LIU W S. Male-specific region of the bovine Y chromosome is gene rich with a high transcriptomic activity in testis development.2013, 110(30): 12373-12378.
[4] GRAVES J A. Sex chromosome specialization and degeneration in mammals., 2006, 124(5):901-914.
[5] KIM T Y, SHIN K D, SONG G M. The male-specific region of the human Y chromosome is a mosaic of discrete sequence classes.,2003, 423(6942): 825-837.
[6] PAYEN E, PAILHOUX E, ABOU M R, GIANQUINTO L, KIRSZENBAUM M, LOCATELLI A, COTINOT C. Characterization of ovine SRY transcript and developmental expression of genes involved in sexual differentiation.,1996, 40(3): 567-575.
[7] FENG J, LAJIA C, TAYLOR D J, WEBSTER M S. Genetic distinctiveness of endangered dwarf blue sheep (schaeferi): evidence from mitochondrial control region and Y-linkedintron sequences.,2001, 92(1): 9-15.
[8] LAWSON L J, HEWITT G M. Comparison of substitution rates inandintrons of sheep and goat related species supports the hypothesis of male-biased mutation rates.2002, 54(1), 54-61.
[9] MEADOWS J R, HAWKEN R J, KIJAS J W. Nucleotide diversity on the ovine Y chromosome.2004, 35(5): 379-385.
[10] 刘帅兵. 绵羊基因干扰载体筛选及性控效果的验证[D]. 石河子: 石河子大学, 2014.
LIU S B. The RNAi vector screening and verification of the effects of sexual control fromgene[D]. Shihezi: Shihezi University, 2014. (in Chinese)
[11] 熊勇, 徐亚欧. 藏系绵羊/基因片段进化分析. 生物技术2010, 20(6): 1-5.
XIONG Y, XU Y O. Evolution analysis the Tibetan sheepgene fragment.2010, 20(6): 1-5. (in Chinese)
[12] 蒋利, 王康环, 王海, 任冰冰, 刘光伟, 徐亚欧. 阿勒泰羊、藏绵羊(贾洛类群、欧拉类群)基因序列分析及亲缘关系研究. 西南民族大学学报(自然科学版)2013, 39(4): 487-494.
JIANG L, WANG K H, WANG H, REN B B, LIU G W, XU Y O. Sequence analysis of the Sry gene and genetic relationship of Altay Sheep, Tibetan sheep(Jialuo population, Oula population).2013, 39(4): 487-494. (in Chinese)
[13] HUGHES J F, SKALETSKY H, PYNTIKOVA T, GRAVES T A, DAALEN S K M V, MINX P J, FULTON R S, MCGRATH S D, LOCKE D P, FRIEDMAN C. Chimpanzee and human Y chromosomes are remarkably divergent in structure and gene content.2010, 463(7280): 536-539.
[14] HUGHES J F, ROZEN S. Genomics and genetics of human and primate y chromosomes.2012, 13(1): 83-108.
[15] SOH Y Q S, ALFÖLDI J, PYNTIKOVA T, BROWN L, GRAVES T, MINX P, FULTON R, KREMITZKI C, KOUTSEVA N, MUELLER J. Sequencing the mouse Y chromosome reveals convergent gene acquisition and amplification on both sex chromosomes.2014, 159(4): 800-813.
[16] SAMBROCK J, RUSSEL D., 3rd ed. New York: Cold Spring Harbor Laboratory Press, 2001: 463-485.
[17] LI R, WANG S Q, XU S Y, HUANG J P, WANG F Q, MA Z J, DANG R H, LAN X Y, CHEN H, LEI C Z. Novel Y-chromosome polymorphisms in chinese domestic yak.2014, 45(3): 449-452.
[18] NIJMAN I J, VAN BOXTEL D C J, VAN CANN L M, MARNOCH Y, CUPPEN E, LENSTRA J A. Phylogeny of Y chromosomes from bovine species.2008, 24(5): 723-726.
[19] CHENG H, SHI H, ZHOU R, GUO Y, LI L, LIU J, YONG J, KUDO T, SUTOU S. Characterization of Bovidae sex-determining gene.2001, 33(6): 687-694.
[20] VERKAARE L, NIJMAN I J, BEEKE M, HANEKAMP E, LENSTRA J A. Maternal and paternal lineages in cross-breeding bovine species. Has wisent a hybrid origin?2004, 21(7): 1165-1170.
[21] BONFIGLIO S, DE G A, TESFAYE K, GRUGNI V, SEMINO O, FERRETTI L. A novelpolymorphism allowing a rapid and unambiguous classification ofY chromosomes into haplogroups.2012, 43(5): 611-613.
[22] GÖTHERSTRÖM A, ANDERUNG C, HELLBOR L G, ELBURG R, SMITH C, DAN G B, ELLEGREN H. Cattle Domestication in the Near East Was Followed by Hybridization with Aurochs Bulls in Europe.2005, 272(1579): 2345-2350.
[23] GINJA C, TELO D G L, PENEDO M C. Y chromosome haplotype analysis in Portuguese cattle breeds using SNPs and STRs.2015, 100(2): 148.
[24] ANDREW P B, SINCLAIR H, PALMER M S, HAWKINS J R, GRIFFITHS B L, SMITH M J, FOSTER J W, FRISCHAUF A F, LOVELL-BADGE R, GOOGFELLOW P N. A gene from the human sex-determining region encodes a protein with homology to a conserved DNA-binding motif.1900, 346(6281): 5.
[25] MEADOWS J R, KIJAS J W. Re-sequencing regions of the ovine Y chromosome in domestic and wild sheep reveals novel paternal haplotypes.2009, 40(1): 119-123.
[26] AO A, ERICKSON R P, WINSTON R M, HANDYSIDE A H. Transcription of paternal Y-linked genes in the human zygote as early as the pronucleate stage.1994, 2(4): 281-287.
[27] NAGAMINE C M, CHAN K, KOZAK C A, LAU Y F. Chromosome mapping and expression of a putative testis-determining gene in mouse.1989, 243(4887): 80-83.
[28] 毛德才, 徐亚欧, 熊勇, 曹特, 苏畅. 牦牛与其他物种基因片段间的进化关系. 中国生物化学与分子生物学报2007, 23(6): 450-456.
MAO D C, XU Y O, XIONG Y, CAO T, SU C. Evolution analysis ofgene fragments between yak and other species., 2007, 23(6): 450-456. (in Chinese)
[29] POLOUMIENKO A. Cloning and comparative analysis of the bovine, porcine, and equine sex chromosome genesand.2004, 47(1): 74-83.
[30] AASEN E, MEDRANO J F. Amplification of theandgenes for sex identification in humans, cattle, sheep and goats.1990, 8(12): 1279.
[31] SATOU K, SUTO J I. Effect of the Y chromosome on testis weight in mice.2015, 77(6): 753.
[32] 马志杰, 胡双龙, 李瑞哲, 陈生梅, 雷初朝, 晁生玉. 柴达木黄牛Y染色体单倍型组构成及父系起源——基于基因多态性的分析. 中国畜牧杂志2017, 53(6): 36-39.
MA Z J, HU S L, LI R Z , CHEN S M, LEI C Z, CHAO S Y. Y Chromosome haplogroups and paternal origin of qaidam cattle: Based on the polymorphism analysis of USP9Y gene., 2017, 53(6): 36-39. (in Chinese)
[33] LI R, XIE W M, CHANG Z H, WANG S Q, DANG R H, LAN X Y, CHEN H, LEI C Z. Y chromosome diversity and paternal origin of Chinese cattle.2013, 40(12): 6633-6636.
[34] 肖红梅, 刘志红, 张文广, 李金泉. 绒山羊基因的BAC筛选与鉴定. 中国畜牧兽医2012, 39(4): 31-34.
XIAO H M, LIU Z H, ZHANG W G, LI J Q. Screening and identification of cashmere goatsgene., 2012, 39(4): 31-34. (in Chinese)
[35] LAASER I, THEIS F J, DE ANGELIS M H, KOLB H J, ADAMSKI J. Huge splicing frequency in human Y chromosomalgene.2011, 15(3): 141.
[36] IVANOV R, HOL S, AARTS T, HAGENBEEK A, SLAGER E H, EBELING S.-specific TCR-transfer generates potential graft-versus-leukaemia effector T cells.2005, 129(3): 392.
(责任编辑 林鉴非)
Screening of Y Chromosome Specific Primers and Y-SNPs in Sheep
CAO XueTao1, PEI ShengWei1, ZHANG Jin3, LI FaDi1,2, Li Gang3, LI WanHong1, YUE XiangPeng1
(1State Key Laboratory of Grassland Agro-ecosystems, Lanzhou University/Key Laboratory of Grassland Livestock Industry Innovation, Ministry of Agriculture and Rural Affairs/ College of Pastoral Agriculture Science and Technology, Lanzhou University, Lanzhou 730020;2Engineering Laboratory of Mutton Sheep Breeding and Re-production Biotechnology in Gansu Province, Minqin 733300, Gansu;3Animal Breeding Centre of Gansu Province, Wuwei 733000, Gansu)
【Objective】The male specific region of the mammalian Y chromosome (MSY)does not recombine with X chromosome during meiosis process, which is an important genetic resource for analyzing paternal genetic diversity due to its strict father to son inherited character. In addition, most of genes on MSY exclusively or predominantly express in the testis, indicating they may play essential roles in spermatogenesis and male reproduction. Since it is extremely difficult to sequence of entire Y chromosome, many species have very few Y chromosome sequences. Therefore, the current study was conducted to select ovine Y chromosome specific primers and Y-SNP based on the previous primer information used in bovidae species, and to compare the Y-fragment sequences similarity among sheep, bovine, goat, yak and bharal. Meanwhile, the Y-SNP was associated with sheep scrotal circumference to supply scientific basis for constructing ovine Y-haplotypes, identifying molecular markers for embryo sex and male reproductive traits in the future.【Method】Based on the investigation of available references about bovidae Y chromosome, 29 pairs of Y-primers reported in cattle, yak, goat were selected to amplify rams DNA using ewes DNA and ddH2O as negative controls. Subsequently, the Y-SNPs within ovine Y-specific fragment identified of different sheep breeds were investigated by DNA sequencing of DNA pooling and PCR-RFLP methods, including Suffolk sheep (n=146),White Suffolk sheep (n=91),East Friesian sheep (n=6), Texel sheep (n=72), South African Mutton Merino (n=17), Dorper sheep (n=32), Hu sheep (n=55),Tibetan sheep (n=34), Tan sheep (n=43), and bharal (n=14). Chromas and DNASTAR were used to analyze the results of DNA-pool sequencing, and DNAman was used for homology analysis of yak, goat, cattle and bharal. Meanwhile, the correlation analysis between the11 gene fragment polymorphisms and the testis size was performed by SPSS 19.0.【Result】The results showed that 6 out of 29 pairs of primers analyzed were ovine Y-specific, which could amplify3,6,,,11 and6 fragments, respectively. However, 17 pairs of primers failed to show amplification bands, and 6 pairs of primers showed amplified bands in the DNA of the ewes. The similarity of them among sheep, bharal, cattle, goat and yak ranged from 81.51% to 98.84%. In addition, a Y-SNP (G>A) with in11 fragment was first identified in the Suffolk and white Suffolk sheep.According to RCR-RFLP analysis, two genotypes (AA, GG) were detected in the Suffolk sheep and white Suffolk sheep, while only the GG genotype was found in the other seven sheep breeds. The genotypic frequencies of the GG and AA were 0.747 and 0.253 in White Suffolk sheep, respectively, while they were 0.986 and 0.014 in Suffolk sheep, indicating the dominant genotype was GG genotype in White Suffolk sheep and Suffolk sheep. Association analysis suggested that the testis size of the GG genotype was significantly higher than those of the AA genotype in the white Suffolk sheep population (=0.029).【Conclusion】In this study, six pairs of ovine Y-specific primers were identified, and the Y-linked fragment identified in ovine showed a high similarity with cattle, goats and yaks, indicating certain conservation in the evolutionary process. In addition, a Y-SNP was found to be specific in White Suffolk sheep and Suffolk sheep, which was closely associated with the testis size of white Suffolk sheep.
Y chromosome; sheep; single nucleotide polymorphism (SNP)
2017-12-25;
2018-04-17
甘肃省农业生物技术研究与应用开发项目(GNSW-2015-24,GNSW-2016-22)、甘肃省重点研发计划(17YF1NA066)、甘肃省农业科技创新项目(GNCX-2014-41)、国家肉羊产业技术体系(CARS-38)和长江学者和创新团队发展计划(IRT13019)
曹学涛,E-mail:caoxt16@ lzu.edu.cn。信作者乐祥鹏,E-mail:lexp@lzu.edu.cn
10.3864/j.issn.0578-1752.2018.15.0014

