不同环境基于高密度遗传图谱的稻米外观品质QTL定位
彭 强 李佳丽 张大双 姜 雪 邓茹月 吴健强 朱速松
不同环境基于高密度遗传图谱的稻米外观品质QTL定位
彭 强**李佳丽**张大双 姜 雪 邓茹月 吴健强 朱速松*
贵州省农业科学院水稻研究所, 贵州贵阳 550006
为解析稻米外观品质遗传基础, 挖掘稳定存在的控制稻米外观品质性状的QTL, 本研究以籼稻品种V20B和爪哇稻品种CPSLO17作为亲本, 构建包含150个重组自交家系(recombinantion inbred line, RIL)的RIL作图群体, 进行外观品质性状QTL定位分析。利用特定位点扩增长度测序(SLAF-seq)技术, 构建了一个由12个连锁群包含8602个标记, 平均间距为0.29 cM的高密度遗传图谱。采用IciMapping 4.0软件的ICIM-ADD方法在3种环境(贵阳、贵定、三亚)对4个外观品质性状(粒长、粒宽、垩白度和垩白粒率)进行QTL (quantitative trait locus)定位分析。结果表明: 3种环境共检测到9个粒长QTL、6个粒宽QTL、3个垩白度QTL和4个垩白粒率QTL; 有5个QTL在多个环境被重复检测到, 其中3种环境都定位到的粒宽QTL和垩白度QTL为同一定位区间(第5染色体的Marker1642127-Marker1514505); 此外, 垩白度QTL的定位区间(Marker1554573-Marker1554589)和垩白粒率QTL也是一样的。序列比对发现QTL定位区间仅51.5 kb, 是新的垩白性状主效QTL。本研究结果不仅为挖掘新的外观品质性状基因奠定基础, 也有助于开发新的分子标记进行水稻外观品质性状遗传改良。
水稻; 外观品质; QTL; 高密度遗传图谱; 高通量测序
近年来, 随着人们对优质稻米的需求越来越多, 如何改善我国稻米品质, 选育出适应市场需求的优质稻新品种, 是水稻育种家们进行基础研究和遗传改良的首要解决问题。外观品质是稻米品质性状最重要的评价指标和构成因素, 严重影响稻米的蒸煮食味品质和营养品质, 如高垩白米饭粒的蓬松性差, 食味口感也差[1-2]。因此, 进行稻米外观品质的遗传基础研究, 对稻米品质遗传改良和优质水稻品种选育具有重要的理论价值和实际意义。
粒形和垩白是衡量稻米外观品质的重要指标, 也是中国水稻品种审定时稻米品质的定级指标[3-4]。得益于水稻全基因组学的快速发展和新型分子标记的开发, 学者们利用不同类型遗传群体(BIL、RIL、DH、IL等)定位到大量的粒形和垩白性状QTL[5-7]。其中, 粒长基因[8-9]、粒宽基因[10-11]及多个粒形调控基因([12-13]、[14]、[15-16]、[17]等)被相继分离克隆; 虽然也有很多的垩白性状QTL[18-21]被检测到, 但只有少数QTL (如[22]、[23]和[24])被精细定位,[25]基因是被成功分离克隆的垩白主效QTL。由于粒形和垩白性状都是多基因控制的数量遗传性状, 且受制于传统分子标记(SSR、InDel、RFLP等)构建的水稻连锁图谱存在标记数量少和覆盖密度低等缺陷, 仍有很多遗漏的粒形和垩白性状QTL未被定位到[26]。因此, 开发遗传稳定性高、分布广、数量多的SNP标记和构建高密度遗传图谱进行QTL定位分析越来越受到学者们的青睐。SLAF-seq技术是一种基于RRGS策略和高通量pair- end sequencing的大规模开发SNP及其基因型鉴定的新技术, 已应用于多个物种的遗传连锁图谱构建和QTL定位[27-29]。
亲本间遗传背景多样性是开发多态性分子标记的前提基础, 有助于挖掘新的粒形和垩白性状QTL。目前水稻遗传连锁图谱主要是籼籼交、籼粳交等遗传背景, 鲜有用籼爪交遗传背景绘制高密度遗传连锁图谱的报道。本研究利用籼爪交遗传背景材料(V20B/CPSLO17)创建高代RIL群体, 在不同环境(贵阳、贵定和三亚)下测定RIL群体的稻米外观品质, 借助SLAF-seq技术开发的多态性SLAF标签和构建的高密度遗传连锁图谱, 进行水稻外观品质性状QTL定位分析, 鉴定出受环境影响小、遗传稳定、贡献率高的稻米粒形和垩白性状主效QTL, 为挖掘新的水稻外观品质性状基因奠定理论基础, 也为开发新的分子标记进行水稻外观品质性状遗传改良提供科学依据。
1 材料与方法
1.1 试验材料
本研究使用的遗传群体是以籼型水稻品种V20B为母本, 爪哇稻品种CPSLO17为父本, 杂交得F1代, 在贵州贵阳和海南三亚多年自交种植, 采用单粒传法于贵州贵阳获得150份F10代重组自交系(recombinant inbred line, RIL)群体材料。
1.2 田间试验及表型数据测定
于2014年至2016年在贵州贵阳、贵州贵定和海南三亚三地试验田对亲本和RIL群体进行种植, 每个材料种植20株, 成熟时混收, 晾干保存, 共收2份亲本和150份RIL材料。对于每份供试样品表型数据测定如下: 随机挑选50粒整精米置于玻璃板上, 目测挑选出有阴影的米粒, 并记录米粒数量, 计算出该样品的垩白粒率(%); 随机挑选10粒有垩白的米粒, 逐粒目测垩白面积占整个米粒面积的百分率并取平均值, 即为该样品的垩白大小; 该样品的垩白度(%)为垩白粒率(%)与垩白大小的乘积; 随机挑取10粒饱满的种子, 纵向或横向排列, 摆成一排, 不重叠, 不留隙, 用电子游标卡尺测量10粒种子总长度和总宽度, 重复3次取平均值, 计算粒长和粒宽。
1.3 叶片DNA提取
在植株八叶期对RIL群体分家系混取成熟叶片组织, 采用 CTAB 法提取基因组 DNA。加800 μL 1.5×CTAB研磨叶片; 65℃水浴30 min; 加800 μL氯仿/异戊醇(24∶1, v/v); 12 000 r min–1离心8 min; 吸400 μL上清液至新管, 加800 μL 95%乙醇, 混匀,-20℃冷冻20 min; 12 000转min–1离心10 min; 弃上清, 加500 μL 75%乙醇洗DNA沉淀, 8000 r min–1离心5 min; 弃上清, 吹干, 加150 μL ddH2O溶解即可。
1.4 高密度遗传连锁图谱的构建
参考日本晴(L)基因组采用III酶切基因组DNA, 经过3¢端加A处理、连接测序接头Dual-index[30]、PCR扩增、纯化、混样等步骤构建SLAF文库(264~314 bp的DNA片段); 再基于Illumina HiSeq 2500平台(Illumina, Inc., San Diego, CA, USA)高通量测序, 在基因组范围内筛选多态性SLAP标记。参照Sun等[27]的方法进行SLAF-seq数据分析和基因型编码(RIL群体的分离方式为aa×bb); 根据高质量SLAF标签间的mLOD值进行连锁分群, 采用HighMap软件[31]的最大似然估计法构建高密度遗传图谱。
1.5 QTL定位
采用 IciMapping 4.0进行稻米外观品质性状QTL定位分析。所需要的文件格式按Manual的要求编辑。完备区间作图(ICIM)法对RIL群体的稻米外观品质性状进行QTL作图及遗传效应分析, 作图步长为1.00 cM, PIN设置为0.001, LOD阈值设置为 3.0。并计算每个QTL对稻米外观品质性状的贡献率和加性效应, QTL的命名遵循McCouch原则。加性效应为正值表示增效等位基因来源于高垩白亲本V20B, 为负值表示来源于低垩白亲本CPSLO17。
2 结果与分析
2.1 稻米外观品质性状的表型分析
150份V20B/CPSLO17 RIL群体种植于3个不同环境(贵阳、贵定、三亚), 每份材料收种保存3个月后进行稻米垩白粒率、垩白度、粒长和粒宽性状考种。3种种植环境(贵阳、贵定、三亚)下150份RIL家系的稻米外观品质检测结果表明, RIL群体中4个外观品质性状(垩白粒率、垩白度、粒长和粒宽)都表现出连续变异(图1), 说明水稻的垩白和粒形性状表现出受多基因控制的数量性状遗传特征。
两个亲本和150份RIL群体在3种种植环境(贵阳、贵定、三亚)下外观品质性状表型数据如表1所示。表型数据表明, V20B和CPSLO17两个亲本间在垩白度和垩白粒率上的差异较大, 在粒长、粒宽上也存在差异, V20B在垩白度、垩白粒率、粒宽上为高值亲本, CPSLO17在粒长上为高值亲本。RIL群体中稻米粒形性状在3个不同生态地点变化不明显, 而稻米垩白性状在3个不同生态地点变化明显, 说明垩白易受环境影响。
2.2 高密度遗传图谱构建
以水稻品种V20B/CPSLO17组合衍生的150份重组自交家系作为作图群体, 利用特定位点扩增长度测序(SLAF-seq)技术开发SLAF标签。高通量测序结果表明, V20B亲本的SLAF数为58 473, 平均测序深度为49.41×; CPSLO17亲本的SLAF 数为51 837, 平均测序深度为53.25×; SLAF标签亲本平均测序深度为51.21× (表2)。本文共开发67 017个高质量SLAF标签, 其中15 853个SLAF亲本间有多态性, 符合aa×bb基因分离类型的有效标签为13 275个, 占比19.81%; 达到绘制高质量遗传图谱要求(测序深度10 ×以上, 亲本纯合)的高质量多态性SLAF标签为8657个, 占比12.92%。

图1 3种环境下RIL群体中稻米外观品质性状分布
GL: 粒长; GW: 粒宽; CGP: 垩白粒率。
GL: grain length; GW: grain width; CGP: chalky grain percentage.
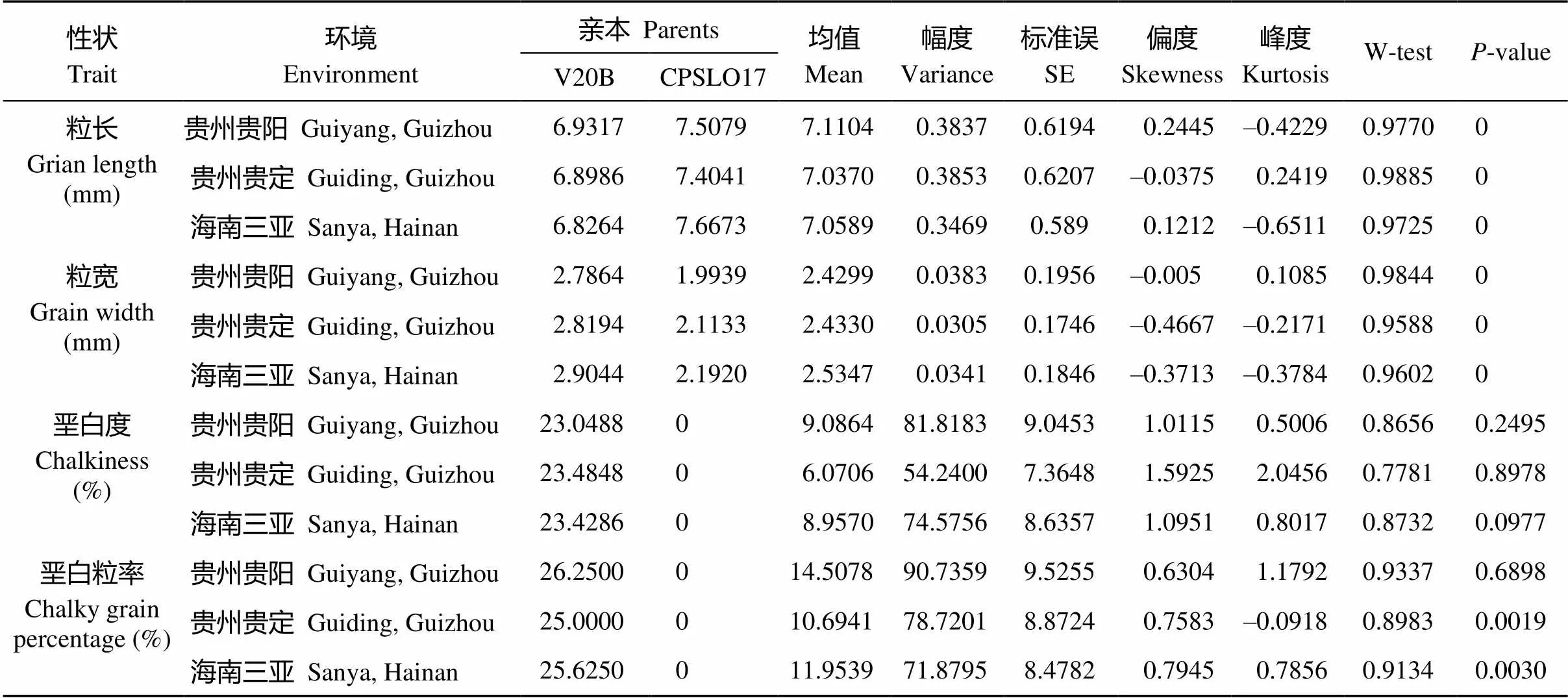
表1 RIL群体在3个环境下的外观品质性状表现
采用HighMap软件的最大似然估计法成功构建了一个包含12个连锁群, 总图距为2508.65 cM, 8602个上图标记, 标记间平均距离为0.29 cM的高密度遗传图谱(表3)。Chr.1连锁图上图SLAF标记数最多(1334个), 图距也最长(365.11 cM), 标记间平均距离为0.27 cM; Chr. 8连锁图上图SLAF标记数最少(386个); Chr.10连锁图的图距最短, 仅103.98 cM, 上图504个SLAF, 标记间平均距离为0.21 cM; SLAF标记间最大距离为10.5 cM, 位于Chr.3连锁图; 12个连锁图的SLAF标记平均间距最低为Chr.5连锁图的0.21 cM, 最高为Chr.3连锁图的0.60 cM (表3)。该图谱可用于重要农艺性状QTL定位。

表2 亲本的SLAF数量及其覆盖深度
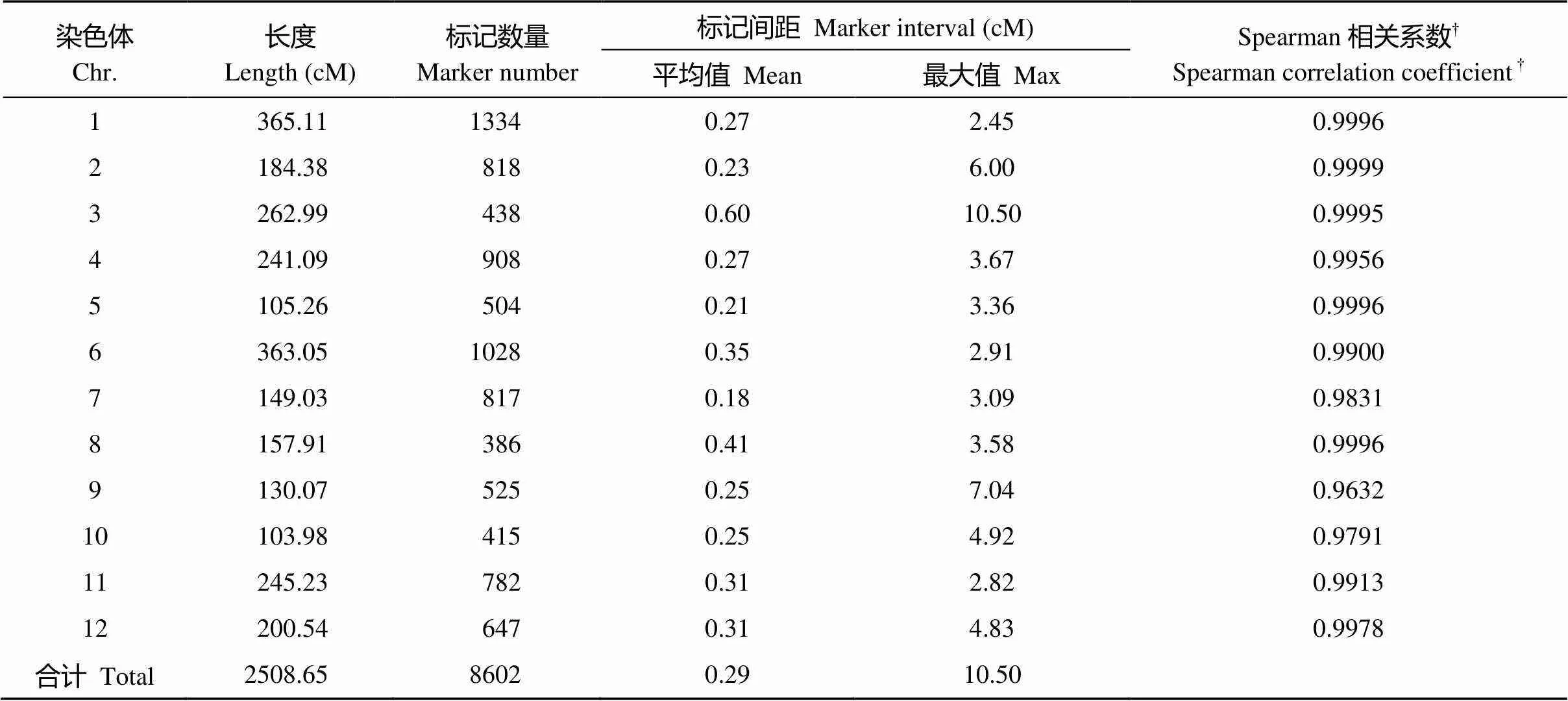
表3 12连锁群基本信息统计
†Spearman相关系数, 系数越接近于1, 表示共线性越好。
†Spearman correlation coefficient. The coefficient closed to 1 means the better the collinearity.
2.3 RIL群体中的QTL定位分析
利用软件IciMapping 4.0的ICIM-ADD方法对水稻外观品质性状进行QTL分析, 结果表明, 在3种不同的种植环境(贵阳、贵定、三亚)中共检测到22个控制水稻粒长、粒宽、垩白度、垩白粒率4个性状的QTL (图2和表4), 分布在除了第6、第9染色体以外的其他染色体上, 3种环境下检测到的水稻外观品质性状QTL在高密度遗传图谱上的位置如图2所示。
3个地点共检测到9个控制粒长的QTL, 分布在第1、第3、第7、第8和第10染色体上, LOD值在3.05~17.97之间, 单个QTL的表型贡献率(percentage of variance explained, PVE)变幅为5.42%~31.18% (表4)。其中贵阳检测到7个QTL, 贵定检测到2个QTL, 三亚检测到2个QTL, 只有和在贵阳和三亚2个环境被重复检测到, 其余均为单一环境检测, 而效应较大的QTL分布在第8染色体Marker201080–Marker118092区间内, 其LOD值为17.97, 表型贡献率31.18%。

表4 不同环境下稻米外观品质性状QTL分析
GL: grian length; GW: grain width; Cha: chalkiness; CGP: chalky grain percentage; PVE: percentage of variance explained; Add: additive effect.

图2 稻米外观品质性状的QTL在遗传图谱上的分布
3个地点共检测到6个控制粒宽的QTL, 分布在第5、第7、第11和第12染色体上, LOD 值在4.09~14.34之间, 单个QTL的表型贡献率变幅为7.81%~28.66% (表4)。其中贵阳检测到3个QTL, 贵定检测到2个QTL, 三亚检测到3个QTL, 而定位于第5染色体Marker1642127-Marker1514505区间内的在三亚、贵阳和贵定3个环境中被反复检测到, 且对应的LOD值(14.34、8.46和5.84)和表型贡献率(28.66%、16.96%和12.59%)都比较高(表4)。
3个地点共检测到3个控制垩白度的QTL, 分布在第4和第5染色体上, LOD 值在3.75~8.55之间, 单个QTL的表型贡献率变幅为7.46%~17.83% (表4)。其中贵阳检测到1个QTL, 贵定检测到1个QTL, 三亚检测到3个QTL, 而3种环境(贵阳、贵定、三亚)都稳定检测到位于第5染色体Marker1642127-Marker1514505区间内的, 且对应的LOD值(4.69、5.05和8.55)和表型贡献率(12.63%、14.77%和17.83%)都比较高(表4)。
控制垩白粒率的QTL仅在贵阳和三亚2个地点检测到4个, 贵定没有检测到, 分布在第2、第4和第5染色体上, LOD 值在3.42~5.82之间, 单个QTL的表型贡献率变幅为9.15%~12.50% (表4)。其中贵阳检测到2个QTL, 三亚检测到3个QTL, 而在三亚和贵阳2个环境中被同时检测到, 且对应的LOD 值为3.42和5.28, 对应的表型贡献率为9.15%和11.15%。
对定位结果分析发现, 在第5染色体上存在2个同时控制2个外观品质性状的定位区间, 其中粒宽QTL和垩白度QTL1都定位于Marker1642127-Marker1514505区间内, 而控制垩白度的QTL2和控制垩白度的QTL2都分布在Marker1554573-Marker1554589区间内。
3 讨论
通过多种不同环境对同一作图群体进行QTL定位, 选择受环境影响小、遗传稳定、贡献率高的QTL来改良作物的某些性状的研究思路越来越受到研究人员的关注, 而且稻米外观品质性状受环境条件的影响较大, 因此在不同生态环境下进行水稻外观品质性状的QTL定位分析, 获得稳定表达的外观品质性状主效QTL, 具有重要的理论研究和实践利用价值。
本文利用亲本V20B和CPSLO17构建的RIL群体, 借助SLAF-seq技术构建的高密度遗传图谱(8602个上图标记, 总图距为2508.65 cM, 标记间平均距离为0.29 cM), 在3种环境(贵阳、贵定、三亚)共检测到22个水稻外观品质(粒长、粒宽、垩白度和垩白粒率)性状QTL。5个QTL (和)被多个环境重复检测到, 其中和在贵阳、贵定、三亚3种环境被反复检测到, 且对应的LOD值和表型贡献率都比较高, 这表明粒宽QTL和垩白度QTL是受环境影响小、遗传稳定、贡献率高的主效QTL。此外,和都位于第5染色体Marker1642127-Marker1514505区间, 说明和之间可能受同一QTL或基因调控, 具有“一因多效”遗传效应, 也可能是两个独立基因的紧密连锁, 这需后续的精细定位和基因功能验证来确认。2个粒长QTL (和)和垩白粒率QTL都是在贵阳和三亚被同时检测到, 而与垩白度QTL的定位区间一样, 都位于第5染色体Marker1554573-Marker1554589区间, 表明垩白粒率与垩白度性状QTL存在紧密连锁, 可能受同一QTL或基因调控。
研究表明, 水稻的第5染色体上垩白主效基因()与粒宽基因()紧密连锁, 而且通常宽粒的籼稻普遍是高垩白, 因此在育种实践中很难实现增加粒宽的同时减少垩白。利用SLAF标签Marker1642127和Marker1514505序列在NCBI网站进行blast比对, 以Nipponbare ()基因组序列作为参照, 发现QTL定位在51.5 kb区间内。在RGAP 7网站检索发现该51.5 kb区间仅包含5个候选基因, 且都不是已知的垩白基因(LOC_Os05g 06480), 说明QTL为新的水稻垩白主效QTL, 为后续的候选基因挖掘奠定了基础。
[1] 张昌泉, 赵冬生, 李钱峰, 顾铭洪, 刘巧泉. 稻米品质性状基因的克隆与功能研究进展.中国农业科学, 2016, 49: 4267–4283 Zhang C Q, Zhao D S, Li Q F, Gu M H, Liu Q Q. Progresses in research on cloning and functional analysis of key genes involving in rice grain quality., 2016, 49: 4267–4283 (in Chinese with English abstract)
[2] 李一博, 赵雷. 水稻品质性状的遗传改良及其关键科学问题. 生命科学, 2016, 28: 1168–1179 Li Y B, Zhao L. Genetic improvement and key scientific questions of grain quality traits in rice., 2016, 28: 1168–1179 (in Chinese with English abstract)
[3] 江良荣, 李义珍, 王侯聪, 黄育民. 稻米外观品质的研究进展与分子改良策略. 分子植物育种, 2003, 1: 243–255 Jiang L R, Li Y Z, Wang H C, Huang Y M. Research progresses on appearance quality of rice grain and strategies for its molecular improvement., 2003, 1: 243–255 (in Chinese with English abstract)
[4] 王忠华, 方振华, 干建彗. 稻米外观品质性状遗传与分子定位研究进展. 生命科学, 2009, 21: 444–351 Wang Z H, Fang Z H, Gan J H. Advances in genetic research and molecular mapping of the rice grain appearance quality., 2009, 21: 444–351 (in Chinese with English abstract)
[5] Qiu X, Chen K, Lv W, Ou X, Zhu Y, Xing D, Yang L, Fan F, Yang J, Xu J, Zheng T, Li Z. Examining two sets of introgression lines reveals background-independent and stably expressed QTL that improve grain appearance quality in rice (L.)., 2017, 130: 951–967
[6] Wan X Y, Wan J M, Weng J F, Jiang L, Bi J C, Wang C M, Zhai H Q. Stability of QTLs for rice grain dimension and endosperm chalkiness characteristics across eight environments., 2005, 110: 1334–1346
[7] 杨亚春, 倪大虎, 宋丰顺, 李泽福, 易成新, 杨剑波. 不同生态地点下稻米外观品质性状的QTL定位分析. 中国水稻科学, 2011, 25: 43–51 Yang Y C, Ni D H, Song F S, Li Z F, Yi C X , Yang J B. Identification of QTLs for rice appearance quality traits across different ecological sites., 2011, 25: 43–51 (in Chinese with English abstract)
[8] Fan C C, Xing Y Z, Mao H L, Lu T T, Han B, Xu C G, Li X H, Zhang Q F., a major QTL for grain length and weight and minor QTL for grain width and thickness in rice, encodes a putative transmembrane protein., 2006, 112: 1164–1171
[9] Mao H, Sun S, Yao J, Wang C, Yu S, Xu C, Li X, Zhang Q. Linking differential domain functions of the GS3 protein to natural variation of grain size in rice., 2010, 107: 19579–19584
[10] Shomura A, Izawa T, Ebana K, Ebitani T, Kanegae H, Konishi S, Yano M. Deletion in a gene associated with grain size increased yields during rice domestication., 2008, 40: 1023–1028
[11] Weng J F, Gu S H, Wan X Y, Gao H, Guo T, Su N, Lei C L, Zhang X, Cheng Z J, Guo X P, Wang J L, Jiang L, Zhai H Q, Wan J M. Isolation and initial characterization of, a major QTL associated with rice grain width and weight., 2008, 18: 1199–1209
[12] Che R, Tong H, Shi B, Liu Y, Fang S, Liu D, Xiao Y, Hu B, Liu L, Wang H, Zhao M, Chu C. Control of grain size and rice yield by-mediated brassinosteroid responses., 2015, 2: 15195
[13] Hu J, Wang Y, Fang Y, Zeng L, Xu J, Yu H, Shi Z, Pan J, Zhang D, Kang S, Zhu L, Dong G, Guo L, Zeng D, Zhang G, Xie L, Xiong G, Li J, Qian Q. A rare allele ofenhances grain size and grain yield in rice., 2015, 8: 1455–1465
[14] Song X J, Huang W, Shi M, Zhu M Z, Lin H X. A QTL for rice grain width and weight encodes a previously unknown RING type E3 ubiquitin ligase., 2007, 39: 623–630
[15] Wang S K, Li S, Liu Q, Wu K, Zhang J Q, Wang S S, Wang Y, Chen X B, Zhang Y, Gao C X, Wang F, Huang H X, Fu X D. Theregulatory module determines grain shape and simultaneously improves rice yield and grain quality., 2015, 47: 949–954
[16] Wang Y X, Xiong G S, Hu J, Jiang L, Yu H, Xu J, Fang Y X, Zeng L J, Xu E B, Xu J, Ye W J, Meng X B, Liu R F, Chen H Q, Jing Y H, Wang Y H, Zhu X D, Li J Y, Qian Q. Copy number variation at thelocus contributes to grain size diversity in rice., 2015, 47: 944–948
[17] Wang S K, Wu K, Yuan Q B, Liu X Y, Liu Z B, Lin X Y, Zeng R Z, Zhu H T, Dong G J, Qian Q, Zhang G Q, Fu X D. Control of grain size, shape and quality byin rice., 2012, 44: 950–954
[18] Peng B, Wang L, Fan C, Jiang G, Luo L, Li Y, He Y. Comparative mapping of chalkiness components in rice using five populations across two environments., 2014, 15: 49
[19] Zhao X, Daygon V D, McNally K L, Hamilton R S, Xie F, Reinke R F, Fitzgerald M A. Identification of stable QTLs causing chalk in rice grains in nine environments., 2016, 129: 141–153
[20] 邱先进, 袁志华, 陈凯, 杜斌, 何文静, 杨隆维, 徐建龙, 邢丹英, 吕文恺. 用全基因组关联分析解析籼稻垩白的遗传基础. 作物学报, 2015, 41: 1007–1016 Qiu X J, Yuan Z H, Chen K , Du B , He W J, Yang L W, Xu J L, Xing D Y, Lyu W K. Genetic dissection of grain chalkiness inmini-core germplasm using genome-wide association method., 2015, 41: 1007–1016 (in Chinese with English abstract)
[21] Xian J Q, Kai C, Wen K L, Xiao X O, Ya J Z, Dan Y X, Long W Y, Fang J F, Jie Y, Jian L X, Tian Q Z, Zhi K L. Examining two sets of introgression lines reveals background independent and stably expressed QTL that improve grain appearance quality in rice (L.)., 2017, 130: 951–967
[22] Zhou L, Chen L, Jiang L, Zhang W, Liu L, Liu X, Zhao Z, Liu S, Zhang L, Wang J, Wan J. Fine mapping of the grain chalkiness QTLin rice (L.)., 2009, 118: 581–590
[23] Guo T, Liu X, Wan X, Weng J, Liu S, Liu X, Chen M, Li J, Su N, Wu F, Cheng Z, Guo X, Lei C, Wang J, Jiang L, Wan J. Identification of a stable quantitative trait locus for percentage grains with white chalkiness in rice ()., 2011, 53: 598–607
[24] Gao Y, Liu C, Li Y, Zhang A, Dong G, Xie L, Zhang B, Ruan B, Hong K, Xue D, Zeng D, Guo L, Qian Q, Gao Z. QTL analysis for chalkiness of rice and fine mapping of a candidate gene for.(N Y), 2016, 9: 41
[25] Li Y, Fan C, Xing Y, Yun P, Luo L, Yan B, Peng B, Xie W, Wang G, Li X, Xiao J, Xu C, He Y.encodes a vacuolar H(+)-translocating pyrophosphatase influencing grain chalkiness in rice., 2014, 46: 398–404
[26] 胡苗, 孙志忠, 孙学武, 谭炎宁, 余东, 刘瑞芬, 袁贵龙, 丁佳, 袁定阳, 段美娟. 利用高密度SNP标记定位水稻粒形相关QTL. 杂交水稻, 2015, 30(5): 54–58 Hu M, Sun Z Z, Sun X W, Tan Y N, Yu D, Liu R F, Yuan G L, Ding J, Yuan D Y, Duan M J. Mapping of rice grain shape relevant QTLs using high-density SNP markers., 2015, 30(5): 54–58 (in Chinese with English abstract)
[27] Sun X, Liu D, Zhang X, Li W, Liu H, Hong W, Jiang C, Guan N, Ma C, Zeng H, Xu C, Song J, Huang L, Wang C, Shi J, Wang R, Zheng X, Lu C, Wang X, Zheng H. SLAF-seq: an efficient method of large-scale de novo SNP discovery and genotyping using high-throughput sequencing., 2013, 8(3): e58700
[28] Wei Q, Wang Y, Qin X, Zhang Y, Zhang Z, Wang J, Li J, Lou Q, Chen J. An SNP-based saturated genetic map and QTL analysis of fruit-related traits in cucumber using specific-length amplified fragment (SLAF) sequencing., 2014, 15: 1158
[29] Zhang Y, Wang L, Xin H, Li D, Ma C, Ding X, Hong W, Zhang X. Construction of a high-density genetic map for sesame based on large scale marker development by specific length amplified fragment (SLAF) sequencing., 2013, 13: 141
[30] Kozich J J, Westcott S L, Baxter N T, Highlander S K, Schloss P D. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform., 2013, 79: 5112–5120
[31] Liu D, Ma C, Hong W, Huang L, Liu M, Liu H, Zeng H, Deng D, Xin H, Song J, Xu C, Sun X, Hou X, Wang X, Zheng H. Construction and analysis of high-density linkage map using high-throughput sequencing data., 2014, 9(6): e98855
QTL Mapping for Rice Appearance Quality Traits Based on a High-density Genetic Map in Different Environments
PENG Qiang**, LI Jia-Li**, ZHANG Da-Shuang, JIANG Xue, DENG Ru-Yue, WU Jian-Qiang, and ZHU Su-Song*
Guizhou Rice Research Institute, Guizhou Academy of Agricultural Sciences, Guiyang 550006, Guizhou, China
To analyze the genetic basis of appearance quality of rice, explore QTL which controlled rice appearance quality related traits stably existing, a mapping population of 150 lines (recombination inbred lines, RIL), derived from a cross between rice varieties V20B and CPSLO17, was applied to analysis QTL location of appearance quality trait. The specific locus amplified fragment sequencing (SLAF-seq) technology was employed to construct a high-density genetic map in rice (L.). A genetic map included 8602 markers on the 12 linkage groups was successfully constructed, which with an average distance of 0.29 cM between adjacent markers. The ICIM-ADD method of IciMapping 4.0 software was used to analyze QTL location for four appearance quality traits, including grain length (GL), grain width (GW), chalky grain rate, and chalkiness degree in three environmental conditions (Guiyang, Guiding, and Sanya). A total of nine QTLs for grain length (GL), six QTLs for grain width (GW), four QTLs for chalkiness size, three QTLs for chalkiness degree traits were detested in three environments. Five QTLs were repeatedly detected in multiple environments, of which the QTLandwith the same localization interval (Marker1642127-Marker1514505 on chromosome 5) were repeatedly located in all three environments. In addition, the positioning interval of chalkiness QTL(Marker1554573-Marker1554589) is the same as the chalkiness size QTL. The sequence alignment found that the location interval of QTLwas only 51.5 kb, which was a new main effect QTL for chalkiness trait. These results lay the foundation for further exploiting candidate gene of appearance quality, which also contribute to the development of new molecular markers for rice appearance quality traits genetic improvement.
rice; appearance quality; QTL; high-density genetic map; high throughput sequencing
2018-06-12;
2018-06-19.
10.3724/SP.J.1006.2018.01248
朱速松, E-mail: susongzhu@139.com, Tel: 0851-83762727
**同等贡献(Contributed equally to this work)
彭强, E-mail: 450058876@163.com, Tel: 0851-83762727; 李佳丽, E-mail: lilylijiali@163.com, Tel: 0851-82285571
): 2018-03-14;
本研究由贵州省科技支撑计划项目(黔科合支撑[2018]2298号), 贵州省科研机构服务企业行动计划项目(黔科合服企[2014]4005号), 贵州省科技计划项目(黔科合平台人才[2017]5719号), 贵州省现代产业体系项目(GZCYTX2017-0602)和贵州省农业科学院青年基金项目(黔农科院青年基金[2018]18号)资助。
This study was supported by the Science and Technology Support Project of Guizhou Province(QKHZC20182298), the Research Institution Service Enterprise Action Plan Project of GuizhouProvince (QKHFQ20144005), the Science and Technology Plan Project of Guizhou Province(QKHPTRC20175719), the Modern Industry System Project of Guizhou Province(GZCYTX2017-0602), and the Youth Fund Project of Guizhou Academy of Agricultural Sciences (QNKYQN201818).
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20180616.0828.002.html

