水稻抗稻瘟病基因Pi47的精细定位和候选基因分析
肖湘谊 史学涛 盛浩闻 刘金灵 肖应辉
湖南农业大学农学院 / 水稻油菜抗病育种湖南省重点实验室 / 南方粮油作物协同创新中心, 湖南长沙 410128
稻瘟病是由子囊真菌(Magnaporthe oryzae)引起的水稻最严重病害之一, 至少分布在85个国家, 每年导致水稻减产10%~30%[1]。使用化学药剂和推广抗病新品种是防治稻瘟病的主要方法。大量施用化学药剂不仅污染环境, 危及环境生物和食品安全,还对人类健康构成潜在威胁。因此, 利用抗病基因选育抗病水稻新品种是防治稻瘟病最有效、经济和安全的措施[2]。然而, 由于稻瘟菌致病性迅速和频繁变异, 导致抗病品种种植3~5年后就丧失了抗性[3]。因此, 不断挖掘新的抗病种质和克隆新的抗病基因是水稻抗稻瘟病育种的基础和核心。
其中, 克隆稻瘟病抗性基因是解析水稻抗病分子遗传机制的重要基础。近年, 通过图位克隆技术分离了 28个稻瘟病抗性基因, 分别是编码 NBSLRR 蛋白的Pb1[4]、Pia[5]、Pib[6]、Pid3[7]、Pik[8]、Pik-h/Pi54[9]、Pik-m[10]、Pik-p[11]、Pish[12]、Pit[13]、Pita[14]、Piz-t[15]、Pi1[16]、Pi2[15]、Pi5[17]、Pi9[18]、Pi25[7]、Pi36[19]、Pi37[20]、Pi56[21]、Pi63[22]、PiCO39[23]、Pi50[24]、Pi65(t)[25]和Pigm[26]; 编码 B-Lectin 蛋白激酶的Pid2[27]; 和编码富含脯氨酸蛋白的唯一隐性抗性基因pi21[28]。通过等位基因挖掘技术克隆了Pid3-A4、Pi54of和Pi54rh[29-31]。这些稻瘟病抗性基因分布于除第 3染色体外的所有水稻染色体上, 其中第6染色体Pi9位点、第11染色体Pik位点和第12染色体Pita位点是稻瘟病抗性基因成簇分布的3个热点区域。Pik位点位于水稻第11染色体长臂近端粒处, 是水稻一个主效抗稻瘟病位点, 该位点具有Pik、Pikm、Pikp、Pikh、Pi1、Piks等多个抗稻瘟病等位基因。研究表明,Pik位点等位基因介导的抗性由2个紧密连锁、具有独立功能的NBS-LRR基因协同作用[10]。NBS包含3个高度保守的功能基序激酶1a、激酶2a和激酶3a, 它们与ATP或GTP结合后获得能量用于抵抗病原菌; 不同的抗病基因其LRR域也相差较大, 差别主要体现在重复序列的长度、数目和位置上; LRR域与病原小种的特异识别相关, 不同的抗病基因拥有不同的抗谱, 这跟 LRR域的不同有较大的关系[32]。最近, Zhang等[33]从全基因组范围内132个位点的332个NBS-LRR基因中鉴定到98个抗稻瘟病新基因, 揭示了水稻基因组存在丰富的 NBS-LRR类抗病基因从而对不同稻瘟菌生理小种产生抗性。
湘资3150是湖南省一个地方水稻品种, 在几十年种植过程中一直保持对稻瘟病广谱、持久抗性[34]。本实验室 Huang等[34]利用湘资 3150与高感品种CO39杂交构建的重组自交系群体(RIL)定位了 2个主效抗瘟基因Pi47和Pi48, 其中Pi47被定位于第11染色体的SSR标记RM206和RM224之间的33.5 cM染色体区域。本研究利用Pi47单基因系与感病品种CO39构建F2分离群体, 旨在对Pi47进行精细定位和候选基因预测分析, 明确Pi47与定位区间已有稻瘟病抗性基因的等位关系, 为进一步克隆分离该基因奠定基础。
1 材料与方法
1.1 植物材料与种植方法
抗性基因供体材料湘资3150 (XZ), 高感稻瘟病材料CO39,Pi47单基因系CX18、CX52和CX279,以及单基因系与CO39的F2群体。种子均由湖南农业大学水稻基因组实验室提供。CO39背景的近等基因系 IRBLk-Ka (Pik)、IRBLkh-K3 (Pikh)、IRBLkm-Ts(Pikm)、IRBLkp-K60 (Pikp), 由国际水稻研究所提供。
在室温下浸种, 露白后播种于装有泥土的塑料育秧盘中(18 cm × 10 cm × 5 cm), 以 CO39 为感病对照。育苗土是预先经过酸化处理, 并添加草炭、N、P、K肥和杀菌剂敌克松的田间土壤, 或草炭蛙石(2∶1)混合的基质。冬春季育苗在温室内进行, 夏秋季在田间进行。于稻苗二叶期浇施(NH4)2SO4一次(2 g 盘–1), 促使秧苗嫩绿、健壮。
1.2 供试菌系
供试稻瘟菌株共17个, 包括来自中国湖南的318-2、CHL473、X2007A-3、CHL440、CHL438、X2007A-7、195-2-2、87-4、193-1-1、110-2、236-1;来自中国广东的RB4、RB6; 来自中国福建的RB18、RB19; 来源不详的中国小种 RB12; 以及来自日本的KOH。菌种均由湖南农业大学水稻基因组实验室收集保存。
1.3 接种与病害调查
称取燕麦片30 g磨成粉状, 约200 g成熟番茄压榨成汁, 琼脂粉 12 g, 混匀后加蒸馏水定容至 1 L。灭菌并分装于培养皿中, 凝固后用薄膜封藏备用。
将纯化保存的稻瘟病菌接种于培养基上, 于 27℃培养箱中暗培养7 d左右。待其菌落直径达4~5 cm,使用无菌水在超净工作台上洗掉培养基表面的气生菌丝, 晾干后置弱光下培养3~4 d即产孢。用1%的Tween 20洗下培养基表面的分生孢子, 双层纱布过滤收集, 10×10倍显微镜下调至每视野30~40个孢子为宜。
将三叶一心期的水稻幼苗移至接种箱中(50 cm × 45 cm × 35 cm), 用高压喷雾器将配制好的孢子悬浮液均匀喷施于水稻叶片上。接种后, 在温度26~28℃、相对湿度85%的黑暗条件下保湿 24 h; 再在相同温度及相对湿度75%条件下光照培养 5~7 d后, 参照国际水稻研究所0~9级发病评判标准[35]调查发病情况。
1.4 STS标记和CAPS标记的开发
从水稻基因组计划(Rice Genome Project, RGP)网站(http://rgp.dna.affrc.go.jp/)下载水稻品种日本晴(Nipponbare)参考基因组对应目标区域的克隆序列[36]。利用 Soft Berry网站(http://www.softberry.com/)的FGENESH软件对目标区域序列进行基因预测, 选取注释基因内含子序列作为标记引物设计靶序列。利用 Primer5.0设计了 34对引物, 引物扩增长度在500~1500 bp之间。利用这些引物在亲本间进行多态分析, 表现为共显性扩增多态的引物则作为 STS标记, 对扩增无多态的引物进行随机酶切分析, 酶切呈现多态的引物作为CAPS标记。
1.5 DNA提取、PCR扩增及电泳检测
采用CTAB法提取DNA[34]。将DNA稀释成 20 ng μL–1, 作为 PCR 扩增的模板。PCR 体系 10 μL, 包含 DNA 模板 1 μL (20 ng μL–1)、SSR 引物 1 μL (含正反向引物, 2 pmol μL–1)、10 × buffer (含 Mg2+) 1 μL、dNTP (2.5 mmol L–1) 0.2 μL、TaqDNA 聚合酶0.1 μL (5 U μL–1, 购自宝生物工程(大连)有限公司)、ddH2O 6.7 μL。用美国Thermo公司PCR1000 DNA热循环仪进行扩增反应, 反应程序为 94℃预变性 4 min, 94℃变性 30 s、55℃退火 30 s、72℃延伸 1 min、35个循环, 72℃延伸7 min。扩增产物经8%非变性聚丙烯酰胺凝胶(PAGE)电泳分离, 银染显色, 用WD-9406型胶片观察仪读胶记录基因型数据。
1.6 遗传与物理图谱的构建和候选基因预测
采用隐性群体分析法(RCA)进行遗传图谱的构建, 标记重组率r= (N1+N2/2)/N, 其中N1为各标记带有抗病亲本纯合基因型的个体数,N2为带有双亲杂合基因型的个体数,N为感病群体的总株数。各标记与基因间的遗传距离按每1%的重组率合1 cM的图距计算。最后根据各标记间的遗传距离绘制Pi47基因位点的遗传连锁图谱。
利用遗传图谱构建中连锁标记信息, 在水稻日本晴参考基因组 IRGSP-1.0版本数据库中进行BLAST比对, 确定各连锁标记在参考基因组上的物理位置, 进而绘制Pi47位点的物理图谱。并查找各标记所在BAC克隆, 构建Pi47位点的BAC克隆重叠群。
利用 RGP在线基因预测软件 RiceGAAS(http://ricegaas.dna.affrc.go.jp/), 结合在线数据库GRAMENE、TIGR、NCBI的基因注释, 对精细定位区间进行基因预测。
1.7 序列比对和系统进化树构建
使用Muscle (https://www.ebi.ac.uk/Tools/msa/muscle/)程序比对 DNA和蛋白序列, 使用相关参数默认值。利用Bioedit (http://bioedit.software.informer.com/)软件分析 DNA和蛋白序列相似性。利用MEGA7软件(http://www.megasoftware.net/)构建Neighbor-joining系统进化树, 采用 Bootstrap (Boot strap replications = 5000)方法计算, 采用p-distance氨基酸替换模式。
2 结果与分析
2.1 Pi47单基因系筛选与精细定位群体的构建
采用分别与Pi47连锁的标记 RM206、RM224和 RM5926, 以及与Pi48连锁的标记 RM3102、RM1337和RM7102对CO39/XZ RIL F10群体各株系进行基因型分析, 筛选出Pi47连锁标记 RM206、RM224和RM5926为抗病亲本XZ基因型、Pi48连锁标记 RM3102、RM1337和 RM7102为感病亲本CO39基因型的12个株系, 记为Pi47单基因系(表1)。
为了进一步对Pi47基因精细定位, 将12个单基因系分别与感病亲本CO39杂交并自交构建F2群体,选取种子量较多的 CX18/CO39、CX52/CO39、CX155/CO39与CX279/CO39的F2群体进行接种鉴定。结果表明除 CX155/CO39外, 其他 3个群体抗感比例都接近3∶1, 符合单基因分离规律(表2)。因此, 以这3个群体接种后存活的1687株感病单株作为精细定位作图群体, 其中CX18/CO39群体308株,CX52/CO39群体247株, CX279/CO39群体1132株(表 2)。
2.2 Pi47的精细定位
利用初定位的 2个侧翼标记 RM224、RM5926对CX52/CO39 F2群体中247个感病单株(表2)进行基因型分析, 用RM224鉴定到2个重组子, 重组率为0.4%; 而用RM5926鉴定到不同于RM224的11个重组子, 重组率为 2.23% (表 3)。表明Pi47位于RM224和RM5926之间, 与前述的初定位结果吻合(图 1)。
根据文献报道[15-16], 筛选获得在抗感亲本湘资3150和CO39中表现多态的STS标记K134、K107和CAPS标记K33 (表4); 并根据定位区间参考基因组序列信息, 设计开发了4个新的CAPS标记S4、S10、S11 与 S32 (表 4)。利用 K134 在 CX52/CO39 F2群体247个感病单株中鉴定到来自RM5926的1个重组子, 重组率为 0.2% (表 3), 表明 K134与RM5926位于Pi47同一侧, 而K134距离基因更近。Pi47被进一步定位在RM224和K134之间(图1)。
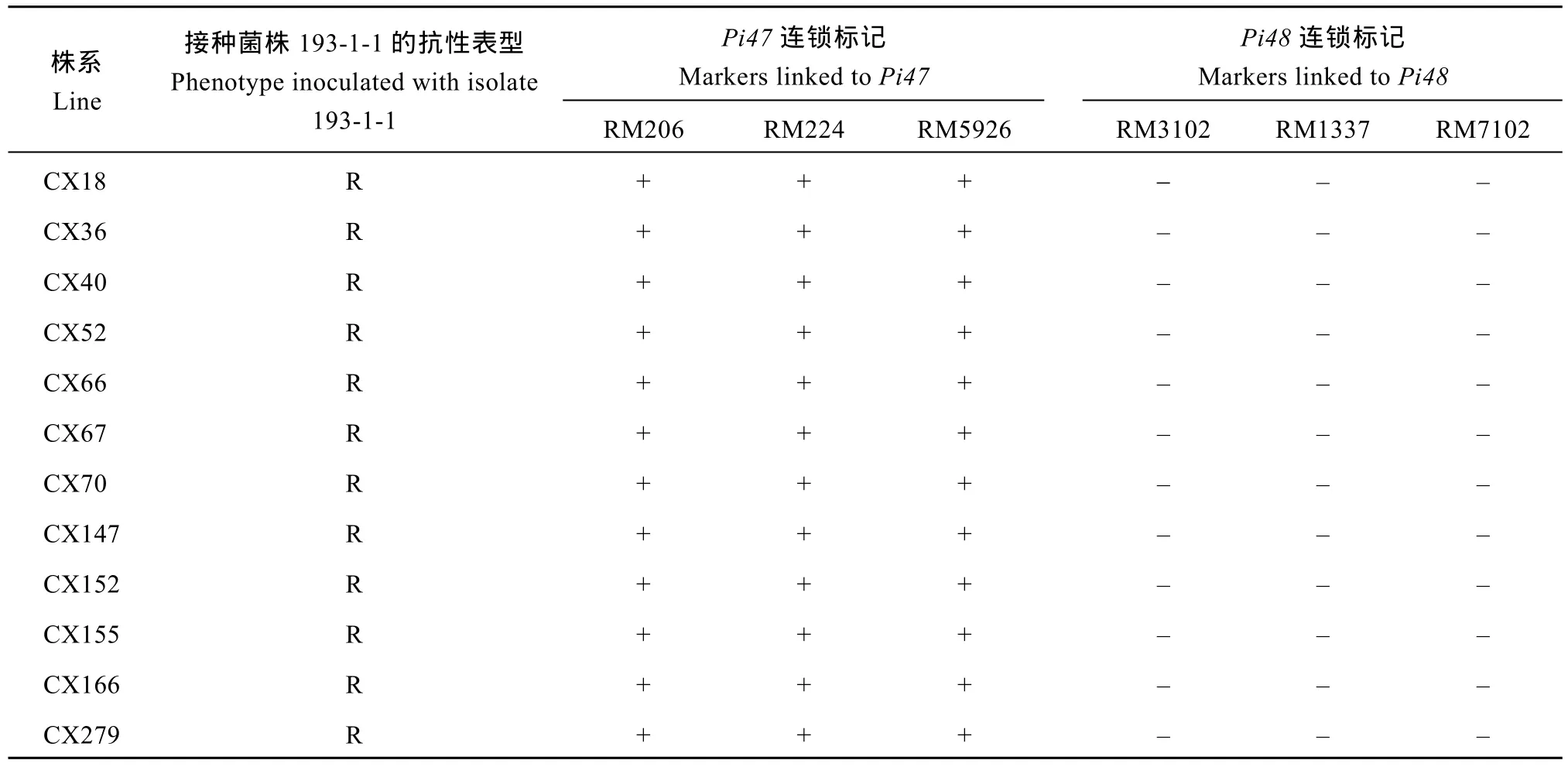
表1 CO39/湘资3150 RIL中Pi47单基因系的鉴定Table 1 Screening of Pi47 monogenic lines in CO39/Xiangzi 3150 RILs
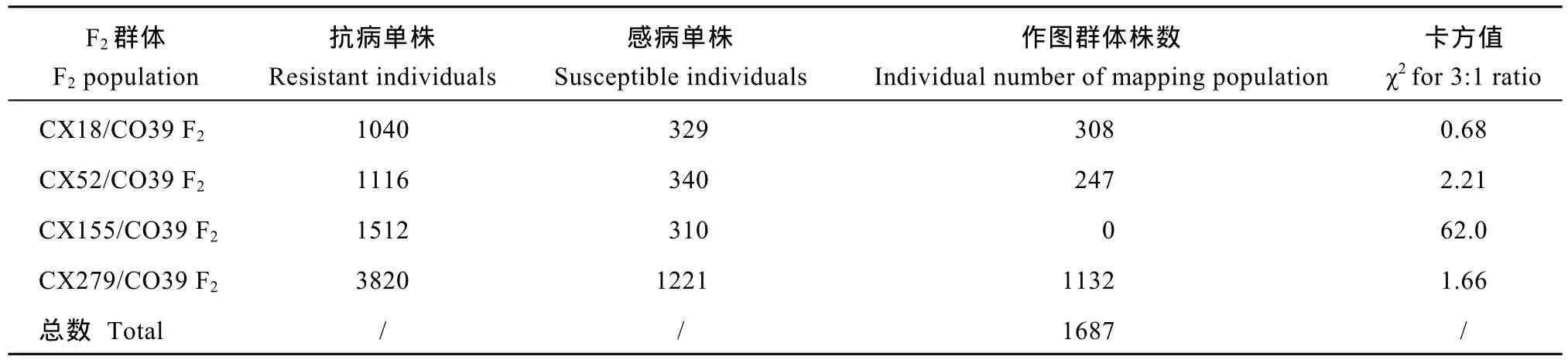
表2 各F2群体抗稻瘟病表型分离分析Table 2 Segregation analysis of resistance to rice blast for each F2 population

表3 所用标记在3个F2群体中鉴定到的重组子数量Table 3 Number of recombinants identified with molecular marker in the three F2 populations
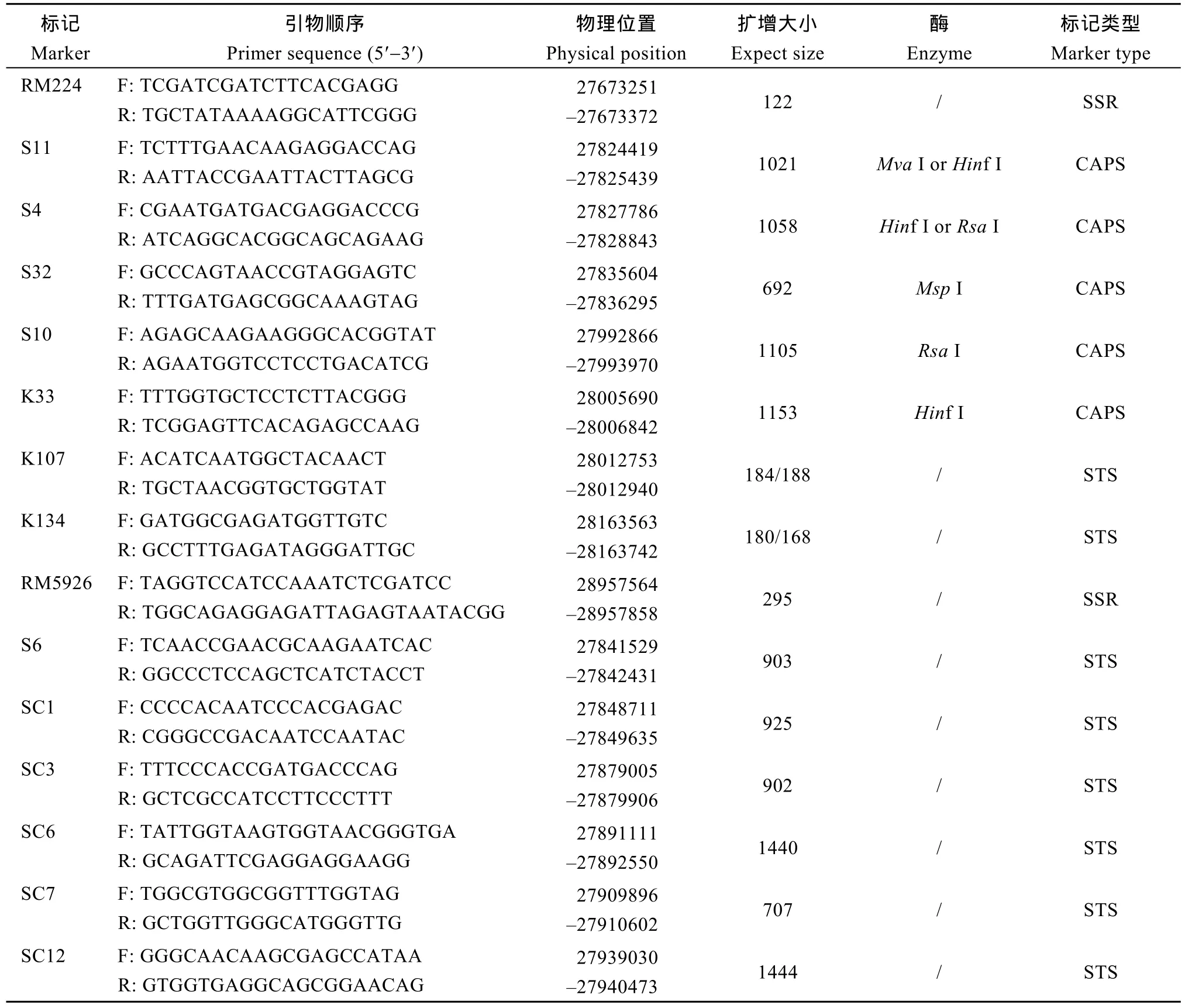
表4 Pi47精细定位使用的标记信息Table 4 Molecular markers used for Pi47 fine mapping
进一步利用RM224和K134对CX18/CO39 F2与CX279/CO39 F2群体总共1440个感病单株进行分析, 以RM224分别鉴定到来自2个群体的5个和20个重组子, 以K134分别鉴定到 1个和 8个重组子(表 3)。因此, 以 RM224和 K134在 3个 F2感病群体总共1687个感病单株中分别鉴定到27个和10个重组子, 对应的重组率分别为0.8%和0.3%, 即与Pi47的遗传距离分别为0.8 cM和0.3 cM (图1)。
再利用 K33、K107、S4、S11和 S32标记对 3个群体 1687个感病单株进行分析, 结果用 K33和K107鉴定到来自K134位点的1个重组子, 用S4、S11和S32鉴定到来自RM224位点相同的7个重组子, 而用S10未鉴定到重组(表3), 表明RM224、S4、S11和S32位于Pi47基因的同一侧, K33、K107和K134位于基因的另一侧, 与目标基因的遗传距离分别为0.21 cM和0.03 cM, 而S10则与目标基因共分离。因此,Pi47被精细定位在S32和K33之间0.24 cM的区间内(图1)。
2.3 遗传图谱与物理图谱的整合及候选基因区间的确定
根据精细定位各标记与Pi47之间的遗传距离,构建了Pi47位点区域的标记连锁图谱(图 1-A)。为了明确Pi47位点在水稻Nipponbare参考基因组中的物理图谱, 利用各标记在参考基因组中的位置信息,获得 5个 BAC重叠群 OSJNBa0064H09、OsJNBa0085H07、OsJNBb0049B20、OsJNBb0047M04、OsJNBa0036K13, 覆盖了Pi47基因位点 RM224至K134之间约 490 kb的物理图谱, 其中重叠群OsJNBb0047M04和OsJNBa0036K13之间存在1个间隙(图 1-C)。Pi47基因被精细定位在 S32和 K33之间 0.24 cM 的遗传距离区间, 对应的物理距离约为 171.2 kb (图 1-D)。

图1 Pi47位点区域遗传图谱与物理图谱的构建Fig. 1 Genetic and physical map construction of Pi47 locus
为了进一步缩小Pi47候选基因区间, 在S32和K33之间开发了6个在抗病亲本湘资3150和感病亲本CO39中表现多态的分子标记, 即S6、SC1、SC3、SC6、SC7和SC12 (表3)。基因型分析结果表明,Pi47单基因系CX279、CX18和CX52上述6个标记座位均表现为感病亲本 CO39的基因型(图 1-E), 表明 3个单基因系中S6-SC12间的基因组片段来自感病亲本CO39, 该区间对应参考基因组物理距离约为97.5 kb (图1-E)。利用标记S10、K33分析3个单基因系基因型, 发现 2个标记位点均表现出与抗病亲本湘资3150相同的基因型(图1-E)。综合上述试验结果,推测Pi47被进一步限定在SC12和K33之间约67.8 kb的区域内(图1-E)。
2.4 候选基因的预测与克隆
根据上述精细定位的结果, 以 Nipponbare参考基因组IRGSP-1.0版本为参照, 对标记SC12和K33间67.8 kb的DNA序列进行了基因预测和注释。结果表明, 该区间编码 8个结构基因(表 5)。其中,LOC_Os11g46180、LOC_Os11g46190和LOC_Os11g462403个基因编码转座子或逆转座子蛋白;LOC_Os11g46150和LOC_Os11g46220编码功能未知的表达蛋白或假想蛋白;LOC_Os11g46200和LOC_Os11g46210则编码 NBS-LRR抗病类似蛋白,因此将后两者作为Pi47的候选功能基因(表5)。

表5 Pi47位点标记SC12和K33间参考基因组区域内候选基因预测Table 5 Candidate gene prediction between SC12 and K33 in Pi47 locus
利用同源基因 PCR克隆法, 根据LOC_Os11g46200和LOC_Os11g46210这2个基因参考基因组序列设计同源PCR引物, 对湘资3150中同源基因进行了 PCR扩增和测序。进一步利用湘资 3150中同源基因序列的测序结果在NCBI DNA序列数据中比对, 发现Pi47的2个候选基因中,Pi47-1(对应LOC_Os11g46200)与已克隆的Pik位点等位基因Pik1-KA、Pi7-1、Piks-1、Pik-2、Pikp-1、Pikm1-TS在DNA序列上的相似度在97.5%~99.8%之间, 在蛋白序列上的相似度达94.6%~99.5% (表6);Pi47-2(对应LOC_Os11g46210)与对应等位基因Pik2-KA、Pi7-2、Piks-2、Pik-1、Pikp-2、Pikm2-TS在DNA和蛋白水平上的相似度分别为 99.8%~99.9%和99.6%~99.9% (表 7)。而Pi47-1、Pi47-2与Pi1的 2个基因Pi1-5、Pi1-6在DNA水平上的相似度分别为 99.7%和 100%, 蛋白水平上的相似度也分别达到99.5%和100% (表6和表 7)。利用全长蛋白序列构建进化树结果表明Pi47与Pi1明显地聚集到一起(图2)。综上说明,Pi47与Pi1可能是同一个基因。
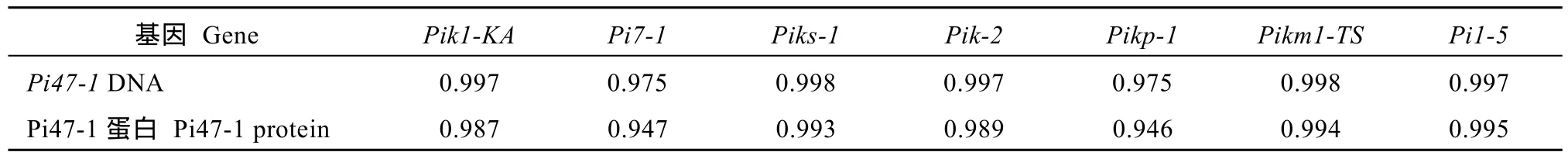
表6 Pi47-1与Pik位点对应的已克隆基因在DNA和蛋白水平上的序列相似性Table 6 Sequence similarity of cloned genes at the DNA and protein levels between Pi47-1 and Pik sites

表7 Pi47-2与Pik位点对应的已克隆基因在DNA和蛋白水平上的序列相似性Table 7 Sequence similarity of cloned genes at the DNA and protein levels between Pi47-2 and Pik sites
2.5 Pi47与其等位基因的抗谱比较
在Pi47定位区间 RM224~RM5926区段, 已有Pikm[37]、Pi46[38]、Pik[39]、Pikp[40]、Pikh[41]5 个抗病基因被精细定位或者克隆, 表明Pi47可能为这些基因的等位基因。为进一步明确Pi47与上述基因的关系和抗谱的差异, 利用来自不同地区的 17个稻瘟病菌小种, 对Pi47基因的单基因系CX18和Pik、Pikm、Pikh及Pikp4个基因 CO39背景的近等基因系(NIL)[42]进行接种鉴定。结果发现, 6份材料均对日本小种 KOH表现高感,Pik与Pikp对来自福建的小种RB18表现感病, 而Pi47、Pikm和Pikh对其表现抗性。Pi47和Pikp对来自湖南的小种195-2-2表现感病, 而其余3个基因对该小种表现抗性(表8), 表明Pi47与这4个基因抗谱不同。
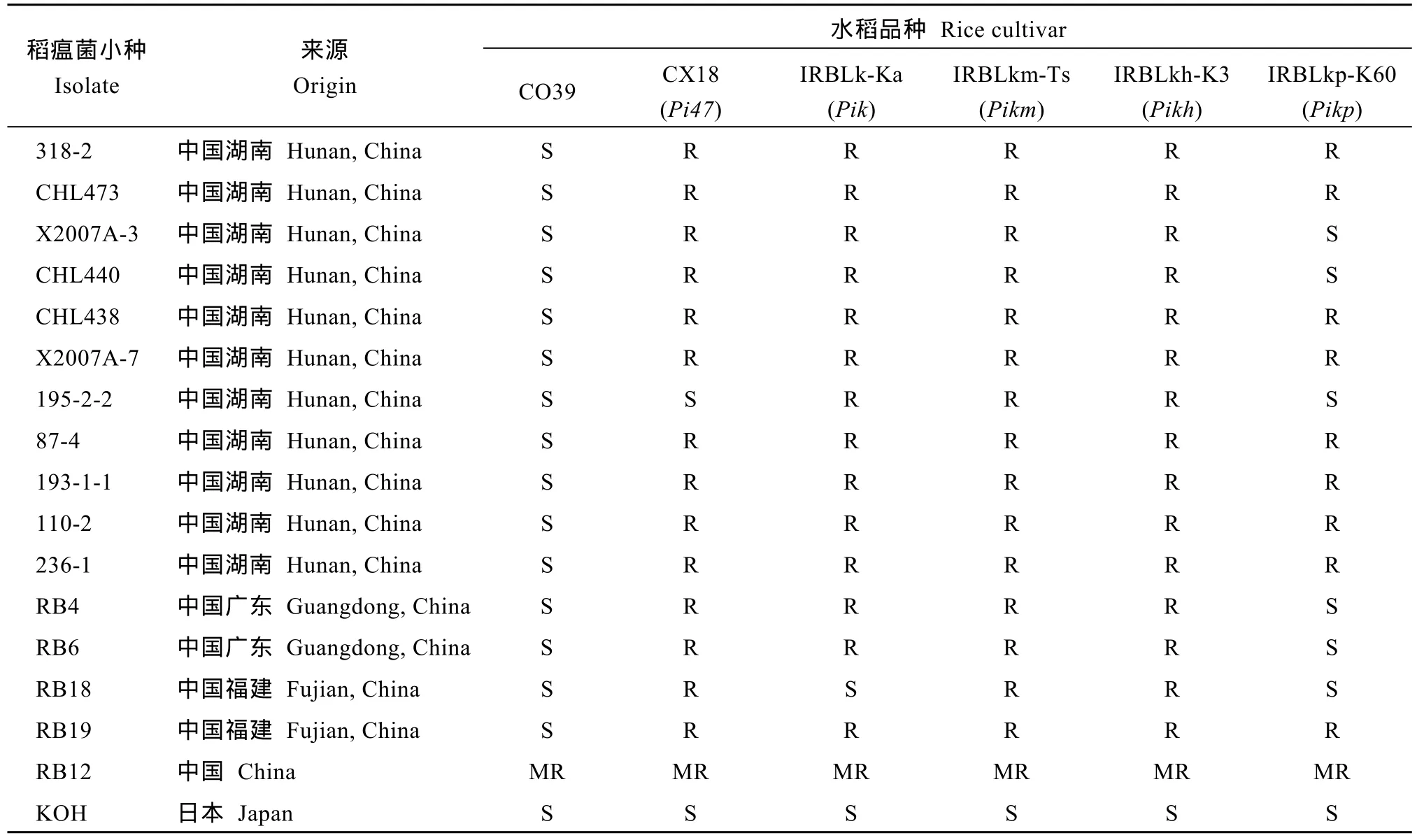
表8 Pi47及其附近位置抗稻瘟病基因的抗谱分析Table 8 Resistance spectrum analysis of Pi47 and the nearby genes

图2 Pi47与Pik位点已克隆基因的系统进化分析Fig. 2 Phylogenetic analysis of Pi47 and cloned genes at Pik locus
3 讨论
稻瘟病是水稻生产中危害最严重的真菌病害之一。由于稻瘟菌自然群体的快速变异导致致病性不断改变的特点, 利用单一的抗病基因很难从根本上持久解决稻瘟病防治的问题, 必须不断挖掘和克隆新的抗病基因, 以适应稻瘟菌群致病性的不断变化。本实验室在前期研究中从世界各地收集的种质资源中鉴定了多份广谱、持久高抗稻瘟病的新种质,并通过构建 F2和重组自交系群体对其中湘资3150[43]、天津野生稻[44]、魔王谷[45]、龙 S[46]和Jefferson[47]5份种质中的抗病基因进行了定位。Huang等[32]利用重组自交系群体从湘资3150中初定位了分别位于水稻第11、第12染色体上的2个抗稻瘟病基因Pi47和Pi48, 本研究进一步对Pi47基因进行了精细定位和候选基因的克隆。
从重组自交系群体筛选的4个Pi47单基因系构建的F2分离群体用于精细定位(表2)表明, 3个群体(CX18/CO39、CX52/CO39与 CX279/CO39)抗感比例符合 3∶1单基因分离规律(表 2), 可以用于进一步的精细定位。而 CX155/CO39F2群体抗感分离不符合单基因分离规律(表2), 不适用于下一步精细定位; 造成这个结果的原因可能一是接种条件不适导致发病不充分, 二是该株系实际可能不是单基因系,因为用于鉴定单基因系的连锁标记与目的基因还有较大的遗传距离, 遗传重组导致鉴定不准确。
在利用3个分离群体中1687个感病单株对Pi47进行精细定位的过程中, 我们在目标区段内筛选了56个SSR标记才得到1个多态标记, 表明抗病亲本湘资3150和感病亲本CO39在目标区段的多态性极低, 这可能是群体双亲都为籼稻导致基因组序列差异小。我们进一步在该区段开发了11个新标记, 包括4个 CAPS标记(S4、S10、S11和 S32)和 7个STS标记(S6、SC1、SC12、SC3、SC6、SC7和 SC12)(表3和图 1), 最终将Pi47基因精细定位于标记 SC12和K33之间约67.8 kb的区域内(图1)。这些标记不仅可用于Pi47基因的分子标记辅助选择育种, 还可以用于其他相关的水稻种质资源基因型鉴定、基因定位和克隆。
通过比对日本晴参考基因组在Pi47精细定位区间的基因组信息, 构建了Pi47基因在 RM224和K134标记间约490 kb的BAC克隆重叠群(图1), 并对SC12和K33间67.8 kb区间内日本晴基因组序列进行了注释, 发现该区间编码8个结构基因, 其中2个编码 NBS-LRR抗病蛋白类似基因(表 5), 可能为Pi47的候选基因。利用同源PCR克隆, 测定了抗病亲本湘资3150中这2个NBS-LRR基因的序列, 比对和注释发现这2个基因是由同一个启动子驱动的方向相反的串联基因。进一步对数据库比对发现,Pi47两个候选基因(Pi47-1和Pi47-2)与已鉴定的Pik位点多个等位基因在 DNA序列和蛋白序列上相似度非常高(表6、表7和图2)。同时, 发现Pi47-2与Pi1-6基因在 DNA和蛋白序列上相似度达到 100%(表7), 而Pi47-1与Pi1-5在DNA和蛋白序列上相似度也达到了99.7%和99.5% (表6), 且Pi47和Pi1编码蛋白在系统进化树中聚集在一起(图 2), 表明Pi47与Pi1可能为同一功能基因。此前研究表明Pi1只在来自西非利比里亚的 LAC23及其衍生品种C101LAC和 IRBL1-CL、以及来自东南亚国家越南的 Tetep等极少数籼稻品种中存在[16]。本研究克隆的Pi47基因供体材料湘资3150是中国湖南的一个地方品种, 这为拓展Pi1基因的起源进化提供了重要的材料。然而, 在地理位置相距甚远的 3个栽培稻品种中均具有同一个抗病基因,Pi47/Pi1基因在自然选择和人工培育过程中的演化进程有待进一步研究。
目前定位和克隆的抗稻瘟病基因中, 多数抗病基因往往成簇分布在少数几个染色体位点[48], 如第6染色体靠近着丝粒附近的Pi2/Pi9位点, 至少已发现了10个具有广谱抗性的等位基因[48-49]。本研究中Pi47所在的第11染色体长臂末端Pik位点也定位了至少 9个等位基因, 如Pik、Pikh、Piks、Pikm、Pikp、Pi1、Pi7、Pi47、Pi49等, 其中Pik、Pikh、Pikm、Pikp、Pi1等 5个基因已被克隆[8-11,16]。我们比较了Pi47单基因系与Pik、Pikm、Pikh、Pikp4个基因近等基因系的抗谱, 发现Pi47与这4个基因的抗谱有差异(表8)。这些不同抗谱的等位基因是在对抗不断变异的稻瘟菌的过程中进化而成, 如Pik对应的无毒基因Avr-Pik中鉴定出5种变异类型Avr-Pik-A、B、C、D和E, 可由Pik位点的5个等位基因Pik、Pikp、Pikm、Piks和Pikh分别识别[50]。这些抗御不同无毒基因的抗病等位基因, 为不同地区利用抗性基因培育抗病品种提供了重要资源。本研究发掘的Pik位点的等位基因Pi47,在湖南地区连续多年的病圃鉴定中表现出高水平持久抗性, 可望作为重要的抗性基因应用于抗病育种。此外, 我们在携带Pi47基因的水稻品种湘资3150中还定位了Pi48基因[34], 2个基因Pi47和Pi48在湘资3150广谱、持久抗性中的作用机制有待进一步研究。
4 结论
采用3个Pi47单基因系与感病亲本CO39构建的F2群体的1687个感病单株, 将Pi47精细定位在S32与K33间0.24 cM区域的171.2 kb物理区间内,背景分析进一步缩小至SC12和K33间67.8 kb的区间内。该区间编码 8个结构基因, 其中 2个编码NBS-LRR抗病类似蛋白基因, 为Pi47的候选功能基因。Pi47可能与Pi1为同一基因。Pi47与Pik的4个等位基因Pik、Pikm、Pikh及Pikp的抗谱不同。

