利用SNP芯片构建我国冬油菜参试品种DNA指纹图谱
赵仁欣 李森业 郭瑞星 曾新华 文 静 马朝芝 沈金雄涂金星 傅廷栋 易 斌,*
1华中农业大学植物科学技术学院 / 作物遗传改良国家重点实验室, 湖北武汉 430000; 2中国农业科学院油料作物研究所, 湖北武汉430062
目前可在中国种业网的大数据平台(http://139.129.194.173:9100/home/ManageOrg)上查询到的油菜保护品种有634个, 其中登记品种有185个, 往年国家冬油菜的区试品种约有 180个。油菜种质基础的狭小导致育成的新品种间的遗传差异越来越小[1],而如今油菜品种数目越来越多, 如何有效管理和鉴别成为油菜品种管理与市场化的首要难题。2017年5月1日《非主要农作物品种登记办法》颁布实施, 油菜被列入第一批非主要农作物登记目录, 油菜由审定品种的事前监管变成了登记品种的事中事后监管[2],只要符合DUS基本条件, 经试验确定品种特征特性和适宜推广范围, 且不存在严重安全问题的油菜品种皆可登记, 由此更将增大油菜品种市场化规范管理的难度。开发基于SNP标记的、满足国家或行业标准的、能为政府品种管理部门提供标准样品的甘蓝型油菜DNA指纹图谱显得十分重要。
SNP标记作为第三代分子标记, 不仅具有数量多、分布广、分型简单、适用于大规模试验及等位基因频率易估计等优势[3], 而且能与DNA芯片技术相结合进行高通量的检测分析, 使试验结果更加准确可靠[4]。SNP标记已广泛运用于种质资源的遗传多样性、类群划分及品种鉴定的研究, 史亚兴等[5]用1059个SNP标记将39份不同基因型的糯玉米自交系划分五大类群, 明确了其亲缘关系; Singh等[6]采用SC基因50K水稻SNP芯片, 研究了包括野生稻、传统水稻品种和遗传改良水稻品种在内的 128种不同种质资源间的遗传多样性和系统发育关系;Zhao等[7]用44 100个SNP标记对82个国家的413份不同的水稻品种进行基因分型, 将其按连续两季的表型、生长情况、农艺性状等系统性地划分为34类; 侯莉娟等[8]对获得的 41 843个 SNP标记建立HRM基因分型方法将羊草的48份种质区分开并绘制了相应的指纹图谱; 北京市农林科学院玉米研究中心利用Maize SNP384 芯片完成4632份审定玉米品种的SNP指纹库构建[9]; Chen等[10]利用Rice 6K芯片对从东北市场收集的19份可能为“稻花香”的品种进行基因指纹检测, 发现有6个品种与稻花香2号差异较大, 13个品种为稻花香2号或其近似品种。众多研究都表明, 利用SNP标记构建甘蓝型油菜新品种 DNA指纹图谱是可行且有效的, 本研究利用Illumina公司联合世界16家科研单位发行的含有52 157个SNP位点的油菜60K SNP芯片[11]对2016—2017年度国家冬油菜区试参试品种(系)做分析鉴定,以期构建我国冬油菜参试品种DNA指纹图谱, 为甘蓝型油菜品种 DNA指纹数据库的建立奠定良好的基础。
1 材料与方法
1.1 油菜品种
参试材料为2016—2017年度国家冬油菜参试品种187份, 包含续试品种33份(表1)。所用实验材料皆由中国农业科学院油料作物研究所2016年12月提供。其中, 随机选取8个品种华油杂29、圣光158、秦优 1618、陕油 1609、阳光 161、蓉油 29、中油153、杂优 15进行 2次重复点样, 对照进行实验误差分析, 即2016—2017年度的冬油菜试验品种191份材料, 2015—2016年度的冬油菜试验品种33份材料, 共224份样品同时进行DNA提取。
1.2 实验方法
1.2.1 DNA的提取 使用新型植物基因组 DNA提取试剂盒(离心柱型, TIANGEN DP320)进行DNA提取, 取油菜的幼嫩子叶 300~500 mg, 用液氮急冻后充分研磨, 按照试剂盒说明书的操作步骤, 提取油菜基因组DNA, 置于-20℃保存备用。
1.2.2 DNA凝胶电泳检测 用1%的琼脂糖凝胶电泳对提取的DNA进行检测, 每孔点样10 μL, 200 V电压电泳15 min左右, 取出胶体在凝胶成像仪上照相留存。琼脂糖电泳须有大于 10 kb的明亮单一条带, 对主带不明显或者没有主带的样品重新取样提取DNA。
1.2.3 DNA浓度测定 用NanoDrop 2000测定提取的基因组 DNA质量, 确保基因组总 DNA的A260/A280值在 1.8~2.0之间, A260/A230值在 1.8~2.2之间, 稀释 DNA 浓度至 50 ng μL–1。
1.2.4 油菜60K Illumina Infinium SNP芯片基因分型 使用高密度的油菜 60K Illumina Infinium SNP芯片进行基因分型分析, 严格按照芯片操作手册的步骤要求进行DNA样品预处理(扩增、片段化、富集、回收、变性)、DNA样品与芯片杂交、洗脱、单碱基延伸、染色、洗涤、包埋。然后利用Illumina公司的芯片扫描仪HiScan扫描分型。
1.2.5 数据处理与分析 用 Genome Studio genotyping software v2011软件对扫描所得原始数据进行分析、筛选。使用Center for System Genomics
在线网址(http://visualization.ritchielab.psu.edu/home/index)绘制所选 SNP标记在油菜全基因组的位置分布图。用PowerMarker V3.25软件分析筛选的SNP标记的遗传多态性信息指数(PIC)、最小基因频率(minor allele frequency, MAF), 并计算Nei遗传距离,结合MEGA软件绘制NJ聚类图。

表1 2016–2017我国冬油菜试验品种名称和编号Table 1 2016–2017 winter-type rapeseed varieties test name and number
(续表 1)
2 结果与分析
2.1 筛选SNP标记与构建指纹图谱
油菜60K芯片共含有52 157个SNP标记位点,均匀分布在油菜全基因组中。使用 Genome Studio genotyping software v2011软件进行原始数据处理,224份样品中 Call Rate值大于 0.80的样品数量占92%。剔除AA或BB基因型频率为0、最小基因型频率(minor freq)小于0.05、SNP得率(call freq)小于0.80、缺失率大于5%的SNP标记后, 得到7457个SNP位点。最后根据分型图手动剔除分型离散杂乱、3种基因型分型边界不明显的(图1-A)或杂合群与纯合群相互重叠的(图1-B)及出现偏移的(图1-C) SNP标记, 选取出分型清晰、分型质量高的(图1-D) SNP标记 5374个。用 PowerMarker V3.25软件计算得,5374个 SNP标记的 PIC均值为 0.27, 变化范围是0.09~0.41, 其中A基因组PIC均值为0.26, C基因组PIC均值为0.27, 略高于A基因组, PIC值大于0.25的占55.94%。
将筛选出的5374个SNP标记进行连锁群比对,有5143个SNP标记比对到了A1~A10、C1~C9连锁群上, 使用Center for System Genomics的在线网址绘制SNP位点物理位置分布图可见, 所选SNP标记比较全面地覆盖了油菜的基因组(图2)。
利用5374个核心SNP标记组合, 以标记名称为前缀, 品种在该标记处芯片分型所得的碱基类型为后缀, 得到品种该标记的特征编号。固定标记顺序,分别串联每个品种的标记特征编号, 构建 191份我国冬油菜参试品种的特征 DNA指纹图谱(本文未列出)。
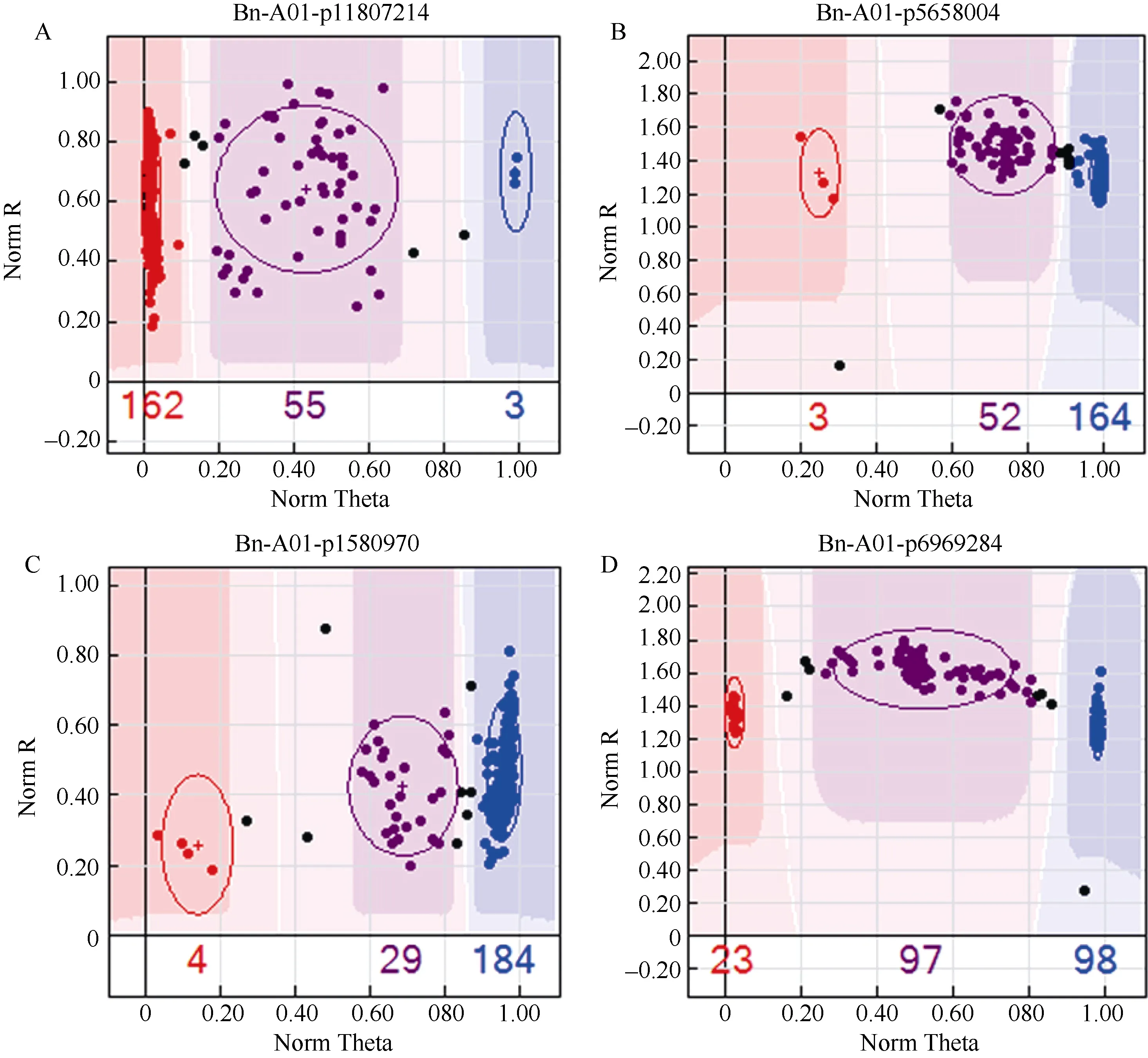
图1 基于Genome Studio分析的不同类型的AA、AB、BB基因型的分型图Fig. 1 Different patterns of clustering of AA, AB, and BB genotypes based on Genome Studio analysisA: 3种基因型分型边界不明显; B: 杂合群与纯合群相互重叠; C: 3种基因型向一侧偏移; D: 3种基因型分型界限明显。A: SNPs did not show three clearly defined clusters; B: AB cluster overlapped with AA/BB cluster; C: the genotype cluster intensities are shifted to one side of the theta space clusters shift; D: SNPs show three clearly defined clusters.
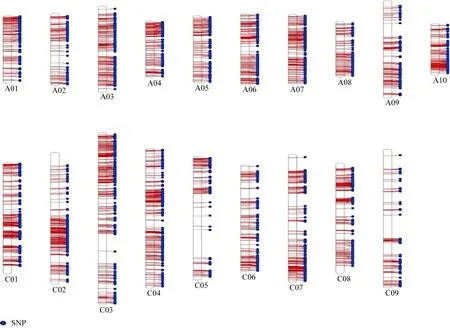
图2 5143个SNP标记在油菜基因组的位置分布图Fig. 2 Genome-wide distribution of 5143 SNPs in physical map
2.2 误差分析
随机选取 8个品种华油杂 29、圣光 158、秦优 1618、陕油 1609、阳光 161、蓉油 29、中油 153和杂优 15进行 2次重复点样对照实验, 利用Power Marker V3.25软件计算其Nei遗传距离, 取8组对照的平均值作为本次实验的技术误差, 如表2所示, 得出Infinium芯片的技术误差为0.36%,与Illumina公司公布的0.1%的技术误差十分接近,误差分析结果表明, 本次研究的基因分型结果准确可靠。
2.3 品种间遗传相似系数分析
用Power Marker V3.25软件计算191份甘蓝型油菜材料的Nei遗传距离获取到18 145个遗传相似系数, 变幅为52.17% (大地192和黔油37) ~99.98%(华油杂12和华油杂12, 不同参试小区的同一品种),平均遗传相似系数为76.20%。绘制18 145个遗传相似系数的正态分布图(图 3)可知, 相似系数值在90%~93%区间的有 16个, 在 93%~95%区间没有,在 95%~100%区间的有 22个(其中 2个来自不同品种间, 20个来自同一品种的对照实验)。可见, 在《油菜品种区域试验技术规程》中规定的SSR标记的品种特异性的鉴定阈值93%并不适合于SNP标记的品种特异性鉴定, 而农业部颁布的《水稻品种鉴定SNP标记法(NY/T 2745-2015)》中是以品种间遗传相似系数 95%作为阈值, 即品种间遗传相似系数≤95%判定为“不同品种”, 品种间遗传相似系数>95%判定为“近似品种或疑同品种”, 故在本研究中也以遗传相似系数 95%作为品种特异性及一致性鉴定的阈值。
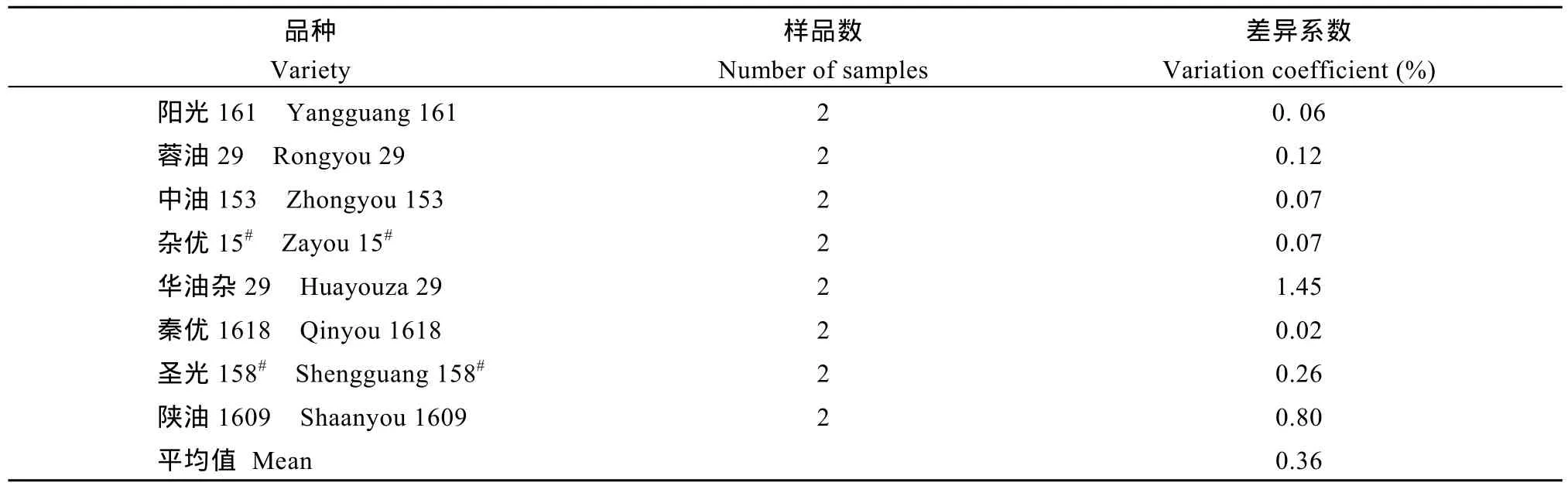
表2 8组点样对照品种的差异系数Table 2 Variation coefficient of eight groups of varieties with two replicates
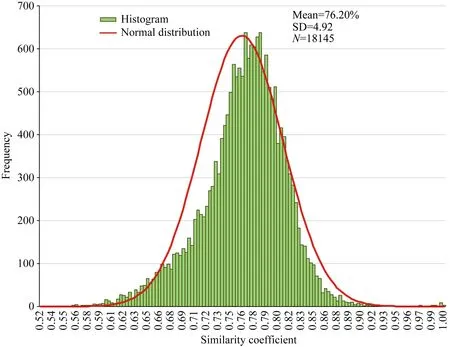
图3 2016–2017年度国家冬油菜参试品种相似系数分布Fig. 3 Distribution of genetic similarity coefficients for the 2016–2017 national winter-type rapeseed variety trial
2.4 2016—2017年度国家冬油菜区试品种(系)特异性分析
利用筛选的 5374个 SNP标记分析 187份2016—2017年度国家冬油菜参试品种(系)共 191份材料的遗传多样性, 绘制 NJ聚类图(图 4)。由图 4可以看出, 2016—2017年度国家冬油菜区试品种(系)的大部分品种的遗传相似系数差异明显, 且同一品种都能聚到一起, 误差小, 可信度高; 以相似系数95%为阈值(以红线标注), 191份材料中有2对品种的相似系数大于95%, 分别为秦荣5号和秦优1699(96.76%)、湘杂油373和湘油422 (98.12%), 此4个品种特异性低, 可判定为疑似近似品种, 其余品种间相似系数均小于 95%, 可判定为不同品种。总体而言, 2016—2017年度国家冬油菜参试品种(系)具有较高的特异性。
2.5 2016—2017年度国家冬油菜续试品种(系)一致性分析
将续试品种的种子与前一年送检的种子同时进行DNA提取, 计算品种年度间的相似系数, 检测品种是否存在替换现象。2016—2017年度国家冬油菜参试品种中包括 33份续试品种, 利用筛选获得的5374个SNP标记对33份续试品种共66份材料进行聚类分析(图5), 大多数品种的2年材料都聚到了一起, 且不同品种间差异度大。33个品种中有4个品种的相似系数小于 95% (图 5中用红色方框圈出),分别为德徽油 88、浙油杂 319、华油杂 701、浙油杂315, 这4个品种一致性低, 可判定为稳定性较差或疑似更换品种, 其余品种间相似系数均大于 95%,一致性高, 稳定性好。
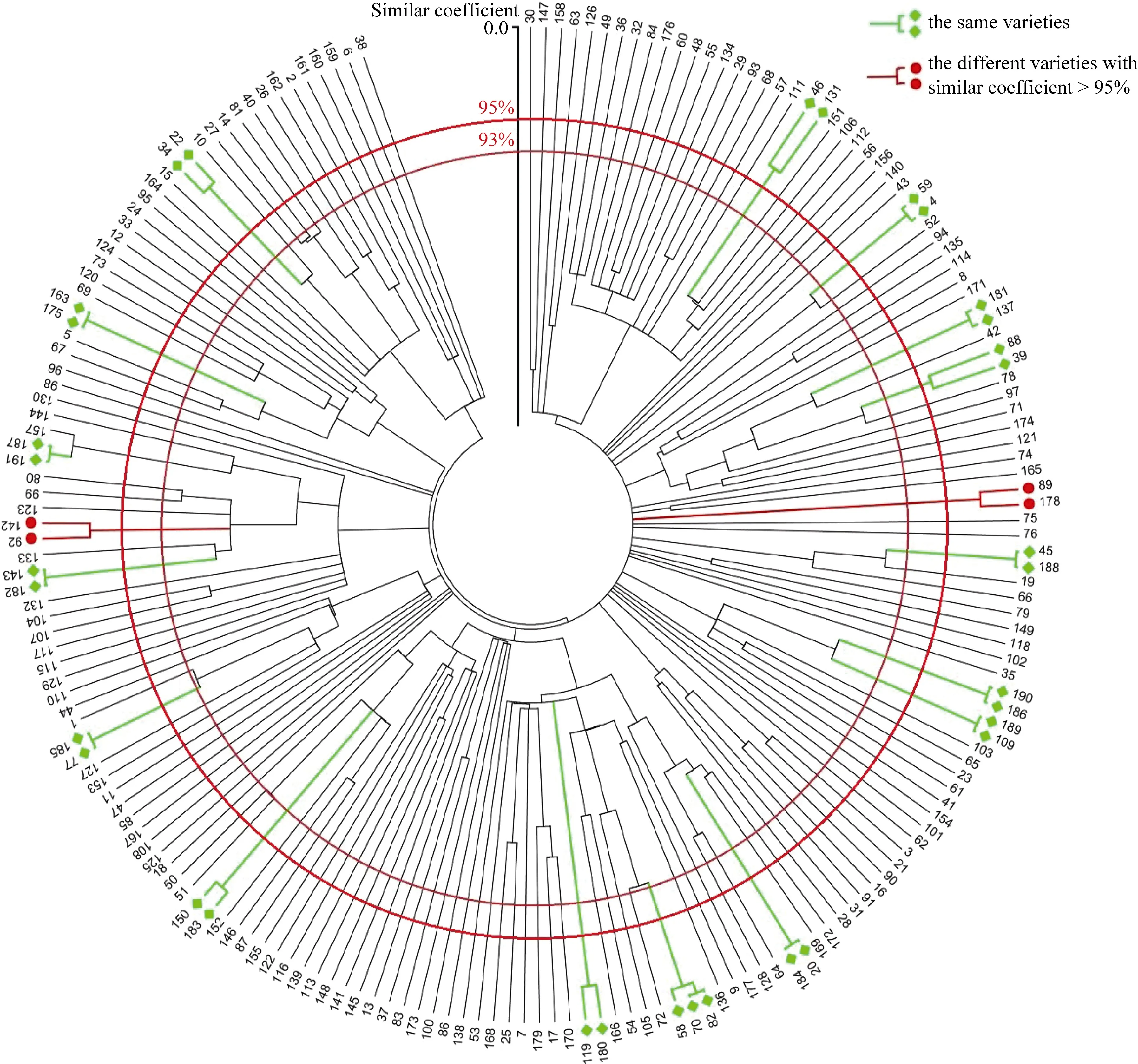
图4 2016–2017年度国家冬油菜区试品种聚类分析结果Fig. 4 Cluster analysis of varieties participated in the 2016–2017 national winter-type rapeseed variety trial编号对应的品种名称见表1。Numbers for varieties correspond with those given in Table 1.
3 讨论
SNP标记是最具发展潜力的建库标记之一, 其通常为二等位变异位点, 在基因组中密度高且分布均匀[12], 易于筛选一套高质量的核心SNP位点组合,实现数据间整合比较[13]。本研究借助于 Illumina公司推出的高通量的SNP芯片分型平台, 一次可检测52 157个SNP标记位点, 不仅可快速、高通量地检测油菜品种的遗传多样性, 而且具有良好的重现性[14]。
为满足构建甘蓝型油菜品种 DNA指纹图谱的要求, 需从52 157个SNP标记位点中, 挑选一套高质量的核心SNP标记。结合国内外对于油菜60K芯片SNP标记的筛选要求[15-19], 本研究剔除AA或BB基因型频率为0的SNP标记及最小基因型频率小于0.05, SNP得率小于0.80的SNP标记; 保留缺失率小于 5%的 SNP标记; 选择分型图分型清晰且分型边界明显的SNP标记。最终筛选出5374个覆盖油菜全基因组的SNP标记, 使用PowerMarker V3.25软件计算出所选SNP标记的PIC均值为0.27, 变化范围是0.09~0.41, PIC值大于0.25的占55.94%, 其中A基因组PIC均值为0.26, C基因组PIC均值为0.27,略高于 A基因组, 与 Delourme等[20]的研究结果一致。因此, 此套SNP标记具有很高的多态性和可信度, 可用于油菜品种的指纹图谱构建与鉴定分析。
本研究随机选取8个品种做2次重复点样对照,得出Infinium芯片的技术误差为0.36%, 与Illumina公司公布的 0.1%的技术误差十分接近, 证明了本次芯片实验用于基因分型的精准性。191份甘蓝型油菜材料的 18 145个遗传相似系数的变幅为 52.17%~99.98%, 平均遗传相似系数为 76.20%, 相似系数值在90%~93%区间的有16个, 在93%~95%区间没有,在 95%~100%区间的有 22个(其中 2个来自不同品种间, 20个来自同一品种的对照实验)。参照国家有关标准, 本研究将遗传相似系数 95%作为品种特异性及一致性鉴定的阈值。
聚类分析可见, 大部分品种的遗传相似性差别明显, 具有很高的区分度, 且相同品种都能聚到一处, 证明利用筛选的5374个SNP标记对油菜品种进行特异性、一致性分析是可行的, 可用于构建我国冬油菜参试品种 DNA指纹图谱, 也表明 2016—2017年度我国冬油菜参试品种种质资源丰富、品种间遗传差异度大, 年度间一致性高。
利用 SNP标记可以为甘蓝型油菜品种构建DNA指纹图谱, 从而定制品种的基因指纹, 使每个品种都有唯一的指纹信息, 可为种子监管部门打击假劣种子、准确鉴定油菜新品种提供有效的技术手段, 进而可保护育种家的知识产权, 达到规范市场行为的目的[21]。
4 结论
本研究筛选了5374个可用于构建甘蓝型油菜品种 DNA指纹图谱的 SNP标记, 并用其构建了2016—2017年度的我国冬油菜品种的 DNA 指纹图谱和进行了品种间特异性与品种年度间一致性分析,在后续的研究中我们将着重进行标准品种及SNP标记的筛选, 规范试验操作程序、软件分析流程、数据库系统开发管理, 构建以SNP标记为基础的甘蓝型油菜品种DNA指纹数据库[22-23]。

