氨基酸产业现状及生产菌株选育技术
,,
(1.中国生物发酵产业协会,北京 100833;2.莲花健康产业集团股份有限公司 博士后科研工作站,河南 项城 466200;3.天津科技大学 生物工程学院,天津 300457)
氨基酸是蛋白质合成的原料,同时其自身及其代谢产物调节机体内许多重要的生命活动,如参与脂代谢、糖代谢等,为机体提供能量,维持机体内环境的稳态,合成激素和神经递质等[1].自Udeka和Kinoshita于1957年发现谷氨酸棒杆菌天然可以分泌谷氨酸以来[2],科学家们通过诱变育种获得了一系列氨基酸生产菌株[3].近年来通过基因工程理性设计极大的加速了育种进程和扩展了可发酵生产的氨基酸品种[4].目前,大多数氨基酸通过发酵法生产,氨基酸发酵产业已经成为整个发酵行业的支柱产业之一[5].
1 我国氨基酸产业现状
我国2017年氨基酸行业总体运行情况良好,主导产品谷氨酸、赖氨酸、苏氨酸等生产状态基本稳定,其他品种氨基酸产量略有增长.据统计,2017年我国氨基酸发酵产品总产量约为542万吨,同比增长17.87%[6](表1).
由此可见,目前我国氨基酸产能已经过剩,尤其大宗氨基酸产量达全球第一,企业间在技术提升和产业创新上差异不突出,同质性竞争较为激烈,氨基酸行业急需在高科技和新理念方面长久规划和全面提升.同时,由于缺乏自主知识产权,部分产品在进入国际市场时受到严重限制.比如,2017年我国谷氨酸钠(味精)的产量已达到供销平衡产销饱和状态,预计今后味精产量不会有太大增加.目前,企业间在规模上已拉开了一定的差距,但由于缺乏技术优越性,产品价格仍然不够理想,企业的经济效益不好.赖氨酸及其盐作为饲用氨基酸,约一半产量进入国际市场.受其影响,今后一两年内还会有一些企业加入赖氨酸生产行列,产能增幅较大,产量也会随之增长,国际市场的竞争会更加激烈.因此,提高产品层次,扩大内需,建立知识产权体系是今后发展的重点.
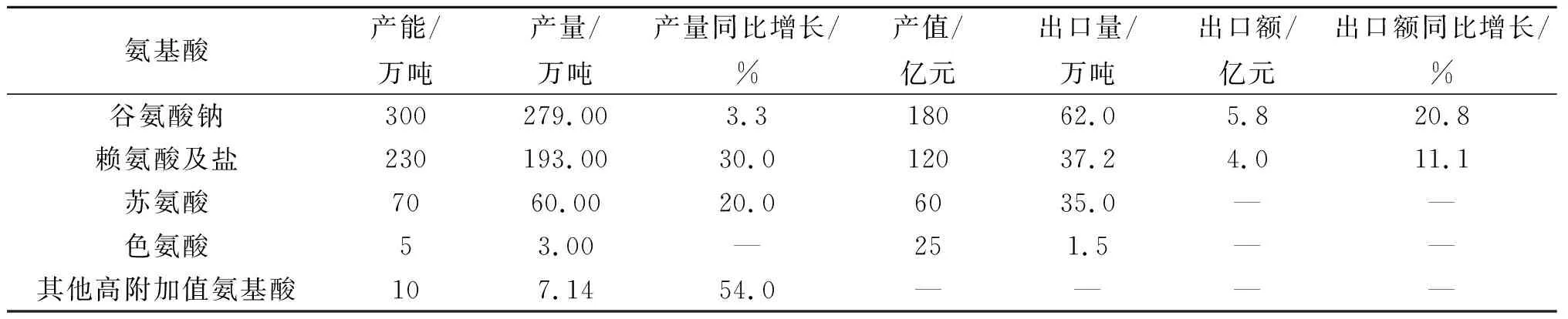
表1 2017年我国氨基酸产量及出口情况Table 1 Amino acid production and export status in China in 2017
另一方面,其他高附加值氨基酸品种开发力度还不够,氨基酸衍生物品种较少,产量还处于较低水平.大宗氨基酸主要用作调味品和饲料添加,而小品种氨基酸及衍生物的应用可以扩展到医疗保健、食品营养和美容化妆等诸多领域.随着人民生活水平的提高,人们对健康的渴望和理解也在不断进步.目前大健康理念已经被人们熟知和接受,迫切需要加强高附加值氨基酸衍生产品的研发.这些品种市场需求较小,但利润可观,其特点是打“技术战”,而非靠产能取胜.多数氨基酸企业已经意识到这一点,不断强化与研发机构的合作,主动提升自身的技术创新能力,其核心是选育具有自主产权的优良生产菌株.
2 常规育种技术
常规育种技术,也叫传统育种技术,包括诱变、杂交和驯化等.诱变育种是人们利用人工诱变手段使得微生物基因发生突变,进而选育具有特定性状的菌株.常用的诱变因子有物理因子和化学因子,前者包括紫外线、激光、X-射线和γ-射线等,后者包括甲基磺酸乙酯(EMS)、硫酸二乙酯(DES)和亚硝基胍(NTG)等.原核细胞的杂交方法有接合、转化和转导,真核细胞杂交包括单核菌丝体杂交和原生质体融合等.驯化也叫适应性进化,是在实验室中人为设置选择压力、对微生物自发突变进行有效筛选和富集的过程.近年来,由清华大学开发的常压室温等离子体(ARTP)诱变育种技术由于条件温和、操作简便、正突变率高等特点,被广泛应用于各种微生物的诱变育种中.
采用常规育种技术,如诱变育种可以在短时间内获得大量突变体,如何筛选具有目标性状的菌株是关键.在氨基酸和核苷生产菌株选育中,在诱变基础上往往采用产物的结构类似物作为选择压力,可以快速高效筛选解除反馈抑制的突变株.另外,在足够库容量的基础上,采用高通量筛选可以节省时间,提高选择效率.实验室常采用孔板培养、流式细胞仪(FACS),甚至应用微流控和机械臂来实现超高通量.本研究室采用ARTP对一株尿苷生产菌BacillussubtilisTD12np进行多轮诱变,依次在添加尿嘧啶结构类似物2-硫脲嘧啶、6-氮杂脲嘧啶和5-氟尿嘧啶培养基上获得耐性菌落,并通过多孔板高通量筛选最终获得解除尿苷酸反馈抑制的氨甲酰磷酸合成酶大亚基突变体,获得的突变株尿苷产量达到30.3 g/L,较出发菌株提高8.7倍[7].
由此可见,常规育种技术并没有过时,在现阶段菌株选育过程中还应该受到足够的重视.其虽然不定向、周期长,但结合合适的筛选体系也可以快速获得特定的性状.比如解除关键酶的反馈抑制,获得不代谢某一目标产物的菌株,获得某一物质细胞膜摄取/输出途径突变的菌株,并通过对关键基因或全基因组测序,解析关键酶的突变,揭示目标产物的降解途径和跨膜运输途径.由于目前对重要氨基酸生产菌种谷氨酸棒杆菌的一些关键基因还没有解析,比如色氨酸的代谢途径,色氨酸和组氨酸的运输途径尚不清楚,这严重制约了理性代谢工程手段的使用.然而,传统育种技术获得目标性状的同时,势必会引入大量不利突变,导致菌体生长变慢、抗逆性差和遗传操作困难等.因此,笔者认为现阶段传统育种技术的重要贡献在于获得目标性状,然后通过反向代谢工程手段解析有用突变,并在出发菌株中重构得到高产菌株.本研究室通过应用上述在枯草芽孢杆菌中获得的氨甲酰磷酸合成酶突变体,并结合系统代谢工程手段构建了一株高产尿苷的重组大肠杆菌(未发表).Ohnishi等[8]通过对一株诱变获得的赖氨酸高产菌株CorynebacteriumglutamicumB-6进行测序,成功解析了3个关键突变位点homVal59Val,lysCThr311Ile和pycPro458Ser,并在标准菌株C.glutamicum13032中重构途径,工程菌株发酵27 h赖氨酸产量达到80 g/L,与诱变菌株产量相当,但发酵周期缩短一半[8-9].
3 代谢工程育种技术
代谢工程育种技术是通过基因工程手段理性改造宿主细胞的基因组,获得高产目标产物的菌株.目前常用的代谢工程(常规代谢工程)研究围绕产物合成途径开展,也可以叫途径工程.其核心育种策略可以归纳为五个字:进、通、节、堵、出,简单描述就是促进底物吸收、打通合成途径、消弱支路代谢、阻断产物降解、促进产物排出,对其详细的阐述参见文献[10].
常规代谢工程往往通过试错的办法,一次只改造一个或几个基因,通过发酵测试保留有益操作摈弃负面操作,然后进行下一轮的基因工程改造.围绕产物合成途径的基因操作没有考虑到细胞整体生理平衡,单个基因改造其实是对细胞造成了一个扰动,可能会对菌体生长和产量积累造成负面影响.在此情况下,即便一些非常关键的基因位点也会被认为是无效的,因而严重阻碍了代谢工程育种进程.现阶段,随着氨基酸生产菌种大肠杆菌和谷氨酸棒杆菌各类组学数据的爆发,以组学为基础的建模已经得到了很好的发展.由于模型综合考虑了细胞的整体代谢水平,不但能够给出系统的产物合成途径基因的操作指导,还可以预测出一些途径无关的改造位点.在全局模型指导下,可以快速构建出氨基酸及衍生物高产菌株,该方法被称作系统代谢工程[11].比较典型的案例有大肠杆菌苏氨酸高产菌株[12]和谷氨酸棒杆菌赖氨酸高产菌株[13]的构建.
前面提到的实验室适应性进化虽然比自然进化效率要高,但还是具有操作周期长、定向性差等缺点.近年来,也出现了一些代谢工程辅助的进化策略,可以命名为进化代谢工程.第一类为全局转录因子进化工程,其工作原理是对全局转录因子进行突变建库,在培养过程中筛选目标性状.Wang等[14]通过全局转录因子改造显著提高了大肠杆菌半纤维素水解液的耐性.第二类为生物传感进化工程,是构建某种底物、产物或中间代谢物的生物传感系统,应用于进化工程的高通量筛选中.Fang等[15]通过构建色氨酸生物传感仪,采用“推”和“拉”两阶段策略构建了脱氧紫色杆菌素生产菌株.第三类为代谢逆境进化工程,是首先利用代谢工程手段设置微生物生长的逆境条件,然后通过适应性进化获得需要的表型.最近,Schwentner等[16]敲除谷氨酸棒杆菌丙酮酸和PEP羧化酶建立选择压力,然后以葡萄糖为底物对菌株进行驯化,获得生长速率达到野生菌80%的突变柱,通过基因组测序比对后发现进化菌株异柠檬酸脱氢酶ICD发生突变,导致乙醛酸循环增强为细胞生长补充草酰乙酸,进一步在野生菌中重构这些突变,同时过表达缬氨酸合成基因,构建了缬氨酸生产菌株.由于丙酮酸是缬氨酸合成的前体物,该菌株丙酮酸和PEP羧化途径的阻断,非常有利于目标产物缬氨酸的积累.
4 基因组编辑技术
工业化氨基酸生产菌株需要具有良好的生产性能、良好的遗传稳定性和对抗生素敏感.因此,目前质粒过表达的方式通常用于基因功能研究,真正构建工业生产菌株均在基因组上直接操作.这就要求非常高效的基因组编辑技术,包括基因敲除、插入和替换,以及点突变.自2000年问世以来,λ-Red重组技术就广泛应用于大肠杆菌的基因组编辑[17].近年来,CRISPR基因组编辑技术带来了基因工程领域的革命性变革,CRISPR/Cas9技术已经成功应用于大肠杆菌的基因组编辑[18].
对于高GC含量的谷氨酸棒杆菌,目前代谢工程操作仍采用传统的非复制型质粒pK18mobsacB介导的基因组编辑方法,CRISPR技术虽然取得了很大进展,但还不够成熟.Cho等[19]利用CRISPR技术首次实现了谷氨酸棒杆菌的基因编辑,发现Cas9蛋白对谷氨酸棒杆菌细胞生长有抑制作用,通过多次尝试发现一个针对放线菌密码子优化的基因可以正常表达,然后通过优化感受态制备方法实现了单链(ssDNA)介导的基因敲除,然而敲除效率极低(菌落数为个位数).Liu等[20]对Cas9表达的启动子进行了优化,采用质粒携带同源片段的方法实现了基因敲除和替换/插入,然而获得的转化子还停留在个位数,制约了实验的可重复性,但是其团队利用ssDNA实现了高效的单碱基突变编辑(90%).几乎同时,Peng等[21]报道了谷氨酸棒杆菌CRISPR/Cas9编辑方法,也取得了类似的结果.此外,Jiang等[21]报道了利用CRISPR/Cpf1对谷氨酸棒杆菌进行基因组编辑,Cpf1表达对细胞没有毒害作用,且其crRNA的设计相对简单.他们实现了高效的单碱基突变,但由于Cpf1的剪切作用不严谨,当进行基因敲除和插入时,效率很低.最近,Wang等[22]利用dCas9携带胞苷脱氨酶的方式,实现了自动高通量谷氨酸棒杆菌单碱基突变体库的构建方法.综上所述,目前利用CRISPR技术可以对谷氨酸棒杆菌进行有效的单碱基突变,但由于谷氨酸棒杆菌内源同源重组效率低和Cas9/Cpf1切割效率的不严谨性,致使进行大片段基因敲除和插入时效率很低,所能获得的菌落数极少,导致实验重复性差,另外需要将模板片段连接到载体上也增加了操作的复杂性.
5 精确代谢调控技术
随着对氨基酸生产菌株大肠杆菌和谷氨酸棒杆菌代谢工程研究的不断深入,科学家们发现基因组水平直接操作带来的菌株虽然遗传稳定,但难以照顾到菌体生长和产物合成之间的矛盾.目前代谢工程研究逐渐强调对代谢途径进行动态调控,称为精确代谢调控技术.该技术包括两方面的内容:一是基因表达的诱导,涉及的多为一些产物合成的关键基因;二是基因表达的关闭或弱化,可以在DNA,mRNA和蛋白质多个层次进行调控,被弱化基因可以催化支路代谢或产物降解途径,也可能是中心代谢途径的关键步骤,后者可以使菌体生长向产物合成切换,实现两阶段发酵工艺.诱导基因表达的因素有:1) 细胞所处的物理环境因素,包括pH、温度、溶氧和渗透压等;2) 化学小分子诱导物,如乳糖、木糖和抗生素等;3) 代谢途径相关的诱导,包括代谢产物自诱导和特定生长阶段转录因子调控.基因弱化方法有:1) 诱导关键蛋白的动态降解;2) 诱导阻遏蛋白表达;3) 基因组弱化方法,包括RNAi和CRISPRi等.
本研究室通过常规代谢工程手段构建了一株高产四氢嘧啶的大肠杆菌工程菌株,关键合成基因ectABC为质粒表达[23].为了提高菌株的稳定性和发酵性能,将ectABC整合到基因组上,同时构建了木糖诱导的基因表达模式.在发酵过程中先积累一定量的菌体后,添加木糖诱导促使代谢流向产物合成途径.将该策略应用于摇瓶发酵,四氢嘧啶产量由13.6 g/L提高到15.0 g/L.在此基础上,笔者团队利用CRISPRi技术对磷酸戊糖途径和TCA循环的关键基因进行干扰,大幅提升了四氢嘧啶的产量和转化率(未发表).
6 结 论
氨基酸生产是我国发酵行业重要支柱产业之一,目前需要提升大宗氨基酸生产的技术水平,同时大力开发小品种氨基酸及衍生物.其中,菌株选育技术是关键,通过代谢工程构建遗传背景清楚、生产性能稳定的工程菌株必将替代现有的传统诱变菌株.未来氨基酸菌株构建除了注重发酵指标,如产酸、转化率和生产强度等,还需要进行诸多方面的考虑.开发廉价的生物质废弃物发酵替代原料,不仅有利于降低发酵成本,而且具有重要的社会意义.提高生产菌株逆境胁迫能力,如耐酸、耐高温、耐渗透压能力,能够极大地提升氨基酸发酵性能,同时可能变革发酵过程控制和产品分离提纯工艺.
参考文献:
[1] 王洪荣,季昀.氨基酸的生物活性及其营养调控功能的研究进展[J].动物营养学报,2013,25(3):447-457.
[2] EGGELING L, SAHM H. L-glutamate and L-lysine: traditional products with impetuous developments[J].Applied microbiology and biotechnology,1999,52(2):146-153.
[3] 陈宁,范晓光.氨基酸生产菌株的研究热点及发展动向[J].发酵科技通讯,2016,45(1):1-6.
[4] 张权威,林高扬,马倩.组学技术在氨基酸生产菌株选育中的研究现状与发展趋势[J].发酵科技通讯,2017,46(4):205-211.
[5] 陈宁,范晓光.我国氨基酸产业现状及发展对策[J].发酵科技通讯,2017,46(4):193-197.
[6] 中国生物发酵产业协会.中国生物发酵产业协会二届六次理事会暨二届六次常务理事会文件汇编[C].北京:中国生物发酵产业协会,2017,北京.
[7] FAN X G, WU H Y, LI G L, et al. Improvement of uridine production ofBacillussubtilisby atmospheric and room temperature plasma mutagenesis and high throughput screening[J].Plos one,2017,12(5):e0176545.
[8] OHNISHI J, MITSUHASHI S, HAYASHI M, et al. A novel methodology employingCorynebacteriumglutamicumgenome information to generate a new L-lysine-producing mutant[J].Applied microbiology and biotechnology,2002,58(2):217-223.
[9] IKEDA M, OHNISHI J, HAYASHI M, et al. A genome-based approach to create a minimally mutatedCorynebacteriumglutamicumstrain for efficient L-lysine production[J].Journal of industrial microbiology and biotechnology,2006,33(7):610-615.
[10] LI Y J,WEI H B,WANG T,et al. Current status on metabolic engineering for the production of l-aspartate family amino acids and derivatives[J].Bioresource technology,2017,245:1588-1602.
[11] MA Q, ZHANG Q W, XU Q Y, et al. Systems metabolic engineering strategies for the production of amino acids[J].Synthetic and systems biotechnology,2017,2(2):87-96.
[12] LEE K H, Park J H, Kim T Y, et al. Systems metabolic engineering ofEscherichiacolifor L-threonine production[J].Molecular systems biology,2007,3(1):2025.
[13] BECKER J, ZELDER O, HFNER S, et al. From zero to hero-design-based systems metabolic engineering ofCorynebacteriumglutamicumfor L-lysine production[J].Metabolic engineering,2011,13(2):159-168.
[14] WANG J, ZHANG Y, CHEN Y, et al. Global regulator engineering significantly improvedEscherichiacolitolerances toward inhibitors of lignocellulosic hydrolysates[J].Biotechnology and bioengineering,2012,109(12):3133-3142.
[15] FANG M, WANG T, ZHANG C, et al. Intermediate-sensor assisted push-pull strategy and its application in heterologous deoxyviolacein production inEscherichiacoli[J].Metabolic engineering,2016,33:41-51.
[16] SCHWENTNER A, FEITH A, MÜNCH E, et al. Metabolic engineering to guide evolution-Creating a novel mode for L-valine production withCorynebacteriumglutamicum[J].Metabolic engineering,2018,47:31-41.
[17] DATSENKO K A, WANNER B L. One-step inactivation of chromosomal genes inEscherichiacoliK-12 using PCR products[J].Proceedings of the national academy of sciences of the United States of America,2000,97(12):6640-6645.
[18] LI Y F, LIN Z Q, HUANG C, et al. Metabolic engineering ofEscherichiacoliusing CRISPR-Cas9 meditated genome editing[J].Metabolic engineering,2015,31:13-21.
[19] CHO J S, CHOI K R, CPS P, et al. CRISPR/Cas9-coupled recombineering for metabolic engineering ofCorynebacteriumglutamicum[J].Metabolic engineering,2017,42:157-167.
[20] LIU J, WANG Y, Lu, Y J, et al. Development of a CRISPR/Cas9 genome editing toolbox forCorynebacteriumglutamicum[J].Microbial cell factories,2017,16(1):205.
[21] PENG F, WANG X Y, SUN Y, et al. Efficient gene editing inCorynebacteriumglutamicumusing the CRISPR/Cas9 system[J].Microbial cell factories,2017,16(1):201.
[22] WANG Y, LIU Y, LIU J, et al. Macbeth: multiplex automatedCorynebacteriumglutamicum, base editing method[J].Metabolic engineering,2018,47:200-210.
[23] NING Y K, WU X J, ZHANG C L, et al. Pathway construction and metabolic engineering for fermentative production of ectoine inEscherichiacoli[J].Metabolic engineering,2016,36:10-18.

