3种果肉颜色桃原花青素积累
严 娟, 宋志忠, 蔡志翔, 沈志军, 马瑞娟, 俞明亮
(江苏省农业科学院果树研究所/江苏省高效园艺作物遗传改良重点实验室,江苏 南京 210014)
原花青素广泛存在于植物树干、叶、花、果和种子中,对植物的生长发育和人体动物健康均有重要作用[1-2]。通过对拟南芥、西班牙三叶草和葡萄等几个模式植物的研究,原花青素合成机制已经比较明晰[3]。从苯丙烷途径开始,通过无色花青素还原酶(Leucoanthocyanidin reductase,LAR)和花青素还原酶(Anthocyanidin reductase,ANR)2个途径形成原花青素,因此LAR和ANR是直接调控原花青素单体以及聚合结构单元形成的关键酶基因。
原花青素在果品中含量极为丰富,其生理作用显得尤为突出[1]。一是可为果树防御生物及非生物伤害,保证其生长发育和产量;二是与果实品质和风味密切相关,赋予鲜食果品果肉以及果汁、葡萄酒等加工产品独特的口感;三是原花青素还是影响果品与葡萄酒等储藏期的关键因素之一;四是相较于果品中维生素和花色素苷等有益成分,其具有更高的抗氧化能力,为人类抵御各种疾病。因此,原花青素现已成为研究与培育富含有益成分果品的重要指标。
桃[Prunuspersica(L.) Batsch]是全球重要的蔷薇科果树,栽培广泛,生产中心国包括中国、意大利、西班牙和美国等。桃种质资源丰富,果实按肉色主要分为白肉桃、黄肉桃、红肉桃(包括bfbf-基因型和DBF-基因型)3种类型[4]。桃果实富含抗氧化成分,与可可、葡萄酒、茶及苹果、草莓等一样,桃,特别是DBF-基因型红肉桃果实中原花青素含量丰富,鲜果中总含量约为 90~700 mg/kg[5],现已分离出儿茶素和表儿茶素单体以及各种低聚和高聚产物,对其中主要成分作了定性定量分析[6-7]。桃果实中原花青素随发育期的积累和代谢机理的相关报道甚少,且相关研究均是以白肉桃为材料[8-10],红肉桃和黄肉桃相关研究尚未涉及,LAR和ANR等关键酶的酶学特性亦未见报道。
作者在前期研究中发现,白、黄、红3种肉色桃成熟果肉中的原花青素单体儿茶素和表儿茶素含量存在显著差异[11]。因此,本研究将以瑞光19号(白肉)、浙金3号(黄肉)、红肉大红袍(DBF-基因型,红肉)为材料,进行3种果肉颜色的桃原花青素积累动态的生理数据采集,测定调控原花青素合成的关键酶LAR和ANR的酶活力;克隆关键酶编码基因(LAR、ANR),并分析其在果实发育过程中的表达。旨在从生理、生化(酶)和转录水平解析不同肉色桃原花青素积累动态及其机制,为探讨桃原花青素合成机理提供基础。
1 材料与方法
1.1 试验材料及取样
本研究材料瑞光19号(白肉)、浙金3号(黄肉)、红肉大红袍(DBF-基因型,红肉)均取自国家果树种质南京桃资源圃,按照常规栽培方法种植,统一田间管理。每个品种从盛花后30 d开始,每隔7 d取1次样品,每次分别取18个果,分为3组,称量每个果实大小,快速削皮,果肉切碎混合液氮冻存。参照Lombardo等[12]的方法,根据每个品种每个发育期的果实大小平均值拟合各桃品种发育的S曲线(图1),确定各品种的果实发育阶段和样品点(表1)。

图1 桃果实发育曲线图Fig.1 The growth curve of peach fruit

表1 桃果实发育期采样点
S1、S2、S3、S4、H分别表示果实发育天数。
1.2 原花青素的提取与测定
将适量样品烘干至恒质量,粉碎,过40目筛之后,称取约0.05 g,加入1.25 ml 60%乙醇提取液,用超声提取法进行提取,超声功率300 W,破碎5 s,间歇8 s,提取30 min, 12 000 r/min,25 ℃,离心10 min,取上清液,用提取液定容至1.25 ml,待测。 吸取40 μl待测液和160 μl工作液(工作液试剂盒购自苏州科铭生物技术有限公司),混匀,30 ℃水浴30 min,在酶标仪(Berthold TriStar LB941, 德国)上进行500 nm 单波长测定。以40 μl待测液和160 μl H2O的混合液为对照。原花青素含量单位为mg/g(干质量,DW),计算公式如下:
标准曲线:Y=0.019 4x+0.000 6,R2=0.999
1.3 ANR和LAR的提取和酶活力测定
取1 g果肉加入等量的PVPP,液氮研磨后加入pH7.4 的 PBS 缓冲液 4 ℃离心,上清液即为粗酶液。参考Xie等[13]和杨琴等[14]的方法,并作一定修改。200 μl 反应体系包括 0.1 mol/L的柠檬酸磷酸缓冲盐(pH6.5)、粗酶液、反应底物[其中无色花青素还原酶的反应底物为二氢斛皮素(0.6 mol/L),花青素还原酶的反应底物为矢车菊素(0.6 mmol/L)]、辅酶NADPH( 2 mmol/L) 和 pH7.0 的 Tris·HCl 缓冲液(0.1 mol/L),于40 ℃下分别反应30 min, 10 倍体积乙酸乙酯终止反应,经萃取浓缩后,甲醇定容至0.5 ml。在酶标仪(Berthold TriStar LB941, 德国)上进行280 nm单波长测定。利用原花青素标准品做标准曲线计算相对酶活力,单位为mg/g(鲜质量,FW),试验重复3次。
1.4 果肉总RNA的提取和荧光定量PCR
果肉总RNA提取利用EASYapin Plus Plant RNA Kit试剂盒(钟鼎,RK16-20T),提取步骤按照试剂盒说明书进行,总RNA经琼脂糖凝胶电泳以及紫外分光光度计检测纯度合格后备用。第一链cDNA合成采用AMV First Strand cDNA 合成试剂盒(钟鼎,PC01-50T),方法参考说明书,反应条件设置:25 ℃ 10 min,42 ℃ 30 min,85 ℃ 5 min。荧光定量RT-PCR引物和内参基因参考 Daniela等[9]和Tong等[15]的研究。引物序列(表2)委托生工生物工程(上海)股份有限公司合成。荧光定量RT-PCR在ABI 7500型荧光定量PCR仪中进行,利用HiScript QRT SuperMix同引物(正反向引物各0.4 μl)和cDNA模板(2 μl)配置20 μl反应体系,设置3次重复。荧光定量RT-PCR扩增条件设置:扩增程序为94 ℃ 30 s;94 ℃ 20 s,55 ℃ 20 s,72 ℃ 30 s循环45 次,72 ℃单点检测信号。溶解程序为:95 ℃ 0 s,60 ℃ 15 s,95 ℃ 0 s,连续检测信号。采用2-ΔΔCt法对数据进行相对定量分析。
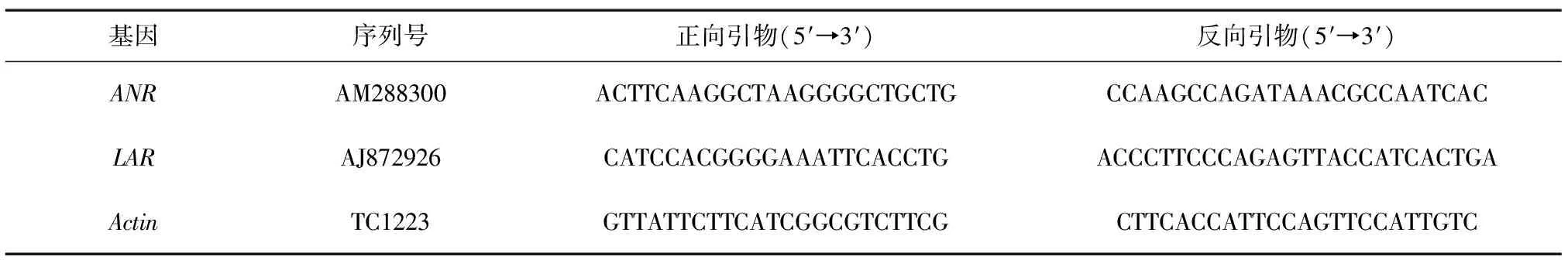
表2 荧光定量PCR扩增引物
2 结果与分析
2.1 桃果实发育过程中果肉原花青素的积累变化
桃果肉原花青素含量随果实发育的积累变化见图2。3个桃品种原花青素含量差异很大,在发育过程中含量分布分别为 0.22~0.53 mg/g, DW、0.14~0.34 mg/g, DW 和 0.10~0.90 mg/g, DW;原花青素积累变化趋势差异明显。瑞光19号含量变化趋势呈倒V形,初期较低,在S4时期急剧积累(P<0.01),达到0.53 mg/g, DW,在成熟时又快速下降,含量为0.22 mg/g, DW。浙金3号初期含量最高,为0.34 mg/g, DW,S2到S3时期快速降低(P<0.01),在果实发育的后期含量趋于平稳,成熟时含量为0.15 mg/g, DW。红肉大红袍的原花青素含量在果实发育前期均处于很低的水平,甚至在S2时期明显下降,但随着果实发育,其含量急剧增加,成熟时达到最高,高达0.90 mg/g, DW。在S1时期,原花青素含量表现为浙金3号>瑞光19号>红肉大红袍,其中浙金3号是红肉大红袍的2倍(P<0.01);到了H时期,原花青素含量则为红肉大红袍>瑞光19号>浙金3号,红肉大红袍是瑞光19号和浙金3号的5倍(P<0.01)。
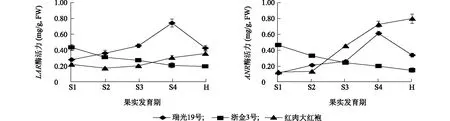
2.2 桃果实发育过程中LAR和ANR的酶活力
LAR和ANR随桃果实发育的酶活力变化见图3。同一桃品种中LAR和ANR的酶活力大小有差异,但变化趋势基本一致;而3个桃品种之间LAR和ANR酶活力变化趋势差异明显,在发育不同时期活力差异大。
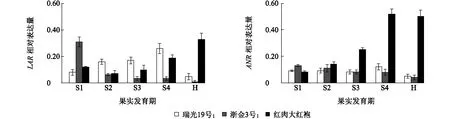
瑞光19号LAR和ANR的酶活力变化趋势较一致,均是在S1至S3时期升高(P<0.05),在S4时期急剧升高(P<0.01),然后急剧下降(P<0.01);但LAR的酶活力(0.28~0.74 mg/g, FW)在各个发育阶段均略高于ANR(0.11~0.61 mg/g, FW)。浙金3号LAR和ANR的酶活力(0.20~0.43 mg/g, FW 和 0.15~0.46 mg/g, FW)和变化趋势均基本相同,表现为随着果实发育逐渐降低,H时期达到最低(P<0.01)。红肉大红袍LAR和ANR的酶活力分别为 0.17~0.36 mg/g, FW和 0.12~0.80 mg/g, FW,除了S1和S2时期LAR酶活力略高于ANR外,其他时期,特别是果实成熟期,ANR酶活力远远大于LAR;2个酶的酶活力变化趋势也有轻微差异,LAR酶活力在S1至S3时期没有显著差异,在S4和H时期则显著升高(0.01 S1、S2、S3、S4、H见表1。图2 桃果肉中原花青素含量变化Fig.2 The changes of proanthocyanidin content in flesh of peach S1、S2、S3、S4、H见表1。图3 桃果实发育过程中LAR和ANR酶活力Fig.3 The activities of LAR and ANR during fruit development period 在S1时期,LAR的活性表现为浙金3号>瑞光19号>红肉大红袍,其中浙金3号是红肉大红袍的2.0倍;到了H时期,LAR的活力则为瑞光19号>红肉大红袍>浙金3号。ANR的活力和LAR相似,即在S1时期,ANR的活力为浙金3号>瑞光19号>红肉大红袍,浙金3号的ANR酶活力是瑞光19号和红肉大红袍的5.0倍;到了H时期,ANR的活力则为红肉大红袍>瑞光19号>浙金3号,红肉大红袍是瑞光19号的2.5倍、浙金3号的7.0倍。 定量PCR结果见图4。结果表明同一桃品种中LAR和ANR基因在转录水平的表达量有差异,但变化趋势基本一致。瑞光19号LAR和ANR表达量均是在S4时期达到最高(P<0.05),然后在H时期急剧下降(P<0.01)。浙金3号LAR基因在转录水平的表达量在S1时期最高,并在S2时间急剧降低(P<0.01),之后,随着果实发育变化不显著;然而,ANR基因在S1至S4各时期的表达量没有显著差异,但显著高于H时期(P<0.05)。红肉大红袍LAR和ANR表达量变化趋势有轻微差异,LAR基因在S1至S3时期的表达水平没有显著差异(P>0.05),但从S4时期开始至H时期则显著升高(P<0.01);ANR基因的表达水平则随着果实不同发育时期而显著升高,在S4时期达到最高水平(P<0.01)。 3个桃品种之间LAR和ANR表达量变化趋势差异明显,在发育不同时期表达量差异大。在S1时期,LAR基因在转录水平的表达量由高到低依次为浙金3号>红肉大红袍>瑞光19号,其中浙金3号LAR的表达量是红肉大红袍和瑞光19号的2倍(P<0.01);而在H期,LAR的表达量由高到低依次为 红肉大红袍>瑞光19号>浙金3号。然而,ANR的表达量在S1时期表现为浙金3号> 瑞光19号>红肉大红袍,但到H时期,则恰好相反,表现为 红肉大红袍>瑞光19号>浙金3号,其中红肉大红袍的ANR表达量约为瑞光19号和浙金3号的10倍(P<0.01)。 S1、S2、S3、S4、H见表1。图4 桃果实发育过程中LAR和ANR基因表达Fig.4 Relative expression of LAR and ANR during fruit development period 原花青素的相关研究在其他果品中已非常深入和广泛,如葡萄[16]、柿子[17]、苹果[18]、草莓[19]、杏[20]、黑莓[21]等,相关报道涉及原花青素在果实中的组成和随发育期的含量变化,各种栽培措施、激素和贮藏条件对其组分的影响,生物合成关键酶LAR和ANR的酶学特性和基因表达、以及功能验证等。相较而言,桃果实中原花青素随发育期的积累以及代谢机理研究报道鲜少,特别是关于红肉或黄肉桃果实中原花青素的积累尚未报道。 在前人研究中,白肉桃晚蜜随着果实成熟,原花青素总量逐渐降低[8];白肉桃Stark Red Gold和Baifeng的原花青素总量随着果实发育先积累,随后又逐渐下降[9-10]。本研究以3种肉色桃果实为试验材料,发现原花青素在不同肉色桃的果实发育过程中积累趋势完全不同。在白肉桃中表现为先升高后降低,在黄肉桃中表现为逐渐下降,说明在所试验的白肉桃和黄肉桃中原花青素的积累都伴随着果实成熟而降低,这与前人研究结果一致[8-10],也与葡萄、草莓和黑莓等其他果品研究结果相似[21-22]。而DBF-基因型红肉桃则恰好相反,随果实发育急剧增加,在成熟时达到最高,且远远高于白肉桃和红肉桃。这一结果在前期研究中也有所体现,即白肉桃和黄肉桃成熟果肉中原花青素单体儿茶素和表儿茶素的含量均远远低于红肉桃中儿茶素和表儿茶素的含量[11]。在桃育种实践工作中,红肉桃果实在口感上普遍存在涩味,这可能与其成熟时含有大量原花青素有关。针对红肉桃中原花青素有别于在白肉桃、黄肉桃以及其他果品中的积累规律,其相关机制需要进行进一步深入研究和探讨。 LAR和ANR基因是直接调控果品原花青素单体以及聚合结构单元形成的关键酶基因[23-24]。具体地,LAR途径合成2,3-反式黄烷-3-醇,如儿茶素,ANR途径生成2,3-顺式黄烷-3-醇,如表儿茶素。Daniela 等[9]提出CHS、CHI、FSH、DFR、LDOX、UFGT、ANR、LAR、FLS1结构基因的表达与原花青素合成有关,调控因子PA1对ANR和LAR的表达具有调控作用;而 Zhou等[10]的研究则着重提出调控因子MYB7调控LAR基因的表达,而非ANR。本研究则以3种肉色桃为材料,探讨了原花青素积累与关键酶LAR、ANR的酶活力和酶基因表达量的关系。结果表明,在同一材料中,LAR、ANR的酶活力及其编码基因在转录水平的表达情况与原花青素积累趋势一致。本研究结果很好地证实LAR和ANR是桃果实中原花青素合成的关键调控基因。此外,本研究发现在白肉桃和黄肉桃中,LAR的酶活力和基因表达量与原花青素的积累相关性更为密切,而在红肉桃中,ANR与原花青素的积累更为密切,这一结果可部分解释严娟等[11]报道的儿茶素含量在白肉桃和黄肉桃高,而表儿茶素在红肉桃中高。本研究结果初步明确了不同果肉颜色的桃原花青素积累与LAR、ANR酶活力及其编码基因之间的内在联系,为进一步探讨桃原花青素合成机理提供了基础。 参考文献: [1] DIXON R A, XIE D Y, SHASHI B S. Proanthocyanidins-a final frontier in flavonoid research? [J]. New Phytologist, 2005, 165: 9-28. [2] 李先良,赵志常,高爱平,等.芒果ANR基因的克隆及其表达分析[J]. 江苏农业科学,2017,45(4):22-25. [3] ZHAO J, PANG Y Z, DIXON R A. The mysteries of proanthocyanidin transport and polymerization [J]. Plant Physiology, 2010, 153:437-443. [4] SHEN Z J, CONFOLENT C, LAMBERT P P,et al. Characterization and genetic mapping of a new blood-flesh trait controlled by the single dominant locus DBF in peach [J]. Tree Genetics & Genomes, 2013, 9:1435-1446. [5] 严 娟,沈志军,蔡志翔,等. 桃果实中酚类物质研究进展 [J]. 果树学报,2014,31(3):477-485. [6] ASAMI D K, HONG Y J, BARRET D M,et al. Processing-induced changes in total phenolics and procyanidins in clingstone peaches [J]. Journal of the Science of Food and Agriculture, 2003, 83:56-63. [7] HONG Y J, DIANEL M, BARRET A E,et al. Liquid chromatography/Mass spectrometry investigation of the impact of thermal processing and storage on peach procyanidins [J]. Journal of Agricultural and Food Chemistry, 2004, 52:2366-2371. [8] 周 君,陈宗玲,张 琼,等. 套袋对桃果实成熟过程中酚酸类和类黄酮类物质积累的影响[J]. 园艺学报,2009,36 (12) :1717-1724. [9] DANIELA R, RICHARD V E, REBECCA A H,et al. Transcriptional regulation of flavonoid biosynthesis in nectarine (Prunuspersica) by a set of R2R3 MYB transcription factors [J]. BMC Plant Biology, 2013, 13:68. [10] ZHOU H, WANG K L, LIAO L,et al. Peach MYB7 activates transcription of the proanthocyanidin pathway gene encoding leucoanthocyanidin reductase, but not anthocyanidin reductase [J]. Frontiers in Plant Science, 2015, 6:908. [11] 严 娟,蔡志翔,沈志军,等. 三种肉色桃中10种酚类物质测定及比较[J]. 园艺学报,2014b,41(2):319-328. [12] LOMBARDO V A, OSORIO S, BORSANI J,et al. Metabolic profiling during peach fruit development and ripening reveals the metabolic networks that underpin each developmental stage [J]. Plant Physiology,2011, 157:1696-1710. [13] XIE D Y, SHARMA S B, DIXON R A. Anthocyanidin reductases from medicago truncatula and arabidopsis thaliana [J]. Archives of Biochemistry and Biophysics, 2004, 422:91-102. [14] 杨 琴,赵 磊,刘亚军,等. 茶树花青素还原酶的酶学特性研究[J]. 茶叶科学,2013,33(3):221-228. [15] TONG Z, GAO Z, WANG F,et al. Selection of reliable reference genes for gene expression studies in peach using real-time PCR [J]. BMC Molecular Biology, 2009, 10:71. [16] SETH D C, JULIE M T, GREG A G,et al. Impact of diurnal temperature variation on grape berry development, proanthocyanidin accumulation, and the expression of flavonoid pathway genes [J]. Journal of Experimental Botany, 2012,63(7):2655-2665. [17] TAKASHI A, AYAKO K, KEIZO Y. Proanthocyanidin biosynthesis of persimmon (DiospyroskakiThunb.) fruit [J]. Scientia Horticulturae, 2011, 130:373-380. [18] REBECCA A, HENRY-KIRK T K, MCGHIE,et al. Transcriptional analysis of apple fruit proanthocyanidin biosynthesis [J]. Journal of Experimental Botany, 2012, 63(15):5437-5450. [19] JAN G S, CHRISTIAN D, IRENE R D L F,et al. Identification and characterization of MYB-bHLH-WD40 regulatory complexes controlling proanthocyanidin biosynthesis in strawberry (Fragaria × ananassa) fruits [J]. New Phytologist, 2013, 197(2):454-67. [20] CLAUDIA N, SYLVAIN G, NATHALIE M,et al. Characterization of plum procyanidins by thiolytic depolymerization [J]. Journal of Agricultural and Food Chemistry, 2008, 56:5188-5196. [21] CHEN Q, YUA H W, TANGA H R,et al. Identification and expression analysis of genes involved in anthocyanin and proanthocyanidin biosynthesis in the fruit of blackberry [J]. Scientia Horticulturae, 2012, 141:61-68. [22] KENNEDY J A, HAYASAKA Y, VIDAL S,et al. Composition of grape skin proanthocyanidins at different stages of berry development [J]. Journal of Agricultural and Food Chemistry, 2001, 49: 5348-5355. [23] BOGS J, MARK O D, JOHN S H,et al. Proanthocyanidin synthesis and expression of genes encoding leucoanthocyanidin reductase and anthocyanidin reductase in developing grape berries and grapevine leaves [J]. Plant Physiology, 2005, 139:652-663. [24] LIAO L, VIMOLMANGKANG S, WEI G,et al. Molecular characterization of genes encoding leucoanthocyanidin reductase involved in proanthocyanidin biosynthesis in apple [J]. Frontiers in Plant Science, 2015, 6:243.
2.3 桃果实发育过程中LAR和ANR的基因表达
3 讨 论

