培养调控激活卡伍尔氏链霉菌NA4产frontalamides类物质的研究
张 盈, 潘华奇, 于素亚, 胡江春*
(1.中国科学院 沈阳应用生态研究所,辽宁 沈阳 110016;2. 中国科学院大学,北京 100049)
植物真菌病害是植物病害中最严重的病害之一,严重威胁着农业生产、粮食安全及生物多样性[1]。主要粮食作物水稻、小麦、马铃薯、玉米等均遭受真菌病害带来的经济损失。据报道,全球每年因稻瘟病造成的水稻产量损失约占总产量的10%~15%,经济损失达数十亿美元[2]。由禾谷镰孢菌和黄色镰孢菌引起的小麦赤霉病可使小麦在大流行年份病穗率达到50%~100%,减产5%~15%[3]。面对严重的植物真菌病害,目前最常用的就是化学防治,但是化学农药的长期大面积使用带来了水土环境污染、农产品农药残留等严重问题。同时新发生的真菌病害和超级耐药病菌不断出现[4]。因此,研制安全和高效的新型生物杀菌剂迫在眉睫。微生物来源的天然产物在药物先导物发现及其新药开发中起着重要作用,是农用抗生素的主要来源之一[5-6]。特别是随着基因组学的快速发展,人们发现微生物基因组中(尤其是放线菌)蕴涵了丰富多样的次级代谢产物合成基因簇。如Salinisporatropica基因组中参与形成次级代谢产物的基因蔟有17个,约占基因组总量的9.9%[7]。著名的阿维菌素产生菌Streptomycesavermitilis中则有25个次级代谢基因簇,占整个基因组的6.4%[8]。然而,目前仅仅从其中分离鉴定了几类次级代谢产物,大多数产物因其合成基因为“隐性”并没有被发掘,因此,基于基因组序列的genome mining技术应运而生[9]。相对于异源表达、过表达正调控基因、敲除负调控基因以及全局性启动子的改造等激活策略,培养调控不需要进行任何遗传操作,简单有效而被研究者广泛使用[10]。这种OSMAC (One strain many compounds) 思想指导下的培养调控,强调通过改变培养基组分、培养条件、添加生物合成前体及关键酶激活或抑制剂等方式,可以有效挖掘微生物菌株次级代谢潜能,提高其代谢产物的多样性,从而实现对“隐性”次级代谢产物的激活[11]。如中国科学院南海海洋研究所张长生等,利用培养调控对深海链霉菌SCSIO 03032的次级代谢产物进行了深入的挖掘,从7个不同培养基条件下共分离获得21个次级代谢产物,含15个新化合物。在改良的ISP3培养基中,该菌产生了3个新的大环内酰胺类抗生素heronamides D-F,而使用改良的A1BFe+C培养基时,该菌则产生了结构新颖独特的双吲哚螺环生物碱spiroindimicins A-D[12-14]。前期对分离自南海深海沉积物的卡伍尔氏链霉菌NA4基因筛选发现其具有产生PKS、NRPS类化合物的潜力,但只分离到了PKS I型的bafilomycins B1和C1[15]。为了全面、深入认识菌株NA4合成次级代谢产物的潜能,挖掘更多抗真菌先导化合物,采用Illumina Hiseq2500二代高通量测序技术测序,并组装获得其基因组扫描图,进一步生物信息学分析发现,其除了能产生已经发现的bafilomycins,还具有产生lantipeptide AmfS、alkylresorcinol、valinomycin、desferrioxamine_B、frontalamides、nonactin、isorenieratene、ectoine、rabelomycin和lassopeptide SRO15-2005等多种结构类型化合物的潜力。经文献调研,该菌株中尚未被发现的polycyclic tetramate macrolactam (PTM) 类化合物frontalamides[16]、valinomycin[17]和alkylresorcinol[18]具有良好的抗真菌活性。为了深入挖掘这些具有抗真菌活性的次级代谢产物,通过改变培养基碳氮源组分,添加氨基酸前体,添加不同金属离子等培养调控的手段尝试对这些合成抗真菌活性物质的“隐性”基因组进行激活,并使用活性追踪和HPLC-DAD-MS进行分析以期发现新的衍生物,为进一步进行目标化合物的分离和鉴定提供指导。
1 材料与方法
1.1 材料
1.1.1 菌种来源 卡伍尔氏链霉菌NA4分离自中国南海(E 111°36.160′, N 17°59.928′)水深1 464 m的海底沉积物中。
1.1.2 培养基 ①人工海水高氏一号培养基:可溶性淀粉20.0 g,KNO31.0 g,K2HPO40.5 g,MgSO4·7H2O 0.5 g,FeSO4·7H2O 0.01 g,人工海水(2.5%海盐水)1.0 L,琼脂20.0 g,pH 7.0~7.2;②ISP2培养基:酵母膏 4.0 g,麦芽汁10.0 g,葡萄糖4.0 g,蒸馏水1.0 L,琼脂20.0 g,pH 7.0;③NM2培养基(Novel Medium 2):葡萄糖1.0 g,乳糖10.0 g,甘油20.0 mL,大豆蛋白胨5.0 g,硝酸铵1.5 g,酵母粉3.0 g,1倍微量元素液(FeSO4·7H2O 0.1 g,MnCl2·4H2O 0.1 g,ZnSO4·7H2O 0.1 g,蒸馏水1.0 L) 0.2 mL,蒸馏水1.0 L,pH 6.0; ④TSBY培养基:胰蛋白胨15.0 g,大豆蛋白胨5.0 g,NaCl 5.0 g,K2HPO42.5 g,葡萄糖2.5 g, 蔗糖103.0 g,蒸馏水1.0 L, pH 7.3~7.5;⑤PDA培养基:新鲜去皮土豆200.0 g,切成小块,加1.0 L自来水煮至土豆松软,用8层纱布过滤得滤液,补自来水至1.0 L,加入葡萄糖20.0 g,琼脂20.0 g,自然pH。
1.1.3 试剂与仪器 DHZ-C恒温摇床(大仓市实验仪器有限公司); YXQ-LS-50S11立式压力蒸汽灭菌锅(上海博讯实业有限公司医疗设备厂);BSA2202S电子天平(赛多利斯);RE52-99旋转蒸发仪(上海亚荣生化仪器厂);Ultimate-3000高效液相色谱仪(美国Dionex);Waters 2695.ZQ4000液相色谱质谱联用仪(美国Waters公司)。色谱乙腈及色谱甲醇(国药集团试剂有限公司);其他化学试剂均为国产AR纯。
1.2 方法
1.2.1 菌株NA4基因组DNA的提取 将菌株在TSBY液体培养基中培养2 d,5 000×g离心10 min,弃上清,收集菌丝体,用STE缓冲液清洗菌体2次,然后用5 mL的STE缓冲液重悬菌体,加入溶菌酶至终浓度为1 mg/mL,37 ℃温育45 min,每隔10 min颠倒混匀;加入蛋白酶K至终浓度为0.5 mg/mL,混匀;加入600 μL的10% SDS,混匀;50 ℃温育2~5 h,20~30 min颠倒混匀1次;加入2 mL 5 mol/L NaCl和5 mL氯仿,室温静置30 min以上;4 500×g 离心10 min,取上清,加0.6倍体积的异丙醇,颠倒混匀,用灭菌的玻璃棒搅出总DNA,用70%乙醇洗涤DNA沉淀2次,小心弃上清,挥干,将基因组DNA溶于1~2 mL TE (pH 8.0) 中,置于-20 ℃备用。
1.2.2 菌株NA4全基因组测序 利用第二代测序技术,illumina 公司的Hiseq2500进行NA4的基因组序列扫描。基因组序列测定和组装由上海凌恩生物科技有限公司完成。
1.2.3 菌株NA4基因组预测的次级代谢产物 将NA4的基因组草图序列提交次级代谢产物在线分析工具antiSMASH 3.0[19],预测NA4次级代谢产物合成基因簇,并进行人工校对。
1.2.4 培养基的设计 以NM2培养基为基础培养基,通过改变培养基碳氮源组分,添加不同金属离子及氨基酸前体,新设计了19种发酵培养基(表1)。
1.2.5 菌株NA4的发酵培养和发酵产物分析 从活化好的NA4平板上,刮取1环孢子接种于ISP2培养基(250 mL三角瓶),于28 ℃ 180 r/min条件下摇床培养3 d制备种子液,按6%的接种量接种于上述20种发酵培养基中,28 ℃ 180 r/min摇床培养8 d,5 000×g离心10 min,发酵上清液用等体积的丁酮萃取,萃取3次后,将萃取液用旋转蒸发仪减压浓缩获得粗提取物样品,用无水甲醇将其配成10 mg/mL的浓度备用。采用滤纸片法,测试发酵粗提物对病原真菌黄瓜枯萎病菌(Fusariumoxysporumf.sp.cucumerinum)、立枯丝核菌(Rhizoctoniasolani)和番茄灰霉病菌(Botrytiscinerea)的抑制效果。将病原真菌接种到PDA平板的中央,待菌落生长至直径5 cm时,将浸有粗提物的滤纸片放在四周。萃取液质量浓度为10 mg/mL,滤纸片直径为8 mm,上样20 μL,对照用无水甲醇。将病原真菌继续培养约2 d,用游标卡尺测抑菌圈的大小。对具有抗病原真菌活性的粗提物进行HPLC-UV分析,判断是否有新的次级代谢产物激活。HPLC-UV检测条件:流动相A相为100%色谱甲醇,含0.05%三氟乙酸,流动相B为超纯水,含0.05%的三氟乙酸;分析程序:0~30 min,A/B(体积比)线性梯度从10∶90到94∶6;30~42 min,A/B(体积比)线性梯度从94∶6到100∶0;42~47 min,A /B(体积比)100∶0等度;47 min后,A /B(体积比)到起始洗脱浓度10∶90。检测波长为210、238、254、280 nm。
1.2.6 HPLC-DAD-MS识别激活的次级代谢产物 经过活性测试和HPLC-UV分析,对激活次级代谢产物的粗提物进行HPLC-DAD-MS分析和结构识别。检测条件:流动相A相为100%色谱乙腈,流动相B为超纯水,含0.05%的三氟乙酸,正负离子同时扫描模式,检测分子量200~1 600; 进样程序:0~20 min,A/B(体积比)线性梯度从10∶90到60∶40;20~30 min,A/B(体积比)线性梯度从60∶40到80∶20;30~40 min,A/B(体积比)线性梯度从80∶20到100:0;40~50 min,A/B(体积比)100∶0等度,50 min后,A /B(体积比)到起始浓度10∶90。流速为0.53 mL/min,检测波长为全波长扫描。
2 结果与分析
`2.1 菌株NA4的基因组草图和生物信息学分析
对菌株NA4进行Hiseq2500测序,平均测序深度超过200×,经组装获得NA4基因组草图。估算该基因组序列大小约为8.02 Mbp,GC%为72.07%。组装获得59个超过1 000 bp的scaffolds,Scaffold N50 286 752 bp,总碱基大小为7 767 524 bp。antiSMASH分析显示其蕴含了丰富的次级代谢产物合成基因簇,预测它们编码合成bafilomycins、lantipeptide AmfS、alkylresorcinol、valinomycin、desferrioxamine_B、frontalamides、nonactin、isorenieratene、ectoine、rabelomycin、lassopeptide SRO15-2005及其他未知结构类型的化合物。
2.2 发酵粗提物的抗真菌活性筛选
抗真菌活性测定结果显示,当用不同碳源替代甘油后,除果糖替代甘油其相应粗提物无抗真菌活性外,其他都具有抗真菌活性。其中蔗糖替代甘油后抗真菌活性变化不大,玉米面和玉米淀粉替代甘油后对特定真菌的抑制强度有所改变,而麸皮替代甘油后抗真菌活性均显著提高,提示其次级代谢产物可能有较大变化(表1)。当用不同氮源替代大豆蛋白胨后,除胰蛋白胨替代其相应粗提物的抗特定真菌(镰刀菌)活性增强外,其他均无抗真菌活性。当添加氨基酸前体供应后,Ala添加能增强其抗真菌活性,Orn添加则降低其抗真菌活性。当添加不同浓度的金属离子后,抗真菌活性均显著降低。
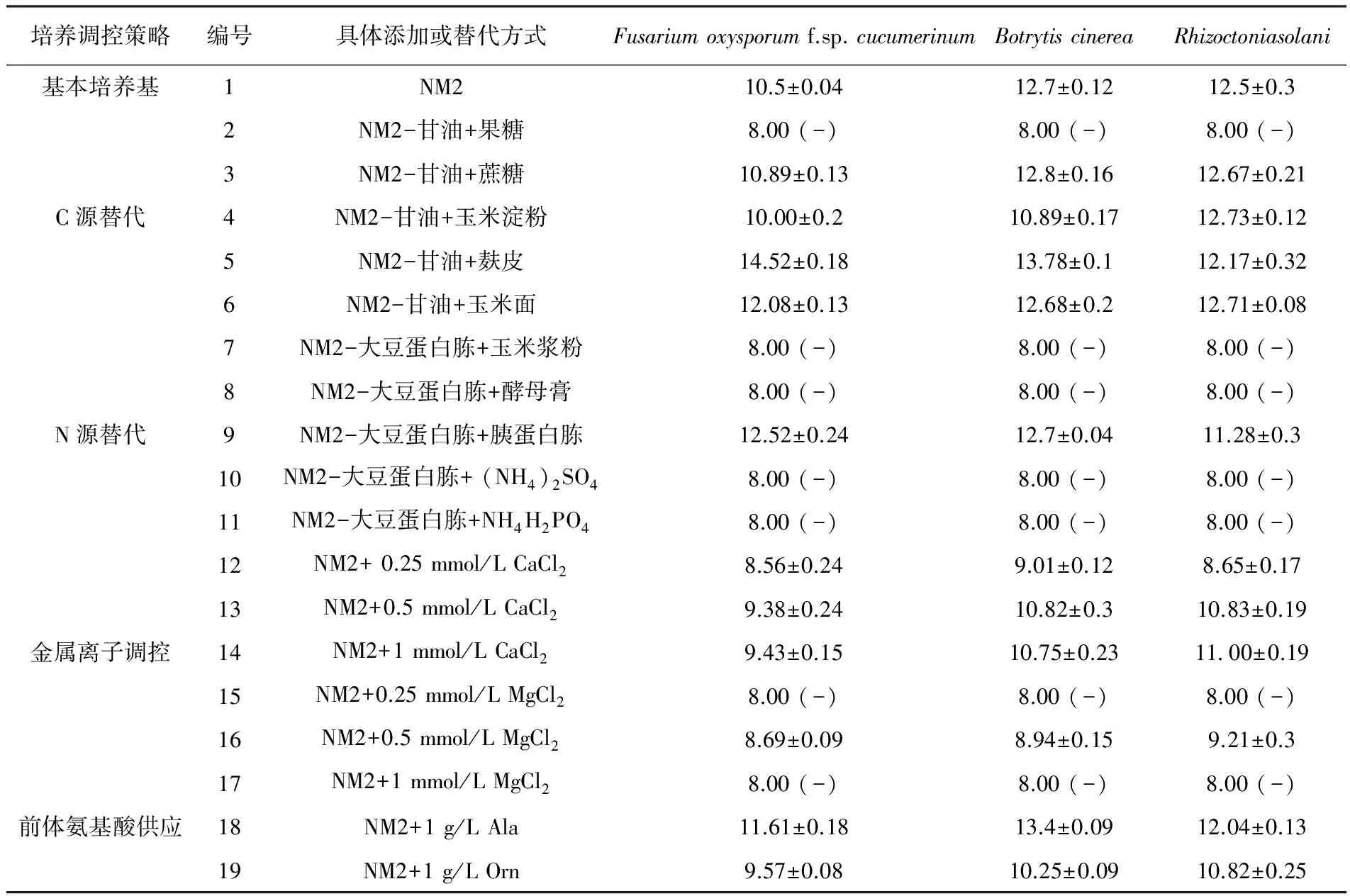
表1 培养基的设计及发酵粗提物对病原真菌的活性测试Table 1 The design of medium and the inhibitory activities against pathogenic fungi
注:表中数据代表抑菌圈直径(mean±s.d,mm);萃取液质量浓度为10 mg/mL;滤纸片直径为8 mm;上样20 μL;“-”代表“替代”;“+”代表“添加” (如:“NM2-甘油+果糖”代表“将NM2培养基中的甘油用等量的果糖替代”)
2.3 抗真菌活性的发酵粗提物HPLC-UV分析
对具有抗真菌活性的粗提物,尤其是抗菌活性增强的粗提物,进行HPLC-UV检测,分析代谢产物的变化情况。比较使用不同碳源替代甘油培养NA4研究发现,在产生抗真菌活性的发酵培养基中,除麸皮替代甘油外均产生了bafilomylins,它们对特定病原菌的抗菌强度变化可能因为bafilomycins各同系物相对产量不同引起。而麸皮替代后抗真菌活性增强,但bafilomycins色谱峰消失,在RT 45~50 min出现了一系列色谱峰,提示其已经激活了其他类型抗真菌次级代谢产物(图1)。氮源替代结果显示,胰蛋白胨替代大豆蛋白胨对特定真菌的抑制强度发生变化主要是因为bafilomycins的几种主要产物的相对含量变化造成(图2)。而添加前体氨基酸引起的抗真菌活性变化主要是bafilomycins产量提高(添加Ala)或bafilomycins产量降低(添加Orn)引起。添加二价金属离子导致抗真菌活性的降低也主要是bafilomycins产量减少的原因(图3)。综上,结合抗真菌活性和HPLC-UV图谱分析结果,选择麸皮替代甘油的发酵产物进行LC-DAD-MS结构分析和识别。

图1 碳源替代方式下不同培养基中具有抗真菌活性粗提物的HPLC-UV图谱Fig.1 The HPLC-UV fingerprint profiles of the antifungal activity of crude extracts in different media with
2.4 HPLC-DAD-MS分析和识别麸皮替代甘油后激活的抗真菌次级代谢产物
从HPLC-DAD图谱可以看出,麸皮替代甘油后激活产生了一系列紫外最大吸收波长约为230 nm、320 nm的化合物(图4),推测它们应为同一类型的化合物。采用正负离子扫描模式对其主要的7个产物进行LC-MS分析,推测其分子量,结合其DAD紫外吸收特征、抗真菌活性以及生物信息学分析结果可推断它们是PTM化合物家族中的frontalamides及其衍生物。MS数据分析显示(表2),化合物6对应的分子量为508 Da,推测为frontalamide B,化合物2和5相应分子量均为512 Da,为HSAF(heat-stable antifungal factor)的同分异构体,化合物1和3分子量为526 Da,而目前尚未发现该分子量的PTM类化合物,推测它们可能为新的frontalamide A二氢还原产物。
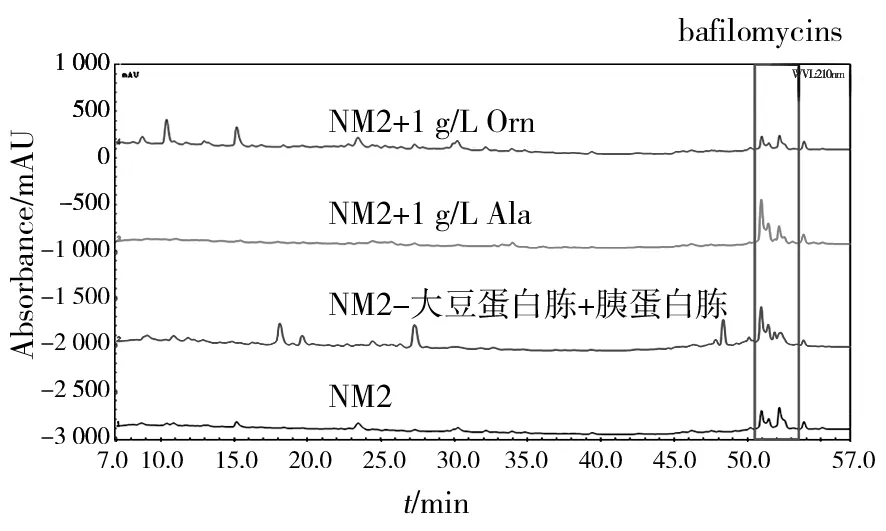
图2 氮源替代和添加氨基酸前体方式下不同培养基中具有抗真菌活性粗提物的HPLC-UV图谱Fig.2 The HPLC-UV fingerprint profiles of the antifungal activity of crude extracts in different media with nitrogen source substitution and adding amino acid precursor

图3 添加金属离子条件下不同培养基中具有抗真菌活性粗提物的HPLC-UV图谱Fig.3 The HPLC-UV fingerprint profiles of the antifungal activity of crude extracts in different media with adding metal ion

图4 链霉菌NA4“NM2-甘油+麸皮”培养基发酵粗提物的HPLC-DAD图谱Fig.4 The HPLC-DAD fingerprint profiles of the fermentation crude growing in the NM2 medium replacing glycerol with bran
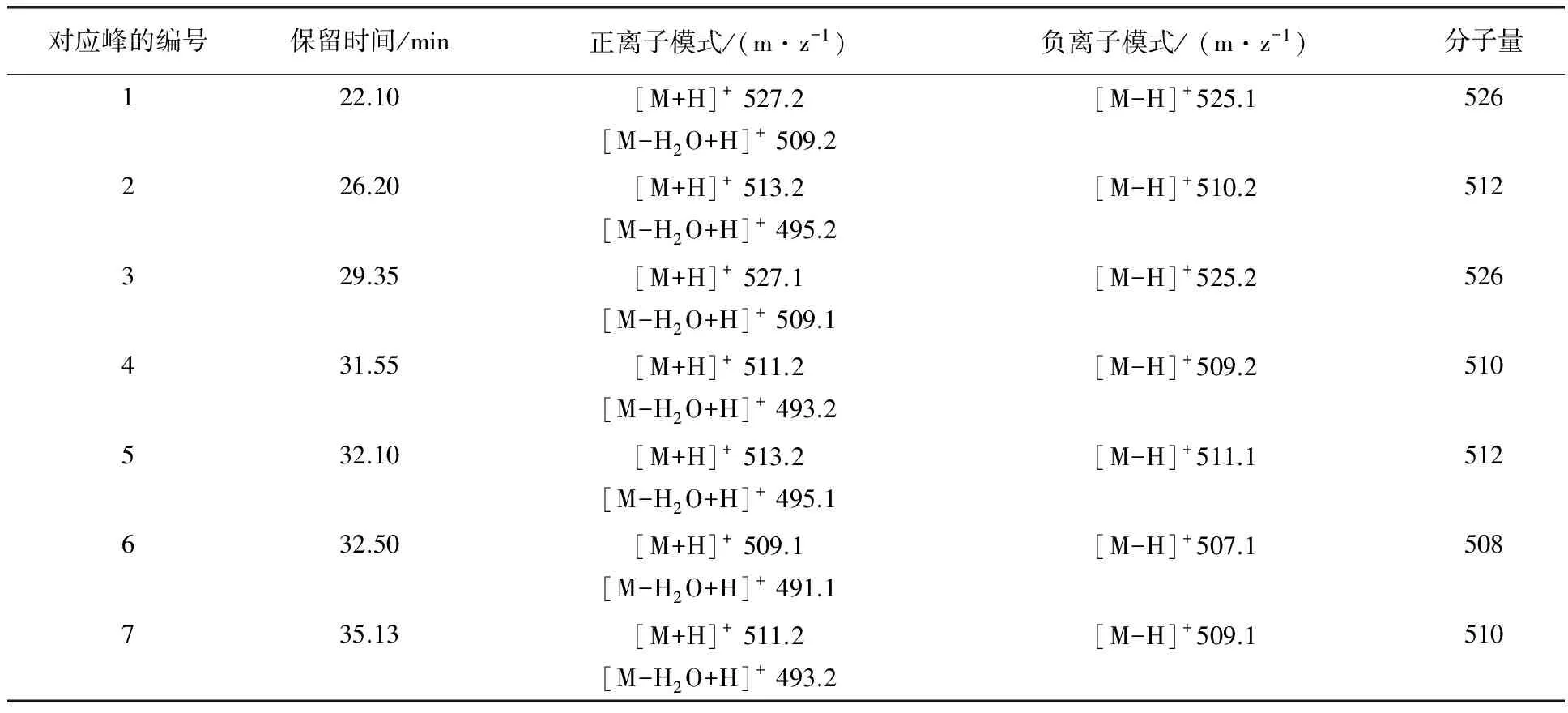
表2 基于LC-MS分析获得的“NM2-甘油+麸皮”培养基发酵粗提物中主要的PTMs化合物Table 2 The main PTM compounds of the fermentation crude of strain NA4 growing in the NM2 medium replacing glycerol with bran using LC-MS detection
3 讨 论
卡伍尔氏链霉菌 NA4分离自中国南海1 464 m深的海底沉积物,是挖掘抗真菌抗生素的新资源。为了深入挖掘NA4中抗真菌活性的次级代谢产物,本研究采用培养调控的手段尝试对上述预测化合物的合成基因簇进行激活。研究发现,碳源改变对次级代谢产物影响很大,甘油被果糖替代后抗真菌活性完全消失,不产生bafilomycins(经HPLC-UV分析证明,数据未显示);甘油被麸皮替代产生了PTM类型的抗真菌活性物质。甘油是三碳延伸单位的前体,它的添加有利于大环内酯类化合物的形成[20]。据报道bafilomycins骨架合成的结构延伸单元甲氧基丙二酰CoA是以甘油为前体合成的[21]。因此将基本培养基中的甘油用其他碳源替代,将不利于bafilomycins的产生,而有利于激活其他非PKS类型的基因簇。而其他碳源替代则引起了bafilomycins产量及其组成相对含量的变化。蛋白胨作为重要的氮源,其存在与否及含量的改变都对微生物初级代谢和次级代谢有很大的影响[22]。因此将基本培养基中的大豆蛋白胨用其他氮源替代,探索对链霉菌次级代谢的影响,以期激活初始条件下难以产生的化合物。氮源替代实验表明,将基础培养基中大豆蛋白胨替换后,除了胰蛋白胨外,其余都不再产生bafilomycins,这也间接证明蛋白胨有利于微生物初次和次级代谢产物的产生[25]。Orn是PTM类化合物NRPS模块腺苷酰化结构域识别的特异性氨基酸,Ala是valinomycin中结构单元L-Lac的前体,也是转氨酶常用的氨基供体。期望Orn、Ala的添加有利于PTM和valinomycin化合物合成基因簇的激活,但很遗憾没有发现这些物质的产生。Ca2+和Mg2+等二价金属离子是微生物代谢中许多关键酶的辅因子,它们参与细胞代谢并影响着细胞内的能量供应、还原-氧化水平等[23]。但添加不同浓度二价离子后,发现bafilomycins产量减少,抗真菌活性降低,并没有出现其他抗真菌物质。以上实验提示,虽然Orn、Ala和二价金属离子对链霉菌NA4的次级代谢有很大影响,但要产生有利预期的调控,其添加的浓度和方式还需要优化。
PTM是PKS-NRPS杂合的具有多环稠合的大环内酰胺类化合物,目前发现的这类化合物有40多种,包括frontalamides、alteramide A、ikarugamycin等[23]。PTM生物合成基因簇保守且广泛存在,该家族的化合物具有多种生物活性,包括抗真菌、抗原虫、抗细菌、抗肿瘤、抗氧化等活性[24]。但是许多PTM合成基因簇往往是沉默的,因此越来越多的科学家通过激活沉默的PTM合成基因簇以挖掘更多结构衍生物[26,30]。在PTM家族化合物中存在5/5/6或5/6/5环两个大类,许多研究表明PTM类物质的合成基因簇基因构成决定了其复杂的成环机制[26,28-29]。在ikarugamycin的生物合成基因簇中FAD依赖的氧化还原酶IkaB催化形成外部5/6环,并最终在NADPH依赖的脱氢酶IkaC催化下形成ikarugamycin的内部五元环[29](图5)。在frontalamides、HSAF以及pactamides等具有5/5/6环的PTM合成基因簇中由脱氢酶PtmB2/FtdC和氧化还原酶PtmB1/FtdB先后催化形成外部5/5环,并在双功能乙醇脱氢酶PtmC作用下最终形成5/5/6环结构[30](图5)。链霉菌NA4的PTM基因簇orf 3和orf 4的基因编码蛋白分别与PtmB1和PtmB2同源,推测其应该合成5/5/6环结构的PTM类化合物。菌株NA4的PTM基因簇与frontalamides基因簇都有P450氧化酶(Orf6/FtdF),具有更相似的结构,其是否参与6元环上的氧化值得深入研究。
将NM2培养基中的甘油用麸皮替代后进行的NA4发酵,激活了最大紫外吸收波长约为230 nm和320 nm特征性PTM类化合物。其中化合物4和7分子量为510 Da,可能是已知PTM maltophilin、alteramide A、xanthobaccin A、lysobacteramide B和FI-2等中的1个或2个;化合物1和3分子量为526 Da,MS图谱中具有较强的脱水离子峰,提示在5/5/6环中存在羟基,符合frontalamide A的结构特征,由于化合物1和3与frontalamide与其分子量相差2,所以推测它们可能为frontalamide A 5/5/6环中双键还原或者羰基还原的产物(图5);化合物2和5为HSAF的同分异构体,目前分子量为512 Da的PTM只有1个HSAF,因此化合物2或5中至少有1个是新的PTM类化合物。若想确定化合物2或5中具体有几种新的PTM类化合物,还需要进一步实验证明。
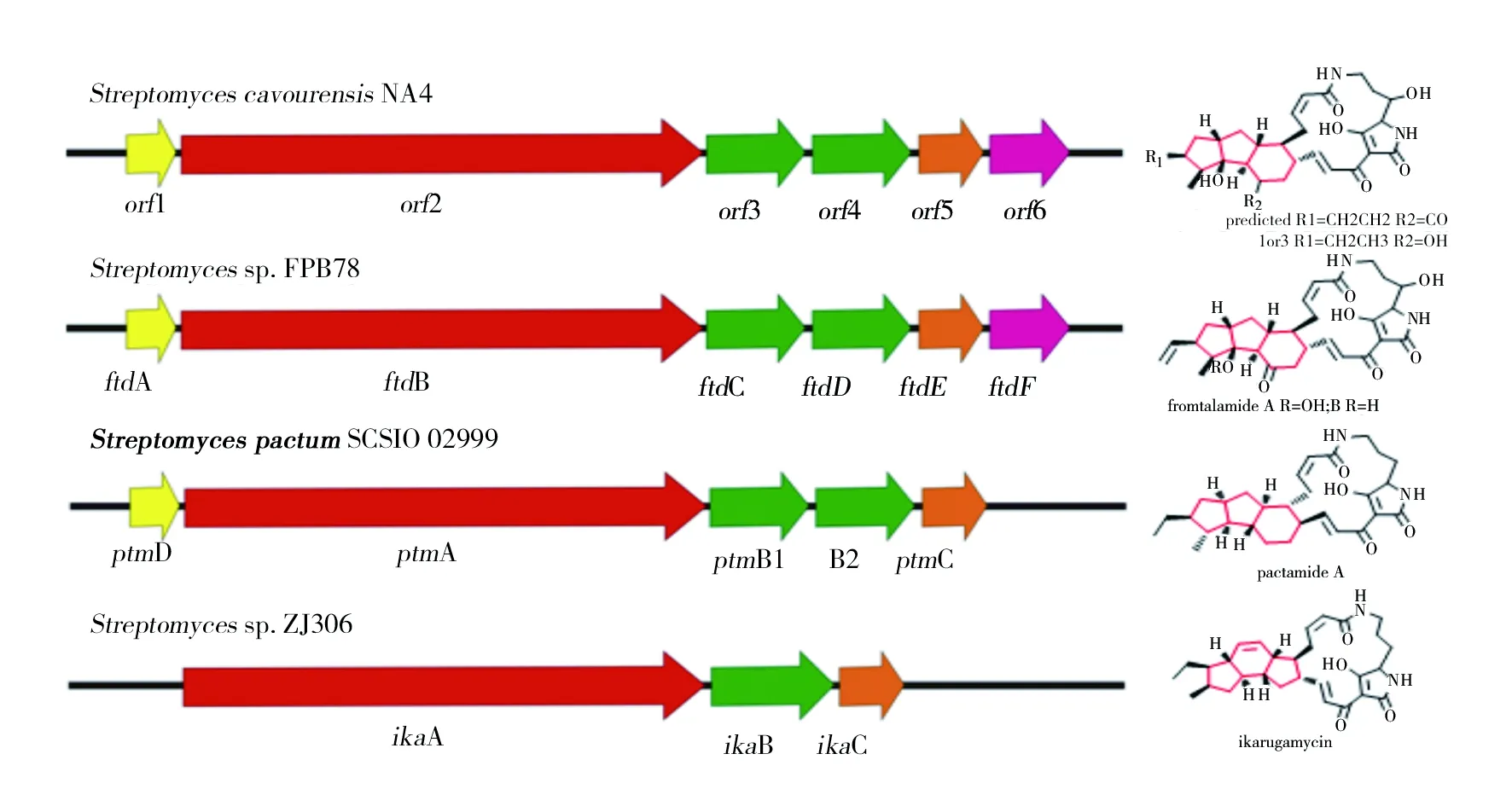
图5 5/5/6环和5/6/5环PTM化合物生物合成基因簇Fig.5 The biosynthetic clusters of the compounds of PTM about the ring of 5/5/6 and 5/6/5
参考文献:
[1] Fisher MC,Henk DA,Briggs CJ,et al.Emerging fungal threats to animal, plant and ecosystem health[J]. Nature, 2012, 484 (7393): 186-194.
[2] 李扬, 王耀雯, 王育荣, 等. 水稻稻瘟病菌研究进展[J]. 广西农业科学, 2010, 41(8): 789-792.
[3] 张洁, 伊艳杰, 王金水, 等. 小麦赤霉病的防治技术研究进展[J]. 中国植保导刊, 2014, 34(1): 24-28.
[4] Thaden JT,Lewis SS,Hazen KC, et al. Rising rates of carbapenem-resistant enterobacteriaceae in community hospitals: a mixed-methods review of epidemiology and microbiology practices in a network of community hospitals in the southeastern United States[J]. Infection Control & Hospital Epidemiology, 2014, 35(8): 978-983.
[5] 武英, 王辂. 开发微生物来源天然产物的新技术[J]. 国外医药抗生素, 2010, 31(1): 1-6.
[6] Katz L,Baltz RH. Natural product discovery: past, present, and future[J]. Journal of Industrial Microbiology & Biotechnology, 2016, 43(2): 155-176.
[7] Udaany DW,Zeigler L,Asolkar RN,et al. Genome sequencing reveals complex secondary metabolome in the marine actinomyceteSalinisporatropica[J]. Proceedings of the National Academy of Sciences, 2007, 104(25): 10376-10381.
[8] Omura S, Ikeda H, Ishikawa J, et al. Genome sequence of an industrial microorganismStreptomycesavermitilis: Deducing the ability of producing secondary metabolites[J]. Proceedings of the National Academy of Sciences, 2001, 98(21): 12215-12220.
[9] Eerikly M,Challis G. Strategies for the discovery of new natural products by genome mining[J]. Challis ChemBioChem, 2009, 10(4): 625-633.
[10] 钟晶晶,赫卫清. 链霉菌隐性次级代谢产物生物合成基因簇的激活[J]. 生命的化学, 2016, 36(2): 231-236.

[12] Zhang W, Liu Z, Li S, et al. Spiroindimicins A-D: new bisindole alkaloids from a deep-sea-derived actinomycete[J]. Organic Letters, 2012, 14(13): 3364-3367.
[13] Saurav K,Zhang W,Saha S, et al. In silico molecular docking, preclinical evaluation of spiroindimicins A-D, lynamicin A and D isolated from deep marine sea derivedStreptomycessp. SCSIO 03032[J]. Interdisciplinary Sciences: Computational Life Sciences, 2014, 6(3): 187-196.
[14] Zhang W,Li S,Zhu Y, et al. Heronamides D-F, polyketide macrolactams from the deep-sea-derivedStreptomycessp. SCSIO 03032[J]. Journal of Natural Products, 2014, 77(2): 388-391.
[15] Pan HQ,Yu SY,Song CF, et al. Identification and characterization of the cntifungal substances of a novelStreptomycescavourensisNA4[J]. Journal of Microbiology and Biotechnology, 2015, 25(3): 353-357.
[16] Ramadhar TR,Beemelmanns C,Currie CR, et al. Bacterial symbionts in agricultural systems provide a strategic source for antibiotic discovery[J]. Journal of Antibiotics, 2013, 67(1): 53-58.
[17] Matter AM,Hoot SB,Anderson PD, et al. Valinomycin biosynthetic gene cluster inStreptomyces: conservation, ecology and evolution[J]. Plos One, 2009, 4(9): 761-768.
[18] Kanda N,Ishizaki N, Inoue N, et al. DB-2073, a new alkylresorcinol antibiotic. I. taxonomy,isolation and characterization[J]. Journal of Antibiotics, 1975, 28(12): 935-942.
[19] Weber T, Blin K, Duddela S, et al. AntiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene cluster[J]. Nucleic Acids Research,2015,43(W1):W237-W243.
[20] Walton LJ,Corre C,Challis GL. Mechanisms for incorporation of glycerol-derived precursors into polyketide metabolites[J]. Journal of Industrial Microbiology & Biotechnology, 2005, 33(2): 105-120.
[21] Zhang W, Fortman JL,Carlson JC, et al. Characterization of the bafilomycin biosynthetic gene cluster fromStreptomyceslohii[J]. ChemBioChem, 2013, 14(3): 301-306.
[22] 陈竹, 李万钧, 储炬, 等. 工业羽毛蛋白胨对红霉素基因工程菌ZL1004发酵过程的影响[J]. 中国抗生素杂志, 2011, 36(10): 751-757.
[23] Dobson LF, O′Shea DG. Antagonistic effect of divalent cations Ca2+and Mg2+on the morphological development ofStreptomyceshygroscopicusvar.geldanus[J]. Applied Microbiology and Biotechnology, 2008, 81(1): 119-126.
[24] Zhang G, Zhang W, Saha S, et al. Recent advances in discovery, biosynthesis and genome mining of medicinally relevant polyayclic totramate macrolactams[J]. Current Topics in Medicinal Chemistry, 2016, 16(15): 1727-1739.
[25] Jomon K,Kuroda Y,Ajisaka M, et al. A new antibiotic, ikarugamycin[J]. Journal of Antibiotics, 1972, 25: 271-280.
[26] Luo Y,Huang H,Liang J, et al. Activation and characterization of a cryptic polycyclic tetramate macrolactam biosynthetic gene cluster[J]. Nature Communications, 2013, 4(1):1-8.
[27] Blodgett JA,Oh DC, Cao S, et al. Common biosynthetic origins for polycyclic tetramate macrolactams from phylogenetically diverse bacteria[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(26): 11692-11697.
[28] Lou L,Qian G, Xie Y,et al. Biosynthesis of HSAF, a tetramic acid-containing macrolactam fromLysobacterenzymogenes[J]. Journal of the American Chemical Society, 2011, 133(4): 643-645.
[29] Zhang G,Zhang W,Zhang QZ, et al. Mechanistic insights into polycycle formation by reductive cyclization in ikarugamycin biosynthesis[J]. Angewandte Chemie International Edition, 2014, 53(19): 4840-4844.
[30] Saha S,Zhang W,Zhang G et al. Activation and characterization of a cryptic gene cluster reveals a cyclization cascade for polycyclic tetramate macrolactams[J]. Chemical Science, 2017, 8(2): 1607-1612.

