基于反转录环介导等温扩增技术检测大肠杆菌O157
梁玉林 刘秀 周鹏飞 周振森 尹建军
(中国食品发酵工业研究院,北京 100015)
大肠杆菌是人肠道中正常的革兰氏阴性菌,大多数大肠杆菌没有致病性。正常情况下这些细菌寄生在人的消化道中,并随粪便排出体外,因此大肠杆菌广泛存在水和土壤中[1-4]。如果人体免疫机能降低或者大肠杆菌入侵肠道外组织时,它们会成为条件致病菌从而引起人类疾病。根据不同的生物学特性将致病性大肠杆菌分为5类:致病性大肠杆菌(Enteropathogenic Escherichia coli,EPEC)、肠产毒性大肠杆菌(Enterotoxigenic Escherichia coli,ETEC)、肠侵袭性大肠杆菌(Enteroinvasive Escherichia coli,EIEC)、肠出血性大肠杆菌(Enterohernorrhage Escherichia coli,EHEC)和肠黏附性大肠杆菌(Enteroadhesive Escherichia coli,EAEC)。肠出血性大肠杆菌主要包括O157、O26、O111和O104等几种血清型[5-7]。其中大肠杆菌O157是最主要的食源性致病菌之一,它除了能引起腹泻、出血性肠炎外,还可发生溶血性尿毒综合症(Hemolytic uremic syndrome,HUS)、血栓性血小板减少性紫癜(Thrombotic thrombocytopenic purpura,TTP)等并发症,后者病情严重,病死率高[8]。感染性腹泻是近年新发现的危害严重的肠道传染病。自1982年美国首次发现该病以来,在许多国家相继爆发和流行,其流行已成为全球性公共卫生问题之一[9]。
大肠杆菌O157检测方法主要是传统培养分离法[10],分离出的病原菌还要对其进一步分型确认。该方法费时费力,当某种致病菌的检测需要快速出具结果时,传统培养分离法的严重滞后性就会凸显出来。近年来,致病菌分子检测方法层出不穷,分子检测不但省时省力而且具有比较高的特异性和灵敏度[11]。张微等[12]利用荧光定量PCR技术,建立快速、有效的检测乳中大肠杆菌O157的方法。尽管该方法操作简单,灵敏度达到67 CFU/mL,但需要专业人员和高精密的仪器设备,不易在基层推广。因此,需要开发一种快速、灵敏、准确的方法来现场检测大肠杆菌O157。
环介导等温扩增(Loop-mediated isothermal amplification,LAMP)技术是一种新的核酸扩增方法[13],其扩增过程中的DNA聚合酶采用具有链置换活性的DNA聚合酶,整个DNA扩增是在60-65℃恒温条件下进行的快速扩增反应。由于DNA双链结构稳定,在死菌中能长时间存在,以DNA为检测模板容易检出死菌中残留的核酸而出现假阳性的后果,不能体现样本中的活菌数量[14]。RNA易降解,不能长时间存在于死菌当中,反转录-环介导等温扩增(RT-LAMP)技术是以RNA为检测模板的扩增反应[15-16],除了具有常规LAMP的优势外,还具有有效区分死菌和活菌的独特优势。本研究以大肠杆菌O157的特异性保守基因序列设计多组引物并筛选,构建基于RNA为检测模板的RT-LAMP技术体系。
1 材料与方法
1.1 材料
1.1.1 材料与试剂 大肠杆菌O157等26株细菌如表1所示;溶菌酶,天根生物科技公司;RNeasy Mini kit,QIAgen 公司;Isothermal Master Mix(IMM),英国OptiGene Limited公司;焦炭酸二乙酯(DEPC),中科瑞泰(北京)生物科技有限公司;平板计数琼脂(PCA),北京路桥技术股份有限公司;One Step PrimeScript RT-PCR kit,宝生物工程(大连)有限公司。

表1 RT-LAMP反应特异性
1.1.2 仪器与设备 微量移液器,德国Eppendorf公司;高压灭菌锅(SX-700),日本Tomy Digital Biology公司;精密天平(CPA323S),德国Sartorius公司;超纯水仪(Mili-Q Advantage A10),德国默克公司;离心机(5804R型,5424型),德国Eppendorf公司;超微量核酸蛋白分析仪(Biodrop),英国柏点公司;GenieⅢ,英国OptiGene Limited公司;生物安全柜,新加坡Esco公司。ABI 7900荧光PCR仪,美国Thermo Fisher Scientific公司。
1.2 方法
1.2.1 引物设计 根据GenBank中的rfbE基因(GenBank Accession S83460.1)的序列,利用LAMP Primer Explorer 4在线软件(http://primer explorer.jp/elamp4.0.0/index.htm l)设计8组引物,每组引物分别包括外引物F3、B3,内引物FIP、BIP和环引物LoopF、LoopB。将8组引物进行RT-LAMP实验,选取稳定性强、扩增时间快和荧光值相对较高的引物组进行下一步试验,每组引物平行进行3次RTLAMP测试。
1.2.2 纯培养细菌的RNA提取 在平板上取大肠杆菌O157单菌落接入LB液体培养基中,培养12 h到对数期。取1 mL菌液5 000×g离心10 min,轻轻倒掉液体。向沉淀加入10 μL蛋白酶K,100 μL TE(含终浓度15 mg/mL溶菌酶),充分混匀沉淀,在室温下反应30 min,接下步骤严格按照RNeasy Mini kit操作步骤提取。
1.2.3 rRT-PCR和RT-LAMP反应 rRT-PCR扩增引 物 rfbE-F :5′-TCCTCAGCTATAGGGTGCTTTG-3′,rfbE-R :5′-ATCGAAACAAGGCCAGTTTTTTAC-3′,rfbE-BJP :5′-ATACGATAACATCCACGGCTCTGGCT GG-3′。按照 One Step PrimeScript RT-PCR kit操作步骤进行反转录和进一步核酸扩增反应。扩增条件为50℃ 2 min,95℃ 10 min,95℃ 15s,60℃ 1 min,共45个循环。对于Ct值≤40的扩增结果判定为阳性。
经优化后在 12.5 μL 体系中,包括 7.5 μL Mix、2.5 μL混合引物(外引物、内引物和环引物含量分别为2.5 pmol、10 pmol和 5 pmol)、1.5 μL 模 板。RT-LAMP反应温度65℃,反应时间30-60 min。
1.2.4 大肠杆菌O157死菌中RNA降解情况 为验证大肠杆菌O157死菌中RNA降解情况,避免死菌中DNA对RT-LAMP检测结果的干扰。我们选取104、103、102、101CFU/mL 四个菌液浓度。121℃下20 min高压蒸汽灭菌,放置一段时间,模拟死菌状态,对死菌进行平板培养,验证是否灭菌完全。分别对灭菌前后各浓度梯度的大肠杆菌O157菌液进行RNA提取,并进行RT-LAMP反应。
1.2.5 RT-LAMP反应的特异性和灵敏度 提取大肠杆菌O157等26株细菌RNA,并以RNA为检测模板进行RT-LAMP反应验证该方法的特异性。
将提取的大肠杆菌O157原菌液总RNA测定浓度后,对总RNA进行稀释,最后选取3 fg、30 fg、300 fg、3 pg、30 pg、300 pg、3 ng/μL 共 7 个 浓 度梯度,从各浓度梯度各取1.5 μL作为检测样本进行RT-LAMP反应检测纯培养大肠杆菌O157灵敏度。
1.2.6 rRT-PCR和RT-LAMP反应在人工污染脱脂乳中的灵敏度检测 为保证检测结果的准确性,经国标GB/T4789.2-2016方法证实试验所用脱脂乳中不含大肠杆菌O157,取25 g脱脂乳置于225 mL灭菌生理盐水中样品。取新鲜培养的12 h细菌,用生理盐水充分洗涤培养基,10倍梯度系列稀释,取各稀释度菌液1 mL加入脱脂乳匀浆中,制备人工污染脱脂乳匀浆,使人工污染脱脂乳匀浆中大肠杆菌O157含量达到10-1-105CFU/mL,充分混合,作为人工污染脱脂乳样品。
取各菌液浓度的人工污染脱脂乳样品1 mL,提取人工污染脱脂乳样品中的RNA,同时取不加大肠杆菌O157的脱脂乳样品1 mL作为阴性对照平行进行RNA提取。以提取的RNA为检测模板分别进行rRT-PCR和RT-LAMP反应,比较两种方法的检测灵敏度。每个浓度的RNA模板分别进行5次独立rRTPCR和RT-LAMP试验,当某个浓度梯度的RNA模板的5次试验中有1次无法观察到扩增曲线,该浓度的上一稀释度才能被认定为最低检测灵敏度。
2 结果
2.1 八组大肠杆菌O157引物筛选
根据引物间距离、GC含量、引物序列Tm值、引物末端稳定性和二级结构5个因素,实验共设计了8组LAMP引物。如图1在整个实验室不含大肠杆菌O157核酸样品情况下,对8组引物进行RTLAMP扩增反应发现3号、4号和8号引物均发生扩增反应,可以断定该扩增反应是非特异性的,可能是由于引物间的碱基互补配对引起的扩增反应。如图2在含有大肠杆菌O157核酸样品情况下,对8组引物进行RT-LAMP扩增反应,2号引物出现扩增曲线的时间最短。综合3次测试结果,2号引物具有较高的稳定性,其在反应时间上明显比其它引物组都快且荧光值相对较好。选取2号引物作为RT-LAMP反应引物进行下一步实验,2号引物序列如表2。
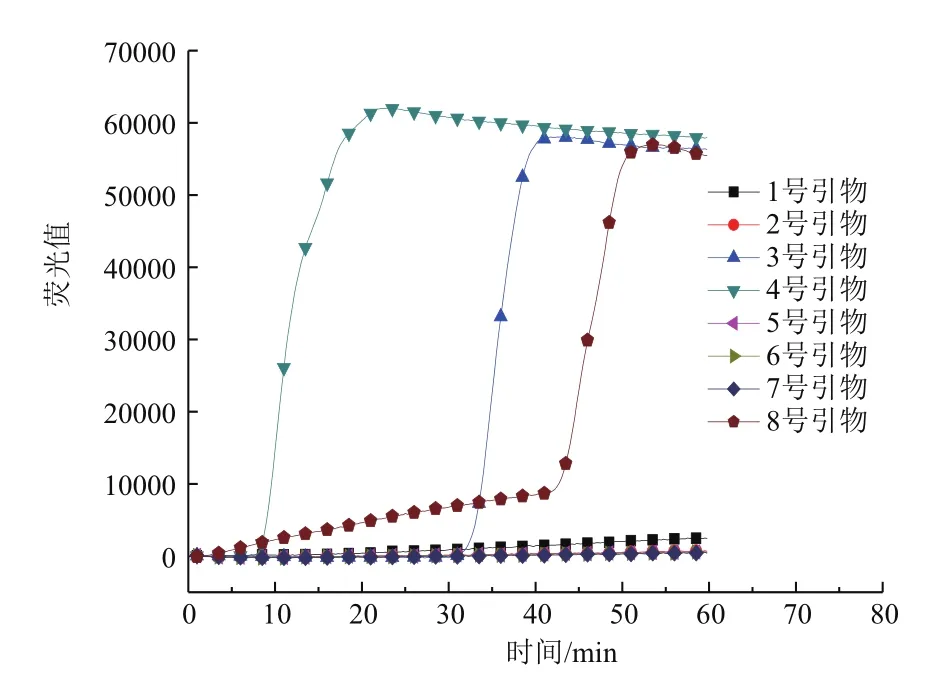
图1 大肠杆菌O157引物筛选(不含检测模板)
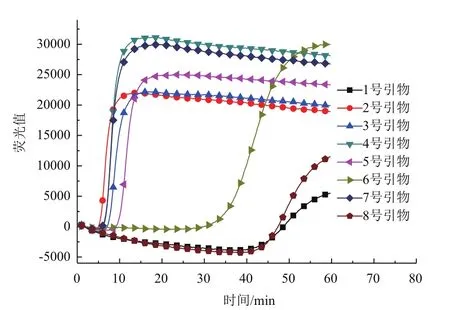
图2 大肠杆菌O157引物筛选(含检测模板)
2.2 大肠杆菌O157死菌中RNA降解情况
在121℃下20 min高压蒸汽灭菌处理的大肠杆菌O157,经平板培养观察未发现形成菌落,灭菌后的细菌均无繁殖能力。对灭菌前后不同浓度的大肠杆菌O157菌液进行RNA提取,并经RT-LAMP检测。图3为灭菌前大肠杆菌O157 RT-LAMP扩增反应,可以看出提取101CFU/mL菌液RNA进行RTLAMP反应仍能出现荧光扩增,说明反应体系中存在RNA。图4为灭菌后大肠杆菌O157 RT-LAMP扩增反应,可以得出各菌液浓度的核酸提取物中未含有RNA。结合图3和图4进一步证实104CFU/mL及其以下死菌菌液中RNA降解完全,以RNA为检测模板进行RT-LAMP反应可以鉴别死活菌。
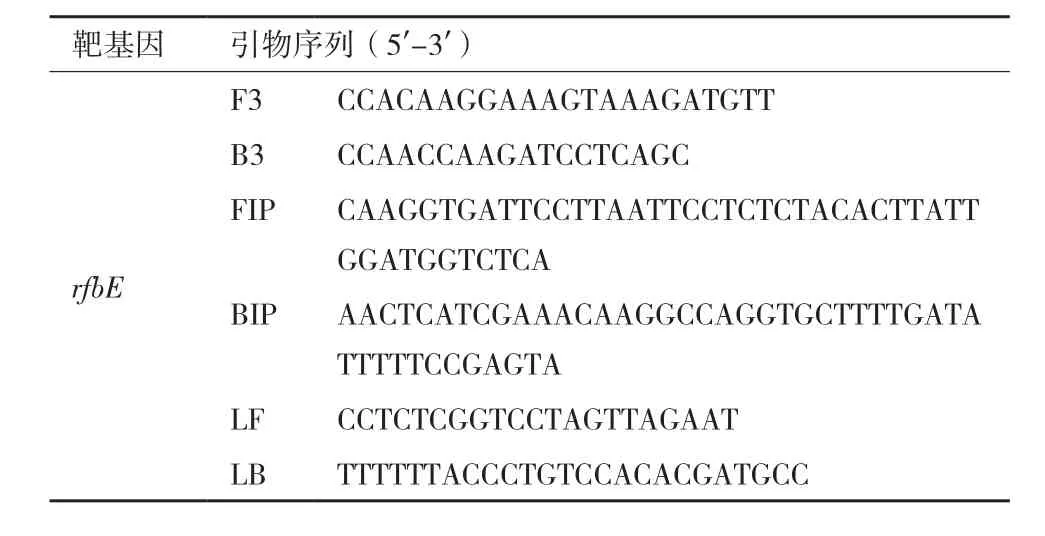
表2 大肠杆菌O157靶基因引物序列
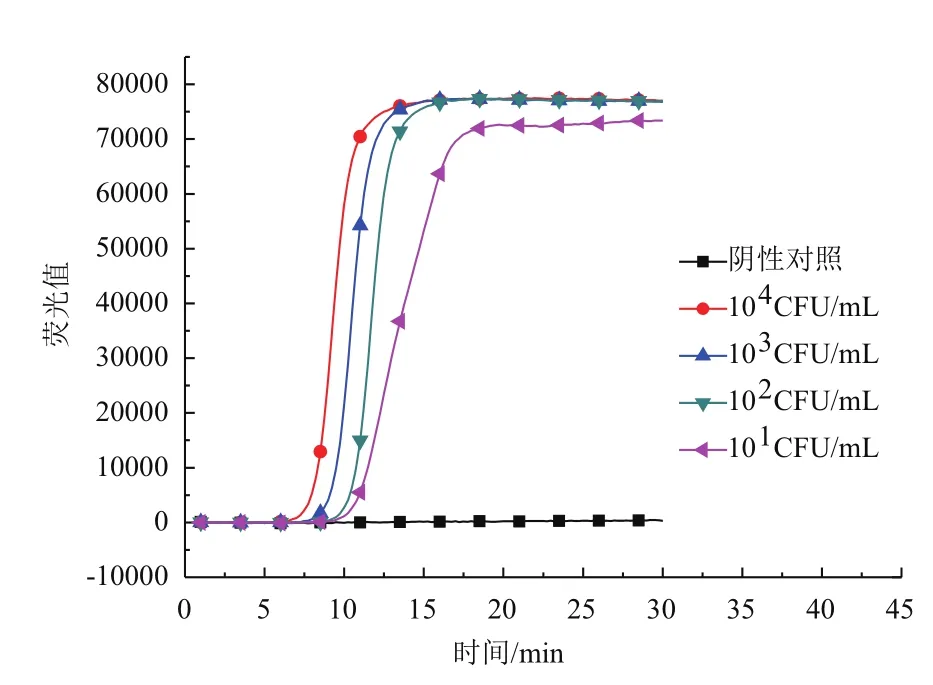
图3 活菌RNA检测
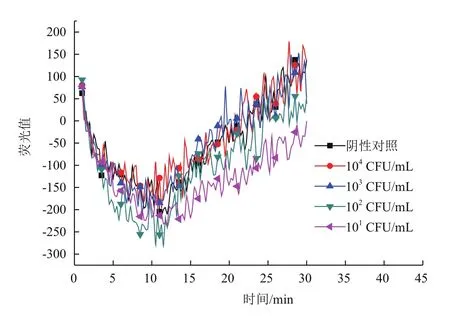
图4 死菌RNA检测
2.3 RT-LAMP反应特异性和灵敏度
本研究共对26株细菌进行RT-LAMP试验,其中只有2株大肠杆菌O157产生特异性扩增反应,其他大肠杆菌血清型和非大肠杆菌菌株均未产生特异性扩增反应(表1)。说明筛选到的引物具有较高的特异性,只能特异性检出大肠杆菌O157。
对提取的纯培养大肠杆菌O157的总RNA进行10倍梯度稀释,验证建立的方法对纯培养大肠杆菌O157的检测灵敏度。结果如图5所示,当总RNA稀释到30 fg/μL时,RT-LAMP反应仍能产生扩增曲线,而继续稀释核酸将不会产生扩增曲线,表明纯培养大肠杆菌O157检测灵敏度达到30 fg/μL。
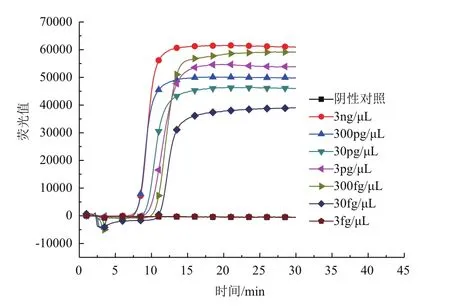
图5 RT-LAMP反应灵敏度
2.4 rRT-PCR和RT-LAMP反应在人工污染脱脂乳中的灵敏度检测
将提取的人工污染脱脂乳样品RNA溶解到50 μL不含RNase的无菌水中,取其中1.5 μL作模板,分别进行rRT-PCR和RT-LAMP反应。同时取1 mL各浓度梯度的人工污染脱脂乳样品进行平板菌落计数,记录各浓度梯度人工污染脱脂乳样品中大肠杆菌O157的准确含量。从图6可以看出rRT-PCR检测灵敏度达到20 CFU/mL,换算成固体脱脂乳污染样品检测灵敏度为200 CFU/g。从图7可以得出RTLAMP反应灵敏度达到2 CFU/mL,换算成固体脱脂乳污染样品检测灵敏度为20 CFU/g。所建立的RTLAMP方法灵敏度高,比rRT-PCR反应灵敏度高10倍。
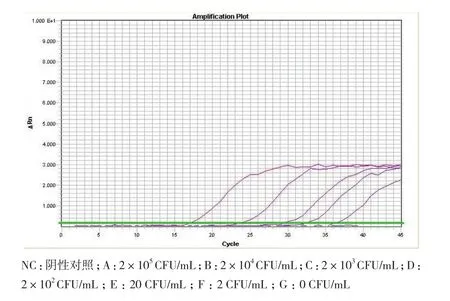
图6 人工污染脱脂乳rRT-PCR反应灵敏度
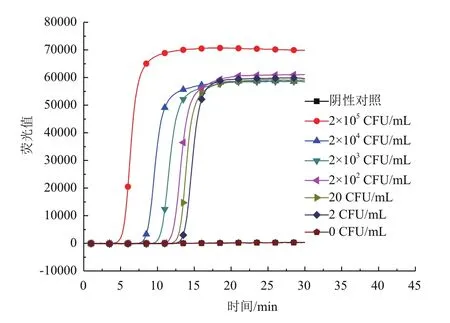
图7 人工污染脱脂乳RT-LAMP反应灵敏度
3 讨论
目前国内外文献报道的大肠杆菌O157的LAMP检测方法多以stex、eae、wzy和rfbE等基因作为特异性扩增靶基因,其中stex是编码志贺毒素毒力因子基因[17-19],但有些大肠杆菌O157缺失stx基因,以stex基因序列设计LAMP引物可能不会扩增出目的基因条带进而造成假阴性的问题。wzy是编码O抗原聚合酶的基因[20],eae是编码紧密黏附素的基因[21],然而eae和wzy基因除了存在于大肠杆菌O157外,也少量存在于一些肠科细菌,以这两种基因序列设计的LAMP引物可能会检测出大肠杆菌O157以外的肠科细菌,进而造成假阳性的后果。研究表明大肠杆菌的rfb基因作为靶基因具有很好的保守性[22],大肠杆菌rfb基因是O抗原的编码基因,不同序列的rfb基因编码产生不同的O抗原,其中大肠杆菌O157的O抗原特异性编码基因为rfbE基因[23]。本研究以rfbE基因设计引物,以RNA为检测模板进行RT-LAMP反应,结果能够证实所检测的大肠杆菌O157是否处于活的增殖状态[24-26]。此外实验依靠GenieⅢ平台使得反转录过程和扩增过程同时进行,大大降低了反应时间,有利于现场快速检测工作的开展。
在人工污染脱脂乳样品实验中,当样品中大肠杆菌O157浓度稀释到2 CFU/mL时,建立的实时荧光RT-LAMP方法每次均能特异性检出,当菌液继续稀释到更低时,该方法有时不能检出。对于细菌含量很少的样品,尤其是量少也易致病的大肠杆菌O157,如果不经过前增菌很有可能造成漏检的严重后果,为保证结果的可靠性可以适当加上前增菌等步骤。因为受大肠杆菌O157污染的食品种类很多,实验只进行了人工污染脱脂乳实验,后期需要扩大人工污染食品种类,验证方法的可靠性。
4 结论
本研究对大肠杆菌O157设计多组LAMP引物并筛选,确定反应体系配比,从而建立了检测大肠杆菌O157的实时荧光RT-LAMP方法。该方法只特异性检出大肠杆菌O157,其他大肠杆菌血清型和非大肠杆菌菌株均未产生特异性扩增反应,对固体脱脂乳污染样品检测灵敏度达到20 CFU/g。
[1]Chen ZZ, Li C, Chen MY. Indirect immunofluorescence detection of E. coli O157:H7 with fluorescent silica nanoparticles[J].Biosensors and Bioelectronics, 2015, 66(4):95-102.
[2]Kanki M, Seto K., Harada T. Comparison of four enrichment broths for the detection of non-O157 Shiga-toxin-producing Escherichia coli O91, O103, O111, O119, O121, O145 and O165 from pure culture and food samples[J]. Lett Appl Microbiol, 2011, 53(2):167-173.
[3]Rayan H, Amandadi M, Sanadgol N. A highly specific and sensitive loop-mediated isothermal amplification method for the detection of Escherichia coli O157:H7[J]. Microbial Pathogenesis, 2016,91:161-165.
[4]金婷婷, 马福金, 袁耀武. 免疫捕获LAMP法快速检测大肠埃希氏O157:H7的研究[J]. 食品科技, 2013, 33(8):1027-1031.
[5]Ohtsuka K, Tanaka M, Ohtsuka T. Comparison of detection methods for Escherichia coli O157 in Beef Livers and Carcasses[J].Foodborne Pathogens and Diseases, 2010, 7(12):1563-1567.
[6]Wang DG, Liu F, Huo GC, et al. Development and evaluation of a loop-mediated isothermal amplification method for detecting Escherichia coli O157 in raw milk[J]. Key Laboratory of Dairy Science, 2009, 17(1):55-66.
[7]朱海, 吕敬章, 范放, 等. E. coliO157:H7 LAMP检测方法的建立[J]. 分子诊断与治疗杂志, 2010, 2(2):98-101.
[8]Yin FG, Zhu Y, Koutchma T, et al. Inactivation and potential reactivation of pathogenic Escherichia coli O157:H7 in apple juice following ultraviolet light exposure at three monochromatic wavelengths[J]. Food Microbiology, 2015, 46 :329-335.
[9]Wang LJ, Li P, Zhang ZH, et al. Rapid and accurate detection of viable Escherichia coli O157:H7 in milk using a combined IMS, sodium deoxycholate, PMA and real-time quantitative PCR process[J]. Food Control, 2014, 36(1):119-125.
[10]Gabriel A. Combinations of selected physical and chemical hurdles to inactivate Escherichia coli O157:H7 in apple and orange juices[J]. Food Control, 2015, 50 :722-728.
[11]党苗苗, 李芳菲, 费楠, 等. 肠出血性大肠杆菌O157:H7的分子生物学检测及PCR检测[J]. 食品安全质量检测学报,2015, 6(2):523-527.
[12]张微, 姚笛, 侯婷婷, 等. 乳中大肠杆菌O157:H7的荧光定量PCR检测方法的建立[J]. 中国乳品工业, 2015, 43(7):52-54.
[13]Notomi T, Okayama H, Masubuchi AH, et al. Loop-mediated isothermal amplification of DNA[J]. Nucleic Acids Res, 2000,28(12):E63.
[14]Yu Y, Ma XY, Zhang W. Detection of Staphylococcus aureus in milk using real-time fluorescence loop-mediated isothermal amplification[J]. Advance Journal of Food Science and Technology, 2015, 8(9):678-684.
[15]Inomata A, KishdaI N, Momoda T, et al. Development and evaluation of a reverse transcription loop-mediated isothermal amplification assay for rapid and high-sensitive detection of Cryptosporidium in water samples[J]. Water Science and Technology, 2009, 60(8):2166-2172.
[16]Zhang GD, Brown EW, Narjol GE. Comparison of Real-time PCR,reverse transcriptase real-time PCR, loop-mediated isothermal amplification, and the FDA conventional microbiological method for the detection of Salmonella spp. in produce[J]. Appl Environ Microbiology, 2011, 77(18):6495-6501.
[17]Wang F, Jiang L, Ge BL, et al. Loop-mediated isothermal amplification assays for detecting shigella toxin-producing Escherichia coli in ground beef and human stools[J].Microbiology. 2012, 50(1):91-97.
[18]Yukiko HK, Jun N, Ikuo G, et al. Surveillance of Shiga toxinproducing Escherichia coli in beef with effective procedures,independent of serotype[J]. Foodborne pathogens and Disease,2008, 1(5):97-103.
[19]易海华, 赵金伟, 徐波, 等. 使用环介导等温扩增技术快速检测食品中肠出血性大肠杆菌O157:H7的初步研究[J]. 中国食品卫生杂志, 2010, 22(3):206-213.
[20]刘道亮, 胡连霞, 赵占民, 等. 改良环介导等温扩增技术快速检测肉类中的大肠杆菌O157:H7[J]. 微生物学通报, 2011,38(3):430-435.
[21]张雪寒, 何孔旺, 叶青, 等. 快速检测大肠杆菌O157的LAMP方法的建立与评价[J]. 中国兽医学报, 2013, 33(7):1027-1031.
[22]Yukiko HK, Noriko K, Kayoko O, et al. Detection of vero toxigenic Escherichia coli O157 and O26 in food by plating methods and LAMP method A collaborative study[J]. International Journal of Food Microbiology, 2008, 122(12):156-161.
[23]张雪寒, 何孔旺, 叶青, 等. 快速检测志贺毒素2基因的LAMP方法的建立[J]. 江苏农业学报, 2013, 29(2):455-457.
[24]Wang F, Jiang L, Yang QR, et al. Rapid and specific detection of Escherichia coli serogroups O26, O45, O103, O111, O121, O145,and O157 in ground beef, beef trim, and produce by loop-mediated isothermal amplification[J]. Appl Environ Microbiology, 2012,78(8):2727-2736.
[25]Techathuvanan C, D’Souza DH. Reverse-transcriptase loopmediated isothermal amplification as a rapid screening/monitoring tool for Salmonella Enterica detection in liquid whole eggs[J].Journal of Food Science, 2012, 77(4):200-205.
[26]Fan FX, Wang SJ, Lou J. Establishment of RT-LAMP assay to detect Salmonella Typhi in blood[J]. Disease Surveillance,2012, 27(4):325-329.

