红壳色中华绒螯蟹群体遗传特征的微卫星分析
王正光,李晓东,姜玉声*,左炳楠,司永国,郑岩,刘谞,孙娜
(1.大连海洋大学水产与生命学院,辽宁 大连 116023; 2.盘锦光合蟹业有限公司研发中心,辽宁 盘锦 124200)
中华绒螯蟹(Eriocheirsinensis)俗称河蟹、大闸蟹,隶属于十足目(Decapoda)、方蟹科(Grapsidae)、绒螯蟹属(Eriocheir),是中国重要的水产经济蟹类[1]。但随着大规模养殖业的发展,中华绒螯蟹的种质出现明显的退化[2-3],开展良种选育工作势在必行。辽宁省盘锦光合蟹业有限公司于2000年开始进行“光合1号”中华绒螯蟹的选育工作,2011年通过了国家级良种审定委员会审定[4]。“光合1号”是以北方水系中华绒螯蟹为基础,以生长快、成活率高为目标的定向选育群体。该组研究人员在“光合1号”人工选育过程中,使用定向交配的方法,经过4代近交筛选,建立了遗传稳定的红壳色中华绒螯蟹家系[5]。
明确育种群体的遗传特征是保证育种计划顺利实施的关键技术环节[6]。随着红壳中华绒螯蟹人工选育工作的广泛开展,因此,有必要对其遗传多样性进行评估。用于鱼类、贝类动物的体外标记识别育种群体谱系的方法并不适用于具有蜕壳习性的虾蟹类,包括中华绒螯蟹,而分子标记技术可以有效地解决这一问题[7-8]。微卫星(simple sequence repeat, SSR)作为第二代分子标记技术,具有符合孟德尔分离定律、多态性信息含量丰富、呈共显性遗传等特点,已广泛应用于蟹类的遗传分化分析[9-12]。本研究采用该技术对3个中华绒螯蟹群体进行遗传多态性和群体间的遗传分化分析,旨在明确各群体的遗传距离,评估红壳色中华绒螯蟹群体的遗传特征,为其选育工作提供科学参考。
1 材料与方法
1.1 材料
实验蟹取自盘锦光合蟹业有限公司研发中心基地,包括中华绒螯蟹红壳色群体(HK)、红壳色群体中正常壳色仔蟹(QK)以及“光合1号”育种群体(GH1),每个群体雌、雄各20只,共取120只进行群体遗传特征分析。
1.2 实验方法
1.2.1 DNA模板制备
实验蟹经过冷冻麻醉,取其足部肌肉。采用常规酚/氯仿法提取基因组 DNA,1%琼脂糖凝胶电泳检测 DNA 质量,紫外分光光度法测定DNA 浓度和纯度,无菌双蒸水稀释至 50 μg/mL后备用[13]。
1.2.2 PCR扩增与SSR引物筛选
参照原振政[14]应用的11对微卫星引物,引物由生工生物工程(上海)股份有限公合成,引物序列及退火温度如表1所示。
PCR扩增按照大连TaKaRa公司试剂盒方法操作,简要介绍如下:PCR 反应体系为10 μL,其中包括10×buffer 1 μL,Mg2+(2.5 mmol/L)0.6 μL,dNTP(2.5 mmol/L)1.0 μL,正反向引物(10 μmol/L)各 0.4 μL,Taq 酶 0.05 U,模板 1.0 μL,最后加 ddH2O补足体积至10 μL。扩增产物用 2%琼脂糖凝胶电泳检测。
1.2.3 聚丙烯酰胺凝胶电泳检测
PCR扩增产物先后通过8%非变性聚丙烯酰胺凝胶电泳和硝酸银染色检验。制备 8%非变性聚丙烯酰胺凝胶,以15 W 恒定功率电泳 60~120 min。电泳结束后,取下凝胶放入AgNO3溶液中染色15 min,双蒸水漂洗后放入显色液(2% NaOH,8 mL 甲醛)中,待条带充分显现后分析电泳图谱判定PCR扩增产物质量。
1.2.4 数据统计与分析
根据每个样品产生的条带位置确定基因型,使用Popgen 32软件(Bayer CropScience AG, Germany)分析各位点的有效等位基因数(Ne)、香农-威纳指数(I)、观测杂合度(Ho)、期望杂合度(He)与遗传分化指数(Fst),计算群体间的 Nei’s 遗传距离[15],并构建UPGMA 系统树[16]。采用固定指数(Fis)评估种群内个体间的近交程度,同时计算Hardy-Weinberg 平衡偏离指数D,D=(Ho-He)/He公式。使用Cervus软件(Field Genetics, Britain)计算引物的多态信息含量(PIC)[17]。

表1 中华绒螯蟹微卫星分子标记及其引物序列Tab.1 Microsatellite markers and their primers sequences from E. sinensis
2 结果与分析
2.1 各基因位点的遗传多态性
如表2所示,11对引物中的10对其多态信息含量PIC>0.5。3个中华绒螯蟹群体的遗传多样性中,各群体的平均Ne在2.403 9~3.694 8之间,GH1、QK与HK群体的平均Ho分别为0.606 5、0.569 2、0.659 9,平均He分别为0.684 8、0.647 8、0.547 8,平均I分别为1.318 5,1.189 5,0.924 4,平均Fis分别为0.102 7、0.109 7、-0.220 8。其中GH1群体的平均He最高,HK群体的平均He最低,且与GH1群体差异显著(P<0.05)。
3个群体的平均D在-0.110 9与0.712 7之间,在NE5、NE16、ES34这3个基因位点上均为负值,这表明3个群体在此3个基因位点上可能存在杂合子缺失。GH1群体的D在NE46与ESA67这两个位点为正数,而在其余9个基因位点均为负数,由此推测,该群体存在严重的杂合子缺失现象。HK群体的平均D高于其他2个群体,但各群体之间差异不显著(P>0.05)。
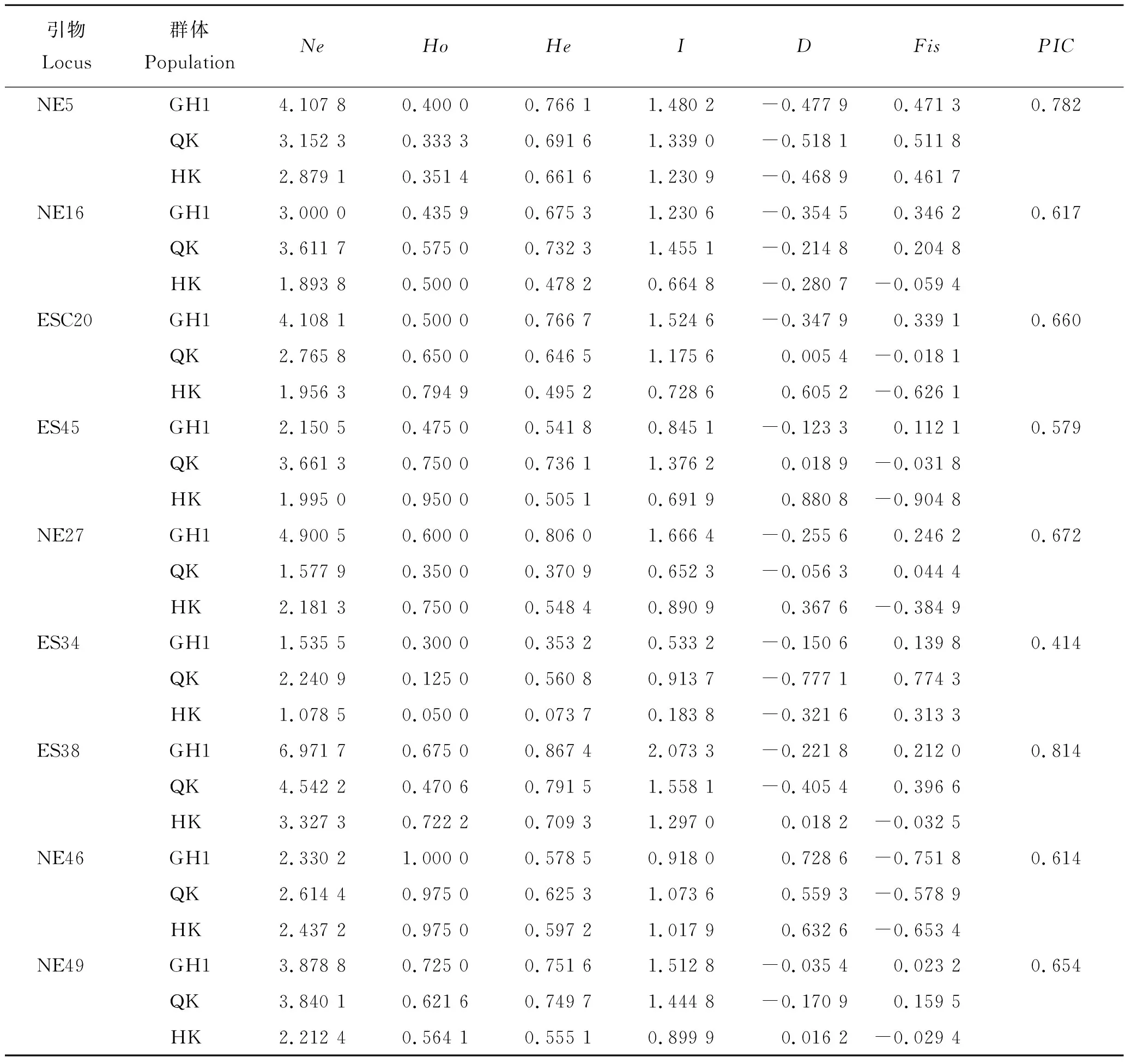
表2 3个群体中华绒螯蟹在11个微卫星位点上的遗传多态性Tab.2 Genetic polymorphism of 11 pairs of microsatellite loci in 3 populations of E. sinensis
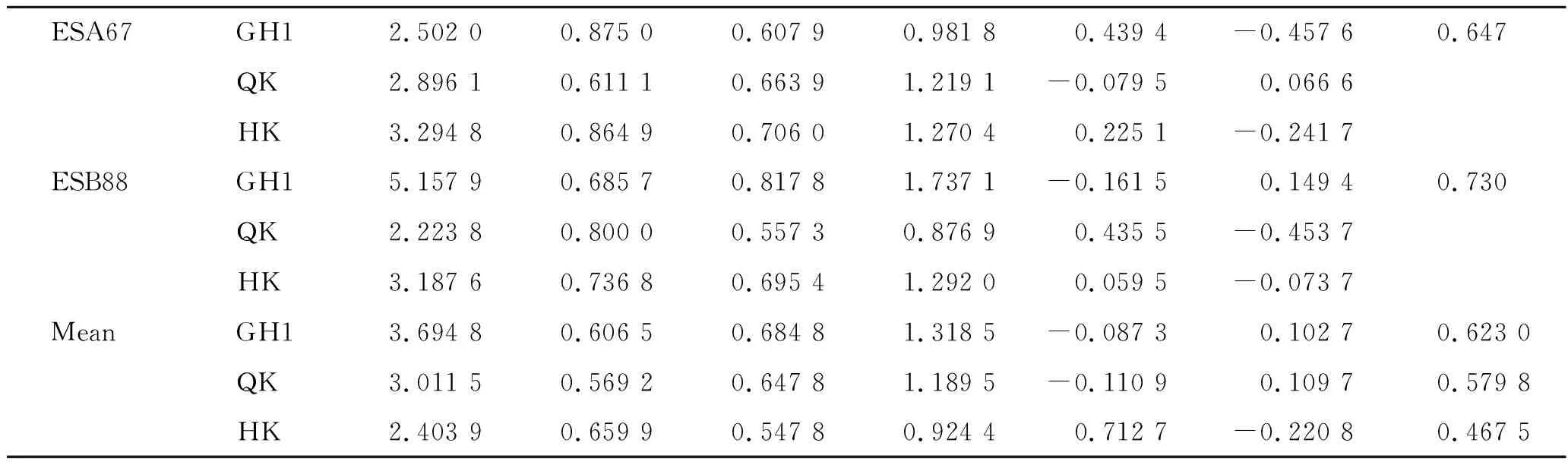
续表2,Tab.2 Continued
2.2 群体间的遗传分化
Fst是用来评估种群内部与种群之间遗传分化的一个重要指标。当Fst<0.05时,群体间遗传分化很小,可以不予考虑;当0.05

表3 3个中华绒螯蟹群体的遗传距离、遗传分化指数与遗传相似系数Tab.3 Genetic distance, genetic differentiationand similarity degree of 3 populations of E. sinensis
注:“—”示无数据。对角线以下为遗传距离/遗传分化指数,对角线以上为相似性系数。

图2 3个中华绒螯蟹群体的UPGMA树Fig.2 UPGMA tree of 3 populations of E. sinensis
3 讨论
3.1 群体的遗传多样性
遗传多样性是生命进化和物种分化的基础,在一定程度上决定了物种在自然或人为条件下对环境改变的适应能力[19]。本研究用11对微卫星引物对中华绒螯蟹3个群体120只个体进行遗传特征分析。根据Botstein等[20]提出的衡量基因变异程度高低的多态信息含量指标,当PIC>0.5时该基因位点为高度多态性位点,0.25≤PIC<0.5时为中度多态性位点,PIC<0.25时为低度多态性位点。本研究选用的11对引物除ES34基因位点外,其余10对引物都有较高的多态信息含量(PIC>0.5),表明所用引物的多态性符合群体遗传分析要求。根据苏龙等[21]的研究报道,当引物多态性含量较高时,10对引物可以准确分析实验群体的遗传结构与聚类关系。本研究选择了前期工作所使用的11对多态性良好的引物[14],分析中华绒螯蟹3个人工选育群体的遗传特性,如此不仅可以进行群体间的横向分析,还可以进行选育群体多代的纵向比较。GH1与QK群体的平均PIC分别为0.623 0与0.579 8(PIC>0.5),而HK群体的平均PIC为0.467 5(0.25≤PIC<0.5),表明所用引物可以较高程度地评估前两个群体的基因变异水平,中等程度评估后一群体的遗传变异水平。相较于黄河、辽河和长江水系中华绒螯蟹野生群体[22-23],本研究中3个群体的平均Ne、平均Ho与平均He均较低,这可能与多代的人工选育有关。长期人工定向交配选育有可能导致非目的性状基因位点缺失,有效群体数量逐渐减少,群体遗传变异水平下降,这也是很多经济种类在长时间、大规模养殖后其遗传多样性降低的主要原因[24]。罗氏沼虾(Macrobrachiumrosenbergii)、中国对虾(Fenneropenaeuschinensis)、菲律宾蛤仔(Ruditapesphilippmarum)、日本对虾(Penaeusjaponicus)、斑节对虾(Penaeusmonodon)与大菱鲆(Psettamaxima)等水产动物人工选育也出现过遗传多样性指数降低的现象[25-30]。人工选育虽然会导致遗传多样性降低,却也利于选育群体基因的纯化,保存有利性状,使得育种性状逐渐趋向稳定。然而,选育群体遗传多样性若低于某一阈值,将会影响其对自然环境的适应能力[31]。因此,日后中华绒螯蟹选育应在保持选择压力获得优良性状的同时,尽可能保持与目的性状不相关基因位点的多样性。另外,还可以通过增加亲本数量降低近交的发生。
HK群体的平均Ne与He低于GH1群体,HK群体的平均I也低于GH1群体且差异显著(P<0.05),表明其遗传多样性低于GH1。这一结果不仅与多代的人工选育有关,也可能由于HK群体选育时间较短,其群体数量远小于经过长期选育的GH1群体。在封闭的小群体中,有效群体减少会增加遗传漂变速度,并且遗传漂变导致的遗传多样性丢失远远高于大群体[32]。HK群体具有小群体属性,经过人工定向选择繁育,导致有效群体减少,最终使其遗传漂变速度加快,遗传多样性丢失的概率提高。这种因亲本数量较少导致的遗传多样性降低被称为“始祖效应”,在大西洋鲑的养殖中也出现过类似现象[33]。而红壳色群体中的正常壳色群体QK的遗传多样性要高于红壳色群体HK,其原因可能与群体的遗传漂变相关,或与本研究使用的微卫星引物其自身特性有关,其中原因还需在未来的研究中进一步明确。
3.2 群体的杂合子的缺失现象
杂合子缺失可能与无效等位基因、基因分型误差、性连锁座位、取样量较少和近交等原因相关[15, 34-35]。本研究中3个中华绒螯蟹群体在NE5、NE16、ES34基因位点上的D为负值,表明在这3个基因位点上出现了杂合子缺失。GH1群体的D仅在NE46与ESA67这两个基因位点上为正数,在其余位点均为负数。虾蟹类育种群体中杂合子缺失现象较为常见[36-38],普遍认为其原因为:长期的人工定向选择导致的基因纯化,从而使某些基因位点选择性消失,最终造成群体的遗传多样性下降。在今后的育种工作中,增加有效群体数量,提高杂合子比例,减少对自交个体的淘汰所导致的稀有等位基因缺失,以及由漂变所导致的杂合子缺失,将有助于防止中华绒螯蟹育种群体的种质衰退。
3.3 红壳色中华绒螯蟹群体的遗传分化
梭子蟹科6种海产蟹的遗传距离为0.200 0~0.939 4,红星梭子蟹与日本蟳遗传距离最大(0.939 4)[39]。本研究中HK群体最大遗传距离为0.479 9,远低于梭子蟹科野生种群,符合人工养殖群体遗传结构特征。Fst指数分析显示,HK群体较GH1、QK群体确实产生了一定程度的遗传分化。基于Nei’s遗传距离构建UPGMA树显示GH1与QK中华绒螯蟹群体之间的遗传距离较小,与HK群体之间的遗传距离较大。QK群体来源于红壳色群体HK中正常壳色的个体,其亲缘关系理应较近。然而,仅就体色性状而言,GH1与QK群体并无差异。养殖过程中也发现两者行为习性并无明显差异,相对HK群体的遗传背景,GH1与QK有可能更相似。另外,微卫星引物和样本的数量等特性也是影响遗传分析的重要因素,增加其数量有助于精确实验结果的分析。引物数量不同会影响遗传距离的精确度,随着引物数量增加,变异系数以及标准差呈下降趋势,遗传距离精确度会相应提升[40]。
固定指数Fis常用来表示群体内个体间的近交系数。当群体内Ho小于He,同时Fis为正值时,表明群体内近交程度较严重;而Fis为负值,群体内Ho大于He,则表示群体内存在远缘繁殖。据此可知,GH1与QK群体内近交情况严重,而HK群体内则存在着远缘繁殖现象。虽然HK群体表现出一定的远缘繁殖,但其遗传多样性却较低,这可能仍然与其明显分化的性状所对应的特殊遗传背景相关。李建林等[41]对鲤(Cyprinidae)6个群体遗传结构的分析也发现,建鲤(CyprinuscarpiovarJian)Ho为0.705大于He的0.702,且Fis为-0.011,表明其群体内存在明显的远缘繁殖,但其等位基因数与等位基因丰富度却低于存在近交的黄河鲤(Cyprinuscarpio)群体,与本研究结果相似。日后在制定选育方案时,可加强GH1与QK群体的人为远缘选育干扰,减少群体内部近交。而HK群体可以作为新的育种家系,通过扩大选育群体数量,适当引入无消极影响的外源基因,增加其基因丰富度,进而提高群体的遗传多样性。
4 结论
3个中华绒螯蟹群体中,GH1群体遗传多样性最高,HK群体遗传多样性最低。各群体中3个微卫星位点可能有杂合子缺失现象。HK群体与GH1、QK群体间产生了一定的遗传分化。建议日后在GH1群体的人工选育中增加有效群体数量,降低近交发生概率。HK群体应在保证体色性状稳定的同时,扩大选育群体数量,适量引入非目的性状相关基因位点,以增加其基因丰富度。
参考文献:
[1] 李晓东. 北方河蟹养殖新技术[M]. 北京: 中国农业出版社, 2006.
[2] 王成辉, 李思发. 中华绒螯蟹种质研究进展[J]. 中国水产科学, 2002, 9(1): 92-96.
[3] 王武. 我国河蟹养殖的现状和发展前景[J]. 当代水产, 1998(4):2-4.
[4] 姜玉声. 辽宁沿海虾蟹类与增养殖[M]. 沈阳:辽宁科学技术出版社, 2015.
[5] 司永国, 李晓东, 姜玉声, 等. 红壳色中华绒螯蟹仔蟹养殖方式研究[J]. 大连海洋大学学报, 2017 ( 2): 139-144.
[6] 孙效文. 鱼类分子育种学[M]. 北京:海洋出版社,2010.
[7] Chang Y M, Liang L Q, Ma H T, et al. Microsatellite analysis of genetic diversity and population structure of Chinese mitten crab (Eriocheirsinensis) [J]. J Genet Genomics, 2008, 35 (3): 171-176.
[8] Sui L Y, Zhang F M, Wang X M, et al. Genetic diversity and population structure of the Chinese mitten crabEriocheirsinensisin its native range [J]. Mar Biol, 2009, 156(8):1573-1583.
[9] 李晓萍,刘萍,李健,等.应用 SSR 分析5个三疣梭子蟹野生群体的遗传多样性[J].中国水产科学,2011, 18(6):132-133.
[10] 舒妙安,周宇芳,朱晓宇,等.中国沿海拟穴青蟹群体遗传多样性的微卫星分析[J]. 水产学报, 2011, 35(7): 977-984.
[11] 宋春妮, 李健, 刘萍. 日本蟳 4 个野生群体遗传多样性的微卫星分析[J]. 水产学报, 2011, 35(7): 985-991.
[12] Ma H T,Chang Y M,Yu D M,et al.Microsatellite variation among four populations ofEriocheirsinensis[J]. Zoolog Res, 2007, 28(2):126-133.
[13] 吕俊宣, 奚雷. 分光光度法测定DNA或RNA含量[J]. 生物化学与生物物理进展, 1984(5): 72.
[14] 原振政. 微卫星标记技术在中华绒螯蟹亲缘关系鉴定中的研究与应用[D]. 大连: 大连海洋大学, 2009.
[15] Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals [J]. Genetics, 1978, 89 (3): 583-590.
[16] Tamura K, Peterson D, Peterson N, et al. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Mol Biol Evol, 2011, 28(10):2731-2739.
[17] 周宇芳, 舒妙安, 赵晓枫, 等. 青蟹野生与养殖群体遗传多样性的微卫星分析[J]. 浙江农业学报, 2011, 23(4):712-716.
[18] 张天时, 王清印, 刘萍,等. 中国对虾(Fenneropenaeuschinensis)人工选育群体不同世代的微卫星分析[J]. 海洋与湖沼, 2005, 36(1):72-80.
[19] Frankham R, Ballou J, Briscoe D. Introduction to conservation genetics[M]. Cambride: Cambrdege University Press, 2010: 260-308.
[20] Botstein D, White R L, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms [J]. Am J Hum Genet, 1980, 32(3):314-331.
[21] 苏龙, 徐志健, 乔卫华, 等. 广西药用野生稻遗传多样性分析及SSR引物数量对遗传多样性结果的影响研究[J]. 植物遗传资源学报, 2017, 18(4):603-610.
[22] 刘青, 刘皓, 吴旭干, 等. 长江、黄河和河水系中华绒螯蟹野生和养殖群体遗传变异的微卫星分析[J]. 海洋与湖沼, 2015, 46(4): 958-968.
[23] 肖起珍, 刘青, 吴旭干, 等. 长江水系中华绒螯蟹野生和人工繁殖大眼幼体的遗传多样性分析[J]. 基因组学与应用生物学, 2017(5):1935-1945.
[24] 刘皓. 长江、黄河、辽河水系中华绒螯蟹野生群体遗传多样性的微卫星分析[D]. 上海:上海海洋大学, 2015.
[25] Chareontawee K, Poompuang S, Na-Nakorn U, et al. Genetic diversity of hatchery stocks of giant freshwater prawn (Macrobrachiumrosenbergii) in Thailand [J]. Aquaculture, 2007, 271(1): 121-129.
[26] 张天时, 刘萍, 李健, 等. 用微卫星DNA技术对中国对虾人工选育群体遗传多样性的研究[J]. 水产学报, 2005, 29(1): 6-12.
[27] 聂鸿涛, 李佳, 霍忠明, 等. 菲律宾蛤仔人工选育群体与野生群体的遗传多样性分析[J]. 中国水产科学, 2016, 23(3): 528-546.
[28] Sordoni V,de Matthaeis E,Sbordoni M C,et al. Bottleneck effects and the depression of genetic variability in hatchery stocks ofPenaeujaponicus(Crustacea,Decpoda)[J] . Aquaculture, l986, 57: 239-251.
[29] Xu Z,Primavera J H,de la Pena,et al. Genetic diversity of wild and cultured black tiger shrimp (Penaeusmonodon) in the Philippines using microsatellites [J]. Aquaculture, 2001, 199: 13-40.
[30] 田岳强. 大菱鲆选育世代遗传多样性分析及生长性状微卫星标记筛选[D]. 上海:上海海洋大学, 2015.
[31] David P. Heterozygosity-fitness correlations: new perspective on old problems[J]. Heredity, 1998, 80(5): 531-537.
[32] 鲁云风, 王茜, 黄文波, 等. 畜禽保种群体遗传多样性的模拟[J]. 中国农业科学, 2012, 45(18): 3849-3858.
[33] Skaala Ø, Høyheim B, Glover K, et al, 2004. Microsatellite analysis in domesticated and wild Atlantic salmon (SalmosalarL.): allelic diversity and identification of individuals[J]. Aquaculture, 240(1/2/3/4): 131-143.
[34] Hedgecock D, Li G, Hubert S, et al. Widespread null alleles and poor cross-species amplification of microsatellite DNA loci cloned from the Pacific oyster,Crassostreagigas[J]. J Shellfish Res, 2004, 23: 379-386.
[35] Zouros E. On the relation between heterozygosity and heterosis: an evaluation of the evidence from marine mollusks [J]. Isozymes Curr Top Biol Med Res, 1987, 15(8):255-270.
[36] 唐刘秀, 许志强, 葛家春, 等. 中华绒螯蟹3个育种基础群体遗传特征的微卫星分析 [J]. 南京师范大学学报(自然科学版), 2013, 36 (2): 84-90.
[37] 刘萍, 孟宪红, 孔杰,等. 中国对虾微卫星DNA多态性分析[J]. 自然科学进展, 2004, 14(3):333-338.
[38] 童馨, 龚世圆, 喻达辉,等. 凡纳滨对虾(Litopenaeusvannanamei)不同世代养殖群体的遗传多样性分析[J]. 海洋与湖沼, 2009, 40(2):214-220.
[39] 金珊, 赵青松, 王春琳,等. 梭子蟹科六种海产蟹的RAPD标记[J]. 动物学研究, 2004, 25(2):172-176.
[40] 张爱玲, 马月辉, 陈秀梅,等. 不等微卫星座位数目对Nei氏标准遗传距离估测精度的影响[J]. 畜牧兽医学报, 2005, 36(5): 431-433.
[41] 李建林, 李红霞, 唐永凯,等. 利用微卫星标记分析6个鲤鱼群体的遗传差异[J]. 福建农业学报, 2012, 27(9):936-940.

