基于16S rRNA序列对4种扇贝4个群体亲缘关系分析
相明玥,曾令坤,李祯,田莹,常亚青,丁君
(农业部北方海水增养殖重点实验室,大连海洋大学水产与生命学院,辽宁 大连 116023)
中国主要的养殖扇贝品种有海湾扇贝(Argopectenirradians)、栉孔扇贝(Chlamysfarreri)以及虾夷扇贝(Patinopectenyessoensis)。目前辽宁省大连市的市场上出现一种被称作“俄罗斯栉孔扇贝”的扇贝,该种扇贝与栉孔扇贝在形态上极其相似,分类地位尚不明确。从养殖角度来说,此种扇贝极有经济价值[1]。但目前对该扇贝的研究主要集中在毒物学[2-3]、生态学[4-7]等方面,而在遗传学及种质资源方面少有报道。单纯通过形态学观察以及内部解剖结构鉴定不足以判定该品种与中国栉孔扇贝的关系。因此,使用深层次的分子生物学技术是有效的解决办法之一。
近年来,随着分子生物学技术迅速发展,一系列新的分子学标记技术开始出现,如限制性片段长度多态性(RFLP)技术、DNA指纹技术等, 克服了以往用同功酶分析多态性位点比率偏低的缺陷。PCR技术和DNA测序技术的出现和广泛使用,使得通过扩增确定基因序列成为可能。已选定的基因或基因片段在高度保守区域互补的通用引物下进行扩增,而后对PCR产物直接测序,分析比对序列数据,可明确基因序列。此技术在哺乳类[8-10]、鱼类[11-12]等研究中广泛应用。本研究正是利用PCR扩增技术获得“俄罗斯栉孔扇贝”的16S rRNA核心片段序列,而后与NCBI数据库序列比对。同时测定中国地区的栉孔扇贝、海湾扇贝、虾夷扇贝,以及美国大西洋地区的海湾扇贝和日本地区的虾夷扇贝等不同地区扇贝线粒体16S rRNA 基因片段序列,与GenBank上已有的其他双壳贝类的同源序列进行比对,分析这4种扇贝在不同分布区自然群体16S rRNA基因序列的多态性以及“俄罗斯栉孔扇贝”在双壳贝类中的遗传地位,以期对其遗传多样性进行更深入的了解,并在分子水平上为研究分析不同地区的4种扇贝群体的遗传背景提供参考。
1 材料与方法
1.1 实验材料
本研究所用的贝类样品包括海湾扇贝(Argopectenirradians)、市场上俗称的“俄罗斯栉孔扇贝”(图1)、虾夷扇贝(Patinopectenyessoensis)、栉孔扇贝(Chlamysfarreri)(图2)全部取自大连智慧渔业集团有限公司(表1)。每个群体各取2枚鲜活个体,活体解剖取闭壳肌,立即放入无水乙醇中固定备用。其中,对“俄罗斯栉孔扇贝”进行简单的形态学测量(表2)。用游标卡尺(精确至0.01 mm)分别测量壳长、壳高、壳宽、绞合线长,用手术刀、剪、镊等无菌解剖并分离软体部、闭壳肌后分别使用电子天平(精确至0.01 g)称量体质量、软体部质量和闭壳肌质量。
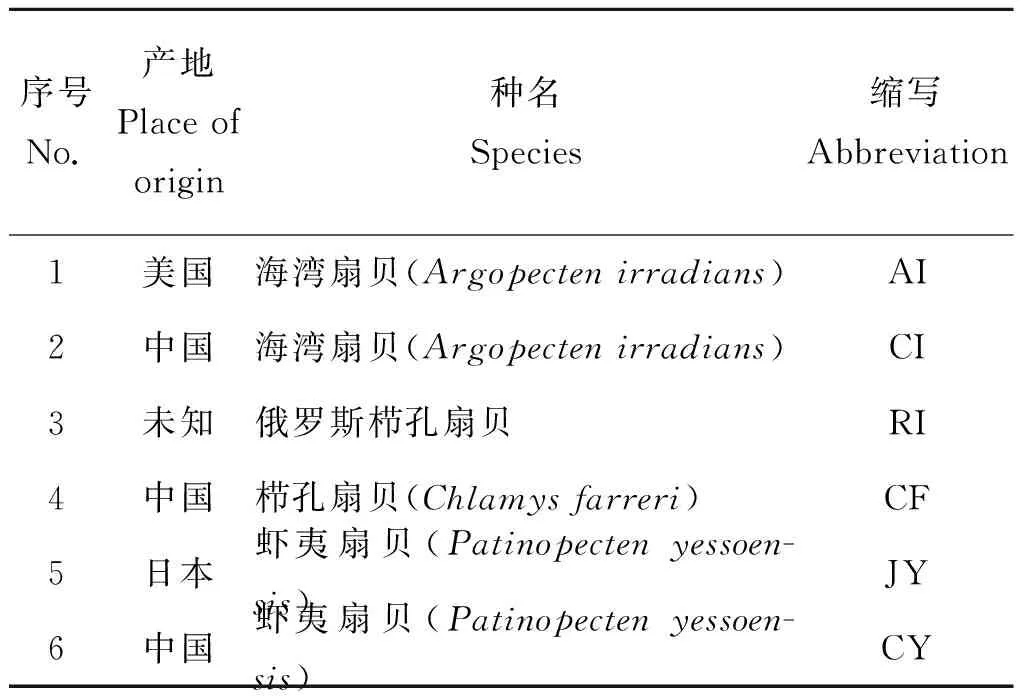
表1 实验材料信息Tab.1 The information of materials in this study

表2 “俄罗斯栉孔扇贝”形态学特征Tab.2 The measureable characters of Russia Chlamys farreri

图1 “俄罗斯栉孔扇贝”的右壳(A)和左壳(B)Fig.1 The right shell (A) and the left shell (B) of Russian Chlamys farreri

图2 栉孔扇贝的右壳(A)和左壳(B)Fig.2 The right shell (A) and the left shell(B) of Chlamys farreri
1.2 实验方法
1.2.1 基因组DNA提取和含量检测
参考DNA提取试剂盒(天根生化科技有限公司http://www.tiangen.com/)提取扇贝的基因组DNA, 用紫外分光光度计(NV3000,美国斯威特)测定其浓度和纯度, 并用8.0 g/L琼脂糖凝胶电泳检测, 之后用ddH2O稀释DNA至50 ng/μL, -20 ℃下保存备用。
1.2.2 16S rRNA基因的扩增及序列测定
利用通用引物16SF(5′-CGCCTGTTTAA CAAAAACAT-3′)16SR(5′-CCGGTTTGAACTCAGA TCACGT-3′)[13]进行常规PCR扩增。PCR反应体系如下:DNA模板(50 ng/μL) 1.0 μL,引物16SF(10 μmol/L) 1.0 μL,引物16SR(10 μmol/L) 1.0 μL,dNTP(2.5 mmol/L)2.0 μL,10×bufer 2.0 μL,Mg2+(25 mmol/L) 1.0 μL,Taq(5 U/μL) 0.1 μL,无菌超纯水补足至20 μL。反应程序为:94 ℃预变性3 min;94 ℃变性30 s,52 ℃退火30 s,72 ℃延伸1 min,如此进行35个循环;最后在72 ℃延伸10 min,4 ℃保存。取PCR产物7 μL与2 μL的6×loading buffer混匀,在20 g/L琼脂糖凝胶(含1.7 μL溴酚蓝)上电泳(电压为95 V),直至溴酚蓝移动到距离胶板下沿约1 cm处时,停止电泳。用凝胶成像系统观察并拍照,初步检测PCR产物。每个扇贝群体选取2枚个体的PCR产物,按照BigDye荧光标记终止底物循环测序试剂盒方法,经DNA 测序仪(ABI3730xl DNA Analyzer,美国Perkin Elmer),获得DNA双向序列,而后对序列和峰图进行人工校对。
1.2.3 序列分析
测得序列用BioEdit(Version 7.2.6, Carlsbad, USA)软件去掉序列两端不可信部分,通过Clustal X(Version 1.83, Conway Institute UCD Dublin, Ireland)软件进行多序列对比和分析,Clustal格式对比文件采用MEGA 5.0(Center for Evolutionary Medicine and Informatics,The Biodesign Institute,USA)软件分析序列的碱基组成以及它们的遗传距离,按照Kimura双参数法计算物种间的DNA序列差异及种间遗传距离。结合GenBank中其他双壳贝类(长牡蛎为外群)16S rRNA同源序列(表3),采用邻接法Neighbor-Joining(NJ)法构建系统发育树,各分支的置信度采用重抽样法(boot-strap),通过1 000次循环评估系统发育树的可靠性。
2 结果
2.1 DNA模板及扩增产物的检测
DNA模板琼脂糖电泳结果显示(图3),DNA条带完整、清晰、无杂带,可用于后续实验。PCR扩增的片段位于500~750 bp。

表3 用于系统分析的扇贝物种及其16S rRNA GenBank注册号Tab.3 Species used for the phylogenetic analysis andGenBank accession number
注:*表示本实验所重点比较的序列。
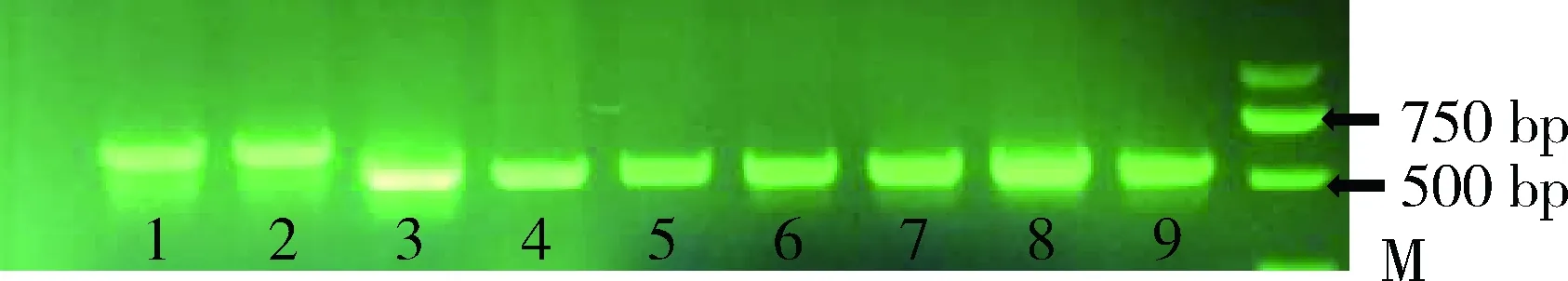
图3 4种扇贝16S rRNA的PCR扩增产物 1、2号DNA条带为中国地区虾夷扇贝16S rRNA基因片段, 3、4号条带为中国地区栉孔扇贝,5号条带为中国地区海湾扇贝, 6号条带为俄罗斯栉孔扇贝,7号条带为日本地区虾夷扇贝, 8、9号条带为美国大西洋地区海湾扇贝。Fig.3 The sequence was amplified via polymerase chain reaction of the 4 species scallops of 16S rRNA gene No.1 and No.2 are the 16S rRNA partial gene sequence of Chinese Patinopecten yessoensis. No.3 and No.4 are the 16S rRNA partial gene sequence of Chinese Chlamys farreri. No.5 is Chinese Argopecten irradians. No.6 is Russian chlamys farreri. No.7 is Japan Patinopecten yessoensis. No.8 and No.9 are the 16S rRNA partial gene sequence of American Argopecten irradians.
2.2 序列特性和碱基组成
将所测得的4种扇贝的16S rRNA序列经BioEdit软件处理,去掉不可信位点后,得到607 bp的基因片段。应用MEGA 5.0计算出6条序列的A、T、G、C的含量(表4),4种扇贝的A+T的含量为52.0%~55.0%,平均为53.5%;而G+C的含量为45.0%~48.0%,平均为46.4%。A+T的含量高于G+C含量。
应用MEGA 5.0软件对序列进行分析,得到555 bp的基因片段。“俄罗斯栉孔扇贝”与中国的栉孔扇贝序列对比中,保守位点521个,变异位点74个,转换位点43个,颠换位点31个;与美国大西洋的海湾扇贝序列比对中,保守位点450个,变异位点106个,转换位点69个,颠换位点37个;与中国的海湾扇贝序列对比中,保守位点405个,变异位点100个,转换位点59个,颠换位点41个;与日本的虾夷扇贝序列比对,保守位点527个,变异位点61个,转换位点35个,颠换位点26个;与中国的虾夷扇贝序列比对中,保守位点531个,变异位点58个,转换位点34个,颠换位点24个。
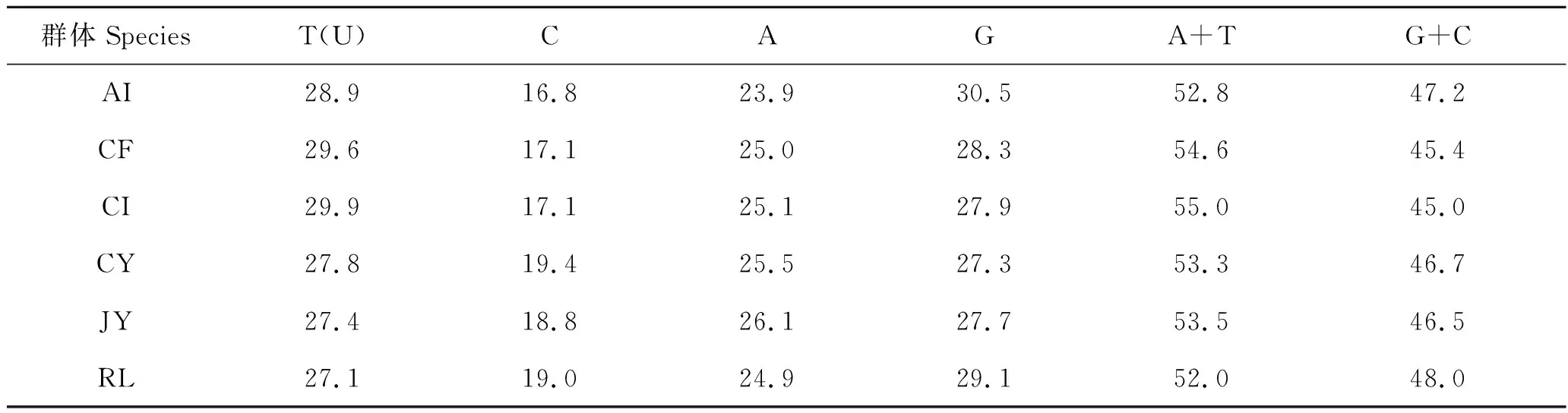
表4 4种扇贝碱基含量Tab.4 Base content of 4 kinds of scallop %
2.3 4种扇贝属物种分化比较
通过计算4种扇贝属物种遗传距离可知(表5):“俄罗斯栉孔扇贝”与中国虾夷扇贝遗传距离最近(0.073);与中国海湾扇贝遗传距离最远(0.221);与栉孔扇贝遗传距离为0.082。物种之间遗传距离平均值为0.154。

表5 4种扇贝属物种遗传距离Tab.5 Genetic distance of 4 species of scallop
注:“—”示无数据。
2.4 系统发生关系
基于扇贝16S rRNA基因序列,用MEGA 5.0软件按照Kimura双参数进行1 000次重复抽样分析,构建NJ系统发育树(图4)。从系统发育树可知,7种扇贝科贝类聚在一起,长牡蛎分为独立的一支,岛扇贝(俄罗斯栉孔扇贝)和虾夷扇贝聚为1支,然后又与栉孔扇贝聚为1支。表现为同种扇贝之间遗传距离最近,且置信度100%。
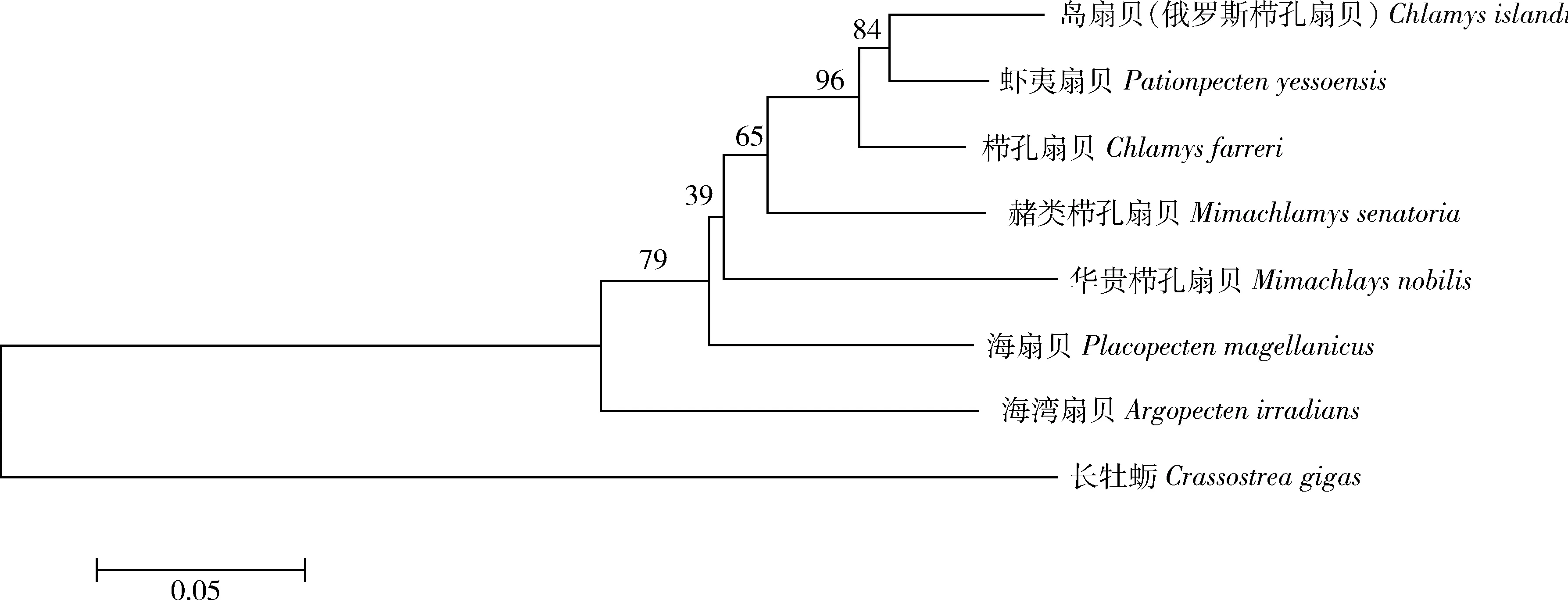
图4 应用MEGA 5.0软件中邻接法(NJ)构建基于16S rRNA序列的8种双壳贝类的系统发育树Fig.4 Neighbor-joining phylogenetic tree of 8 species of bivalve based on the 16S rRNA gene by using MEGA 5.0
3 讨论
3.1 扇贝16S rRNA基因片段碱基序列分析
近年来,16S rRNA已经逐渐应用于海洋生物属间[14-15]、种间的分子系统学和种质鉴定[16]方面的研究,在海洋双壳贝类的分子系统学研究及种群关系的分析方面也有报道,但它在贝类中的分化速度及变异程度有所不同。鉴于研究结果的不同,一般认为,线粒体16S rRNA基因序列分析适用于种尤其是属及属以上阶元的分类[17]。本研究中涉及的“俄罗斯栉孔扇贝”与中国广泛养殖的3种养殖扇贝之间的分类阶元均在属以上,进行16S rRNA序列的比对分析能够有效地揭示这4种扇贝之间的亲缘关系,更加明确“俄罗斯栉孔扇贝”的分类地位。
本研究被检测的样品中,4种碱基(A、T、G、C)各自的含量较为接近,而且这几种扇贝的基因片段中,A+T的含量高于G+C的含量,这个结果与其他研究者在头足类[18-19]、双壳类[20-21]、甲壳类[22]等的16S rRNA对比结果一致。即较高的A+T量是目前观测到的无脊椎动物线粒体DNA序列中普遍存在的现象[17]。
将PCR扩增技术扩增出的“俄罗斯栉孔扇贝”16S rRNA核心片段序列,与NCBI数据库序列比对。结果表明:数据库中序列同源性为100%,确定市场上出现的“俄罗斯栉孔扇贝”其学名为岛扇贝(Chlamysislandica),多分布于极地附近。结合GenBank上其他双壳贝类的同源序列,对其序列变异和系统发育树进行分析,结果显示,与岛扇贝亲缘关系由近及远分别虾夷扇贝、栉孔扇贝、赭类栉孔扇贝、华贵栉孔扇贝、海扇贝、海湾扇贝及长牡蛎。岛扇贝与虾夷扇贝同属于一个分支。种间与种内序列的差异分析显示,16S种内遗传距离在0.000到0.008之间,平均为0.001;种间的遗传距离在0.053到0.309之间,平均为0.231[23]。即与栉孔扇贝(0.082)相比,二者已达到种间差异水平;岛扇贝与虾夷扇贝遗传距离更小,亲缘关系较近(0.073);岛扇贝与海湾扇贝亲缘关系较远(0.221)。系统发育树(NJ)测绘结果与核苷酸变异值分析结果一致,这一结果将为在分子水平上研究不同地区的这4种扇贝遗传背景提供基础理论。
3.2 16S rRNA基因序列于双壳贝类种质鉴定中的应用
利用16S rRNA基因序列对双壳贝类系统发育的研究已有报道。Boulding等[20]用此法分析了虾夷扇贝一个养殖群体和两个野生群体样品,发现了丰富的遗传变异;Saavedra等[24]通过线粒体16S与12S基因序列分析研究了美国多种双壳贝类的系统进化关系;李霞等[17]通过对海扇贝、虾夷扇贝、栉孔扇贝和海湾扇贝16S rRNA同源序列的对比发现,它们分别存在3、7、8、13个差异位点,核苷酸变异位点较为丰富,且碱基颠换和插入变异比例大于碱基转换;胡丽萍等[25]利用16S rRNA序列探讨紫扇贝(Argopectenpurpuratus)在海湾扇贝属中的分类地位,发现紫扇贝和海湾扇贝同源序列间的变异均来自转换或者颠换,无插入或缺失变异,且转换位点多于颠换位点,这些变异位点可用于分子系统学和遗传学的分析。目前,对于岛扇贝的研究在遗传学及种质资源方面鲜有报道,单纯的通过形态学以及简单的内部解剖构造观察不足以将该品种与中国栉孔扇贝进行区分。本研究中利用16S rRNA基因序列,得出了岛扇贝与中国广泛养殖的3种扇贝之间亲缘关系并确定了其分类地位,这一结果为扇贝的种质鉴定工作进行了有效的补充。
4 结论
本研究基于16S rRNA基因片段,利用PCR扩增及测序技术,将人们俗称的“俄罗斯栉孔扇贝”与栉孔扇贝、虾夷扇贝、海湾扇贝进行比对,并结合GenBank中其他双壳贝类16S rRNA序列计算种间遗传距离,构建系统发育树,明确探讨该扇贝的分类地位。结果表明:该“俄罗斯栉孔扇贝”是岛扇贝(Chlamysislandica),与该扇贝亲缘关系由近及远分别是虾夷扇贝、栉孔扇贝、赭类栉孔扇贝、华贵栉孔扇贝、海扇贝、海湾扇贝和长牡蛎。岛扇贝与虾夷扇贝遗传距离最近。种内与种间的差异分析显示,岛扇贝与栉孔扇贝遗传距离已达到种间差异水平。本研究为在分子水平上研究了不同地区的4种扇贝群体遗传背景提供信息。
参考文献:
[1] Wallace J C, Reinsnes T G, 廖海清.各种环境参数对垂挂养殖的冰岛栉孔扇贝生长的重要性[J].水产科学,1985(4):47-51.
[2] Nahrgang J,Brooks S J, Evenset A. et al. Seasonal variation in biomarkers in blue mussel (Mytilusedulis), Icelandic scallop (Chlamysislandica) and Atlantic cod (Gadusmorhua): implications for environmental monitoring in the Barents Sea[J]. Aquat Toxicol, 2013, 127(2):21-35.
[3] Hannam M L, Bamber S D, Moody J A, et al. Immune function in the Arctic Scallop,Chlamysislandica, following dispersed oil exposure[J]. Aquat Toxicol, 2009, 92(3):187-194.
[4] Vahl O.Energy transformations by the Iceland scallop,Chlamysislandica(O. F. Müller), from 70°N. I.b The age-specific energy budget and net growth efficiency[J].J Exp Mar Biol Ecol, 1981,53(2):281-296.
[5] Arsenault D J, Himmelma J H. Size-related changes in vulnerability to predators and spatial refuge use by juvenile Iceland scallopChlamysislandica[J].Mar Ecol, 1996,140(1/2/3):115-122.
[6] Vahl O.Seasonal variatins in seston and in the growth rate of the iceland scallop,Chlamysislandica(O.F. Müller) from balsfjord, 70°N[J].J Exp Mar Biol Ecol,1980,48(2):195-204.
[7] Harvey M, Bourget E, Miron G. Settlement of Iceland scallopChlamysislandicaspat in response to hydroids and filamentous red algae: field observations and laboratory experiments[J].Mar Ecol, 1993,99(3):283-292.
[8] 邵武,于蛟,姜先.人类和14种哺乳类动物DNA的SON基因3'非编码区PCR测序种属鉴定技术的研究[M].北京:中国科学技术出版社, 2001.
[9] 邵武,姜先,于蛟,等.人和动物SON基因3′非编码区的PCR测序分析[J].中国法医学杂志,2003,18(2):87-89.
[10] 张艳,陈彬,陈敏,等.一种简单易行的重组方法——三引物PCR法[J].第三军医大学学报,2008,30(3):268-269.
[11] 岳志芹,刘荭,梁成珠,等.实时定量RT-PCR检测鱼类传染性造血器官坏死病毒方法的建立与应用[J].水生生物学报,2008,32(1):91-95.
[12] 张利峰,张鹤晓,乔卫虹,等.荧光RT-PCR检测鱼类鲤春病毒血症病毒的研究[J].检验检疫学刊,2005,15(6):22-25.
[13] Canapa A,Barucca M,Marinell A,et al.Molecular data from the 16S rRNA gene for the phylogeny ofPectinidae(Mollusca:Bivalvia)[J].J Mol Evol,2000,50:93-97.
[14] 任岗,章群,钱开诚,等.12种石鲈科鱼类线粒体16S rRNA基因的部分序列分析[J].热带海洋学报, 2007(3):48-52.
[15] 郭天慧,孔晓瑜,陈四清,等.三疣梭子蟹线粒体DNA 16S rRNA和COI基因片段序列的比较研究[J].中国海洋大学学报(自然科学版),2004(1):22-28.
[16] Miller A D, Murphy N P, Burridge C P, et al. Complete mitochondrial DNA sequences of the decapod crustaceans Pseudocarcinus gigas (Menippidae) and Macrobrachium rosenbergii (Palaemonidae) [J].Mar Biotechnol, 2005, 7(4):339-349.
[17] 李霞,秦艳杰,初冠囡,等.采用16S rRNA基因序列分析海扇贝与3种扇贝的亲缘关系[J]. 大连水产学院学报,2009,24(3):269-273.
[18] Zheng X, Wang R, Wang X, et al. Genetic variation in populations of the common Chinese cuttlefish Sepiellamaindroni (MolluscaCephalopoda) using allozymes and mitochondrial DNA sequence analysis[J]. J Shellfish Res,2001,20(3):1159-1165.
[19] Anderson F E.Phylogeny and historical biogeography of the loliginid squids (Mollusca:cephalopoda) based on mitochondrial DNA sequence data[J].Mol Phylogenet Evol, 2000, 15(2):191-214.
[20] Boulding E G. Genetic variation in one bottlenecked and two wild populations of the Japanese Scallop(Patinopectenyessoensis):empirical parameter estimates from coding regions of mitochondral DNA[J]. Can J Fish Aquat Sci, 1993, 50(6):1147-1157.
[21] 孔晓瑜.栉孔扇贝和海湾扇贝线粒体DNA16S rRNA基因片段序列研究[C]//贝类学论文集.北京:海洋出版社,2001:59-63.
[22] 高天祥,张秀梅,吉崎悟朗,等.中华绒螯蟹和日本绒螯蟹线粒体12S rRNA基因序列研究[J]. 中国海洋大 学学报, 2000,30(1):43-47.
[23] 冯艳微.珍珠贝亚目和蚶目DNA条形码与系统发生学研究[D].青岛:中国海洋大学,2012.
[24] Saavedra C, Pea J B. Phylogenetics of American scallops (Bivalvia:Pectinidae) based on partial 16S and 12S ribosomal RNA gene sequences[J].Mar Biol, 2006, 150(1):111-119.
[25] 胡丽萍,姜黎明,黄晓婷,等. 基于16S rDNA基因序列探讨引进物种紫扇贝(Argopectenpurpuratus)在海湾扇贝属(Argopecten)中的分类地位[J].海洋与湖沼,2016,47(6):1149-1157.

