闽西地区猪流行性腹泻病毒M基因遗传进化及分子结构特征分析
董 波,安永帅,戴爱玲,李晓华,杨小燕,*
(1.龙岩学院 生命科学学院,福建 龙岩 364012;2.福建省生猪疫病防控工程技术研究中心,福建 龙岩 364012)
闽西地区猪流行性腹泻病毒M基因遗传进化及分子结构特征分析
董 波1,2,安永帅1,戴爱玲1,2,李晓华1,2,杨小燕1,2,*
(1.龙岩学院 生命科学学院,福建 龙岩 364012;2.福建省生猪疫病防控工程技术研究中心,福建 龙岩 364012)
摘 要:为了解2015—2017年闽西地区猪流行性腹泻病毒(PEDV)遗传进化规律,本研究收集了闽西地区12份PEDV阳性样品,对其M基因进行RT-PCR扩增,克隆至pMD18-T载体测序,并与国内外已知参考毒株序列进行比对及遗传进化分析。结果表明,12株闽西毒株M基因氨基酸序列同源性为100.0%;遗传进化分析表明,12株闽西毒株与经典毒株CV777、2010年前中国分离毒株LCZ、以及韩国毒株SM98、virulent DR13的亲缘性较远,而与2010年后国内分离毒株以及2013年美国分离毒株亲缘关系最近,说明闽西PEDV毒株属于目前国内外流行毒株。分子结构特征分析表明,PEDV M基因具备高度保守的特性,可以作为设计疫苗的候选基因。
关键词:猪流行性腹泻病毒;M基因;进化特征;分子特征
猪流行性腹泻(porcine epidemic diarrhea,PED)是由猪流行性腹泻病毒(porcine epidemic diarrhea virus,PEDV)引起猪的一种急性、高度接触性肠道传染病,以水样腹泻、呕吐和脱水为临床特征,主要感染哺乳期仔猪,感染率和病死率极高,给养猪业造成极大损失[1-3]。该病第一次被报道于1971年的英国,之后在欧洲、亚洲相继有该病的出现[4-5]。我国第一次出现该病是在1976年,继而呈现大面积流行趋势[6]。自2010年冬季以来,我国10多个省市暴发PED,对国内养猪业带来了严重影响,甚至出现以前接种过PED疫苗的猪也未能幸免,提示该流行毒株毒力较强,而且与现有疫苗毒株存在较大的差异,导致现有疫苗难以提供免疫保护[7-8]。2011年,张世忠等[9]对福建省各个地区的规模化养猪的顽固性腹泻病例进行检测,PEDV感染率高达52.2%。由此可见,该病一旦暴发流行,其危害十分严重,对养猪业的潜在危害不容忽视。2013年,美国多个洲先后有暴发PEDV,病猪表现出水样腹泻、急性肠炎、呕吐等典型症状,死亡率极高,给美国养猪业造成重创,引起了美国乃至全球养猪业的高度重视[10-11]。
PEDV为单股正链RNA病毒,全长大约28kb,由4种结构蛋白组成,分别为纤突蛋白(spike,S)、膜蛋白(membrane,M)、核衣壳蛋白(nucleocapsid,N)和小膜蛋白(small membrane,E)[12]。其中,M基因全长681bp,编码226个氨基酸,位于病毒表面,是病毒囊膜中含量最多的基因[13]。M基因在PEDV的结构基因中,相对较为保守,然而,近些年的相关文献显示,M基因也呈现了进化和变异的趋势。闽西地区地处闽粤赣三省交界,一旦出现病毒变异并迅速蔓延,对三省乃至全国的养猪业将造成不小的影响。因此,本研究收集了2015年1月—2017年6月闽西地区规模化猪场送检PEDV阳性病例,采用RT-PCR技术,扩增阳性样品全长M基因,旨在从分子水平了解PEDV闽西地区流行毒株的特点,进一步阐明闽西地区流行毒株的遗传变异规律,为我国PEDV地方毒株的遗传演化分析提供基础资料,并为下一步制备PEDV疫苗提供参考依据。
1 材料与方法
1.1 病毒、野外样品、质粒与菌种
PEDV弱毒疫苗购自福州大北农公司;野外阳性样品为2015年1月—2017年6月年期间采自闽西地区规模化养猪场送检腹泻症状仔猪的粪便和小肠组织样品12份;大肠埃希菌DH5α感受态细胞,由本实验室制备、保存;pMD18-T Vector购自宝生物工程(大连)有限公司。
1.2 主要试剂
DL2000 Marker、总RNA提取液(TaKaRa RNAiso Reagent)、病毒RNA/DNA提取试剂盒(TaKaRa MiniBEST Viral DNA/RNA Extraction Kit Ver.3.0)、AMV反转录酶、PrimeSTAR® HS DNA Polymerase 高保真酶、Oligo(dT)18、dNTP、DNA Marker DL 2000、RNA酶抑制剂购自宝生物工程(大连)有限公司;DNA凝胶回收试剂盒(DNA Gel Extraction Kit)及质粒提取试剂盒(Plasmid Miniprep Kit)购自AXYGEN公司产品;感受态细胞制备试剂购自宝生物工程(大连)有限公司;氯仿、异丙醇采用分析纯试剂。
1.3 总RNA提取及逆转录
按照试剂盒提供的操作手册,自粪便和小肠组织以及灭活的PEDV样品(阳性对照)中提取总RNA。取洁净EP管,加入RNA 11μL、Oligo(dT)18 1μL,12000g离心40s,于70℃水浴10min,加入5×M-MLV Buffer 5μL、M-MLV 1μL、dNTP 1μL、DEPC水 6μL,混匀后,42℃水浴30min,95℃水浴10min。cDNA于-80℃下保存。
1.4 M基因的扩增、纯化
参考GenBank上发表的PEDV CV777株M基因序列,使用Premier Premier 5.0生物软件设计合成一对特异性引物,PEDV-M-F:TTCTATTCCCGTTGATGAGG;PEDV-M-R:CATGAAGCACTTTCTCACTATC。以cDNA为模板,扩增病毒M基因。反应条件为:95℃预变性5min;94℃变性30s,56℃退火30s,72℃延伸45s,35个循环;最后72℃延伸10min。产物经电泳分离,凝胶切割纯化,凝胶成像系统下观察结果。
1.5 重组质粒构建和鉴定
取5μL纯化产物与0.5μL pMD18-T载体(50 ng·μL-1)进行连接反应,16℃连接4h后,将连接产物转化入DH5α感受态细胞,涂板过夜,挑取单个白色菌落,每个样品挑选3个菌落,以保证测序准确性。过夜培养,提取质粒,经鉴定正确后,送上海生工生物工程技术服务有限公司测序。
1.6 序列比对、同源性及系统发育分析
使用DNAman、DNAstar、MEGA5.2分子生物学软件,对M基因序列进行同源性及系统发育分析。
2 结果与分析
2.1 M基因的扩增、纯化、克隆及测序
12份阳性样品编号为: FJLY1501、FJLY1502、FJLY1503、FJLY1504、FJLY1601、FJLY1602、FJLY16031、FJLY1604、FJLY1701、FJLY1702、FJLY1703、FJLY1704,对M基因进行PCR扩增。M基因PCR产物经扩增后,使用凝胶电泳进行鉴定,结果显示获得大小约为681bp基因片段(图1)。将纯化后的PCR产物连接到pMD18-T载体,将阳性克隆送往上海生工生物工程技术服务有限公司测序,序列结果正确。
2.2 M基因氨基酸序列比对及同源性分析
将12份阳性样品的氨基酸序列与国内外已知代表毒株M基因序列进行比对及同源性分析。所引用的已知代表毒株信息见表1。12份阳性样品M基因与其他代表性毒株M基因进行氨基酸同源性比较后发现,闽西毒株M基因之间的同源性为100.0%,与经典毒株CV777和LCZ的同源性分别为98.7%和98.6%,与2014年福建分离毒株FJ-ZP和FJ-FQ的同源性分别为99.6%和99.7%。与2010年以后中国分离毒株的同源性均在99%以上,与2013年美国毒株的同源性也均在99%以上。
2.3 M基因系统发育分析
遗传进化分析表明,42株PEDV可以分成两个大组。其中,Ⅰ组包括经典毒株CV777,3株韩国毒株,6株中国毒株;Ⅱ组则包括1株韩国毒株,4株美国毒株,22株2010年后中国毒株以及12株闽西分离毒株。系统发育分析显示,闽西分离毒株与经典毒株CV777、2010年前中国分离毒株LCZ、SM98以及韩国毒株virulent DR13的亲缘性较远,与2010年以后中国分离毒株的亲缘性较高,与2013年美国毒株亲缘性也较高。见图2。
2.4 M基因氨基酸结构特征
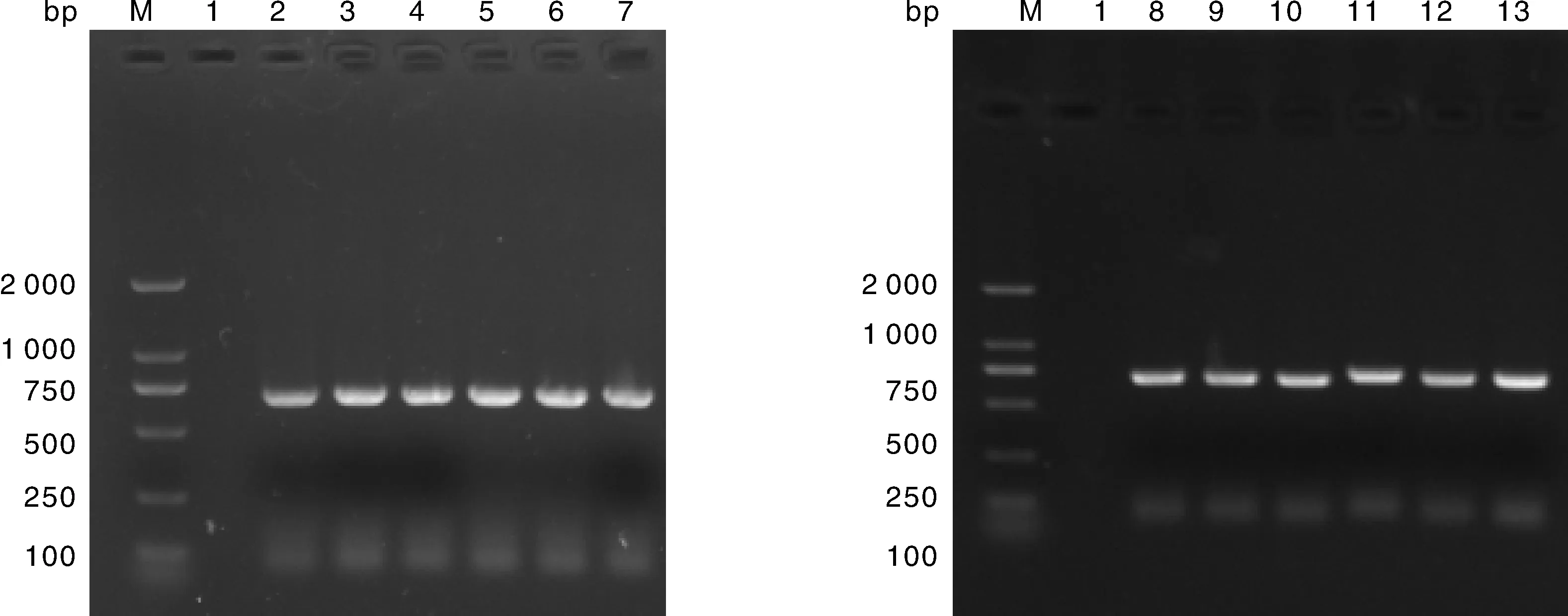
M,DL2000 marker;1,阴性对照;2~13依次为FJLY1501、FJLY1502、FJLY1503、FJLY1504、FJLY1601、FJLY1602、FJLY16031、FJLY1604、FJLY1701、FJLY1702、FJLY1703、FJLY1704。M,DL2000 marker;1,Negative control;2-13,FJLY1501,FJLY1502,FJLY1503,FJLY1504,FJLY1601,FJLY1602,FJLY16031,FJLY1604,FJLY1701,FJLY1702,FJLY1703,FJLY1704.图1 M基因PCR扩增纯化结果Fig.1 PCR amplification result of M genes
表1序列比对及系统发育分析中引用的PEDV毒株
Table1Cited PEDV virus strains in sequence alignment and phylogenetic analysis
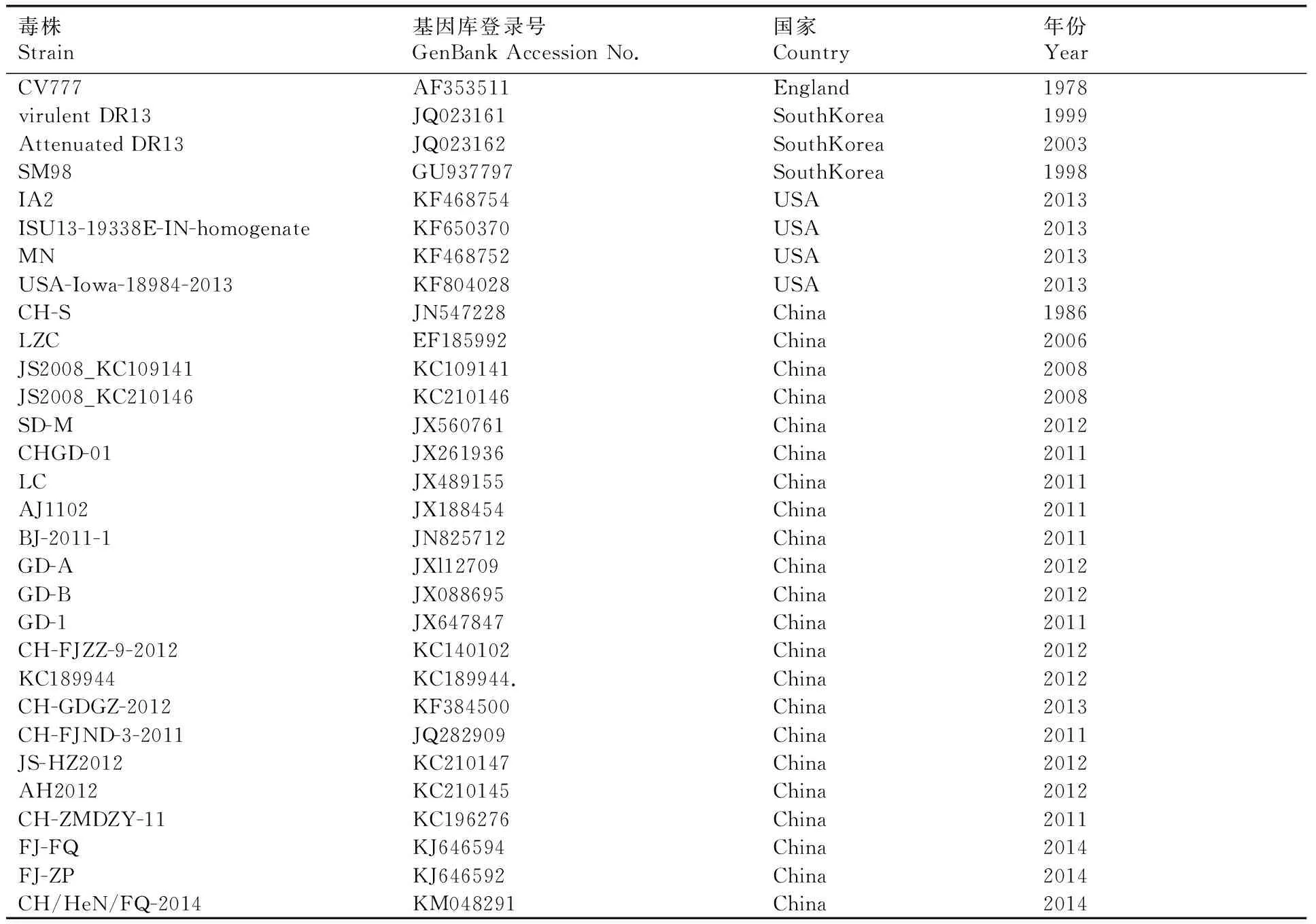
毒株Strain基因库登录号GenBank Accession No.国家Country年份YearCV777AF353511England1978virulent DR13JQ023161SouthKorea1999Attenuated DR13JQ023162SouthKorea2003SM98GU937797SouthKorea1998IA2KF468754USA2013ISU13-19338E-IN-homogenateKF650370USA 2013MNKF468752USA2013USA-Iowa-18984-2013KF804028USA2013CH-SJN547228China1986LZCEF185992China2006JS2008_KC109141KC109141China2008JS2008_KC210146KC210146China2008SD-MJX560761China2012CHGD-01JX261936China2011LCJX489155China2011AJ1102JX188454China2011BJ-2011-1JN825712China2011GD-AJXl12709China2012GD-BJX088695China2012GD-1JX647847China2011CH-FJZZ-9-2012KC140102China2012KC189944KC189944.China2012CH-GDGZ-2012KF384500China2013CH-FJND-3-2011JQ282909China2011JS-HZ2012KC210147China2012AH2012KC210145China2012CH-ZMDZY-11KC196276China2011FJ-FQKJ646594China2014FJ-ZPKJ646592China2014CH/HeN/FQ-2014KM048291China2014
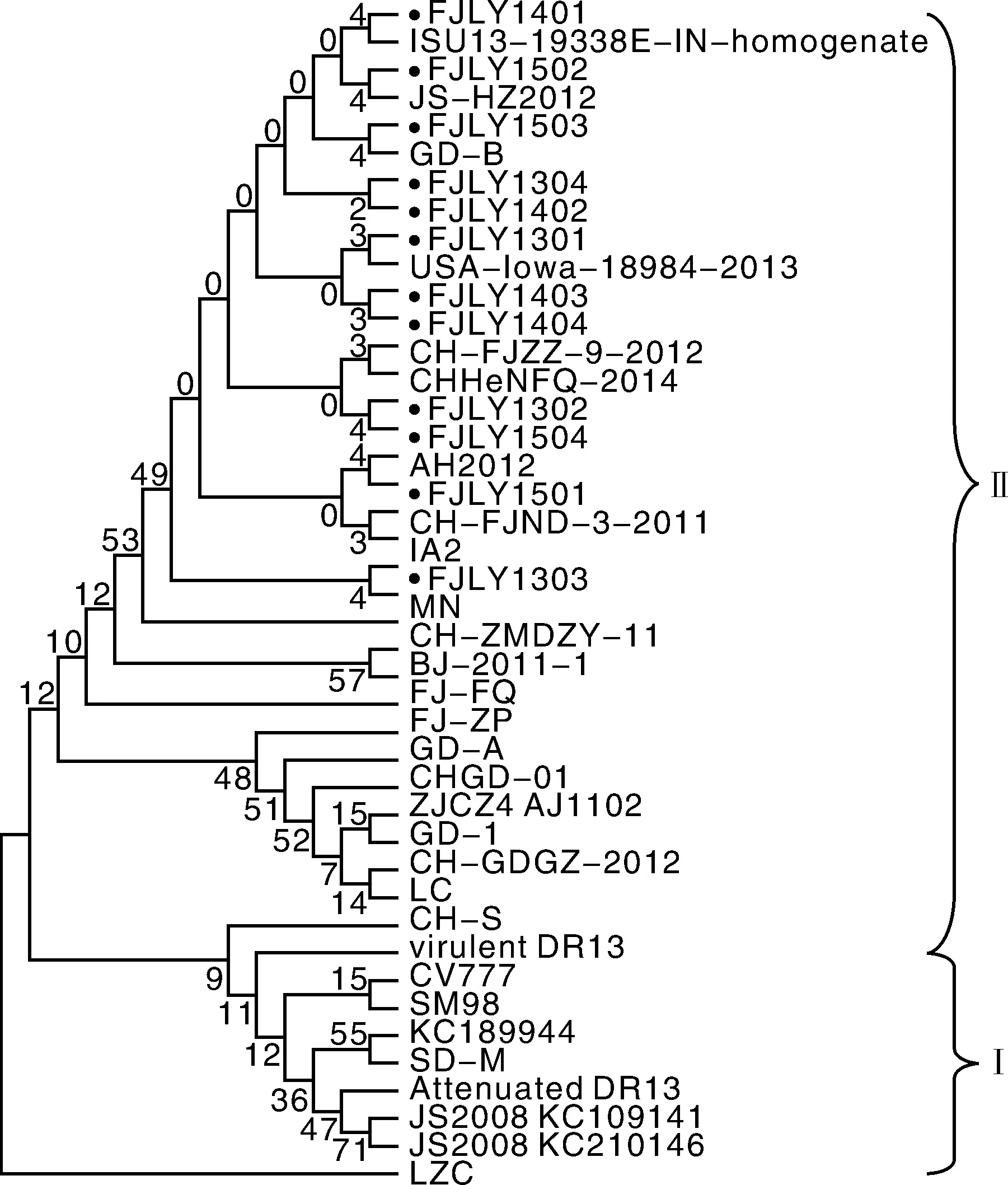
图2 PEDV M基因系统发育分析Fig.2 Phylogenetic analysis based on M genes of PEDV
M基因在PEDV中是相对保守的基因,序列比对结果表明,M基因全长为681bp,编码226个氨基酸,包含一个完整的阅读框架。闽西分离毒株与经典毒株CV777相比,氨基酸序列仅存在3处突变,分别为12E-Q、42A-V、213A-S,见表2。这些突变位置与2010年后中国变异毒株一致。N-糖基化位点预测结果表明,经典毒株CV777含有两个N-糖基化位点,而闽西分离毒株的N-糖基化位点与经典毒株CV777一致,见表3。M基因其他预测结果表明,闽西分离毒株的穿膜肽、疏水性、pI值等方面与经典毒株CV777无明显差异,见表4。

表3M基因潜在糖基化位点比对分析
Table3Alignment of mutant amino acids of potential glycosylation sites on M genes
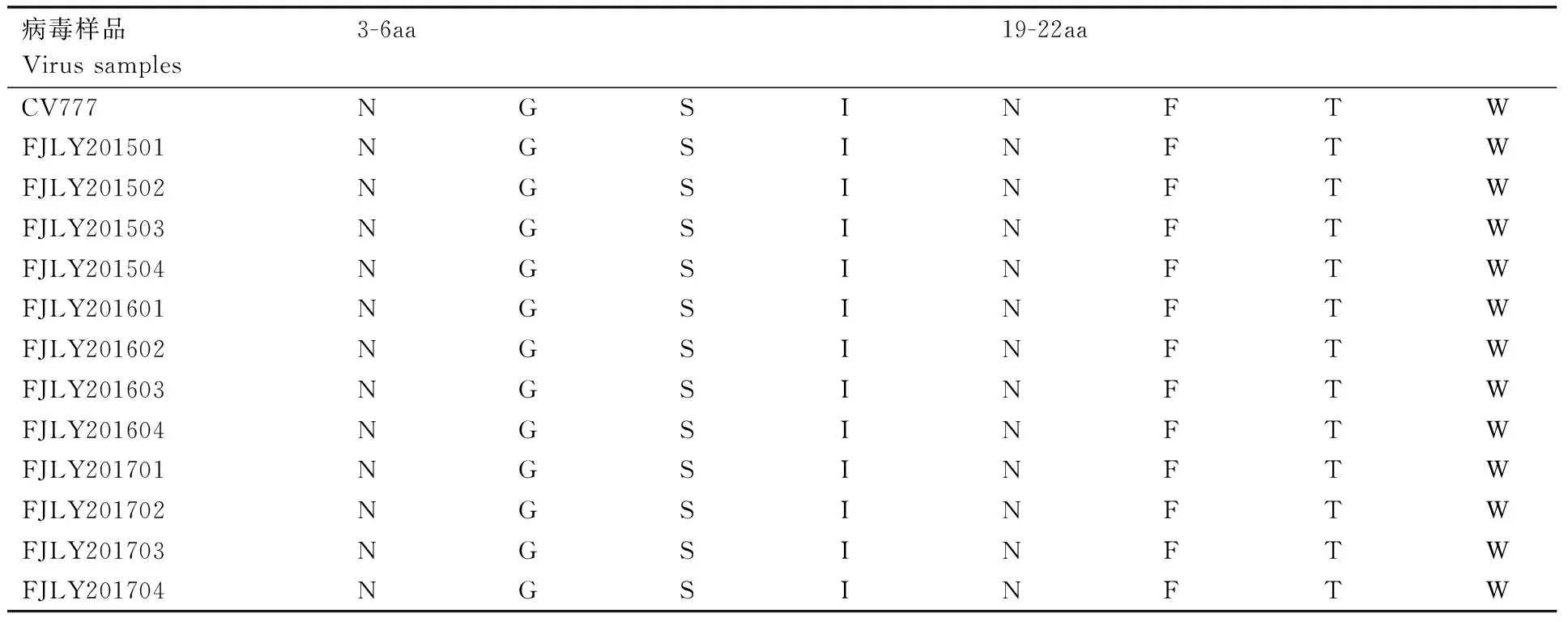
病毒样品Virus samples3-6aa19-22aaCV777NGSINFTWFJLY201501NGSINFTWFJLY201502NGSINFTWFJLY201503NGSINFTWFJLY201504NGSINFTWFJLY201601NGSINFTWFJLY201602NGSINFTWFJLY201603NGSINFTWFJLY201604NGSINFTWFJLY201701NGSINFTWFJLY201702NGSINFTWFJLY201703NGSINFTWFJLY201704NGSINFTW
表4M基因编码蛋白性质预测
Table4Predicted protein characterizations of deduced protein of M gene

病毒样品Virus samples疏水区Hydrophobic region穿膜肽Cell-penetrating peptidepICV77722-33aa;48-64aa;75-95aa;130-135aa;141-145aa20-38aa;43-65aa;75-97aa9.31FJLY20150122-33aa;48-64aa;75-95aa;130-135aa;141-145aa20-38aa;43-65aa;75-97aa9.54FJLY20150222-33aa;48-64aa;75-95aa;130-135aa;141-145aa 20-38aa;43-65aa;75-97aa9.54FJLY20150322-33aa;48-64aa;75-95aa;130-135aa;141-145aa20-38aa;43-65aa;75-97aa9.54FJLY20150422-33aa;48-64aa;75-95aa;130-135aa;141-145aa20-38aa;43-65aa;75-97aa9.54FJLY20160122-33aa;48-64aa;75-95aa;130-135aa;141-145aa 20-38aa;43-65aa;75-97aa9.54FJLY20160222-33aa;48-64aa;75-95aa;130-135aa;141-145aa 20-38aa;43-65aa;75-97aa9.54FJLY20160322-33aa;48-64aa;75-95aa;130-135aa;141-145aa20-38aa;43-65aa;75-97aa9.54FJLY20160422-33aa;48-64aa;75-95aa;130-135aa;141-145aa20-38aa;43-65aa;75-97aa9.54FJLY20170122-33aa;48-64aa;75-95aa;130-135aa;141-145aa 20-38aa;43-65aa;75-97aa9.54FJLY20170222-33aa;48-64aa;75-95aa;130-135aa;141-145aa20-38aa;43-65aa;75-97aa9.54FJLY20170322-33aa;48-64aa;75-95aa;130-135aa;141-145aa20-38aa;43-65aa;75-97aa9.54FJLY20170422-33aa;48-64aa;75-95aa;130-135aa;141-145aa20-38aa;43-65aa;75-97aa9.54
3 讨论
PEDV是PED的致病原,主要感染对象为哺乳期仔猪,致死率高,对养猪业的危害非常严重。研究证实,PEDV只存在一个血清型,然而,分子流行病学调查显示,近十年来我国流行毒株与欧洲流行毒株、原始毒株比较,我国流行毒株的结构蛋白基因(M、N、S)均与欧洲流行毒株及原始毒株亲缘关系较远,可能出现了新的基因型[14]。2011年,刘孝珍等[15]将17株分离得到的PEDV S基因进行遗传进化分析研究,证实我国出现了PEDV新的基因型。M基因能诱导机体产生针对病毒的中和抗体,在病毒粒子的组装和出芽过程中具有重要作用[16]。M基因是高度保守的基因,能介导机体产生干扰素,常用作RT-PCR诊断靶基因和疫苗候选基因[17]。在遗传进化的分析中,选择M基因,有助于更多地了解病毒的流行趋势与进化规律,对于病毒的防控和疫苗的研制具有重要意义。
2011年冬末,广东、福建等地区持续暴发仔猪腹泻,引起了学者的关注,学者们普遍认为,造成这次PEDV暴发的毒株为变异毒株。自此以后,该变异毒株成为我国主要流行毒株。2013年,张志等[18]将分离到的PEDV阳性毒株M基因进行遗传进化分析发现,新分离毒株M基因与2010年后我国新分离毒株亲缘性较高,为国内变异毒株。本研究中,闽西毒株M基因氨基酸序列与2010年以后中国分离毒株以及2013年美国毒株的同源性均在99%以上,证实了闽西流行毒株与国内其他地区流行毒株关系密切,属于2010年后国内分离变异毒株。
2013年,美国暴发了猪流行性腹泻,给美国养猪业造成了严重影响,经济损失巨大。这次暴发的PEDV,与2010年我国暴发的PEDV的流行方式极为相似,并且学者通过进化分析证实,2013年在美国暴发的PEDV与2010年在中国流行的PEDV,在亲缘性上很相近,可以认为来自同一祖先。本研究中,42株病毒可以分成2个大组,Ⅰ组和Ⅱ组。其中,Ⅰ组主要包括CV777、LZC和SM98为代表的的经典株,以及韩国DR13弱毒株和国内早期毒株;而Ⅱ组包含毒株均为2010年后中国分离毒株以及2013年后美国分离毒株,这也证实了闽西毒株与2010年后国内分离毒株和2013年后美国分离毒株进化关系更为紧密,为当前国内外流行毒株。同时,亲缘比较结果表明,闽西毒株与疫苗研发株CV777亲缘关系较远,提示我们现在猪场所使用的疫苗,对于防治PED的流行所起到的作用并不显著,这也成为控制PEDV暴发的主要隐患。
M基因氨基酸结构特征比较显示,与经典毒株CV777相比,闽西毒株无特有的片段缺失和插入,12份毒株存在3处相同突变,分别为12E-Q、42A-V、213A-S,这些突变位置与2010年后分离毒株一致,证实了闽西毒株属于2010年后国内分离变异毒株。闽西毒株N-糖基化位点预测结果与经典毒株CV777一致,且穿膜肽、亲水性、pI值等方面与经典毒株CV777也无差异,这些结果进一步证明了PEDV M基因具备高度保守的特性,可以作为设计疫苗的候选基因。
综上,通过比较分析2015—2017年闽西地区PEDV M基因氨基酸序列发现,闽西毒株M基因与2010年国内分离毒株以及2013年美国分离毒株同源性较高,属于国内外流行毒株。并且,蛋白预测结果表明,虽然闽西毒株M基因与经典毒株CV777相比存在3处基因改变,但是大部分性质没有明显变化,证明M基因是高度保守的。这些结果的获得,有助于从分子水平了解闽西地区PEDV M基因分子结构及遗传变异特征,为进一步研制有效的生物制剂和诊断方法提供资料支持,同时为猪流行性腹泻的防控提供病原学和流行病学参考依据。
:
[1] 刘景华,殷震.动物病毒学 [M].2版.北京:科学出版社,1997:681-688.
[2] 吕茂杰,陈建飞,时洪艳,等.猪流行性腹泻病毒核衣壳蛋白与感染细胞核磷蛋白的共定位分析[J].微生物学报,2011,61(5):643-647.
LYU M J,CHEN J F,SHI H Y,et al.Co-localization analysis between porcine epidemic diarrhea virus nucleocapsid protein and nucleolar phosphoprotein B23.1[J].ActaMicrobiologicaSinica,2011,61(5):643-647.(in Chinese with English abstract)
[3] CAVANAGH D.Nidovirales:a new order comprising Coronaviridae and Arteriviridae[J].ArchivesofVirology,1997,142(3):629-633.
[4] WOOD E N.An apparently new syndrome of porcine epidemic diarrhoea[J].VeterinaryRecord,1977,100(12):243-244.
[5] PENSAERT M B,BOUCK P D.A new coronavirus-like particle associated with diarrhea in swine[J].ArchivesofVirology,1978,58(3):243-247.
[6] 倪艳秀,林继煌,何孔旺,等.猪流行性腹泻研究概况[J].畜牧与兽医,2001,33(1):38-40.
NI Y X,LIN J H,HE K W,et al.A survey of porcine epidemic diarrhea[J].AnimalHusbandry&VeterinaryMedicine,2001,33(1):38-40.(in Chinese)
[7] PAN Y,TIAN X,WEI L,et al.Isolation and characterization of a variant porcine epidemic diarrhea virus in China[J].VirologyJournal,2012,9(1):195.
[8] SUN R Q,CAI R J,CHEN Y Q,et al.Outbreak of porcine epidemic diarrhea in suckling piglets,China[J].EmergingInfectiousDiseases,2012,18(1):161-163.
[9] 张世忠,江斌.2011年福建省猪流行性腹泻的流行特点及其防治措施[J].福建畜牧兽医,2012,34(2):23-25.
ZHANG S Z,JIANG B.Epidemiological characteristics and control measures of swine epidemic diarrhea in Fujian Province in 2011[J].FujianJournalofAnimalHusbandryandVeterinaryMedicine,2012,34(2):23-25.(in Chinese)
[10] STEVENSON G W,HOANG H,SCHWARTZ K J,et al.Emergence of Porcine epidemic diarrhea virus in the United States:clinical signs,lesions,and viral genomic sequences[J].JournalofVeterinaryDiagnosticInvestigation,2013,25(5):649-654.
[11] CHEN Q,LI G,STASKO J,et al.Isolation and characterization of porcine epidemic diarrhea viruses associated with the 2013 disease outbreak among swine in the United States[J].JournalofClinicalMicrobiology,2014,52(1):234-243.
[12] BRIAN D A,BARIC R S.Coronavirus genome structure and replication[J].CurrentTopicsinMicrobiology&Immunology,2005,287:1-30.
[13] VENNEMA H,DE GROOT R J,HARBOUR D A,et al.Primary structure of the membrane and nucleocapsid protein genes of feline infectious peritonitis virus and immunogenicity of recombinant vaccinia viruses in kittens[J].Virology,1991,181(1):327-335.
[14] 陈建飞,冯力,时洪艳,等.猪流行性腹泻病毒CH/S株N蛋白基因的遗传变异及其原核表达[J].中国预防兽医学报,2007,29(11):856-860.
CHEN J F,FENG L,SHI H Y,et al.Genetic variation and prokaryotic expression of N protein gene of porcine epidemic diarrhea virus CH/S strain[J].ChineseJournalofPreventiveVeterinaryMedicine,2007,29(11):856-860.(in Chinese with English abstract)
[15] 刘孝珍,陈建飞,时洪艳,等.2011年猪流行性腹泻病毒的遗传变异分析[J].中国预防兽医学报,2012,34(3):180-183.
LIU X Z,CHEN J F,SHI H Y,et al.Genetic variation analysis of porcine epidemic diarrhea virus isolated in 2011[J].ChineseJournalofPreventiveVeterinaryMedicine,2012,34(3):180-183.(in Chinese with English abstract)
[16] DE HAAN C A,KUO L,MASTERS P S,et al.Coronavirus particle assembly:primary structure requirements of the membrane protein[J].JournalofVirology,1998,72(8):6838-6850.
[17] REN X,LI P.Development of reverse transcription loop-mediated isothermal amplification for rapid detection of porcine epidemic diarrhea virus[J].VirusGenes,2011,42(2):229-235.
[18] 张志,李岚,董雅琴,等.猪流行性腹泻病毒PCR检测及其M基因的遗传变异分析[J].动物医学进展,2013,34(8):13-18.
ZHANG Z,LI L,DONG Y Q,et al.PCR detection of porcine epidemic diarrhea virus and genetic analysis of M gene[J].ProgressinVeterinaryMedicine,2013,34(8):13-18.(in Chinese with English abstract)
EvolutionaryandmolecularcharacteristicsofMgenesofporcineepidemicdiarrheavirusesinwesternFujian,China
DONG Bo1,2,AN Yongshuai1,DAI Ailing1,2,LI Xiaohua1,2,YANG Xiaoyan1,2,*
(1.CollegeofLifeScienceofLongyanUniversity,Longyan364012,China;2.FujianEngineeringResearchCenterforSwineDiseaseControlandPrevention,Longyan364012,China)
Abstract:To elucidate evolutionary characteristics of the memberane(M) genes of porcine epidemic diarrhea viruses in Fujian,China,12 samples collected from Fujian province during 2015 to 2017 were screened by multiplex RT-PCR,M genes of PEDV viruses from positive samples were amplified by RT-PCR and cloned into pMD18-T vectors for sequencing respectively.The sequence data were done alignment and phylogenetic analysis with sequences from known reference strains.The M genes from 12 representative positive samples shared100.0% identity at amino acid level.12 representative positive samples are remote to classical strain CV777,SM98 and virulent DR13.12 representative strains belong to the genotype variant strain.Molecular structure characteristics analysis showed that M gene is highly conservative and could be an optimal candidate protein for vaccine development.
Key words:porcine epidemic diarrhea virus;M gene;evolutionary characteristics;molecular characteristic
中图分类号:S858.28
A
文章编号:1004-1524(2018)06-0939-07
收稿日期:2017-09-28
基金项目:福建省科技厅重大专项专题(2014NZ0002);福建省高校自然基金青年重点项目(JZ160481);福建省教育厅中青年科技项目(JAT160483);龙岩学院博士启动项目(LB2014004);大学生创新创业计划项目(2016017)
作者简介:董波(1983—),男,天津人,博士,讲师,主要从事动物病毒学研究。E-mail:381289930@qq.com
,杨小燕,E-mail:1906834157@qq.com
10.3969/j.issn.1004-1524.2018.06.08
(责任编辑张 韵)

