植物核DNA含量在全球尺度上的纬度变异式样及其气候适应意义
——以菊科植物为例
解 梦,于 晶,郭水良
上海师范大学生命与环境科学学院, 上海 200234
DNA C-值指的是一个物种配子核中没有复制时的DNA 含量,一个物种的单个染色体组的DNA含量称为基因组大小[1]。一个物种的基因组大小等于其DNA C-值除于其倍性水平。传统的观点认为,DNA C-值(或基因组大小)具有物种的特异性,对于物种来讲是高度恒定的,C代表恒定的意思[2]。随着工作的深入,植物核DNA含量不仅在种间存在着巨大的差异,而且亲缘关系近的种类间也存在着巨大的差异[3],并且呈现出分类学、生物地理学、生态适应上的意义[4]。Bennett等发现,一些重要的世界性杂草的基因组要比其它植物的小,恶性杂草的基因组要比一般性杂草的小[3];郭水良等对中国境内被子植物DNA C-值的统计和测定发现,具有入侵性的植物的DNA C-值往往小于亲缘关系相近的非入侵性植物[5- 6]。比利时菊科的Centaurea属植物 DNA C-值与海拔高度存在正相关关系;部分分类群中,植物核DNA含量随着纬度、经度、海拔,以及气温雨量差异呈现有规律的变化,这可能是DNA 对气候条件适应的结果[7]。核DNA含量种间差异,被认为是生态位分化的重要原因。例如与鸟类相比,两栖类不同物种间的 C 值相差更大,这与两栖类动物生活环境具有极大多变化性有关。同样,高等植物分类群间极大的核DNA含量差异,也和高等植物生境高度多样化有关[8]。
但是,关于核DNA含量与地理和气候因素关系的结论并不一致。早期研究表明核DNA含量与纬度呈正相关[9];但是Grime和Mowforth发现英国植物区系中169种植物的DNA C-值和纬度之间存在负相关[10];Razafinarivo等发现,咖啡树的DNA C-值在马达加斯加由北向东南、在非洲大陆由东向西均呈增加趋势[11]; Bottini发现,小檗属植物(Berberis)二倍物种中,核DNA含量小的类群倾向于分布在降雨大、但是水分可利用性低的高海拔地区,而核DNA含量高的类群则倾向于分布在营养生长期较长、水份可利用性高的中海拔森林区域[12]。也有其他的一些研究发现核DNA含量与环境之间没有什么相关性[13-14]。
在大多数早期文献中,核DNA含量与纬度(或海拔)的关系被描述成线性关系。但是,Knight和Ackerly通过分位数回归分析发现,加州植物区系中,DNA 2C-值大的物种在7月极端高温过高或过低地区的分布频度均下降,因此,他们推测核DNA含量与环境因素之间可能存在着非线性关系[15]。李丹丹等发现,中国热带到温带广大区域内稗草(Echinochloacrus-galli)种群的核DNA含量与经度、纬度和10个生物因素均存在明显的非线性关系,大多数情况下均能够用单峰函数进行拟合[16]。Rayburn和Auger发现具有最高核DNA含量的玉米种群倾向分布于中海拔的环境中,随着向高或低海拔变化,玉米的核DNA含量呈现下降趋势[17]。取样范围过窄,会影响到人们对核DNA含量与纬度、气候因素关系的正确认识[16]。但是,核DNA含量与环境因素之间的这种非线性关系是否普通?全球范围内、在更广的分类群中核DNA含量与地理和气候因素之间的关系如何,有待进一步研究。
综合先前有关核DNA含量与环境因子之间关系的报道发现,早期的工作多数仅涉及个别分类群,或取样范围过窄,这可能会影响人们对核DNA含量与环境因子关系的正确认识。菊科是世界性分布的被子植物最大科,该科植物有不少种类已经有核DNA数据和比较详细的地理分布。本文的目的是,以菊科植物为材料,通过核DNA含量在全球范围内变化式样分析,了解大的空间尺度上植物核DNA含量是否存在有规律的非线性的地理梯度变化,以及这种变化是否具有环境适应意义。
1 研究方法
1.1 数据来源
(1)核DNA含量的数据来源
菊科核DNA含量数据来自于被子植物DNA C-值数据库[18]。基因组大小是DNA 2C-值除于倍性水平[3]。在菊科植物中有822种检索到了DNA 1C-值数据,有400种已有倍性信息,计算到得了基因组大小。
(2)为定量分析经纬度及气候因素对菊科植物核DNA含量的影响,在经度方向上分成10条样带,每条样带横跨15度,分别为样带1至10分别为90°—75°W、75°—60°W、60°—45°W、0°—15°E、15°—30°E、30°—45° E、75°—90°E、90°—105°E、105°—120°E和120°—135°E;其中,第1、2、3样带南北方向上涵盖了北美和南美洲,第4、5、6样带涵盖了欧洲和非洲,第7、8、9和10样带涵盖亚洲和大洋洲。对每条样带,再从82.5°N到82.5°S的范围内均分成22个样块,每样块在纬度梯度上的区间为7.5度(图1)。10条样带共分成面积一致的220个样块。
图1 10条样带位置Fig.1 Location of ten transects
(3)物种地理分布数据来源
822种菊科植物在上述220个样块中有无分布的数据来自于全球生物多样性信息网站(www.gbif.org)。
(4)生物气候数据来源
10个样带中,第5样带数据量丰富,随着纬度菊科植物的DNA C-值呈现有规律的变化。以第五样带为对象,进一步分析气候因素对核DNA含量的影响。首先从世界气候数据网站下载1950—2000年时间段的14个气象数据( http://www.worldclim.org,表1),数据的空间分辨率为30 arc- seconds,即每一栅格大致相当于0.86 km2,再应用ArcGIS 9.3获得以上气候数据在第5样带每样块中的平均值。

表1 14个气候因子
1.2 统计分析
首先获得每一个样块中出现的有核DNA含量数据的菊科植物种类,对出现5种以上植物的样块进行统计分析,计算每个样块菊科植物平均的DNA 1C-值、基因组大小及其标准误。
以第5样带上每个样块平均的生物气候因子值为自变量(x),以DNA C-值为因变量(y),应用EXCEL作散点图,发现DNA C-值和纬度,以及和大多数气候因子呈现非线性关系。进一步计算14个气候因子之间的Pearson相关系数,发现部分气候因子之间有很高的相关性,暗示它们之间存在多重共线性(Multicollinearity)问题。由于气候因子与DNA C-值之间是非线性关系,所以没有采用常规的多元逐步线性回归来筛选变量。为避免气候因子之间的多重共线性对数据分析的影响,首先应用主成分分析对数据进行降维,筛选出前几个主成分上载荷量最高的气候因子,再以它们为对象,删除与它们Person相关系数高的气候因子,保留与它们相关系数在-0.7至0.7之间的气候因子。应用XLSTAT软件拟合DNA C-值与筛选出的气候因子之间的关系,应用拟合效果最好的函数来反映气候因子与DNA C-值之间的关系。为反映气候因子对DNA C-值影响的程度,拟合前各气候因子采用了极差标准化,使它们的变化范围均处于0—1之间。
2 结果与分析
2.1 菊科植物核DNA含量的变异范围
822种菊科植物最大的DNA 1C值为26.15 pg(Sececiopendulus),最小的为0.40 pg(Leontodonlongirostris),平均为4.11 pg;基因组最大的为8.5 pg,最小的为0.19 pg, 平均为2.23 pg。
2.2菊科核DNA含量在全球10个样带上的变异趋势
菊科植物在10个样带上的核DNA含量变化分别见图2。图2显示在赤道及附近样块中菊科植物核DNA含量有变小趋势。随着向南、向北纬度的增高,核DNA含量上升。除了第1条带上的基因组数据外,其余样带上DNA 1C-值和基因组大小与纬度关系均能够用多项式拟合,达到显著或极显著关系。在第一样带上,DNA 1C-值与纬度也符合两次函数关系。
图2的第1、2、5三个样带的少数样块,有基因组大小值高于DNA C-值的情况,这可能是样本来源不同造成。即统计的DNA C-值的种类中,仅有部分有染色体倍性数据,恰好这部分多数是二倍体,而且DNA C-值又比较大(基因组也大,因为对于二倍体植物来讲,它们的基因组大小与DNA C-值数据是一致的)。


图2 全球10条样带上菊科植物核DNA含量变化变异情况Fig.2 Variation of nuclear DNA content of Asteraceae species across ten global latitudinal transects
2.3 气候因素对第5样带菊科DNA 1C-值的影响
对样带5上14个样块对应的14个气候因子进行主成分分析。结果第1、2主成分的方差分别占总方差的58.9%和28.6%,累计达85.5%。因此选择前二个主成分作进一步分析。14个气候因子在前二个主成分上的载荷(表2)。

表2 第5样带上14个生物气候因子在前两个主成分上的信息负荷量
气候因子1—14与表1中的相同
第一主成分主要反映了温度因子(C1—7)信息,其中最冷季度平均温度(Mean temp. of coldest quarter,7)具有最大的载荷。第二主成分主要反映了雨量因子(C8—C14)信息,其中最干季节雨量(C12)具有最大的载荷。首先选择这两个因子,再选择其他因子,使参与拟合的因子间的相关系数处于-0.7至+0.7之间。最后筛选出了最冷季度平均温度(C7)、最湿月份雨量(C9)和最干季度雨量(C12)3个气候因子,进一步分析它们对菊科植物DNA C-值的影响。通过拟合发现,二项式比线性关系能够更好地描述这3个气候因子与DNA C-值间的关系(图3)。
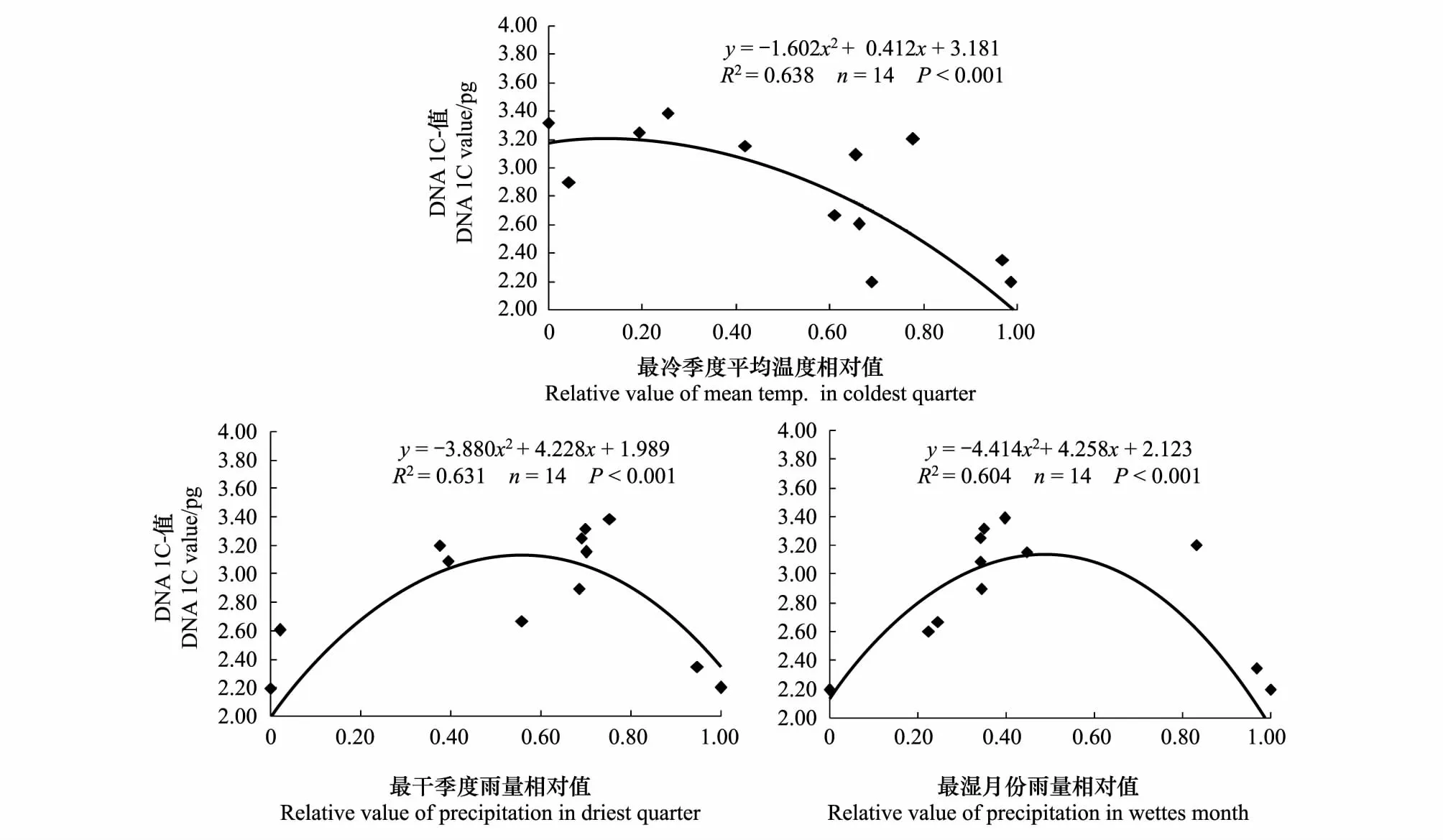
图3 3个生物气候因子对第5样带上菊科植物DNA 1C值的影响Fig.3 Influences of three climatic factors on DNA 1C-values of Asteraceae on Section 5
为比较三个气候因子对核DNA C-值影响的大小,进一步拟合DNA 1C-值与三个气候因子间的非线性关系,得到如下函数:
式中:y为DNA 1C-值,x1、x2和x3分别为最冷季度平均温度、最湿月份雨量和最干季度雨量。

图4 3个生物气候因子对第5样带上菊科植物DNA 1C值影响大小的比较Fig.4 Comparison of influences of three climatic factors on DNA 1C-values of Asteraceae on Section 5
最冷季度平均温度、最湿月份雨量和最干季度雨量均显著地影响到菊科核DNA C值的大小(图3)。图4在同一量纲上比较了第五样带上3个气候因子变化对菊科DNA C-值变化的影响。计算图4中对应于3个气候因子变化形成的DNA C-值差异,发现变化的影响程度也不同(图4),按3条曲线对应数值的极差和标准差,最冷季度平均温度(Ra=0.75, Sd=0.24)对DNA C-值的影响最大,其次是最干季节的雨量(Ra=0.49,Sd=0.15),最湿月份雨量(Ra=0.40,Sd=0.13)的影响相对较小。就气候因素来讲,那些气候条件适中的样块分布的菊科植物往往具有高的DNA 1C-值最高,随着气候条件向两个极端变化,菊科植物DNA 1C-值呈现出有规律的下降趋势,统计上绝大多数达到了极显著的关系。
从以上结果可以发现,在全球尺度上,植物核DNA含量与纬度、气候因素之间存在显著的非线性关系。
3 讨论
有关核DNA含量影响植物的地理分布和生态适应能力已有不少报道。但是关于核DNA含量与纬度、海拔、气候因素间的关系却一直存在着争议,有正相关、负相关或没有相关的不同报道,且绝大多数将这种关系描述成简单的线性关系。先前有关核DNA含量与地理、气候因素之间关系的矛盾性报道,我们推测可能的原因是研究对象没有相对一致的遗传背景,或取样范围较窄有关。除了选样和取材的不足之外,“对核DNA含量与地理气候因素之间”过于简单化地理解为仅是线性关系,进而影响到分析方法的选择,故而得到了相互矛盾的结论,这也可能是原因之一。Knight和Ackerly认为核DNA含量与环境因子间的关系也可能是非线性的[15]。李丹丹等发现,中国境的稗草36个地理种群的核DNA含量与经度、纬度间均存在显著的非线性关系,可以用单峰函数进行拟合。本文以整个菊科具有核DNA含量数据的种类为对象进行统计,在全球范围这样的空间尺度上也证实,植物的核DNA含量随着纬度呈现出有规律的非线性变化[16]。
3个气候因子中,最干季节雨量和最湿月份雨量与DNA C-值呈明显的单峰曲线。随着最冷季节平均温度的增加,DNA C-值似有单调递减的趋势。但是用二项式函数拟合最冷季节平均温度和DNA C-值关系的方程式,判定系数R2为0.638,预测值与实测值在0.001水平上显著相关。但是如果用线性函数进行拟合,判定系数为0.562,拟合效果明显差于非线性方法。因此,最冷季节平均温度对DNA C-值的影响也明显地有非线性特点,曲线呈现出单峰特点。
Levin和Funderburg对335种温带植物和67种热带植物的DNA 4C-值比较发现,温带物种的平均4C-DNA值为27.06pg,而热带的平均仅为12.13pg,两者差异极为显著,但是他们认为热带植物比温带植物核DNA含量小的现象并不普遍[9]。他们认为核DNA含量在热带与温带种类之间的这种差异是由于生物类群发生过程中具有不同核DNA含量大小的类群的地理替代造成,而不是由于适应于环境变化而形成的,也即他们认为核DNA含量的变化并不具有气候适应的意义。但是本文发现3个生物气候因素显著地影响到菊科植物DNA 1C-值,应用单峰函数能够很好地解析生物气候因素与核DNA含量之间的关系。李丹丹等也发现,稗草核DNA含量的种下变化也极明显地受气候因素的影响。因此,核DNA含量这个指标具有明显的生态适应意义[16]。
对10个样带上的菊科植物核DNA含量变化分析发现,位于或接近赤道的样块,其上分布的菊科植物核DNA含量有下降的趋势,这种现象在第2、5样带上比较明显。Bennett也发现热带栽培的豆科和禾草类植物具有较小的基因组[19]。这种现象的原因可能与生态位宽窄有关。Stebbins认为,与温带性种类相比,热带地区的物种往往分布在较窄气候变化范围的环境中,从而要求更少的基因[20]。重复的结构基因使得温带植物具有更宽的生态位,从而加大了核DNA含量。但是在趋向两极的环境中,由于生长季节缩短,或者环境单一,使核DNA含量也有下隆趋势。Vidic等也认为基因组大的植物并不适合于生长在极端环境中[21]。
单峰模型经常用来解析生物分布与环境因子之间的关系[22]。一个从温带到热带的分布的物种往往有最适生长的地理区域和对应气候条件,并有对应的形态性状和核DNA含量。因此,核DNA含量与地理、气候因素之间的关系理论上能够用单峰模型拟合。核DNA含量又是如何影响植物的形态特征的?前人研究表明,核DNA含量小的植物,染色体、细胞核、细胞也均小,细胞分裂速率相对较高。核DNA含量小的植物生活史长更短、种子变大、幼苗生长速率变高[23-24],单位面积叶重增加,代谢速率提高[25]。核 DNA 既可以通过遗传物质的表达,也会通过核 DNA 本身的重量及体积的物理作用影响。显然,细胞大小、体积、重量及发育速率跟非编码的DNA含量变化有关,说明他们在本质上存在因果关系.而且细胞水平上的核型效应是可加成的,可扩展到对多细胞组织结构的大小、重量、发育速率的影响。基因组大小与在细胞水平上的表型(如细胞大小,保卫细胞长度,表皮细胞面积等)存在着较强正相关[26]。因此,核DNA含量的生态适应价值有理论上的基础。
菊科植物是世界性大科,而且是一个相对自然的分类群,具有比较一致的遗传和进化背景,而且本文又是在全球范围的10个样带上进行的数据分析,结果有一定的普遍性。但是作为高度多样化的植物类群,其他类群核DNA含量的地理变化式样,以及与气候因素间的关系,尚需要在更多类群中开展研究,以进一步明确本文发现的菊科植物核DNA含量地理变异规律及其与气候因子之间的关系在整个高等植物中是否具有普遍性。另外,由于数据来源问题,本文主要分析了纬度梯度上菊科植物核DNA含量的变化及其可能的气候因素。对于核DNA含量在全球范围内与经度、海拔因素的关系,也是今后进一步研究的内容。
参考文献(References):
[1] Leitch I J, Bennett M D. Genome downsizing in polyploid plants. Biological Journal of the Linnean Society, 2004, 82(4): 651- 663.
[2] Swift H. The constancy of desoxyribose nucleic acid in plant nuclei. Proceedings of the National Academy of Sciences of the United States of America, 1950, 36(11): 643- 654.
[3] Bennett M D. Plant genome values: how much do we know? Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(5): 2011- 2016.
[4] Chen G Q, Guo S L, Yin L P. Applying DNA C-values to evaluate invasiveness of angiosperms: validity and limitation. Biological Invasions, 2010, 12(5): 1335- 1348.
[5] 郭水良, 陈国奇, 毛俐慧. DNA C-值与被子植物入侵性关系的数据统计分析——以中国境内有分布的539种被子植物为例. 生态学报, 2008, 28(8): 3698- 3705.
[6] 郭水良, 于晶, 李丹丹, 周平, 方其, 印丽萍. 长三角及邻近地区138种草本植物DNA C-值测定及其生物学意义. 生态学报, 2015, 35(19): 6516- 6529.
[7] 李国旗, 安树青, 陈兴龙, 程晓莉, 张纪林. 生物的C值矛盾与其生态适应性的关系初探. 大自然探索, 1999, 18(2): 61- 66.
[8] 苏筱娟. 非编码DNA到底有没有用? 国外科技动态, 2000,(11): 32- 33.
[9] Levin D A, Funderburg S W. Genome size in angiosperms: temperate versus tropical species. The American Naturalist, 1979, 114(6): 784- 795.
[10] Grime J P, Mowforth M A. Variation in genome size-an ecological interpretation. Nature, 1982, 299(5879): 151- 153.
[11] Razafinarivo N J, Rakotomalala J J, Brown S C, Bourge M, Hamon S, de Kochko A, Poncet V, Dubreuil-Tranchant C, Couturon E, Guyot R, Hamon P. Geographical gradients in the genome size variation of wild coffee trees(Coffea) native to Africa and Indian Ocean islands. Tree Genetics & Genomes, 2012, 8(6): 1345- 1358.
[12] Bottini M C J, Greizerstein E J, Aulicino M B, Poggio L. Relationships among Genome Size, Environmental Conditions and Geographical Distribution in Natural Populations of NW Patagonian Species ofBerberisL.(Berberidaceae). Annals of Botany, 2000, 86(3): 565- 573.
[13] Teoh S B, Rees H. Nuclear DNA amounts in populations ofPiceaandPinusspecies. Heredity, 1976, 36(1): 123- 137.
[14] Creber H M C, Davies M S, Francis D, Walker H D. Variation in DNA C value in natural populations ofDactylisglomerataL. New Phytologist, 1994, 128(3): 555- 561.
[15] Knight C A, Ackerly D D. Variation in nuclear DNA content across environmental gradients: a quantile regression analysis. Ecology Letters, 2002, 5(1): 66- 76.
[16] Li D D, Lu Y L, Guo S L, Yin L P, Zhou P, Lou Y X. Nuclear DNA contents ofEchinchloacrus-galliand its Gaussian relationships with environments. Acta Oecologica, 2017, 79: 36- 47.
[17] Rayburn A L, Auger J A. Genome size variation inZeamaysssp.maysadapted to different altitudes. Theoretical and Applied Genetics, 1990, 79(4): 470- 474.
[18] Bennett M D, Leitch I J. Angiosperm DNAC-values database [DB/OL]. http://www.kew.org/cvalues/.2017- 10- 30
[19] Bennett M D. DNA amount, latitude, and crop plant distribution. Environmental and Experimental Botany, 1976, 16(2/3): 93- 108.
[20] Stebbins G L. Chromosomal variation and evolution. Science, 1966, 152(3728): 1463- 1469.
[21] Vidic T, Greilhuber J, Vilhar B, Dermastia M. Selective significance of genome size in a plant community with heavy metal pollution. Ecological Applications, 2009, 19(6): 1515- 1521.
[22] Cui H J, Liu X W, Tan W F, Feng X H, Liu F, Daniel Ruan H. Influence of Mn(III) availability on the phase transformation from layered buserite to tunnel-structured todorokite. Clays and Clay Minerals, 2008, 56(4): 397- 403.
[23] 郭水良, 陈国奇. 根尖分生组织细胞核大小: 一个可能用于植物入侵性评估的新指标. 植物科学学报, 2015, 33(1): 53- 60.
[24] 倪丽萍, 郭水良. 论DNA C-值与植物入侵性的关系. 生态学报, 2005, 25(9): 2372- 2381.
[25] Beaulieu J M, Leitch I J, Knight C A. Genome size evolution in relation to leaf strategy and metabolic rates revisited. Annals of Botany, 2007, 99(3): 495- 505.
[26] Knight C A, Beaulieu J M. Genome size scaling through phenotype space. Annals of Botany, 2008, 101(6): 759- 766.

