基于深度表示的中医病历症状表型命名实体抽取研究*
原 旎,卢克治,袁玉虎,舒梓心,杨 扩,张润顺,李晓东,周雪忠**
(1.北京交通大学计算机与信息技术学院 北京 100044;2.湖北省中医院 武汉 430061;3.中国中医科学院广安门医院 北京 100053)
中医临床记录通常由医生在日常临床诊疗工作中记载,其中包含大量的表型信息、检查记录病人的治疗信息[1]。随着现代化生物医学电子病历的快速发展,应用现代技术实现对临床病历文本中数据的深度挖掘[2],对其进行数据分析,以发现中医诊疗规律是一项非常有价值的工作。但由于中文语言的灵活性和中医临床的个体化表达[3],使得自动从中医电子病历数据中提取有意义的信息和知识具有挑战性。
目前为止,命名实体识别技术在许多领域取得了显著的成果,其F-评测值(F)已经高达90%以上[4],但是在生物医学领域,命名实体识别的研究尚处于初级阶段。因此,对生物医学领域的命名实体识别的相关研究正在引起人们的重视。Wang等[5]比较了条件随机场(Conditional Random Field,CRF),支持向量机(Support Vector Machines,SVM)和最大熵(Maximum Entropy,ME)三种机器学习算法的性能,并应用这三种算法识别中国古代医案中的症状和发病机制,发现CRF更具有优势。Wang等[6]对中医临床文本中的症状名称的识别进行了初步研究。Xu等[7]研究提出一种将分割和命名实体识别相结合的联合模型,以提高对出院数据的命名实体识别的性能。Zhou等[8]在中医方面使用Bootstrapping方法自动识别中医题录文献中的疾病名称和复方名称,取得了很好的效果。Xu等[9]应用CD-REST提取系统从化学诱导疾病的生物医学文献中提取化学关系,该系统使用的是CRFs方法进行的化学和疾病名称的命名实体识别。Zhang等[10]参加2015年BioCreativeV中的化学专利与药物命名实体抽取任务,使用基于词向量(Word embedding)的方法测试了CRF[11]和SSVM[12-13]的性能,其中CRF模型的精确度、召回率、F-测度值达到了(0.860 2、0.884 5、0.872 2),SSVM模型达到了(0.858 8、0.899 9、0.878 9)。袁玉虎等[14]用CRFs模型对中医临床病历中的现病史部分进行症状术语抽取实验,发现CRFs模型可以很好的实现文本中症状术语的序列标注任务。本文将运用深度表示的方法对中医病历文本进行命名实体抽取的实验,并对抽取结果进行比较与评价,实现对临床上现病史数据的自动标识。
1 研究方法
1.1 方法介绍
我们使用CRF和SSVM两种机器学习算法来进行症状词的实体标识。CRF是一个经典的基于概率图模型的序列标识算法。SSVM结合了CRF和SVM的优点[15-18],适用于命名实体抽取的任务[13]。条件随机场和结构支持向量机是目前为止最好的命名实体抽取(NER)机器学习方法。我们首先使用基于规则的方法对现病史句子进行规则化处理,然后基于条件随机场和结构化支持向量机构建机器学习分类器。本文中,使用CRF和SSVM的Web引用作为CRF和SSVM的实现[15]。
条件随机场是自然语言处理领域常用的算法之一,常用于句法分析、命名实体识别、词性标注等。CRF本质上是隐含变量的马尔科夫链和可观测状态的隐含变量的条件概率。以词性变量为例,在词性标注中词性标签就是隐含变量,具体的词语就是可观测状态,词性标注的目的是通过可观测到的各个单词推断出每个单词应该被赋予的词性标签。
SVMHMM是用于序列标签的SSVM的实现,它可以应用于词性标签、命名实体识别、主题查找等[12,19]问题。跟过去的SVM算法相比,SVMHMM算法可以轻松的处理数百万字或数百万标签的标注问题,可以转换和输出任意长度依赖性的更高阶模型,其中包括用于快速近似训练和预测可选的搜索算法。
本文使用词嵌入(Word embedding)方法生成词向量,该词向量作为CRFs和SSVM模型的输入。在自然语言处理中,词嵌入是语言模型集合和特征学习的集合名称,它把字典中的词或者短语映射为实数向量。词嵌入把每个字转化为更低维的数字型连续向量空间,它生成的这种映射方法包括:概率模型[20-22]、词共现矩阵降维[23]、神经网络[24-25]和利用词的上下文信息。利用词嵌入技术作为底层输入表示,可以提高自然语言处理任务的性能。
我们引入了新的基于图的算法学习框架node2vec,使用它来发现节点的连续特征并构建节点向量,以供下游的机器学习方法使用。node2vec最早是在自然语言处理任务中提出的。由于node2vec寻找节点的连续特征的特点,在文本中通过设置上下文窗口的大小,最终形成节点向量。我们将生成的词向量运用于SSVM模型和CRFs模型,将其结果和基于Word embedding生成的词向量的两种模型进行对比分析。在Word embedding方法和node2vec方法的基础上,融合命名实体抽取的模型即CRF模型和SSVM模型形成WENER方法和GENER方法。
1.2 方法框架
本次实验的流程图如图1所示,在本文中引入词嵌入方法:(1)基于规则的数据预处理:由于临床文本本身具有半结构化,口语化的特点,为了使其在进行命名实体抽取时能够直接使用,我们使用Java正则表达式对现病史文本中的数字、时间、单位、标点符号和特殊符号用S、TU、DU或者其组合字母代替,这种方法可以简化句子结构。由于在一些症状词后面常常跟着不同的标点符号或者单位信息,这些信息对于文本的提取是没有用的,因此,在进行统一处理后,由字母S、TU或DU来取代这些无用的信息。(2)分词:实验中使用中科院提供的分词工具(NLPIR)进行分词,在分词中使用的术语列表为湖北中医药大学课题研究组整理出的疾病症状词列表;(3)特征抽取:本实验使用基于skipgram模型的word2vec方法生成的词向量和node2vec生成的词向量,并将这两种词向量特征应用到CRFs模型和SSVM模型上,进行症状词的实体抽取;(4)评价指标:针对不同特征,分析对比CRFs模型和SSVM模型的标注性能。
1.3 评价指标
为了评估文本序列标记策略的可行性和引入的序列分类器在现病史文本中的性能,我们使用了症状名称识别精确度(P)、召回率(R)和F-测评值(F)进行评估。这些评价指标可以评估CRF算法模型和SSVM算法模型序列标记的可行性。
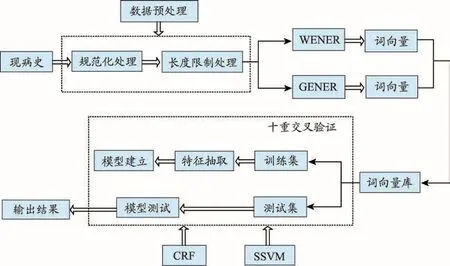
图1 深度表示的命名实体抽取框架流程图
如图2所示,A表示正确识别的症状名称的数量,B表示未正确识别的症状名称,A+B表示从现病史文本中抽取的实体个数,A+C表示标准病历文本中症状实体个数的总数。症状名称正确识别是指该症状词的开始、中间、结尾部分全部被正确标识[26]。
2 实验数据集及相关处理
2.1 数据集
本实验使用的数据是来自于湖北省中医院肝病研究所的2012年到2016年肝病临床电子病历数据,其中包括人口学领域(如年龄、性别)和主要症状诊断数据(如现代疾病诊断,中医症候诊断),我们一共收集整理了10 426条现病史文本数据,并对数据进行规范化处理和长度限制分割,再使用分词工具对现病史句子进行分词。分词以后的现病史句子,在基于word2vec和node2vec两种方法上分别生成词向量。为了避免选取的语料出现最佳与最差偶然性的事件,对生成的词向量用不同的机器学习方法进行十重交叉验证取平均值,选取其中90%的数据量作为训练集,10%的数据量作为测试集。
2.2 数据预处理
2.2.1 数据规范化处理
由于临床病历文本自身具有口语化、专业化、半结构化的特点,因此不能直接对文本进行命名实体抽取。为了让我们能更方便的提取到准确的信息,需要用字符代替一些对文本抽取无用的信息,例如跟时间相关的词语、用药的剂量、标点符号等,这些词会对信息提取造成干扰,因此,我们可以用TU取代时间,S取代标点符号,DU取代剂量单位。通过对现病史的规范化处理,可以大大地提高标注算法的性能。表1所示为进行规范化处理前后的一个现病史文本句子。

图2 评价指标公式
2.2.2 数据长度限制处理
现病史是记述患者病后的全过程,即发生、发展、演变和诊治经过,包含的内容丰富,相对来说,现病史的句子较长。句子过长会导致一个句子中涵盖的信息过多,通过前人的研究了解到,句子过长会对命名实体抽取造成一定的影响。因此本文在进行命名实体抽取时,将现病史句子长度控制在一定的范围之内。本文在对现病史进行规范化处理时采用了以下处理规则:由于前期对文本进行了规范化处理,标点符号用大写英文字母S表示,因此可以用S来分割句子,将长的文本分割成若干个短的字符串。因为字符串的长度大部分都在50字以上,我们就以50为单位来进行分割。如果一个进行过规范化处理的文本句子长度本身就在50个字以内,则不做任何处理。如果文本长度超过了50个字,那么我们就用50来分割。若最后剩下的字符串小于20,则将其和上一个字符串合并为一个字符串。表2是分割现病史长度的具体例子。

表1 现病史数据的规范化处理

表2 现病史文本的长度限制处理

表3 现病史分词结果
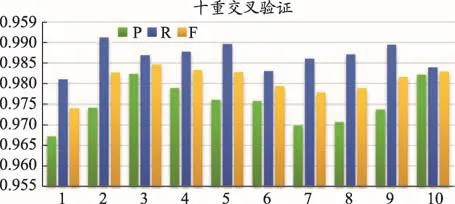
图3 WENER方法的CRF模型标注性能
2.2.3 数据分词处理
基于深度学习的方法是用word2vec和node2vec的方法生成词向量,用生成的词向量表示现病史文本内容,因此我们首先需要对现病史句子进行分词处理,其分词结果如表3所示。
3 基于WENER方法的症状表型命名实体抽取方法研究
对于现病史数据的算法流程描述如下:首先要对现病史数据进行数据预处理,将现病史句子进行句子长度限制处理,将每个现病史句子分割成若干个长度在50个字左右的字符串,并对长度处理以后的句子进行分词,将分词后的结果作为WENER方法中词嵌入方法的输入文件,使用word2vec词嵌入方法生成200维的词向量,将生成的词向量作为CRF模型和SSVM模型的输入文件,测试比较两种模型的算法性能。
如图3所示为WENER方法的CRF模型在十重交叉验证的评价结果,其性能的平均值分别为:精确度(0.975 0)、召回率(0.984 9)、F值(0.979 8),比传统的CRF模型的命名实体抽取的性能提高8.61%,召回率提高19.8%,其F值提高了14.5%。
如图4所示,是基于word2vec的方法生成词向量后,使用SSVM模型在现病史数据文本上十重交叉验证方法的评价结果。其十重交叉验证的平均值分别为:精确率(0.992 8)、召回率(0.988 9)、F值(0.990 8)。这比传统的基于CRF算法的命名实体抽取方法的精确度高出10.39%,召回率高出20.2%,F值高出15.6%。
从以上的结论可以看出,深度表示的命名实体抽取方法比传统的命名实体抽取的算法性能好,其原因分析如下:由于生物医学上命名的主观性,症状名称的命名规则不统一[4,27],并且临床病历的文本数据是由医生手工书写的中文字符,具有灵活多样性,导致其利用较为困难[28],使得传统的CRF模型的标注性能要略差于WENER下的命名实体抽取算法。另外,词嵌入方法能够随意选择特征的维数,并且不需要人为干预,与传统的方法相比,能更好的利用词的上下文特征。但是WENER方法依赖于分词后的结果,而领域知识会影响到分词系统的分词效果,从而影响到命名实体识别系统的性能。
从表4中可以看到,虽然WENER方法中的SSVM模型和CRF模型的均值和标准差相差不大,但是SSVM模型的F值比CRF模型的F值有所提高。SSVM模型的性能比CRF的性能好是因为CRF模型是一种有代表性的序列标注算法,它是一种有辨识度的无向概率图判别模型[28],而SSVM模型是基于结构数据的最大化决策边界模型[12],如序列、二分图和树。SSVM模型结合了CRF算法和SVM算法的优点,适用于序列标注问题[15]。因此,SSVM模型的F值通常要比CRF模型的F值高[15-18,29]。
4 基于GENER方法的症状表型命名实体抽取方法研究
GENER方法是基于网络嵌入方法node2vec生成的词向量,其算法流程如下:首先构建node2vec的网络结构图,通过随机游走的方式生成200维的词向量,然后将生成的词向量作为CRF和SSVM模型的输入文件,最后计算CRF和SSVM两种模型的评价指标。其训练集和测试集同样使用肝病现病史文本数据,并对肝病现病史数据进行长度限制处理和分词处理。在分词过程中,我们根据中文粒度的不同,将分别构建基于字和基于词特征的网络,并测试不同网络之间GENER方法的标注性能。
(1)基于词特征的GENER方法的实验结果分析
从图5所示可知,GENER方法的CRF模型的性能评价结果的平均值分别为精确度(0.950 1)、召回率(0.950 8)、F值(0.950 4)。相比传统的CRF模型的性能精确度、召回率、F值分别提高了6.12%、16.39%、11.56%。
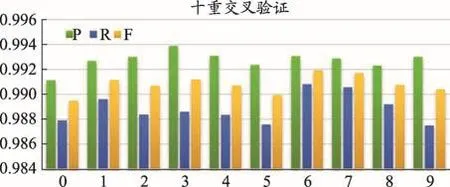
图4 WENER方法的SSVM模型标注性能
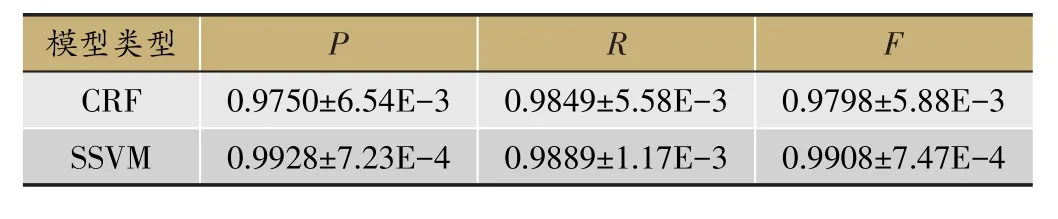
表4 WENER方法下的CRF方法和SSVM方法的性能对比

图5 GENER方法CRF模型基于词特征的标注性能
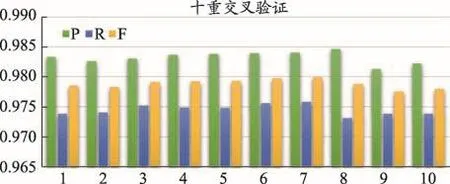
图6 GENER方法的SSVM模型基于词特征的标注性能

表5 GENER方法下基于词特征的CRF模型和SSVM模型的性能对比
从图6中可以看出,GENER方法在SSVM模型上的标注性能结果的平均值为:精确度(0.983 3)、召回率(0.974 5)、F值(0.978 8)。结果比传统的基于CRF的命名实体抽取算法的精确度高出9.44%,召回率高出18.76%,F值高出14.4%。
GENER方法在基于词特征下的CRF模型和SSVM模型的性能的对比结果分析如表5所示。
从表5可以看出,在GENER方法下的基于词特征的CRF模型和SSVM模型的评价指标的对比,发现SSVM模型的F值比CRF模型的F值高0.36%,说明在基于网络嵌入方法提取的特征上,虽然CRF和SSVM的F值相差不大,但是SSVM模型的性能还是要优于CRF模型的性能。
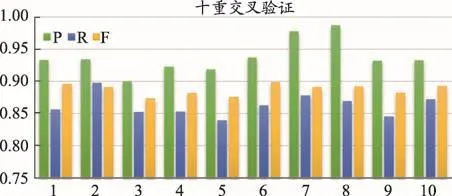
图7 GENER方法的CRF模型基于字特征的标注性能

图8 GENER方法的SSVM模型基于字特征的标注性能
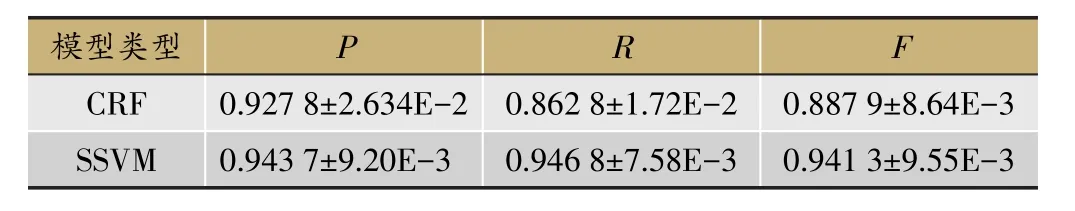
表6 GENER方法下基于字特征的CRF模型和SSVM模型的性能对比
(2)基于字特征的GENER方法的实验结果及分析
在基于字特征的GENER方法下的CRF模型和SSVM模型的评价结果如图7和图8所示。
从表6可以看出,SSVM模型的精确度要比CRF模型的精确度高1.59%,召回率高8.4%,F值高5.34%。
无论是基于字还是基于词的GENER方法,SSVM模型的精确率、召回率和F值与CRF模型相差不大,但是SSVM的性能指标要略高于CRF模型的性能。再次验证了对于SSVM模型和CRF模型在不同领域的比较,SSVM模型的性能通常优于CRF模型的性能。
5 讨论
在本文的研究中,我们提出了基于深度表示的中医病历中症状表示的命名实体抽取方法,CRF和SSVM两种算法模型,实验结果表示,深度表示的命名实体抽取性能要明显高于传统的命名实体抽取算法CRF模型的性能。传统的非深度表示的命名实体抽取算法(CRF算法)的F值为0.834 8,而深度表示的算法的F值分别为(0.979 8,0.990 8,0.975 2,0.887 9,0.978 8,0.941 3),因此,其性能比传统的非深度表示的命名实体抽取的算法性能好;而对于同一种词向量特征下,SSVM模型的F值要比CRF模型的F值高。例如,在WENER方法下,SSVM的F值是0.990 8,而CRF的F值为0.979 8,SSVM的性能要高于CRF模型一个百分点。通过对不同的词向量特征的比较发现,基于word2vec生成的词向量下的CRF和SSVM模型的性能要高于基于node2vec生成的词向量下的CRF和SSVM模型的性能。另外,SSVM模型与CRF模型在基于词的网络嵌入方法特征上的性能比基于字的网络嵌入方法特征上的性能好,基于词的网络嵌入只有两种判别类型,即是否为症状表型术语。而基于字的网络判别类型三种:“B-S”、“E-S”、“O”。基于字的判别类型增加需要做出更多的判断,可能造成性能的下降。而且,基于字的特征使得特征粒度减小,特征信息降低,而基于词的特征粒度比基于字的粒度大,特征信息较强。
1 Jensen P B,Jensen L J,Brunak S.Mining electronic health records:towards better research applications and clinical care.Nat Rev Gen,2012,13(6):395.
2 刘凯,周雪忠,于剑,等.基于条件随机场的中医临床病历命名实体抽取.计算机工程,2014(9):312-316.
3 Feng L,Zhou X,Qi H,et al.Development of large-scale TCM corpus using hybrid named entity recognition methods for clinical phenotype detection:An initial studyComputational Intelligence in Big Data,2015.
4 王世昆,李绍滋,陈彤生.基于条件随机场的中医命名实体识别.厦门大学学报(自然版),2009,48(3):359-364.
5 Wang S K,Shao-Zi L I,Chen T S.Recognition of Chinese Medicine Named Entity Based on Condition Random Field.J Xiam Univ,2009,48(3):359-364.
6 Wang Y,Liu Y,Yu Z,et al.A preliminary work on symptom name recognition from free-text clinical records of traditional chinese medicine using conditional random fields and reasonable features.The Workshop on Biomedical Natural Language Processing,2012.
7 Xu Y,Wang Y,Liu T,et al.Joint segmentation and named entity recognition using dual decomposition in Chinese discharge summaries.J Am Med Inform Ass,2014,21:e84.
8 Zhou X,Wu L Z,Yi F.Integrative mining of traditional Chinese medicine literature and MEDLINE for functional gene networks.Artif Intell Med,2007,41(2):87-104.
9 Xu J,Wu Y,Zhang Y,et al.CD-REST:a system for extracting chemical-induced disease relation in literature.Database the Journal of Biological Databases&Curation,2016,baw036.
10 Zhang Y,Xu J,Hui C,et al.Chemical named entity recognition in patents by domain knowledge and unsupervised feature learning.Database the Journal of Biological Databases&Curation,2016:baw049.
11 Andrew G,Gao J.Scalable training of L1-regularized log-linear models,2007.
12 Tsochantaridis I,Joachims T,Hofmann T,et al.Large Margin Methods for Structured and Interdependent Output Variables.J Mach Learn Res,2005,6(2):1453-1484.
13 Bakir G,Hofmann T,Schölkopf B,et al.Support Vector Machine Learning for Interdependent and Structured Output SpacesInternational Conference on Machine Learning.ACM,2004.
14 袁玉虎,周雪忠,张润顺,等.面向中医临床现病史文本的命名实体抽取方法研究.世界科学技术:中医药现代化,2017,19(1):70-77.
15 Tang B,Feng Y,Wang X,et al.A comparison of conditional random fields and structured support vector machines for chemical entity recognition in biomedical literature.J Cheminformatics,2015,7(S1):1-6.
16 Zhang Y,Wang J,Tang B,et al.UTH_CCB:A report for SemEval 2014-Task 7 Analysis of Clinical Text.International Workshop on Semantic Evaluation,2014.
17 Tang B,Wu Y,Jiang M,et al.A hybrid system for temporal information extraction from clinical text.J Am Med Inform Ass Jamia,2013,20(5):828-835.
18 Balamurugan P,Shevade S,Sundararajan S,et al.An Empirical Evaluation of Sequence-Tagging Trainers.Computer Science,2013.
19 Mikolov T,Sutskever I,Chen K,et al.Distributed Representations of Words and Phrases and their Compositionality.Adv Neur Inform Process Sys,2013,26:3111-3119.
20 Lebret R,Collobert R.Word Emdeddings through Hellinger PCA.Computer Science,2014.
21 Levy O,Goldberg Y.Neural word embedding as implicit matrix factorization.Adv Neur Inform Process Sys,2014,3:2177-2185.
22 Li Y,Xu L,Tian F,et al.Word embedding revisited:a new representation learning and explicit matrix factorization perspectiveInternational Conference on Artificial Intelligence,2015.
23 Globerson A,Chechik G,Pereira F,et al.Euclidean Embedding of Cooccurrence Data.J Mach Learn Res,2007,8(4):2265-2295.
24 Levy O,Goldberg Y.Linguistic Regularities in Sparse and Explicit Word Representations.Eighteenth Conference on Computational Natural Language Learning,2014.
25 AlexRudnicky.Can Artificial Neural Networks Learn Language Models?.Proc Int Conf Spok Lang Proc.2000:202-205.
26 Wang Y,Yu Z,Chen L,et al.Supervised methods for symptom name recognition in free-text clinical records of traditional Chinese medicine:an empirical study.J Biomed Inform,2013,47(2):91-104.
27 胡俊锋,陈蓉,陈源,等.一种松耦合的生物医学命名实体识别算法.计算机应用,2007,27(11):2866-2869.
28 Lafferty J,Mccallum A,Pereira F.Conditional Random Fields:Probabilistic Models for Segmenting and Labeling Sequence Data.Eight Int Conf Mach Learn,2002,53(2):282-289.
29 Lei J,Tang B,Lu X,et al.A comprehensive study of named entity recognition in Chinese clinical text.J Am Med Inform Ass,2014,21(5):808-814.

