谷子PT2基因保守序列的克隆与分析
李 燕,杨致荣,高建华
(1.山西农业大学文理学院,山西 太谷 030801;2.山西农业大学生命科学学院,山西 太谷 030801)
谷子(Setaria italica)又称粟,是起源于我国的一种古老作物,也是我国过去几千年来的主栽作物和中华民族的哺育作物。近年来,随着人们生活水平的不断提高及食品多样化和营养化,小米及其制品备受青睐。提高谷子产量和品质也成为目前研究的热点。
对于水稻(Oryza sativa)等粮食作物而言,覆盖地膜[1],喷施赤霉素[2],不同氮肥组合和施肥方式[3],提高籽粒大小、质量,防止成熟后落粒,均是促进产量的有效手段。目前,已经克隆出了多个籽粒性状相关基因如 GL7[4],GW8[5],GW7[6]和 GW2[7]等;落粒相关基因,如 qSH1[8],SHAT1[9],SH4[10],PT2[11]和 SH5[12]等。其中,水稻PT2(Panicle Traits 2)基因编码蛋白,即生长调节因子Os GRF4(Growth Regulating Factor 4,Os02g47280)是拟南芥At GRF的同源蛋白。研究表明,Os GRF4能够调节细胞分裂素脱氢酶前体基因(CKX5和CKX1),导致细胞分裂素含量提高,进而调节籽粒大小、形状等。当Os GRF4表达受到抑制后,离层发育良好,从而增强了落粒性;反之,若Os GRF4高效表达,离层发育不完全,落粒性减弱[13]。PT2基因的表达受到miRNA的调控,第3个外显子中有一段miR396结合序列。当OsmiR396结合该位点时,PT2基因的表达被抑制;该结合位点的突变,导致OsmiR396无法结合,从而使PT2基因的表达量升高[14-15]。另外,作为一种调节因子,Os GRF4蛋白具有与核酸和其他蛋白质(比如,GRF-Interacting Factors,GIF)互作的结构域——WRC和QLQ。其中,WRC帮助Os GRF4蛋白与核酸互作,QLQ协助Os GRF4与其他蛋白互作[14-15]。另外,水稻Os04g51190基因编码序列与Os GRF4氨基酸序列同源性约70%,但目前未发现其与籽粒性状或落粒相关。
在豫谷1号的基因组数据(Setaria italica v2.2)中,有2个PT2基因的同源基因(Seita.1G287100.1和Seita.7G224500.1)。初步分析发现,这2个基因均含有相应的miR396结合序列,且其编码多肽也具有WRC和QLQ结构域。狗尾草(Setaria viridis)是谷子的近缘野生种,但二者在籽粒大小和落粒性等方面存在显著差异。
为了研究PT2基因与谷子籽粒性状及其落粒性的相关性,本研究首先分析了几种植物PT2基因的亲缘关系;预测了豫谷1号PT2基因启动子序列中的相关元件,并验证了PT2基因在豫谷1号不同部位的表达水平;筛选并比较了43种谷子和3种狗尾草中该基因的保守区域序列,旨在为后期筛选谷子落粒相关基因奠定了基础。
1 材料和方法
1.1 材料
本研究所用到的谷子和狗尾草材料列于表1。所有材料均种植于山西农业大学试验田。
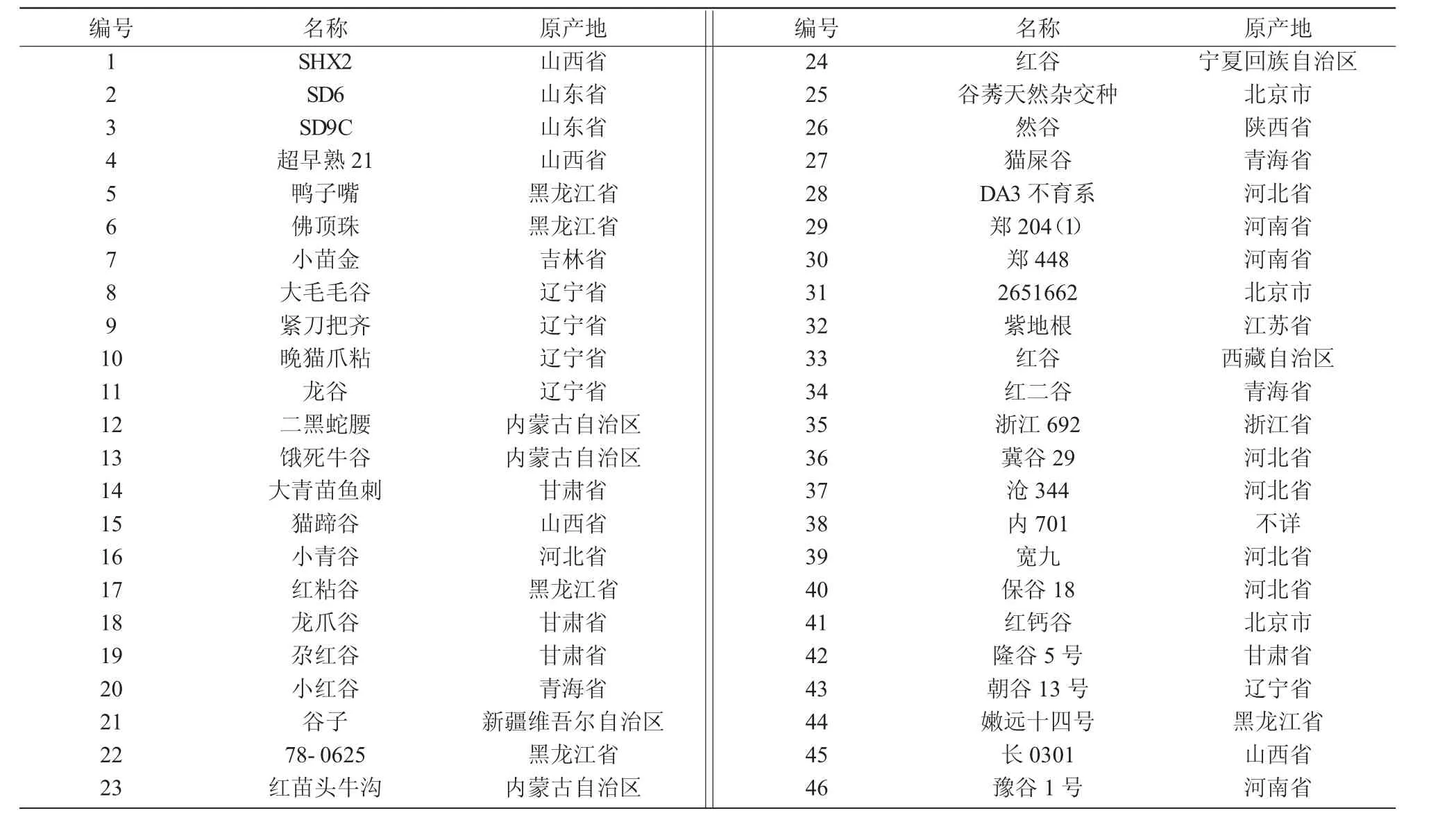
表1 研究所用到的谷子和狗尾草种质资源
1.2 方法
1.2.1 PT2基因及其启动子的生物信息学分析从 phytozome网站(https://phytozome.jgi.doe.gov/)下载水稻PT2基因序列(LOC_Os02g47280),并下载拟南芥、水稻、谷子、狗尾草、高粱和玉米中与之同源性最高的基因序列。对这些基因编码的氨基酸序列进行同源性分析,并利用MEGA 7.0分析其亲缘关系。下载Seita.1G287100基因起始密码子上游1 500 bp的序列,在 PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行启动子在线预测。然后,对谷子和狗尾草PT2基因的启动子进行序列比对。
1.2.2 总RNA提取与PT2基因的检测 使用TaKaRa总RNA提取试剂盒(MiniBEST Plant RNA Extraction Kit,货号 9769S),按照说明书提取豫谷1号苗和穗的总RNA。然后使用PrimeScript RT reagent Kit with gDNA Eraser试剂盒(TaKaRa,货号RR047A)合成 cDNA。使用引物 579F(5′-CAGCGG CTTCCAGAACCACT)和 1179R(5′-GTTCGGCGAC TGAGAGCTTGCCA)检测Seita.1G287100.1基因的表达。
1.2.3 植物基因组DNA的提取 谷子和狗尾草基因组DNA提取使用CTAB法:CTAB提取液(200 mmol/L Tris-Cl,250 mmol/L NaCl,25 mmol/L EDTA和0.5%SDS,pH值7.5)65℃预热;在1.5 mL离心管中加入50mg幼嫩叶片,并加入500μLCTAB提取液,用高通量DNA研磨机(宁波新芝生物科技股份有限公司,型号SCIENTZ-48)充分研磨;放入65℃水浴温浴1 h,期间每隔5 min颠倒混匀;加入500 μL氯仿 /异戊醇(体积比 24∶1),振荡混匀,室温静置10 min;12 000 r/min离心10 min,取上清至新离心管;加入等体积氯仿/异戊醇(体积比24∶1),混匀后12 000 r/min离心10 min;取上清至新离心管,并加入0.7倍体积预冷的异丙醇,混匀后放入-20℃1 h;取出后12 000 r/min离心10 min,弃上清,并用70%乙醇清洗沉淀2次;室温晾干,加入30 μL ddH2O溶解DNA,最后-20℃保存备用。
1.2.4 PT2基因外显子的扩增及序列分析 根据数据库中Seita.1G287100基因序列,设计2对特异性引物,用于分别扩增不同谷子或狗尾草中PT2基因的前3个外显子序列。引物PT21-E12-F(5′-ACAAAGCGGGCAATAAAGGC)和PT21-E12-R(5′-ACACGGCAAGTATTCGGGAG)用于扩增外显子1和 2。引物 PT21-E3-F(5′-GTAGCTGCTCCAC TGTTCGC)和 PT21-E3-R(5′-AGTTATCTGCGTGA CCATCTCTG)用于扩增外显子3。PCR体系为:Takara Ex Taq premix (RR902A)10 μL,DNA 模板0.5μL,上下游引物各 0.5 μL,ddH2O补齐至 20 μL。PCR反应条件为:94℃预变性3 min;94℃,30 s,60 ℃,30 s,72℃,60 s,共 35 个循环;最后 72 ℃延伸5 min。PCR产物使用1%的琼脂糖凝胶电泳检测,并克隆测序。使用MEGA 7.0对不同谷子和狗尾草的PT2基因序列进行同源性和单核苷酸多态性分析。
2 结果与分析
2.1 谷子PT2候选基因的筛选与分析

将数据库中谷子、狗尾草以及其他植物的PT2候选基因编码的蛋白质进行比对,并构建系统发育树。分析发现,已经报道的水稻Os GRF4蛋白(Os02g47280基因编码)[10-12]与谷子 Seita.1G287100、狗尾草Sevir.1G292200编码的蛋白质序列同源性较高(图1-A)。其中,谷子Seita.1G287100和狗尾草Sevir.1G292200编码的蛋白序列完全一致。该结果说明这2个基因分别是谷子和狗尾草的PT2基因。进一步分析3种植物的PT2蛋白质序列发现,QLQ结构域序列完全一致,而WRC结构域也仅存在一个氨基酸差异(图1-B)。该结果说明该生长调节因子中参与核酸或蛋白互作的2个重要位点高度保守,保障其正常执行功能。
2.2 谷子PT2基因启动子元件分析
将豫谷1号的PT2基因(Seita.1G287100)上游1 500 bp的序列进行在线分析,结果表明,该基因启动子较为复杂。除了常见的TATA-box,CAAT-box等核心启动子元件外,还有较多的响应元件,比如,光 应 答 响 应 元 件 ACE,AE-box,BOXI,G-BOX,G-box,GT1-motif,I-box,L-box,sp1,TCT-motif;脱落酸信号转导下游响应元件ABRE和motif-Iib;茉莉酸甲酯 (MeJA) 响应元件 CGTCA-motif和TGACG-motif;赤霉素响应元件 GARE-motif;缺氧特异性诱导响应元件GC-motif;参与防御和应激反应响应元件TC-rich-repeats等(表2)。另外,还有多种其他调控位点,比如调控转录水平的5′UTR Py-rich stretch,MYBHv1 结合位点 CCAAT-box,胚乳表达所需skn-1 motif元件,玉米醇溶蛋白代谢调控响应元件O2-site,参与分生组织表达响应元件CAT-box等。该预测结果说明PT2基因参与多种环境响应及代谢调节,是重要的调节因子。另外,对谷子和狗尾草PT2基因的启动子序列进行对比发现,二者只有8个核苷酸位点有差异(图2)。其中,只有2个位点分别位于2个G-box元件中。该结果暗示在2种植物中,PT2基因的调控基本一致。
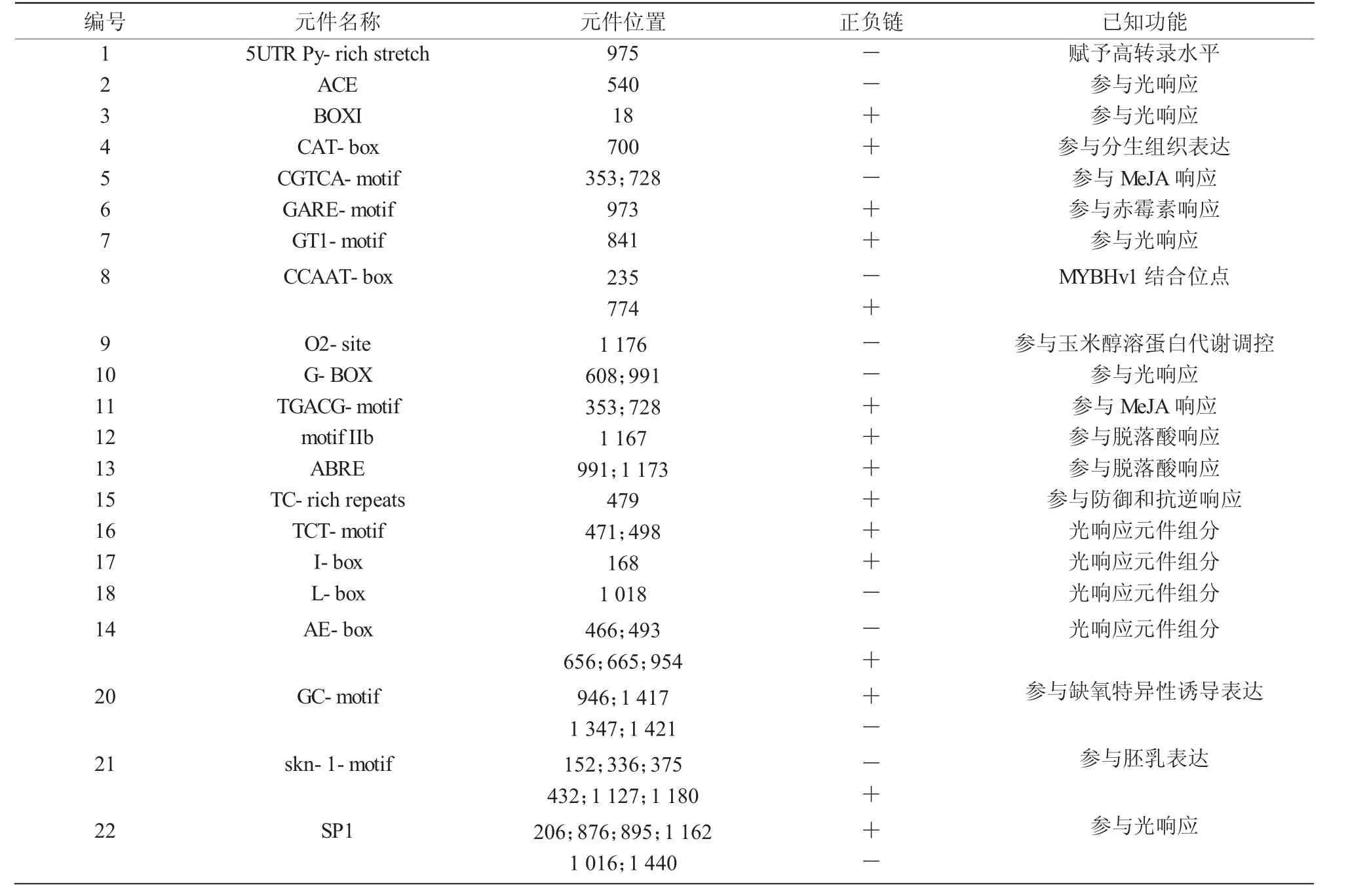
表2 PT2启动子元件分析
2.3 谷子PT2家族基因表达部位分析
提取豫谷1号苗和穗的总RNA,并将其反转录为cDNA。以cDNA为模板,使用引物579F和1179R,检测该基因在不同部位的转录情况,结果表明,该基因在豫谷1号苗、穗部位均有表达(图3)。说明PT2基因可能参与多种代谢调节和环境响应。
2.4 PT2基因保守性分析
为分析PT2基因在不同品系中的保守性,将43种谷子和3种狗尾草的PT2基因前3个外显子(包括OsmiR396结合位点、WRC和QLQ的编码区域)进行了序列测定。
序列比对发现,其核酸序列高度保守,共发现4处SNP(图4),但是只有一处导致氨基酸突变(T190A)。该突变位点(T190A)在谷子和狗尾草中均被检测到,说明其与谷子和狗尾草之间籽粒性状、落粒性的差异无关。
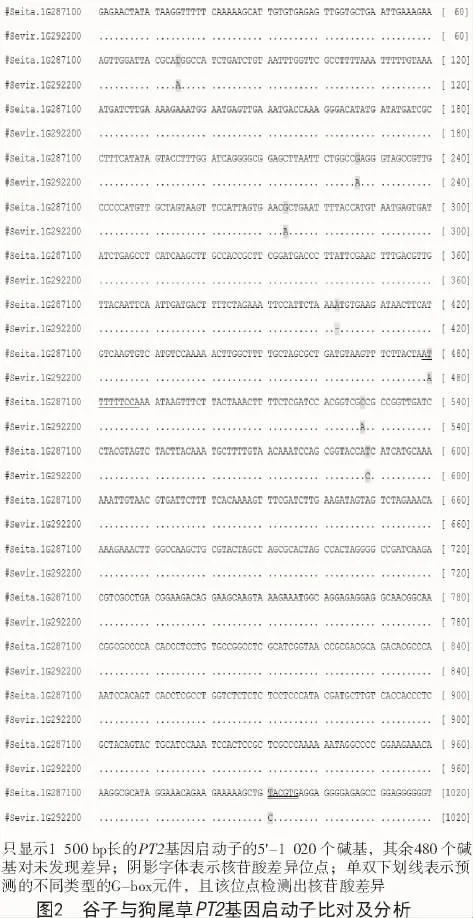


3 讨论
对于水稻、小麦、谷子等禾本科作物而言,其籽粒大小、形状、数量以及落粒等性状是决定其产量高低的重要因素。研究表明,水稻PT2基因编码的Os GRF4蛋白能够调控籽粒形状、大小以及落粒性等多种性状。为研究该基因在谷子籽粒性状调控中的作用,本试验通过生物信息学的方法,确定了谷子和狗尾草中的PT2候选基因。通过对谷子PT2基因启动子中各种相应元件的分析发现,该基因的启动子含有多种环境(如光照、干旱)和激素(如水杨酸、茉莉酸)等响应元件,暗示其参与多种代谢调节。RNA转录检测结果也显示,其在植株不同部位均有表达,进一步确定该基因具有多重功能。但是,本研究并未发现该基因其他形式的转录本。
值得注意的是,谷子与狗尾草的PT2基因编码蛋白序列完全一致,仅在个别品系中发现一个氨基酸的点突变。该点突变在谷子和狗尾草中均存在。该基因启动子序列也基本吻合,仅存在几个核苷酸的差异。其中,2个差异分别位于2个G-box元件中。这2个差异是否影响该基因对某些因素的响应,尚未确定。然而,谷子和狗尾草籽粒性状差异显著,比如籽粒形状明显不同,落粒性完全相反。说明在2种植物体内,其他未知的基因调控了上述差异,其与水稻不同。
本研究通过生物信息学的方法确定了谷子和狗尾草的PT2基因,并且通过二者间的比对,初步判断PT2基因与谷子籽粒大小、形状以及落粒性等性状无关,结果为后续筛选相关基因奠定了坚实的基础。
[1]世佳,任晓利,魏志敏.覆膜对夏播谷子生长发育与产量的影响机制及其相关性分析[J].华北农学报,2016,31(Z):25-30.
[2]郑林林,原向阳,邵冬红.灌浆期喷施赤霉素对谷子农艺性状及产量的影[J].山西农业科学,2014,42(5):455-457.
[3]代小冬,常世豪,杨育峰.不同氮肥组合和施肥方式对谷子生长和产量的影响[J].河南农业科学,2014,43(11):13-16.
[4]WANG Y,XIONG G,HU J,et al.Copy number variation at the GL7 locus contributes to grain size diversity in rice[J].Nature Genetics,2015,47(8):944-948.
[5] WANG S,WU K,YUAN Q,et al.Control of grain size,shape and quality by OsSPL16 in rice[J].Nature Genetics,2012,44(8):950-954.
[6]WANG S,LI S,LIU Q,et al.The OsSPL16-GW7 regulatory module determines grain shape and simultaneously improves rice yield and grain quality[J].Nature Genetics,2015,47(8):949-954.
[7]BEDNAREK J,BOULAFLOUS A,GIROUSSE C,et al.Down-regulation of the TaGW2 gene by RNA interference results in decreased grain size and weight in wheat[J].Journal of Experimental Botany,2012,63(16):5945-5955.
[8] MAGWA R A,ZHAO H,YAO W,et al.Genomewide association analysis for awn length linked to the seed shattering gene qSH1 in rice[J].Journal of Genetics,2016,95(3):639-646.
[9]ZHOUY,LUD,LI C,et al.Genetic control of seed shatteringin rice by the APETALA2 transcription factor shattering abortion1[J].The Plant Cell,2012,24(3):1034-1048.
[10] INOUE C,HTUN TM,INOUE K,et al.Inhibition of abscission layer formation by an interaction of two seed-shattering loci,sh4 and qSH3,in rice[J].Genes&Genetic Systems,2015,90(1):1-9.
[11] HU J,WANG Y,FANG Y,et al.A rare allele of GS2 enhances grain size and grain yield in rice[J].Molecular Plant,2015,8(10):1455-1465.
[12]YOON J,CHO LH,KIM SL,et al.The BEL1-type homeobox gene SH5 induces seed shattering by enhancing abscission-zone development and inhibiting lignin biosynthesis[J].The Plant Journal,2014,79(5):717-728.
[13] SUN P,ZHANG W,WANG Y,et al.OsGRF4 controls grain shape,panicle length and seed shattering in rice[J].Journal of Integrative Plant Biology,2016,58(10):836-847.
[14] LI S,GAO F,XIE K,et al.The OsmiR396c-OsGRF4-OsGIF1 regulatory module determines grain size and yield in rice[J].Plant Biotechnology Journal,2016,14(11):2134-2146.
[15] DUAN P,NI S,WANG J,et al.Regulation of OsGRF4 by OsmiR396 controls grain size and yield in rice[J].Nature Plant,2015,2(1):15203.

