杭州湾入海口沉积物原核生物多样性分析
李双彤,张秋芳,周阳靖,唐方园,程军蕊,郑琦宏,徐继荣
(1.宁波大学 建筑工程与环境学院,宁波 315211; 2. 泉州师范学院 海洋与食品学院,泉州 362000; 3.福建省海洋藻类活性物质制备与功能开发重点实验室,泉州 362000)
位于河流与海水交汇处的河口生态系统,是陆源污染物重要排放口,极易因人类活动导致富营养化而引发赤潮等环境问题[1-2]。杭州湾地处钱塘江入海口,同时也是上海、杭州、宁波三大都市几何中心,但近年来由于周边城市工业和水产养殖业迅速发展,大量营养盐输入后会随悬浮泥沙沉积在沿岸潮滩沉积物中,形成了各种营养盐的蓄积库[3]。与国内典型河口和沿岸潮滩相比,该地区总氮和总磷已处于较高水平,属于严重富营养化状态[4-5],且东海水域潮汐、泥沙和盐度以及人类活动等复杂多变的因素[6]都导致该区域生态系统变得更为脆弱。因此,杭州湾入海口生态环境正面临着巨大挑战。
微生物是生态系统重要组成部分,对维持河口生物地球化学循环和生态系统平衡起着重要作用,其多样性可以作为评估河口生态系统重要生物学指标[7]。然而,作为杭州入海口生态系统重要组成部分的微生物,其多样性及与环境的相关关系等研究迄今为止还很匮乏。与传统方法(DGGE和T-RFLP等)相比,高通量测序(HTS)技术可用于分析丰度较低和复杂的微生物群落[8],该方法具快速、灵敏等优点,已经广泛应用于环境微生物多样性分析中。
因此,为探明杭州湾入海口生态系统原核生物多样性及其与环境因子相关关系,本研究拟通过新一代测序平台Illumina,分析河口沉积物总DNA中16S rRNA基因序列,并将其多样性与环境因子进行相关性分析,了解近海水域微生物分布特征与碳、氮、磷和重金属等环境因子互作关系,为杭州湾入海口环境质量监控提供参考和科学依据。
1 材料与方法
1.1 样品采集
于2013年7月20—23日,应用GPS定位系统采集了杭州湾入海口南北两岸和内海3个区域共13个点位(详见图1和表1)沉积物样品。其中,1)杭州湾北岸:金山工业区排污口上游(HZW1)和上游(HZW2)、海盐县(HZW3)、洋山港码头(HZW10);2)杭州湾南岸:上虞工业区排污口上游距排污口约1 km处(HZW4)、排污口(HZW5)和排污口下游距排污口2 km处(HZW6),杭州湾湿地保护区内距中国林科院亚热带林业研究所北部方向200 m处(HZW7)、新浦镇海滩距防洪堤1.5 km(HZW8)和镇海舟山桥头滩涂(HZW9);3)距南北两岸较远处的内海3个点位:HZW11、HZW12和HZW13。
在每个采样点用掘式采样器采集距沉积物表面0~10 cm 处的沉积物,混匀后,分成两部分,一部分装入无菌采样袋密封后置液氮罐冷冻保存,然后迅速运回实验室冻干后,过1 mm筛,于-80℃下保存,用于提取总DNA;另一部分自然风干后用于理化指标分析。
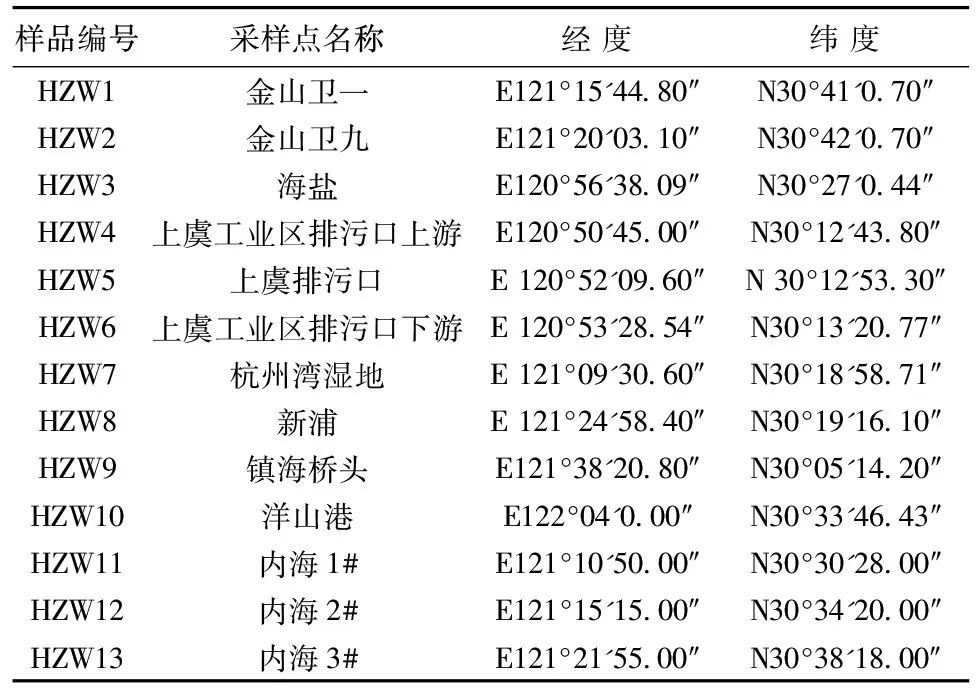
表1 采样点Table 1 Sampling sites
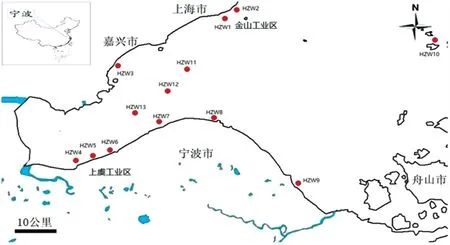
图1杭州湾入海口沉积物采样点
Fig 1 Sampling sites in Hangzhou Bay
1.2 理化性状分析
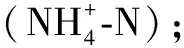
1.3 总DNA提取
称取冻干后沉积物样品0.50 g,采用FastDNA Spin Kit For Soil(Qbiogene公司,美国)试剂盒,应用核酸提取仪(Qbiogene公司,美国)提取,均质条件为40 s和6.0 m/s,提取步骤参照说明书进行。将获得的总DNA用70 μL DES溶液收集,用Nonadrop-1000超微量紫外分光光度计进行含量和纯度测定后,储存于-80℃下备用。
1.4 PCR扩增及高通量测序
用引物338F:5′-ACTCCTACGGGAGGCAGCAG-3′和806R:5′-GGACTACHVGGGTWTCTAAT -3′[11]对提取到的DNA进行 PCR扩增。扩增条件:95℃预变性3 min; 95℃变性30 s,55℃退火30 s,72℃延伸40 s,30个循环;72℃再延伸1 min。PCR仪为ABI Geneamp® 9700型。在Illumina Miseq平台上测序。
1.5 数据分析
采用Flash[12]软件将除引物和barcode后的PE-reads拼接成一条序列,再通过QIIME v.1.7.0(Quantitative insights into microbial ecology, )软件将对其进行过滤和分析[13]。然后用Uclust软件[14]对序列进行聚类,将相似性大于97%的序列归为同一OTU(Operational Taxonomic Unit)。把每个OTU序列提取出来并用RDP(Ribosomal Database Project)Classifier软件[15]和SILVA数据库[16]对序列的分类信息进行注释。利用Mothur v1.30.1软件对相似水平高于97%以上OTUs进行α-多样性分析,包括了评估物种总数(Chao1)、每个样本OTUs数量(Observed species)和物种多样性(Shannon-Wiener)3个指数。应用SPSS19.0软件计算出相对丰度>1%优势菌群与环境因子的相关性(Pearson系数),运用R软件绘制相关性热图(Heatmap),直观地表征原核生物多样性与环境因子相关关系。
高通量测序所获得的数据已收录于NCBI的Sequences Read Archive(SRA),登录号为SRP096130。
2 结果
2.1 沉积物理化指标
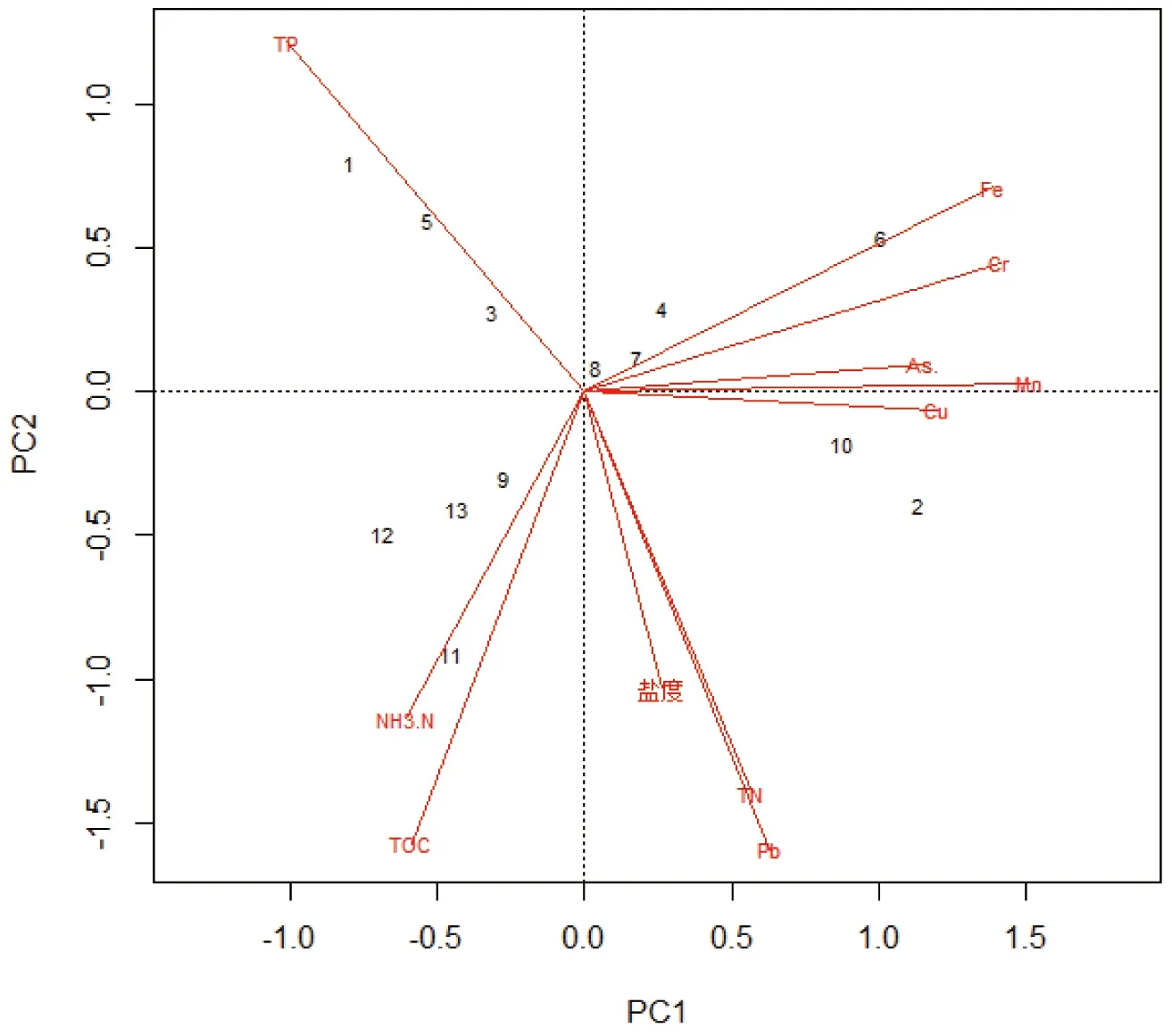
图2 采样点与环境理化因子的主成分分析图Fig 2 PCA ordination graph for the sampling site of patterns and environmental factors
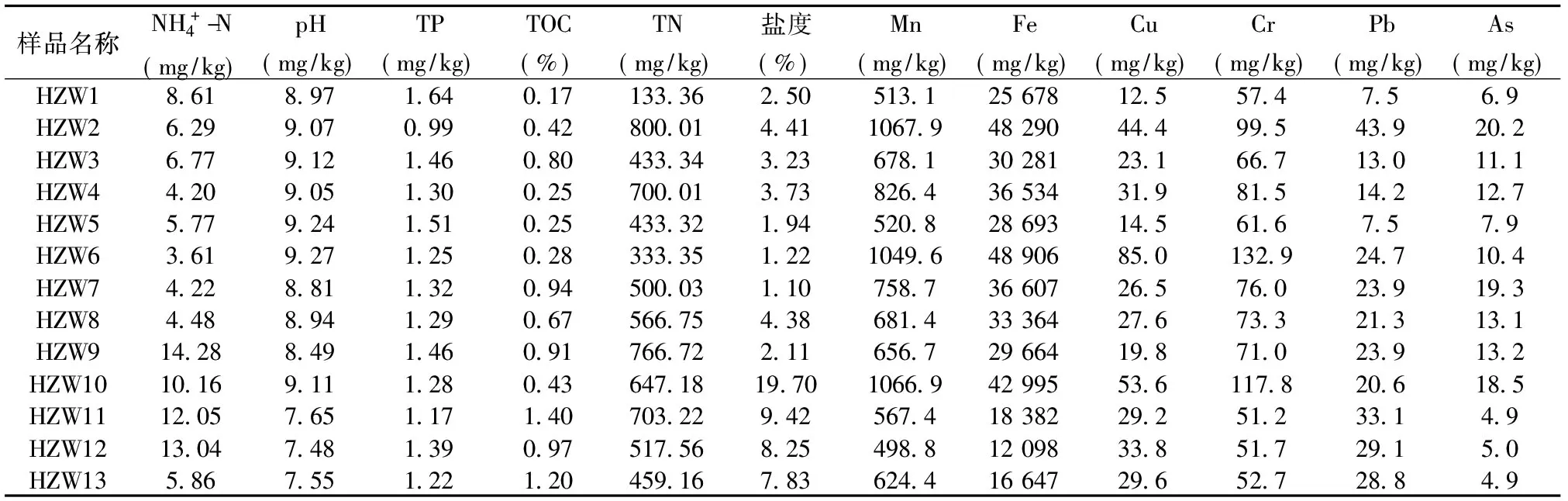
表2 沉积物样品主要理化性质Table 2 Physicochemical properties of sediment samples
2.2 原核生物群落多样性
将长度过短和有潜在错误的序列经筛选删去后,在HZW1~HZW13中分别得到了41 351、34 263、39 716、39 915、29 904、36 632、44 321、33 205、33 878、40 884、33 346、43 798和35 419条序列。将相似度>97%序列进行聚类,共得到15 656个OTU。
α多样性分析结果如表3所示,Observed species最大的是HZW6,其次是HZW9和HZW11;HZW2的Shannon-Wiener指数最大,其物种多样性最高,其次是HZW5和HZW6。Chao1指数中,最高的是HZW2,其次是HZW3和HZW5。
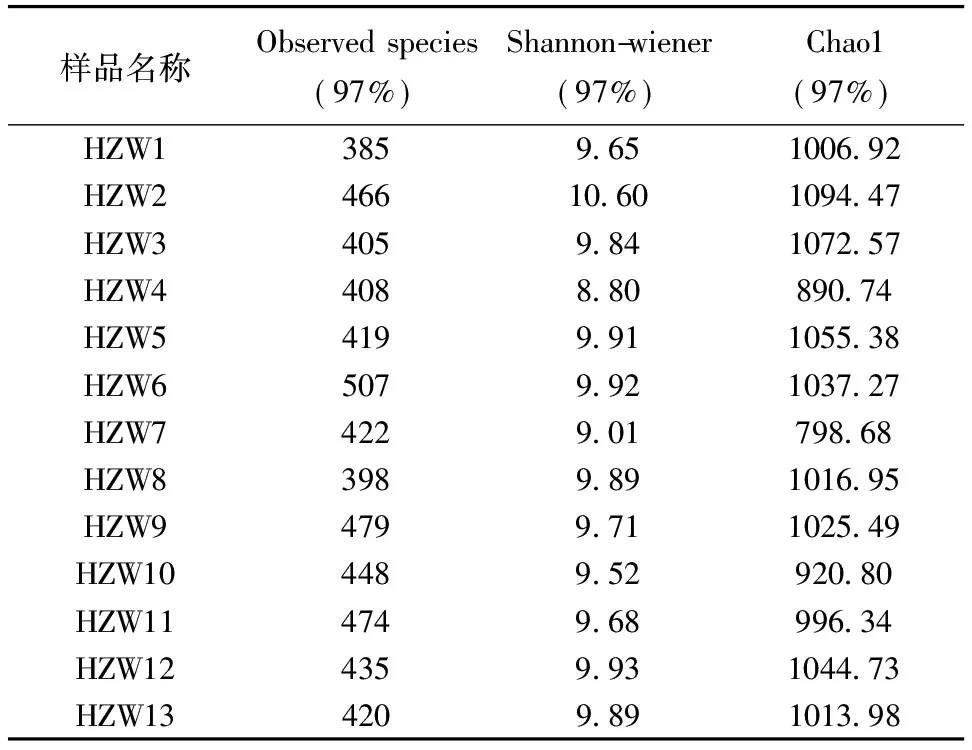
表3 基于97%相似度的α多样性分析Table 3 α-diversity analysis based on 97% similarity
按照众数原则,利用RDP对15 656个OTU进行注释,共有65个门、210个纲、419个目、688个科和1118个属。由于菌门较多,本研究只选取了相对丰度>1%的菌门,在堆积柱状图上做了图例。结果如图3所示,共有19个相对丰度>1%的菌门(纲)。13个样品中相对丰度最高的优势菌门均为变形菌,HZW10中变形菌相对丰度最高,而HZW12中变形菌相对丰度最低。因为每个样品中变形菌门相对丰度都很高(相对丰度均>30%),所以在柱状图上以变形菌门下相对丰度>1%的α、β、γ和δ变形菌纲进行展示。
在科水平下,13个样品中除了HZW4、HZW5、HZW6和HZW7之外,优势菌均为鱼立克次体(Piscirickettsiaceae),其平均相对丰度为8.4%。HZW4的优势菌为地杆菌(Geobacteraceae,6.4%),HZW5的优势菌为丛毛单胞菌(Comamonadaceae,3.6%),HZW6的优势菌为(Comamonadaceae,2.5%),HZW7的优势菌为不动细菌(Acinetobacter,13.8%)。
2.3 原核生物群落与环境因子关系
菌群丰度与环境因子相关性用Pearson系数表示,若基小于0.05,则认为两者显著相关;Pearson系数若为正的,则表示两者之间呈正相关,以红色表示;Pearson系数若为负的,则表示两者呈负相关,以蓝色表示。
3 讨论
α多样性是反映物种多样性的重要指标,通过高通量测序对杭州湾入海口沉积物中原核生物群落多样性分析结果表明Shannon-Wiener指数达9.72,Chao1指数达997,OTU总数达到15 656,这些指数均远高于应用PCR-DGGE和T-RFLP等传统方法研究近岸海域沉积物所获得的结果[17-18],说明了高灵敏度的高通量测序方法能够更准确地反映出环境中原核生物群落多样性特征。
本研究对杭州湾南北两岸以及内海部分区域和洋山港进行了海水表层沉积物原核生物多样性分析,共发现了65个菌门,且优势菌都是变形菌,而变形菌门下的优势菌有α、β、γ和δ变形菌,这与孟加拉湾深海[19]和日本冲绳县伊平屋岛北部海洋[20]的沉积物所获得结果相类似。3个(HZW11、HZW12和HZW13)内海样品中变形菌的丰度相对较小,而绿菌则相反。另外,南北岸微生物多样性的差异可能是由于南北两岸所处的地理位置不同而受海水的潮汐、温度和环境污染物等因素综合影响而导致的,尤其是不同工业区排放的污水虽经处理,但是长期排放而累积于沉积物中不同污染物如重金属等所形成的不同微生态环境可能会对原核生物的生长具有选择性。此外,在海盐县秦山核电站附近采集的样品(HZW3)群落结构特征与邻近HZW1和HZW2的较为相似,说明了核电站的附近滩涂中的微生物多样性受其影响不明显。

4 小结
综上所述,通过高通量测序方法分析了杭州湾入海口沿岸和内海沉积物原核生物多样性特征以及环境因子对其分布的影响,发现南北两岸原核生物种类存在差异,金山工业区和上虞工业区排污口附近水域微生物多样性也存在明显不同,环境因子及累积的重金属等对原核生物多样性皆产生了重要的影响。
[1]MYUNG C E, INHONG S, SOON K K.Estimation of pollutant loads in an estuarine reservoir considering pollution source characteristics and seasonal variation [J].Paddy Water Environment, 2010, 8(4): 347-360.
[2]YOON C G, HAM J H.Prediction of water quality in estuarine reservoir using SWMM and WASP5 [J].Korean J Environ Agric, 2000, 19(3): 252-258.
[3]BOSTROM B, ANDERSON J M, FLEISCHER S, et al.Exchange of phosphorus across the sediment-water interface [J].Hydrobiologia, 1988, 170(1): 229-244.
[4]秦铭俐, 蔡燕红, 王晓波, 等.杭州湾水体富营养化评价及分析 [J].海洋环境科学, 2009, 28 (Supp1): 53-56.
[5]宋祖光, 高效江, 张 弛.杭州湾潮滩表层沉积物中磷的分布、赋存形态及生态义 [J].生态学杂志, 2007, 26(6): 853-858.
[6]叶 然, 刘艳云, 崔永平, 等.东海营养盐结构的时空分布及其对浮游植物的限制 [J].海洋与湖沼, 2015, 46(2): 311-320.
[7]FALKOWSKI P G, FENCHEL T, DELONG E F, et al.The microbial engines that drive earth′s biogeochemical cycles [J].Science, 2008, 320(5879): 1034-1039.
[8]WHEELER D A, SRINIVASAN M, EGHOLM M, et al.The complete genome of an individual by massively parallel DNA sequencing [J].Nature, 2008, 452(7189): 872-876.
[9]HJ 634-201.土壤氨氮、亚硝酸盐氮、硝酸盐氮的测定: 氯化钾溶液提取-分光光度法 [S].北京:环境保护部,2012.
[10]杨锐明, 李 彦.微波消解及ICP-AES同时测定土壤中多种元素含量 [J].实验技术与管理, 2011, 28(8): 26-28.
[11]XU N, TAN G C, WANG H Y, et al.Effect of biochar additions to soil on nitrogen leaching, microbial biomass and bacterial community structure [J].European Journal of Soil Biology, 2016, 74: 1-8.
[13]CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al.QIIME allows analysis of high-throughput community sequencing data [J].Nature methods, 2010, 7(5): 335-336.
[14]EDGAR R C.Search and clustering orders of magnitude faster than BLAST [J].Bioinformatics, 2010, 26(19): 2460-2461.
[15]WANG Q, GARRITY G M, TIEDJE J M, et al.Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy [J].Applied and Environmental Microbiology, 2007, 73(16): 5261-5267.
[16]QUAST C, PRUESSE E, YILMAZ P, et al.The SILVA ribosomal RNA gene database project: improved data processing and web-based tools [J].Nucleic Acids Research, 2013, 41(Database issue): D590-D596.
[17]杜 萍, 刘晶晶, 沈李东, 等.Biolog和PCR-DGGE技术解析椒江口沉积物微生物多样性 [J].环境科学学报, 2012, 32(6): 1346-1444.
[18]蒋 然,王健鑫,黄 备,等.椒江口沉积物中细菌多样性初步研究 [J].海洋与湖沼, 2015, 46(4): 887-900.
[19]VERMA P, RAGHAVAN R V, CHE O J, et al.Complex bacterial communities in the deep-sea sediments of the Bay of Bengal and volcanic Barren Island in the Andaman Sea [J].Mar Genomics, 2017, 31: 33-41.
[20]ZHANG X Y, WANG G H, XU X Y, et al.Exploring fungal diversity in deep-sea sediments from Okinawa trough using high-throughput Illumina sequencing [J].Deep-Sea Research Ⅰ, 2016, 116: 99-105.
[21]WEBSTER G, O′SULLIVAN L A, MENG Y, et al.Archaeal community diversity and abundance changes along a natural salinity gradient in estuarine sediments [J].FEMS Microbiology Ecology, 2015, 91(2): 1-18.
[22]ZHANG W P, BOUGOUFFA S, WANG Y, et al.Toward understanding the dynamics of microbial communities in an estuarine system [J].PLOS One, 2014, 9(4): e94449.
[23]WANG K, YE X S, CHEN H P, et al.Bacterial biogeography in the coastal waters of northern Zhejiang, East China Sea is highly controlled by spatially structured environmental gradients [J].Environmental Microbiology, 2015, 17(10): 3898-3913.
[24]RASMUSSEN L D, ZAWADSKY C, BINNERUP S J, et al.Cultivation of hard to culture subsurface mercury resistant bacteria and discovery of new merA gene sequences [J].Applied and Environmental Microbiology, 2008, 74(12): 3795-3803.
[25]GUO H, NASIR M, LV J, et al.Understanding the variation of microbial community in heavy metals contaminated soil using high throughput sequencing [J].Ecotoxicology and Environmental Safety, 2017, 144: 300-306.
[26]KACI A, PETIT F, FOURNIER M, et al.Diversity of active microbial communities subjected to long-term exposure to chemical contaminants along a 40-year-old sediment core [J].Environmental Science and Pollution Research, 2016, 23(5): 4095-4110.

