Spt蛋白与酿酒酵母压力抗性研究进展
芦志龙,陆琦,陈英,吴仁智,黄俊,陈小玲,陈东,黄日波
1 广西大学 亚热带农业生物资源保护与利用国家重点实验室,广西 南宁 530005
2 广西大学 生命科学与技术学院,广西 南宁 530005
3 广西科学院 国家非粮生物质能源研究工程中心,广西 南宁 530007
Spt蛋白是一系列具有转座子突变回复能力、与酿酒酵母抗性高度相关的转录过程的一大类调控因子[1-5]。Spt蛋白因此已成为全局转录工程的改造对象,并且已取得了一定的成效[6-8]。然而,针对 Spt蛋白的研究还存在一定的盲区。一是没有针对 Spt蛋白调控下的转座子群的行为对酵母适应和进化的影响,尤其是对压力环境下的研究。二是没有针对Spt蛋白在压力环境下通过细胞膜壁重塑进行调控的研究。我们通过耐高温发酵酿酒酵母转录组比较分析和表达差异的溯源发现,一些Spt蛋白 (如Spt23) 的突变体在酿酒酵母高温环境适应性方面发挥了主导作用,而且这种作用可能部分是通过转座子产生的。基于Spt23的全局转录调控将成为一种极具潜力的酿酒酵母改造手段。本文尝试从Spt蛋白通过转录调控、转座突变回复及细胞膜转变这3个方面来增强酿酒酵母抗性的潜在作用作综合阐述,并对当前研究中的不足提出了一些针对性的建议。
1 Spt与SAGA复合体
酿酒酵母的转录调控是个复杂的过程,涉及到大量的全局调控因子,并由它们调控着数目巨大但彼此间联系松散的基因群[9]。它们通常是作为大型蛋白分子复合体的单元发挥作用的:它们有的通过核小体相互作用来调节转录抑制的程度,有的直接与其他转录激活因子相互作用来调节转录,有的作为RNA聚合酶中与DNA相互作用的亚基发挥作用。但大体上,酿酒酵母的转录调控被分为两种不同的机制。
一是TFII参与的转录调控活动。Spt蛋白便是具有这些特征的酵母转录调控因子。这类蛋白通常与组蛋白编码基因或者 TATA结合蛋白(TBP蛋白) 相关联。以酵母Spt15为例,它是由Spt15编码的大小为27 kDa的蛋白,又被称为d因子,是 TFIID的一个亚基,是RNA聚合酶Ⅱ和RNA聚合酶Ⅲ的组成成分,对于转录的起始过程是不可或缺的。Spt15具有结合TBP的功能,结合 TBP的速率关系着转录激活的效率。TFIID参与了约90%的酵母转录活动。
二是SAGA复合体参与的转录调控活动。约有10%的酵母基因转录是通过SAGA复合体(Spt-Ada-Gcn5乙酰转移酶复合体) 来实现转录调控的[10]。SAGA复合体是能够重塑染色质构象,结合TATA区域,协助RNA聚合酶Ⅱ起始转录的多亚基复合体[11]。SAGA的构象依据调控的基因不同而改变。SAGA复合体具有组蛋白的乙酰基转移酶活力和去泛素酶活力[12]。
RNA聚合酶Ⅱ的这两类转录活动呈现出明显的差异性,因此被命名为TFIID-型和SAGA-型。前者调控范围广泛,调控基因多为基本生命活动相关的看家基因,调控的方式较为疏散;后者调控范围较小,调控基因多与环境压力应激反应有关,且调控方式较为紧密。SAGA调控的基因大部分是与酵母的高渗透压、有害代谢物等环境压力应激反应如 MAPK、HOG途径高度相关的[13-17]。对于在环境压力下表达上调的基因,与SAGA调控的关联性尤其显著。随着表达差异分析技术的进步,关于TFIID-型和SAGA-型调控的认识基本明确:两种复合体独立地、全局地参与RNA聚合酶Ⅱ的转录活动,只是前者结合于UAS (上游激活序列区),后者则直接结合于转录起始位点[18-20]。SAGA-型通过激活因子介导而结合到启动子区域,TFIID是直接结合启动子区域的[17]。
SAGA复合体的调控活动是通过修改染色质形态来实现的。核小体结合于 DNA之上,通常使基因的转录处于抑制状态。SAGA复合体通过转乙酰基作用改变核小体中组蛋白与 DNA的结合状态,从而使转录因子能够顺利地结合顺式作用元件,开始转录活动。但是对于复合体发挥作用的完整细节,目前还没有定论。Grant等提出了两种由Ada复合体-Spt复合体相互作用的SAGA转录激活模型[3]。一是Ada复合体和 Spt复合体各自独立但均包含Gcn5,因而都具有组蛋白乙酰基转移酶活力,只不过它们二者均通过未知的Ada蛋白 (或Spt蛋白) 与不同的激活因子相互作用而激活转录;二是只有 Ada复合体包含 Gcn5蛋白而具有了组蛋白乙酰基转移酶活力,但必须通过与Spt复合体的相互作用才能使SAGA复合体结合至 TATA区域,从而启动转录 (图 1)。这两种模型都被认为还有其他未发现的Ada蛋白和Spt蛋白。

图1 SAGA复合体启动转录需要Spt蛋白参与的核小体亚基去乙酰化过程Fig. 1 Initialization of transcription by SAGA complex requires deactylation of nucleosome subunits implemented by Spt proteins. Spt proteins and Ada, Gcn could alternate chromation configuration by deactylaltion of nuclesome subunits and initialize transcription of RNA polymeraseⅡ.
Spt蛋白广泛地参与了酿酒酵母的两类转录调控机制。但 Spt蛋白是个比较庞大的家族,各个成员也存在着巨大的差异。常见的SAGA复合体 Spt蛋白有 Spt3、Spt7、Spt8和 Spt20等。它们相互之间并不调节彼此的转录活性。为了区别于Spt15这样的TBP作用形式,它们被归类为Spt3类 Spt蛋白。依据 Spt蛋白重要程度的不同,Sterner等将它们分成3组。第1组包括Spt7、Spt20和Ada1,它们的突变会导致酵母难以利用大多数碳源,是SAGA复合体中不可或缺的成员。而Spt3和Spt8组成的第 2组与由 Gcn5、Ada2和 Ada3组成的第3组则会导致部分碳源不能利用,其程度较第 1组要轻微得多[21]。Spt蛋白间可能存在功能的重合,这表现在当一个Spt基因被敲除后,酵母并不会死亡。Spt蛋白呈现出的多样性,可以用不同的方式进行分类,比如通过调节方式或转座子抑制活力来区分,此处不再赘述[22-23]。
Spt23则不依赖于SAGA复合体发挥调控作用。Spt23被重新密集研究,是因为它在转座抑制作用之外[24],还对酿酒酵母配型调控发挥着重要作用。MAT基因座左右方向分别为HML和HMR两个基因。在单倍体酵母中,二者只有一个可以被转录,另一个被沉默,从而避免出现非交配性酵母。这个沉默过程是由被沉默基因两翼的E和I沉默子区域,以及一系列转录调控因子相互作用完成。其中,Sir1–Sir4起主要作用。这4种任何一种的敲除均不能导致酵母死亡,但会导致部分或全部的转录沉默现象的消失。Rap1、Abf1以及ORC六亚基复合体介导了Sir蛋白与E或I沉默子区域的结合。通过Sir蛋白与H3、H4组蛋白相互作用,对染色质的构象进行调整。而 Sum1可以抑制因 Sir蛋白突变导致的沉默能力的失效。在SUM1缺失的菌株中开展的染色体缺失实验中,发现有Spt23、MGA2等4个位点的缺失可以补偿Sum1的效应[25]。可见Spt23/MGA2的转录激活能力,与配型基因的沉默机制相互竞争,从而改变一些基因的转录水平。即便如此,Spt23及其同源体MGA2对沉默机制的作用细节并不完全相同。比如,在SIR2敲除的酵母中,Spt23/MGA2与HML的沉默作用形成了拮抗关系;但在HML的沉默子区域突变的酵母中,Spt23/MGA2似乎又强化了沉默作用。这种机制的实现细节尚未明了,但Spt23通过染色质构象调节转录活性的能力无疑得到印证。
当然,SAGA复合体并非唯一的依赖染色质形态重塑进行转录调控的功能体。Swi-Snf复合体同样具有染色质形态重塑、组蛋白乙酰化并调控转录的能力[26-28]。事实上这个系统与SAGA复合体功能有相互重叠,在一定程度上是冗余的。此外,Srb/Mediator复合体也与 SAGA复合体有一定程度的结构和功能冗余[5,29-31]。比如,Spt23也可以抑制由SNF2敲除所导致的转录缺陷现象,而这种功能通常由 Srb/Mediator调控因子复合体来实现[32]。总体上,虽然3个复合体实现机制有相似之处,但它们各自调控的基因各不相同[4]。为此,Roberts等构建了一个 SAGA、Swi-Snf、Srb/Mediator三个复合体共同调节酵母RNA聚合酶Ⅱ转录行为的模型[5]。
2 Spt对转座子的作用
Spt蛋白因为其抑制转座子活力的特性而被发现和命名。酵母转座子Ty是一类具有在基因组中迁移整合能力的多拷贝基因。Ty转座子具有反转录病毒样的蛋白表达序列基因,两侧由正向重复的δ序列包围。δ序列具有多样性,典型δ序列长度约为330 bp,包含有一个带TATA框的启动子和一个终止子[33-34]。δ序列也可以分散地存在于基因组中,成为酵母进化过程中转座子活动的足迹。转座子迁移会产生多样的基因变异,徐岗详尽列举了转座子可产生的遗传效应,如插入、删除、染色体结构变异,多达 20余种[35]。转座子移动性和突变性的特点,催生转座子突变技术在20世纪90年代的兴起,这进而导致越来越多的转座子抑制基因被发现。δ序列具有其自身的TATA框启动子,当它插入到基因上游就可能形成相互竞争的双TATA框结构,导致转录酶结合到错误的位点 (图2A)。如HIS4基因的启动子区域被转座子或δ序列插入后,便失去了转录活性,无法在组氨酸营养缺陷型筛选培养基上存活。当Spt作用于转座子序列时,能够促使转录酶结合到正确的TATA框,抑制了转座子突变性,从而使转录活性失而复得 (图2B)。此时,HIS4基因能够转录出天然长度的mRNA,从而使菌株能够在组氨酸营养缺陷型筛选培养基上存活。后续的研究表明,两个TATA框结构存在竞争作用,而Spt突变蛋白能够起始正确的转录。与此同时,转座子自身的转录产物也会被抑制[36]。依据调控转座子基因转录的强弱程度,Spt蛋白被分为2类。Spt3、Spt7、Spt8和Spt15中的任何一个基因的突变都会导致转座子转录水平的强烈下调。其余Spt蛋白归入一组,对于转座子的调控能力较弱[37]。
不同的 Spt蛋白对转座子突变的抑制作用呈现一定的偏好性。Spt13、Spt14对完整转座子 (Ty)插入突变的回复转录能力极强,但对于δ因子插入突变的回复能力弱[37-39]。Spt23则对δ因子在起始位点上游不同插入位点造成的突变均具有较强的突变回复能力[24]。而 Spt3则对转座子 Ty和 δ因子插入突变引发的转录失活均有回复能力[40-41]。Spt蛋白的这种回复能力存在剂量效应,以Spt23为例,只有多拷贝 Spt23被表达的时候才具有抑制转座子突变的表型;单拷贝表达不具备配型调节能力。
转座子对于生物具有重要的进化意义。转座子是生物获得基因组多样性的重要原因。例如,基因组的重排行为依赖于转座子衍生的重组酶。转座子序列构成了人类基因组的45%左右,与发育和疾病关系密切;植物基因组的大小差异,很大程度上源于TE (转座元件) 数目的差别。转座子对于在恶劣环境下的适应性扮演重要角色[42]。转座子是新基因的进化来源之一[43]。酿酒酵母转座子的丰富度在一定程度上反映了酵母通过转座子突变得到进化优势的能力。因此,通过 Spt蛋白对酿酒酵母转座子群进行调控,从而改变其在压力环境下得到优良性状的能力,无疑将是今后重点探索的方向之一。

图2 Spt蛋白通过竞争性结合TATA框回复转座子突变的原理示意图Fig. 2 Spt proteins reccovery transposon induced mutations by competitive enrollment to TATA boxes. (A) Transposons or δ-sequences randomly insert into promoter regeion of genes to form a dual-TATA box structure. Transcription fails as RNA polymerase binds to the wrong TATA box. (B) Spt mutants enables RNA polymerase subunits to bind the native TATA box with higher affinity and neglect the allian one to start transcription, demonstring the ablity to recovery transposon induced mutations.
3 Spt的调控与酵母菌株改造
如前文所述,Spt调控基因通常与酵母抗性高度相关。多种环境压力诱导的酵母应激反应均会造成细胞膜的变化和细胞壁重塑。以9%乙醇耐受研究中的酿酒酵母细胞为例,细胞膜由于高浓度乙醇的存在导致完整性降低,直接表现是细胞膜厚度的减小,薄的细胞膜进而导致细胞壁的刚性和光滑度剧烈降低。并且,由此产生的细胞壁的重塑作用是独立于细胞壁完整性通路 (CWI) 的[2]。Spt23是一个极为特殊的Spt蛋白,在于其调控过程与细胞膜重塑的高度相关性。它主要参与不饱和脂肪酸合成 (OLE1) 调控。不饱和脂肪酸是组成细胞膜的重要成分,与细胞膜的流动性和形态有密切的关系[44-45]。You等通过基因敲除的方式发现不饱和脂肪酸,尤其是油酸对于酿酒酵母耐受乙醇能力起到至关重要的作用[46]。Spt23通过改变细胞膜不饱和脂肪酸的含量提高了酵母抵御环境压力的能力 (图 3)。不饱和脂肪酸的含量和组成反过来也可以调控Spt23转录活性。Spt23对不饱和脂肪酸的调节机制依赖 Cdc48-Npl4-Ufd1复合体。该复合体在真核生物膜介导的细胞过程中发挥重要作用。它可以指导依赖泛素-蛋白酶体的蛋白裂解,并激活两个内质网锚定蛋白Spt23和它的同源蛋白Mga2。这两个蛋白直接激活OLE1基因转录 Δ-9脂肪酸脱饱和酶。Spt23和MGA1是同源基因,同为人源NF-κB的同源体。若Spt23和MGA1同时敲除时,酵母无法在不含不饱和脂肪酸的培养基上生长[32]。两者功能有重合,但不完全相同。比如,MGA1在胆固醇的合成中,对ERG1基因的转录起到关键作用,而Spt23却不能调控[47]。但单就二者的不饱和酸合成调控能力而言,它们都具有改变细胞膜的组分、提高酵母抗性的潜力。我们可以依据 Spt23及其同源体蛋白的调控特点,设计相应的改造手段来提高酵母的抗性水平,尤其是耐高温发酵的性能。
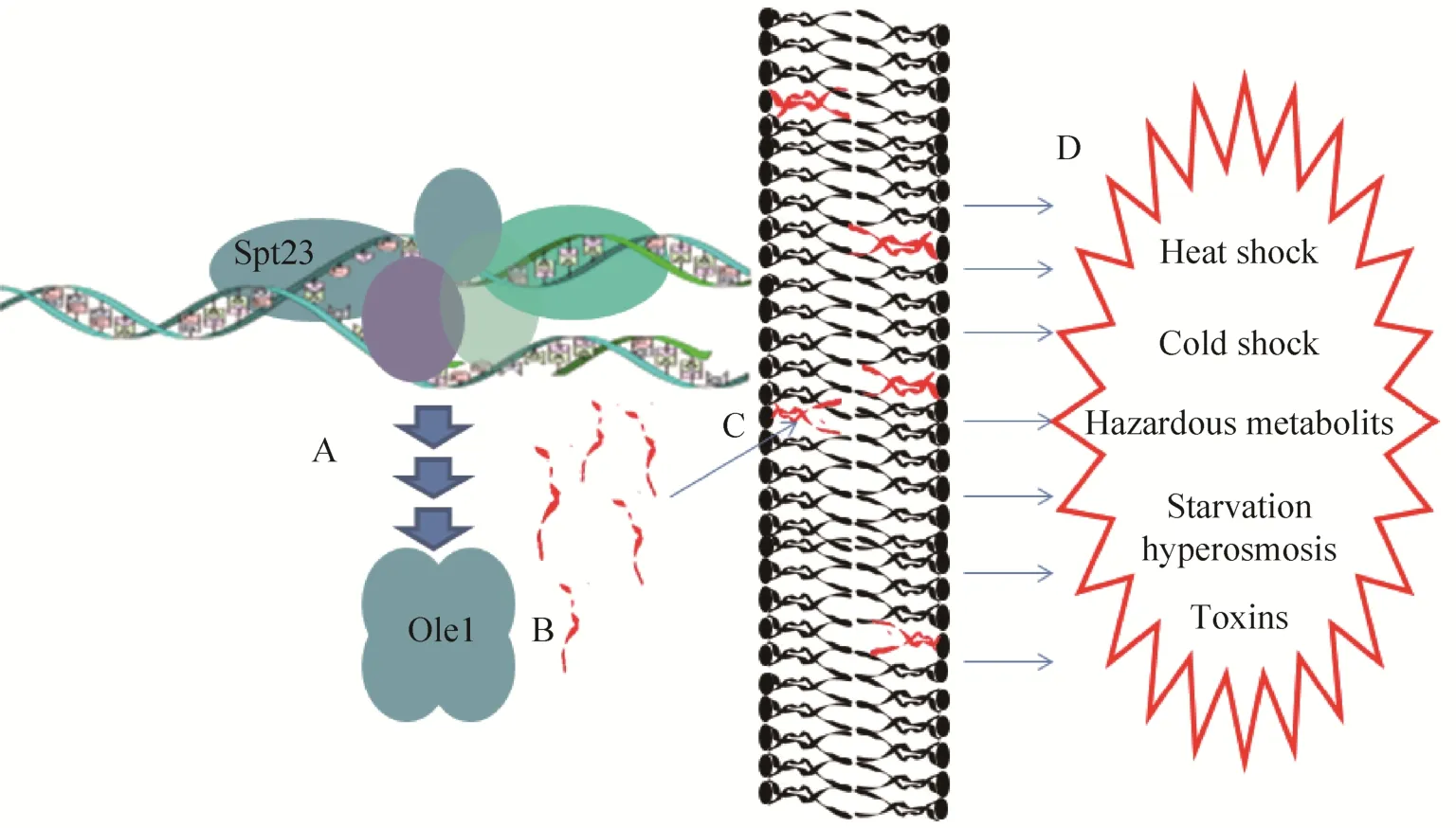
图3 Spt23基因通过调控细胞膜不饱和脂肪酸含量提高酵母抗性Fig. 3 Spt proteins play a roll in stress resistance of budding yeast by regulating the species and ratio of unsaturated lipid acids. A: Spt23 regulates the transcription of OLE1, the key enzyme in unsaturated lipid acids synthesis; B: unsaturated lipid acids synthesis by ole1; C: ratio of unsaturated lipid acids in the phospholipid bi-layer changed accordingly to the environmental turbulence; D: environmental stress resistance strengthened through unsaturated lipid acids constitution change in the cell membrane.
Spt23的活化机制已被阐释清楚。泛素化的Spt23的无活性前体p120最初锚定在内质网上。p120通过二聚体化后具备了活性。但活化后蛋白质分子p90与未活化分子p120并不分离,而是依靠分子间的 IPT结构域结合在一起。p90分子的泛素化修饰特征在此过程中保留。依赖Cdc48-Npl4-Ufd1复合体的作用,p90与其前体p120解离,成为可溶的、定位于细胞核的成熟蛋白分子。在Cdc48-Npl4-Ufd1复合体中, Cdc48对泛素化蛋白具有选择性,同时对于膜的融合发挥重要作用。Ufd1不仅直接参与OLE1基因的调控,而且参与不稳定泛素化分子的降解。Npl4不仅是一种转运蛋白,而且紧密参与不饱和脂肪酸调控的UFD (泛素融合降解蛋白) 途径。p90解离的方式是通过内质网膜定位信号 (C端跨膜区)被降解而实现的。其N端的转录调控区保存完整。p90被运输到细胞核之后,开始调控OLE1基因的表达,从而影响了细胞不饱和脂肪酸的合成。正是由于Spt23基因调控过程中有蛋白酶体的深度参与,Auld等倾向于将其机制归类为蛋白酶体转录调控的范畴。蛋白酶体转录调控主要的调控形式包括转录因子加工和蛋白酶体-染色质关联[1]。
通过 Spt的全局转录工程从细胞整体水平上进行转录水平的扰动,进而获得优良性状是一种有益的尝试。对参与细胞转录起始的所有转录因子,利用包括易错PCR、基因重排等多种手段,得到活力发生变化的转录因子,最终引导代谢流向有利于目标产物的方向。国内有通过对Spt15基因进行饱和突变,得到饱和突变文库,将文库转入酿酒酵母,筛选得到具有葡萄糖木糖共发酵的酿酒酵母菌株[7]。笔者认为,这株独特的酿酒酵母菌株可能通过基因水平转移获得了木糖代谢的基因,但是受碳源经济性的限制而受到转录抑制;通过Spt15的改造,RNA聚合酶Ⅱ的转录谱偏好发生转换,开启了相关基因表达,从而产生了木糖代谢的表型。山东大学通过Spt15基因的删除和导入,探讨对酿酒酵母高渗透压和高乙醇耐受性能力的影响[48]。曹喜涛等针对Spt15和TAF25开展了基于易错 PCR的全局转录工程改造,结果使S-腺苷蛋氨酸酿酒酵母生产菌株的产量提高了1.8−2.0倍[8]。也有针对Spt3开展的全局转录工程改造研究,用于提高酿酒酵母产乙醇水平[6]。
4 Spt通过转座调控酵母抗性
前文已对酿酒酵母转座子的突变性和迁移性作了阐述。正是由于转座子具有迁移性,它们可以随机地开启或关闭一些基因的转录过程。根据广西科学院工业生物科学与技术研究中心的初步分析结果,酿酒酵母转座子和δ序列的数目与酵母乙醇耐受性呈现一定的负相关性。例如,含有381个δ序列的遗传高产菌株1015,其耐高渗和耐乙醇水平要远远高于含有771个δ序列的低产菌株 1415。但两者的 δ序列数目差异是否由Spt23基因的突变所引发还需要进一步的实验支持。通过乙醇高温发酵的表现发现,Spt23蛋白的点突变会导致1015菌株转座子表达量下调,与此同时其高温发酵产酒率 (37 ℃条件下) 提高了2%,甚至能在54 ℃热击后仍可存活,作为对比的出发菌株完全灭活 (未发表)。通过对Spt23蛋白突变前后的转录组比较发现,突变后的Spt23基因促使大部分可调控基因表达量下调 (图 4)。通过对常温和高温乙醇发酵酿酒酵母转录组进行了表达差异分析,并利用基于网络算法的数量性状连锁分析手段将差异表达的诱因追溯到特定的基因突变。eQTL即表达数量性状位点分析,依据的原理是差异表达基因倾向于由相互紧密互作关系的驱动基因串驱动的。其方法是将公开数据库得到的细胞互作数据,依据互作程度强弱形成带权重的有向图,将全部基因表达程度作为节点间重值,从差异表达基因出发,通过二元决策的算法上溯至最可能的驱动突变子[49-50]。结果表明,Spt23是等级最高 (Rank=1)、驱动基因数目最多 (137个)、驱动效应最明显的基因 (绝大部分被调控基因表达下调,见图4)。Rank反映了差异表达基因与突变基因间关联程度的强弱,这与Spt系列基因的全局调控能力是相吻合的。同时,可以从图中看到有3个转座子 (YER137C-A、YMR045W和YIL082W-A) 发生了表达差异是可以追溯到Spt23的基因突变的。推测这3个转座子基因插入位点上下游的基因可能在提高酿酒酵母抗性水平中发挥关键作用,因此有必要开展更深入的研究。将来可以通过Spt23的全局转录工程改造和Spt23突变库的转座子活力干扰来探索提高酿酒酵母对环境压力的抗性。由此可见,Spt23从两个层面上对转录进行了调控:一是作为通用转录调控因子发挥作用;二是调控转座子活性而发挥活性。两种层面都是全局性的、效果显著的转录调控。
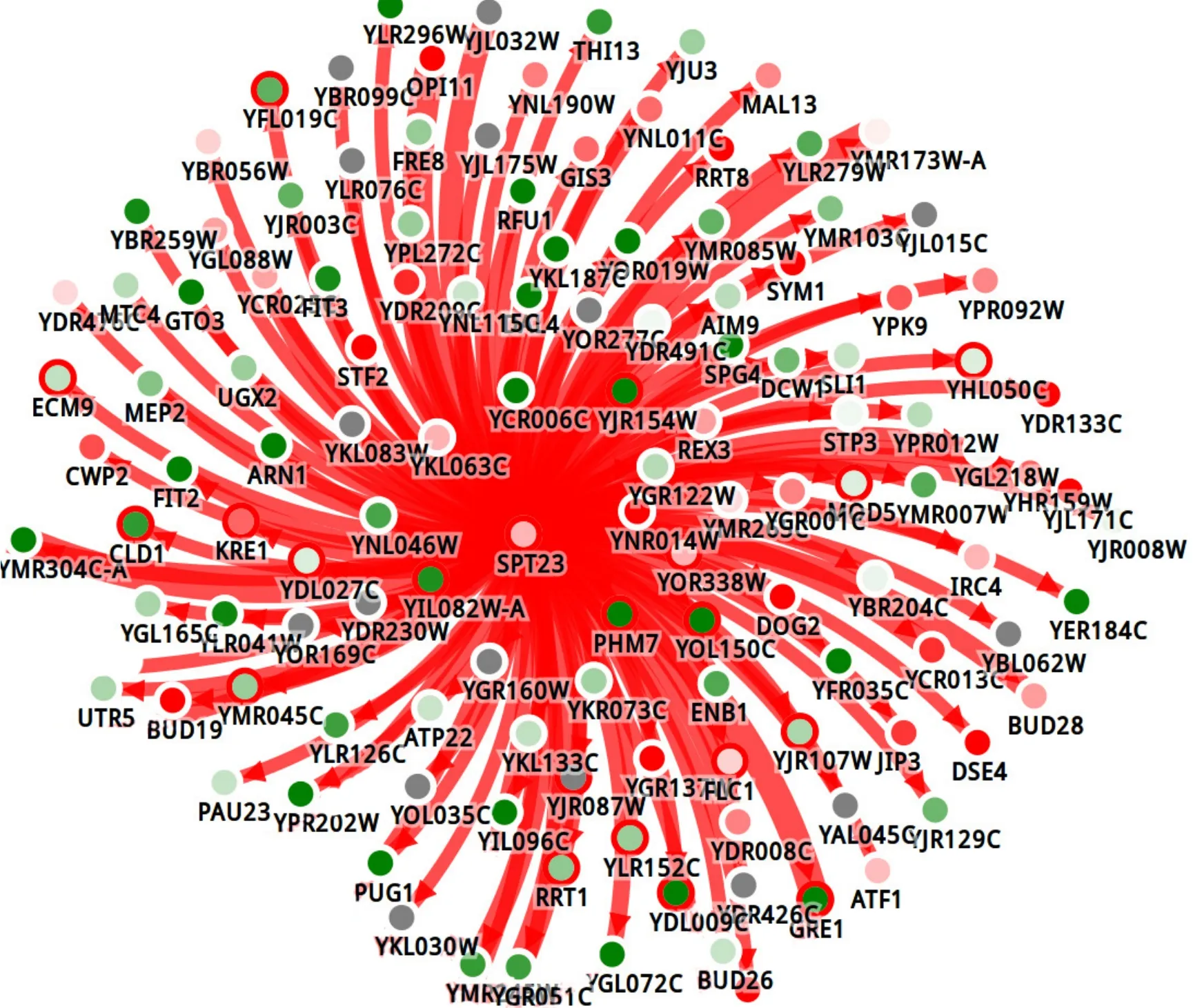
图4 Spt23突变体导致转录下调和高温耐受性提高Fig. 4 Down regulation by Spt23 mutant leads to enhancement of high temperature stress resistance. Tracking of driving mutant for the genes with transcriptional level changed was implemented by PheNetic eQTL. The direction of regulation goes was indicated by arrows. The strength of regulation was indicated by the width of arrows. Different interaction types are represented by the colors of arrows, in which red for DNA-protein interaction. Red in center of the regulated genes indicated up-regulation, while green for down-regulation. The intensity of color indicted the extend of up-/down-regulation.
5 研究方向展望
上文介绍很多针对Spt进行全局转录工程改造的研究。整体上看,这些研究绝大部分注重RNA聚合酶自身亚基的改造,这是合理的,但是有局限性。事实上,影响全局转录调控的因素极其繁多,RNA聚合酶自身亚基只是其中的一部分。我们认为更多的研究应当关注SAGA复合体亚基,尤其是非RNA聚合酶亚基的Spt蛋白 (如非Spt15) 在酿酒酵母环境压力耐受性方面的应用,比如Spt3、Spt4、Spt8等。而Spt23可以从全局转录调控改造、转座子抑制和细胞膜重塑3个方面发挥酵母抗性增强的潜在作用,应当成为优先研究的对象。
除了扩展全局转录调控工程的研究对象外,更重要的是改变 Spt系列蛋白的表达水平。作为酿酒酵母重要的生命机制之一的转录过程,其参与者众多、功能互有重叠,点突变或敲除产生的性状变化往往有限。相比而言,改变启动子产生的影响可能更大。为此,适当采用启动子重排的方法,通过一定的手段改变Spt基因各自的启动子,使其活力有高有低,形成不同的活力谱,从而改变信息的传递路径和通量,最终对性状产生影响。这种策略已经作为一种代谢组研究工具应用于木糖发酵酿酒酵母菌株的开发[51-52]。从实现难度上看,目前有成熟的 DNA组装的方法[53]。如基于Ⅱ型限制性内切酶的“金门”克隆可以快速随机地重排启动子,有助于该策略的开展[54]。
针对 Spt蛋白做酿酒酵母环境压力耐受性研究,除了从全局转录工程入手外,还可以通过Spt的转座子抑制效应来改造酿酒酵母。虽然目前已知 Spt蛋白、转座子和酵母抗性存在着一定的关联性,其作用机理仍然不全面、不详尽,这也将是 Spt蛋白调控研究的一个方向。具体而言,今后的研究应当解决的问题包括但不限于:Spt蛋白对转座子的全局调控机制;Spt蛋白不同突变体和全基因组中转座子数据以及 δ序列数目间的联系;Spt蛋白不同突变体对酿酒酵母总体转座活力的影响;转座总体活力对酿酒酵母抗性的影响;转座总体活力对酿酒酵母基因水平转移能力 (从环境中得到进化优势基因) 的影响;Spt蛋白赋予酵母抗性时各个作用方式的权重等。
[1]Auld KL, Silver PA. Transcriptional regulation by the proteasome as a mechanism for cellular protein.Cell Cycle, 2006, 5(14): 1503–1505.
[2]Schiavone M, Formosa-Dague C, Elsztein C, et al.Evidence for a role for the plasma membrane in the nanomechanical properties of the cell wall as revealed by an atomic force microscopy study of the response ofSaccharomyces cerevisiaeto ethanol stress.Appl Environ Microbiol, 2016, 82(15): 4789–4801.
[3]Grant PA, Sterner DE, Duggan LJ, et al. The SAGA unfolds: convergence of transcription regulators in chromatin-modifying complexes.Trends Cell Biol,1998, 8(5): 193–197.
[4]Winston F, Sudarsanam P. The SAGA of Spt proteins and transcriptional analysis in yeast: past, present, and future. Cold Spring Harbor Symp Quant Biol, 1998,63: 553–562.
[5]Roberts SM, Winston F. Essential functional interactions of SAGA, aSaccharomyces cerevisiaecomplex of Spt, Ada, and Gcn5 proteins, with the Snf/Swi and Srb/mediator complexes.Genetics, 1997,147(2): 451–465.
[6]Zhao XQ, Jiang RJ, Li N, et al. Improving ethanol tolerance ofSaccharomyces cerevisiaeindustrial strain by directed evolution of SPT3 industrial strain by directed evolution of SPT3. Chin J Biotechnol,2010, 26(2): 159–164 (in Chinese).赵心清, 姜如娇, 李宁, 等. 利用 SPT3的定向进化提高工业酿酒酵母乙醇耐受性.生物工程学报,2010, 26(2): 159–164.
[7]Liu HM, Tang W, Lai CG, et al. Effects of mutational spt15 gene to xylose utilization ofSaccharomyces cerevisiae. Chin J Biotechnol, 2009, 25(6): 875–879(in Chinese).刘红梅, 唐雯, 来灿钢, 等. spt15突变基因对酿酒酵母木糖利用的影响.生物工程学报, 2009, 25(6):875–879.
[8]Cao XT, Chen K, Li Y, et al. gTME duction for construction of recombinant yeast for improving the production of S-adenosylmethionine (SAM).Pharmaceut Biotechnol, 2012, 19(5): 386–391 (in Chinese).曹喜涛, 陈凯, 李扬, 等. 全局转录工程法构建产S-腺苷蛋氨酸重组酿酒酵母的研究.药物生物技术,2012, 19(5): 386–391.
[9]Michael H. Molecular genetics of the RNA polymeraseⅡgeneral transcriptional machinery.Microbiol Mol Biol Rev, 1998, 62(2): 465–503.
[10]Huisinga KL, Pugh BF. A genome-wide housekeeping role for TFIID and a highly regulated stress-related role for SAGA inSaccharomyces cerevisiae.Mol Cell, 2004, 13(4): 573–585.
[11]Kornberg RD. Mediator and the mechanism of transcriptional activation.Trends Biochem Sci, 2005,30(5): 235–239.
[12]Koutelou E, Hirsch CL, Dent SYR. Multiple faces of the SAGA complex.Current Opinion in Cell Biology,2010, 22(3): 374–382.
[13]Proft M, Struhl K. Hog1 kinase converts the Sko1-Cyc8-Tup1 repressor complex into an activator that recruits SAGA and SWI/SNF in response to osmotic stress.Mol Cell, 2002, 9(6): 1307–1317.
[14]van Voorst F, Houghton-Larsen J, Jønson L, et al.Genome-wide identification of genes required for growth ofSaccharomyces cerevisiaeunder ethanol stress.Yeast, 2006, 23(5): 351–359.
[15]Basehoar AD, Zanton SJ, Pugh BF. Identification and distinct regulation of yeast TATA box-containing genes.Cell, 2004, 116(5): 699–709.
[16]Sanz AB, García R, Rodríguez-Peña JM, et al.Cooperation between SAGA and SWI/SNF complexes is required for efficient transcriptional responses regulated by the yeast MAPK Slt2.Nucleic Acids Res,2016, 44(15): 7159–7172.
[17]De Jonge WJ, OʼDuibhir E, Lijnzaad P, et al.Molecular mechanisms that distinguish TFIID housekeeping from regulatable SAGA promoters.EMBO J, 2016, 36(3): 274–290.
[18]Taatjes DJ. The continuing SAGA of TFIID and RNA polymeraseⅡtranscription.Mol Cell, 2017, 68(1):1–2.
[19]Baptista T, Grünberg S, Minoungou N, et al. SAGA is a general cofactor for RNA polymeraseⅡtranscription.Mol Cell, 2017, 68(1): 130–143.e135.
[20]Warfield L, Ramachandran S, Baptista T, et al.Transcription of nearly all yeast RNA polymeraseⅡ-transcribed genes is dependent on transcription factor TFIID.Mol Cell, 2017, 68(1): 118–129.e115.
[21]Sterner DE, Grant PA, Roberts SM, et al. Functional organization of the yeast SAGA complex: distinct components involved in structural integrity,nucleosome acetylation, and TATA-binding protein interaction.Mol Cell Biol, 1999, 19(1): 86–98.
[22]Wu PYJ, Winston F. Analysis of Spt7 function in theSaccharomyces cerevisiaeSAGA coactivator complex.Mol Cell Biol, 2002, 22(15): 5367–5379.
[23]Gansheroff LJ, Dollard C, Tan P, et al. TheSaccharomyces cerevisiaeSPT7 gene encodes a very acidic protein important for transcriptionin vivo.Genetics, 1995, 139(2): 523–536.
[24]Burkett TJ, Garfinkel DJ. Molecular characterization of the SPT23 gene: A dosage-dependent suppressor of ty-induced promoter mutations fromSaccharomyces cerevisiae.Yeast, 1994, 10(1): 81–92.
[25]Dula ML, Holmes SG.MGA2andSPT23are modifiers of transcriptional silencing in yeast.Genetics, 2000, 156(3): 933–941.
[26]Phelan ML, Sif S, Narlikar GJ, et al. Reconstitution of a core chromatin remodeling complex from SWI/SNF subunits.Mol Cell, 1999, 3(2): 247–253.
[27]Peterson CL, Tamkun JW. The SWI-SNF complex: a chromatin remodeling machine?Trends Biochem Sci,1995, 20(4): 143–146.
[28]Peterson CL, Dingwall A, Scott MP. Five SWI/SNF gene products are components of a large multisubunit complex required for transcriptional enhancement.Proc Natl Acad Sci USA, 1994, 91(8): 2905–2908.
[29]Narlikar GJ, Fan HY, Kingston RE. Cooperation between complexes that regulate chromatin structure and transcription. Cell, 2002, 108(4): 475–487.
[30]Kadonaga JT. Eukaryotic transcription: an interlaced network of transcription factors and chromatin-modifying machines.Cell, 1998, 92(3): 307–313.
[31]Natarajan K, Jackson BM, Zhou H, et al.Transcriptional activation by Gcn4p involves independent interactions with the SWI/SNF complex and the SRB/mediator.Mol Cell, 1999, 4(4): 657–664.
[32]Zhang SR, Skalsky Y, Garfinkel DJ.MGA2orSPT23is required for transcription of the Δ9 fatty acid desaturase gene, OLE1, and nuclear membrane integrity inSaccharomyces cerevisiae.Genetics, 1999,151(2): 473–483.
[33]Paquin CE, Williamson VM. Temperature effects on the rate of Ty transposition.Science, 1984, 226(4670):53–55.
[34]Fink G, Farabaugh P, Roeder G, et al. Transposable elements (Ty) in yeast. Cold Spring Harb Symp Quant Biol, 1981, 45: 575–580.
[35]Xu G. Yeast transposon: the Ty factors and effects on gene expression. Chin J Cell Biol, 1988(3): 3–12 (in Chinese).徐岗. 酵母转座子——Ty因子及其对基因表达的影响.中国细胞生物学学报, 1988(3): 3–12.
[36]Hirschman JE, Durbin KJ, Winston F. Genetic evidence for promoter competition inSaccharomyces cerevisiae.Mol Cell Biol, 1988, 8(11): 4608–4615.
[37]Winston F. 47 Analysis of SPT genes: a genetic approach toward analysis of TFIID, histones, and other transcription factors of yeast//McKnight SL,Yamamoto KR, Eds. Transcriptional Regulation. New York: Cold Spring Harbor Monograph Archive, 1992,22: 1271–1293.
[38]Fassler JS, Winston F. TheSaccharomyces cerevisiaeSPT13/GAL11 gene has both positive and negative regulatory roles in transcription.Mol Cell Biol, 1989,9(12): 5602–5609.
[39]Eisenmann D, Arndt KM, Ricupero SL, et al. SPT3 interacts with TFIID to allow normal transcription inSaccharomyces cerevisiae.Genes Dev, 1992, 6(7):1319–1331.
[40]Winston F, Minehart PL. Analysis of the yeastSPT3gene and identification of its product, a positive regulator of Ty trarscription.Nucleic Acids Res, 1986,14(17): 6885–6900.
[41]Winston F, Durbin KJ, Fink GR. TheSPT3gene is required for normal transcription of Ty elements inS.cerevisiae.Cell, 1984, 39(3): 675–682.
[42]Patterson K. Transposons: molecular tools for genome investigation. Irvine: University of California, 2016.
[43]Kloeckener-Gruissem B, Freeling M.Transposon-induced promoter scrambling: a mechanism for the evolution of new alleles.Proc Natl Acad Sci USA, 1995, 92(6): 1836–1840.
[44]Stukey JE, McDonough VM, Martin CE. Isolation and characterization of OLE1, a gene affecting fatty acid desaturation fromSaccharomyces cerevisiae.J Biolog Chem, 1989, 264(28): 16537–16544.
[45]Krulwich TA, Quirk PG, Guffanti AA.Uncoupler-resistant mutants of bacteria.Microbiol Rev, 1990, 54(1): 52–65.
[46]You KM, Rosenfield CL, Knipple DC. Ethanol tolerance in the yeastSaccharomyces cerevisiaeis dependent on cellular oleic acid content.Appl Environ Microbiol, 2003, 69(3): 1499–1503.
[47]Rice C, Cooke M, Treloar N, et al. A role forMGA2,but notSPT23, in activation of transcription of ERG1 inSaccharomyces cerevisiae.Biochem Biophys Res Comm, 2010, 403(3/4): 293–297.
[48]Gao CJ. Analysis of ethanol tolerance and metabolic engineering for improved fermentation inSaccharomyces cerevisiae[D]. Jiʼnan: Shandong University, 2010 (in Chinese).高翠娟. 酿酒酵母耐乙醇特性的分析及代谢工程改造研究. 济南: 山东大学, 2010.
[49]de Maeyer D, Weytjens B, de Raedt L, et al.Network-based analysis of eqtl data to prioritize driver mutations.Gen Biol Evolut, 2016, 8(3): 481–494.
[50]de Maeyer D, Renkens J, Cloots L, et al. PheNetic:network-based interpretation of unstructured gene lists inE. coli.Mol BioSyst, 2013, 9(7): 1594–1603.
[51]Lu CF, Jeffries T. Shuffling of promoters for multiple genes to optimize xylose fermentation in an engineeredSaccharomyces cerevisiaestrain.Appl Environ Microbiol, 2007, 73(19): 6072–6077.
[52]Kinkhabwala A, Guet CC. Uncovering cis regulatory codes using synthetic promoter shuffling.PloS ONE,2008, 3(4): e2030.
[53]Schmid-Burgk JL, Xie Z, Benenson Y. Hierarchical ligation-independent assembly of PCR fragments//Valla S, Lale R, Eds.DNA Cloning and Assembly Methods. Methods in Molecular Biology(Methods and Protocols). Totowa, NJ: Humana Press,2013: 49–58.
[54]Engler C, Marillonnet S. Golden gate cloning//Valla S, Lale R, Eds. DNA Cloning and Assembly Methods.Methods in Molecular Biology (Methods and Protocols). Totowa, NJ: Humana Press, 2013:119–131.

