PPR蛋白在植物线粒体和叶绿体中功能的研究进展
王婉珍, 田发安, 任育军, 缪 颖
(福建农林大学生命科学学院分子细胞和系统生物学中心,福建 福州 350002)
三角状五肽(pentatricopeptide repeat, PPR)蛋白有一种三角状五肽重复结构域,具有该结构域的蛋白属于PPR蛋白家族的成员,是植物中最大的蛋白家族之一.以模式植物拟南芥、番茄和水稻为例,它们分别含有450、471和477个PPR蛋白[1-2].PPR蛋白的N末端存在长度可变及非保守的细胞器定位信号,其后连接着串联重复的PPR结构域.虽然PPR蛋白家族的数量庞大,但仍可根据PPR串联重复的基序种类和数量以及排列方式将不同PPR蛋白分为P亚类和PLS亚类[3].为了探讨PPR蛋白的作用机制,对PPR蛋白进行了结构生物学分析[4].目前,研究发现的PPR蛋白主要是在基因转录后对RNA起修饰作用的一类蛋白,特异性识别并结合到细胞器基因组转录的RNA上,并对这些RNA进行进一步的编辑和修饰.玉米叶绿体中的PPR10蛋白是目前结构和生理功能研究最为清楚的PPR蛋白之一.PPR10属于经典的P类蛋白,在串联的PPR序列前端有一个N端结构域,一个PPR的重复序列有35个氨基酸形成的发卡型α螺旋结构,后面还连接一个含有5个氨基酸的loop结构.两个PPR10蛋白形成二聚体后与RNA结合并发挥功能[4-5].目前的研究表明,PPR蛋白主要定位于植物细胞的细胞器中,特别是在叶绿体和线粒体中的定位最多.它们的主要功能是调节细胞器中基因的转录活性以及对转录后的RNA进行修饰,参与这些RNA的剪接、编辑和翻译等,并对植物的生长发育起调控作用,包括呼吸和光合作用、细胞质雄性不育、胚胎发育以及组织的衰老和死亡调控等[6-9].线粒体和叶绿体是植物细胞中半自主性的细胞器,是植物感受发育和逆境信号的重要感受器,在植物响应内外环境变化的逆向信号传导过程中发挥着重要的功能[10-13].近些年来,随着对植物中PPR家族蛋白的发现和认识不断深入,对PPR蛋白的亚细胞定位、生理功能和分子结构,以及与细胞器中RNA的特异性结合和作用方式等都有了较为深入的了解,逐渐揭开了植物中这一超大蛋白家族的神秘面纱,也对它们参与调控植物生长发育的各个方面都有了全新的认识.同时,研究PPR在细胞器中的功能也有利于进一步探究细胞器和细胞核之间的相互作用关系.本文综述了植物中PPR的分布、结构和种类、定位和分子功能,特别是在线粒体和叶绿体中的功能、植物发育以及与环境适应之间的关系,期望能对PPR的研究提供一些参考.
1 PPR蛋白的分布及基因的结构特征
PPR蛋白家族是由两个独立的研究小组对拟南芥基因组数据进行分析后发现的一类新的蛋白家族[14].Small et al[15]运用强大的计算机分析能力筛选与线粒体、叶绿体功能相关的一系列蛋白,出乎意料地发现一个与三角状四肽(TPR)蛋白家族结构相似的蛋白家族,因这一家族的蛋白都含有一种由35个简并氨基酸串联排列组成的三角状五肽重复结构域,且与TPR蛋白的结构相似,因此被命名为PPR.目前,PPR几乎在所有的真核生物中均能被找到,但在植物与非植物之间的数量差异明显[16].PPR在陆生植物中的数量最多,有几百甚至上千个,但在藻类、原生动物、酵母、真菌以及动物中的数量则屈指可数[14,16-17],因此认为PPR基因主要是在陆生植物中不断延伸、进化和发展.然而,同样是陆生植物,苔藓中仅有103个PPR基因,而维管类植物,如水稻和拟南芥中却有四百多个,因此认为植物在从低等到高等进化的过程中,PPR基因的数量也是呈不断上升的趋势并且在不断分化[3,16].根据PPR蛋白家族的结构与分类,人们猜想PPR基因在植物进化的同时也发生了大规模的扩增,但对于PLS类PPR亚家族的起源还需要进一步论证.有研究推测,PLS家族是由P亚族的突变引起的,其C末端的结构域是为了更好地适应植物进化过程中RNA的编辑或者是一些剪接位点增加的需要[18-21].
研究发现,PPR基因很少甚至不含有内含子[3,15].例如,拟南芥中平均每个基因含有5个内含子,但PPR家族80%以上的基因不含内含子.这种情况在水稻中也是一样的[3,14,16].相反,苔藓中PPR基因含有内含子的比例很高,进化分析的结果显示,这些内含子与高等植物中含有内含子的PPR基因的内含子结构相似[3,15].这与之前猜想的PPR基因的起源相互吻合,PPR基因家族的起源类似于反转录转座事件,即有内含子的祖先PPR基因转录加工形成成熟的RNA,再经过反转录成为没有内含子的双链DNA,该DNA片段进一步插入到基因组中形成不含有内含子的拷贝,这种事件重复地多次发生,最后就形成了庞大的PPR基因家族,从而也使得大部分的PPR基因丢失了内含子[14,16,21].
2 PPR蛋白的种类和结构
根据三角状五肽重复结构域基序的种类、数量以及排列方式的不同大体上可以将PPR蛋白分成两大类:P亚类和PLS亚类[3].PPR基序的数量一般为2~27个[3,22-23].P亚类中PPR蛋白的基序是由简单的35个氨基酸重复排列形成的,前人[15,23]将其命名为PPR基序,简称为P基序.P亚类蛋白主要参与细胞器基因的转录过程.Lurin[3]在对拟南芥的PPR基序进行数据分析后发现了另外两个与P基序高度相似的结构域:S基序和L基序,其中,L基序在氨基酸的数目和碱基组成上是可以变化的,因此将这类以P、L和S等3种基序为重复单位的PPR蛋白,简称为PLS亚类[3].这类相对复杂的PPR蛋白被认为主要参与RNA转录后的修饰,如RNA剪接和编辑等[24-25].PLS亚类可根据其C末端序列结构域的不同进一步细分为4个亚子类:典型的PLS亚子类、E亚子类、E+亚子类和DYW亚子类.DYW亚子类的命名是由于其蛋白C末端高度保守的天冬氨酸—酪氨酸—色氨酸而得来的[3].DYW基序被发现含有一个与胞嘧啶脱氨酶相同的催化残基,且胞嘧啶脱氨酶与C→U(胞嘧啶突变成尿嘧啶)之间的转化相关,因此,DYW的功能被认为与RNA的编辑直接或者间接相关[14,24,26].最近,为了更好地探究PPR蛋白特异性识别并结合RNA的机制,Yin et al[27]对PPR蛋白进行了结构生物学分析.玉米叶绿体中的PPR10蛋白属于典型的P亚类,其晶体结构的分析结果显示,PPR的重复序列可以形成右手发夹型α超螺旋结构(螺旋转角螺旋结构),后接一个含有5个氨基酸的loop结构.一个不对称单元中含有两个反向平行的PPR10分子.两个PPR10分子二聚化,一个PPR分子的N末端与另一个PPR分子的C末端相连接.单链RNA的5′和3′末端区域则分别跟一个PPR10分子的N末端以及与之反向平行的另一个PPR10分子的C末端特异性结合[27].
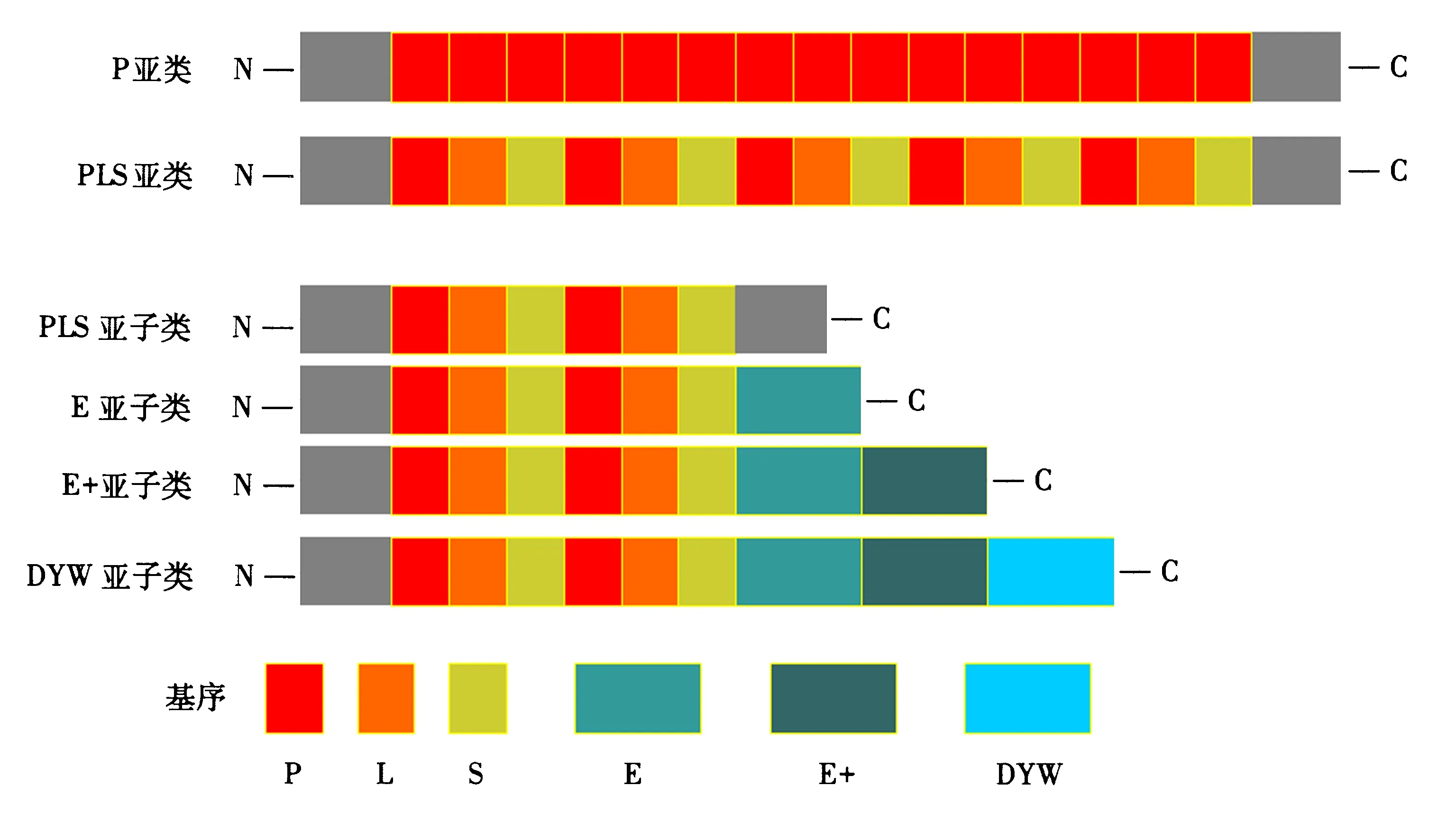
改自于文献[14-15].图1 PPR蛋白家族主要分类Fig.1 The classification of PPR family
3 PPR蛋白在细胞中的定位
目前,蛋白亚细胞定位分析有两种方法:一种是从已经积累的知识出发利用蛋白定位分析软件对未知蛋白质进行预测分析其在细胞中可能的定位和信号肽的位置;另一种是采用实验的方法直接进行定位分析,可实现的技术包括免疫荧光、免疫胶体金和GFP标记融合蛋白荧光显微镜观察等,通过检测免疫或荧光的信号进行目的蛋白的亚细胞定位[1,28].利用第一种方法对已知的PPR蛋白进行亚细胞定位预测,发现大部分PPR蛋白的N末端含有细胞器定位的序列,其中有65%的蛋白定位于线粒体中,17%的蛋白定位于叶绿体中;另外,还有相当数量的PPR蛋白预测存在着双定位,大大超出了之前的预期[15,28].通过第2种方法,已经证明玉米中一个P亚类的PPR蛋白ZmPPR4定位于叶绿体中[23],拟南芥中的MTL1蛋白定位于线粒体中[29].通过免疫印迹实验发现拟南芥中的一个PPR蛋白AtECB2定位于叶绿体的类囊体膜上,它能够影响6个叶绿体基因ACCD、NDHF、NDHG、PETL、RPOA和RPOC1转录本的编辑[30].除了上述单独定位于叶绿体或线粒体中的PPR蛋白,还有一些可以定位在细胞核中的PPR蛋白,如GRP23;同时也存在一部分双定位的PPR蛋白,如PNM1,它同时存在于线粒体和细胞核中[31-32].因此,通过明确PPR蛋白的亚细胞定位将有助于进一步研究PPR蛋白的功能.
4 PPR蛋白的功能
目前对PPR蛋白一些特异的功能已经有所了解.第一种功能是参与RNA编辑,指的是PPR蛋白能特异性地识别细胞器RNA后招募其他的编辑因子,共同对RNA上的胞嘧啶进行编辑为尿嘧啶的过程.如小立豌藓中的PPR78蛋白可以编辑线粒体中的COX1-C755和RP14-C137基因[26].第2种是剪接,指由PPR蛋白和其他剪接体共同对细胞器中转录的前体RNA的内含子进行加工[33-34].第3种是维持RNA的稳定性,是指PPR蛋白与RNA结合后形成空间位阻,从而避免RNA被RNA内切酶所识别和降解,达到稳定RNA结构的作用[35].以下从线粒体和叶绿体中定位的一些PPR出发阐述这些PPR蛋白在植物细胞中的作用以及与植物生长和发育的关系.
4.1 PPR蛋白在线粒体中的功能
线粒体的基因组(mtDNA)有两个典型的特征:一是不同物种间的线粒体基因组大小差异很大;二是不同物种间线粒体基因组的存在形式也不同[36-38].研究表明,植物中线粒体的基因组较大,为200~2 500 kb,明显比动物和真菌中的线粒体基因组要大,这是植物线粒体基因组的一个典型特征.除此之外,高等植物的线粒体基因组还有重排速度快以及具有一些特殊的表达方式等,如顺反剪切、RNA编辑和密码子的使用偏好性等[37,39-40].从目前GenBank上发表的植物线粒体基因组的数据已经知道油菜mtDNA最小,为221 853 bp,而玉米mtDNA最大,为569 630 bp[38].拟南芥和甜菜mtDNA的大小为360 kb,烟草为430 kb[41].拟南芥的mtDNA可以编码32个蛋白、23个tRNA、4个rRNA以及呼吸链上各复合体亚基的一些的关键蛋白[36].拟南芥、水稻和玉米的线粒体基因组可以编码相同的基因产物,如复合体Ⅰ、NADH脱氢酶的9个亚基(NAD1~7、NAD4L和NAD9)、复合体Ⅲ、细胞色素还原酶(COB)、复合体Ⅳ、细胞色素氧化酶(COXⅠ~Ⅲ)、复合体Ⅴ、ATP氧化酶(ATP1、ATP6、ATP8和ATP9)[36,41].此外,烟草和拟南芥的mtDNA还编码呼吸链复合体Ⅱ亚基上的琥珀酸还原酶(SDH)[41].
虽然植物的线粒体基因组只编码30~40个蛋白,但在线粒体中有超过2 000个蛋白[42-44].这些核基因组编码的蛋白质对线粒体的生物功能起重要作用,其中还有部分蛋白在调控线粒体基因组的表达方面起着重要的作用[45].线粒体基因组的表达调控一般发生在转录后修饰的水平,包括RNA的加工、内含子的剪接和RNA的编辑等[45-46].植物线粒体呼吸链的完整性是保证整个植物进行正常生理代谢的关键,进而在整个生长发育过程中发挥正常的生理功能.当植物线粒体的代谢出现缺陷或供能不足时可能会引起植物的早衰或者细胞质雄性不育.而生物线粒体RNA的代谢一般是维持真核生物和原核生物细胞生命活动独有的特性[45].线粒体基因组中第2类内含子的剪接代谢是一个典型的例子.植物中的线粒体因为缺少自我剪接的功能,需要借助一些核基因组编码的蛋白来促进线粒体基因组内含子的剪接[47-49].PPR蛋白就是这类核基因编码参与剪接功能的蛋白,它们在拟南芥中的数量有400多个[50],大部分定位于线粒体或叶绿体中[3,14,21-22,51].目前已知在拟南芥线粒体中部分PPR蛋白的功能如下:OTP43涉及NAD1基因内含子1的剪接,缺失OTP43基因的突变体植株在第4周开始相对于野生型会提前衰老[52];ABO5对NAD2基因的内含子3进行剪接[53];OTP439和TAG2分别参与NAD5基因内含子2和3的剪接[54];BIR6和SG3分别剪接NAD7基因的内含子1和2[55-56].线粒体SLO4蛋白既参与NAD4基因RNA的编辑,同时也影响NAD2基因内含子1的剪接,借此辅助植物的正常生长和发育[34].MTSF1可以稳定NAD4基因的mRNA,保证NAD4基因的正常翻译,从而形成线粒体复合物Ⅰ的亚基[57].另外有些PPR蛋白还参与了细胞色素c氧化酶亚基和ATP合成酶编码基因mRNA的编辑[32,58-60].MTL1是一类P亚型的PPR蛋白,它会影响NAD7基因的翻译起始,导致非正常翻译,使植物中NAD7蛋白不能正常累积,造成发育迟缓,但不影响种子的成熟和萌发[29].拟南芥和玉米中的DEK36基因均编码一种PLS类的PPR蛋白,它的突变会影响线粒体转录本的编辑和种子的发育[9].MEF35蛋白是以线粒体RPL16、NAD4和COB基因的转录本作为靶标进行RNA编辑[61].拟南芥PPR19蛋白在植株的生长和发育过程中起重要作用,它的突变会直接影响线粒体的功能以及NADH脱氢酶1转录本的稳定性[35].另外,欧洲油菜中一个定位在线粒体上的PPR蛋白能够恢复NAP基因突变导致的雄性胞质不育的性状[62](表1).以上研究表明,植物线粒体中定位的这些PPR蛋白一旦缺失就会使植物细胞中线粒体基因组的转录和表达不正确,造成植物生长不良,表现为植株发育延迟、胚胎发育异常、叶形不正常、叶片早衰以及种子产量低等特征[21].因此,以线粒体基因组为靶标的PPR蛋白在保证植物生长发育的过程中对线粒体正常功能的发挥起关键作用.
4.2 PPR蛋白在叶绿体中的功能
叶绿体是植物特有的半自主性细胞器,含有双链环状的DNA分子,主要进行光合作用.叶绿体基因组的基因以功能不同主要分为三大类:与基因表达相关、与光合作用相关以及与脂肪酸等生物合成相关,如ATP合成酶、起始因子、NADH脱氢酶的各个亚基等[63].不同植物的叶绿体基因组大小不同[64].拟南芥叶绿体基因组的大小约为150 kb,含有26 264 bp的一对反向重复序列、87个编码蛋白的基因序列以及编码4个rRNA和37个tRNA基因的序列[63-64].

表1 植物中鉴定的PPR的类型、亚细胞定位和主要功能Table 1 The type of PPR, subcellular lacalization and some function in plant
植物叶绿体自身可以编码60~200个蛋白,参与叶绿体的发育、新陈代谢以及光合作用等,然而叶绿体的发育以及功能行使仍需要受到细胞核基因组的调控[73].当叶绿体基因组进行表达时,一些转录后的加工步骤是必须的,如RNA的加工、编辑和剪接[74-75].在叶绿体中,RNA的加工包括5′和3′末端的成熟和多顺反子转录本的断裂[74].拟南芥HCF152参与了叶绿体RNA的加工[69].RNA的编辑是指在特定的位点上将胞嘧啶腺苷转变为尿嘧啶腺苷的过程[76].一些叶绿体的RNA包含第二类内含子,需要在翻译前进行剪接[77].上述基于RNA的修饰和加工过程需要许多核基因编码蛋白,包括PPR蛋白[15]、MORFs蛋白和CRM蛋白等[71,78]的参与.已有的研究结果显示,PPR蛋白参与了叶绿体基因组表达的每个步骤(表1),如SOT1是叶绿体前体rRNA正确加工所必需的[65];HCF152和PPR10是序列特异的RNA结合蛋白,参与叶绿体前体多顺反子的加工和特殊RNAs的稳定化[69,79].在叶绿体中发现存在一些与叶绿体RNA剪接相关的PPR蛋白,包括5个P类型的蛋白THA8、PPR5、OTP51、PPR4和WSL4[7,23,68,80-81],以及一个PLS类型的蛋白OTP70[33].除了剪接功能,还有许多PPR蛋白参与RNA编辑.AtECB2影响ACCD、NDHF、NDHG和RPOA等基因的编辑[82].另外,许多PPR蛋白的编辑功能同时需要多个细胞器RNA编辑因子MORFs蛋白的参与[71,83],如PDM1与MORFs蛋白相互作用参与ACCD-1基因的RNA编辑[64].参与RNA编辑的PPR蛋白大多属于PLS类,目前比较明确的是这些PPR蛋白选择RNA编辑位点的功能区是在PPR结构域,但E1、E2、E+和DYW结构域的具体功能目前还不是很清楚[76].分子和系统发育的证据认为DYW结构域有胞苷脱氨酶活性,主要负责RNA编辑[26,76].CRR28和OTP85的DYW结构域(5′和3′末端到0区域)与靶C相互作用,使靶目标C脱氨基[70,84],而CLB19主要是对RPOA和CLP1基因转录本进行编辑,其中,E结构域是编辑区域,而不是RNA结合的区域[67].但E结构域在编辑中是怎样起作用的,目前仍不是很清楚,推测可能是与其他的编辑因子相互作用后而激发的功能.如叶绿体NDHD-1基因位点的编辑由CRR4识别,CRR4是含有E结构域的PPR蛋白,然而具有编辑活性的DYW结构域却是由DYW1提供,DYW1是只含有DYW结构域的小分子蛋白[85].RARE1和VAC1都是DYW类型的PPR蛋白,以拟南芥叶绿体ACCD-C794基因位点为编辑目标的PPR蛋白[16,66].除了已知的编辑RNA特异序列的PPR蛋白外,RNA的编辑还需要其他非PPR蛋白的参与,MORFs蛋白就是其中的一种.在拟南芥中MORF蛋白家族有10个成员,均含有保守的蛋白结构域,命名为MORF结构域[71].这10个成员中有7个蛋白定位在线粒体中,MORF9和MORF2定位在叶绿体中,而MORF8则在这两个细胞器中都有定位[86].与PPR编辑因子不同,MORF9和MORF2的突变体都会影响拟南芥叶绿体所有RNA位点的编辑.因此认为NDHD-1基因位点的编辑至少需要4种蛋白:CRR4、DYW1、MORF2和MORF9[60,83,87].定位在线粒体中的MORF蛋白也参与许多RNA位点的编辑,同时MORF蛋白也会与其他RNA编辑蛋白相互作用[88].这些非PPR的编辑因子与PPR蛋白共同组成更高级别的编辑复合体对RNA进行编辑.既然是定位在叶绿体中的PPR蛋白,另一方面也许会与光合作用相关,可能会影响到叶绿体的发育,从而影响植物整个的生长周期,引起植物早衰或者延缓衰老等[30].如水稻的WSL4蛋白主要对叶绿体第二类内含子进行剪接,并且在植株早期的生长过程中会影响叶绿体的发育,引起白斑[81].研究还发现,拟南芥基因组编码的定位于叶绿体中的PPR蛋白与莲座叶片的衰老调控之间存在一定的负相关性[6].目前对PPR在叶绿体中起作用的很多具体机制尚未明确,还需要进一步的深入研究.
5 展望
植物中的叶绿体和线粒体都是携带遗传信息的细胞器,它们的功能同时受到细胞器基因组和核基因组的共同调控.PPR蛋白是核基因组编码的一类含三角状五肽重复序列结构域且成员数目庞大的蛋白质家族,参与细胞器RNA的转录和加工,包括RNA的编辑和剪接等.这些PPR蛋白会持续影响叶绿体和线粒体基因组的表达和功能,然后影响植物的生长发育[21,32,70].
目前已知陆生植物中含有的PPR蛋白的数量都比较庞大[14,16-17],已被证实功能的仅是很小的一部分,未来如何在植物中进一步验证这些数量庞大的PPR蛋白的功能将是一个很大的挑战.虽然已经对PPR蛋白家族的分类、结构和定位等有了较深入的了解,甚至有些PPR蛋白的功能也得到了证实,但对于整个PPR蛋白家族的功能和作用机制还需进一步深入.如虽然已经明确了PPR蛋白的种类和结构,但植物与非植物之间PPR蛋白数量存在巨大差异的原因至今还不是很清楚,还需要进一步探讨.此外,虽然已经发现PPR蛋白对植物线粒体或叶绿体中的RNA加工修饰,但这些PPR是如何特异性识别并结合到RNA上,以及如何进一步对RNA进行剪接和编辑的机制还不太明确,需要进一步利用结构生物学等方法进行分析.除了PPR蛋白外,RNA的剪接和编辑还需要其他非PPR蛋白的参与,那么PPR与哪些非PPR蛋白互作以及如何互作等问题都需要解答.因此将来还需要从两方面进一步深入研究PPR蛋白.一方面,由于PPR家族成员数量庞大,需要进一步发现和证实其他PPR蛋白的存在,也需要尽可能多地对PPR蛋白进行进一步的分子功能和作用机制的研究,加强对该蛋白家族的认识;另一方面,明确该蛋白家族的功能与作用机制,以便更好地开发和利用该蛋白家族.
[1] 丁安明,屈旭,李凌,等.植物PPR蛋白家族研究进展[J].中国农学通报,2014,30(9):218-224.
[2] 丁安明,李凌,屈旭,等.番茄PPR基因家族的鉴定与生物信息学分析[J].遗传,2014,36(1):77-84.
[3] LURIN C. Genome-wide analysis ofArabidopsispentatricopeptide repeat proteins reveals their essential role in organelle biogenesis [J]. Plant Cell,2004,16(8):2 089-2 103.
[4] 李泉秀.PPR蛋白的结构生物学和生物化学研究[D].北京:清华大学,2015.
[5] LI Q, YAN C, XU H, et al. Examination of the dimerization states of the single-stranded RNA recognition protein pentatricopeptide repeat 10 (PPR10) [J]. Journal of Biological Chemistry, 2014,289(45):31 503-31 512.
[6] 朱正火,任育军,李燕云,等.拟南芥PPRs基因与莲座叶衰老之间的关系[J].福建农林大学学报(自然科学版),2016,45(3):282-289.
[7] BEICK S, SCHMITZ-LINNEWEBER C, WILLIAMS-CARRIER R, et al. The pentatricopeptide repeat protein PPR5 stabilizes a specific tRNA precursor in maize chloroplasts [J]. Molecular and Cellular Biology, 2008,28(17):5 337-5 347.
[8] TAN J, TAN Z, WU F, et al. A novel chloroplast-localized pentatricopeptide repeat protein involved in splicing affects chloroplast development and abiotic stress response in rice [J]. Molecular Plant, 2014,7(8):1 329-1 349.
[9] WANG G, ZHONG M, SHUAI B, et al. E+subgroup PPR protein defective kernel 36 is required for multiple mitochondrial transcripts editing and seed development in maize andArabidopsis[J]. New Phytologist, 2017,214(4):1 563-1 578.
[10] SAKAII A, TAKANO H, KUROIMA T. Organelle nuclei in higher plants: structure, composition, function, and evolution [J]. International Review of Cytology—A Survey of Cell Biology, 2004,238:59-118.
[11] ROBLES P, QUESADA V. Emerging roles of mitochondrial ribosomalp proteins in plant development [J]. International Journal of Molecular Sciences, 2017,18(12):2 595.
[13] RICHARDSON L G L, SINGHAL R, DJ S. The integration of chloroplast protein targeting with plant developmental and stress responses [J]. BMC Biology, 2017,15(1):118.
[14] O′TOOLE N, HATTORI M, ANDRES C, et al. On the expansion of the pentatricopeptide repeat gene family in plants [J]. Molecular Biology and Evolution, 2008,25(6):1 120-1 128.
[15] SMALL I, PEETERS N. The PPR motif-a TRR-related motif prelvalent in plant organellar proteins [J]. Trends in BioSchemical Sciences, 2000,25(2):45-47.
[16] IFUJII S, SMALL I. The evolution of RNA editing and pentatricopeptide repeat genes [J]. New Phytologist, 2011,191(1):37-47.
[17] RIVAL E. Formation of theArabidopsispentatricopeptide repeat family [J]. Plant Physiology, 2006,141(3):825-839.
[18] CHOUDHARY M, ZANHUA X, FU Y, et al. Genome analyses of three strains of rhodobacter sphaeroides: evidence of rapid evolution of chromosome Ⅱ [J]. Journal of Bacteriology, 2006,189(5):1 914-1 921.
[19] CAZALET C, GOME-VALERO L, RUSNIOK C, et al. Analysis of the legionella longbeachae genome and transcriptome uncovers unique strategies to cause legionnaires′ disease [J]. Plos Genet, 2010,6(2):e1000851.
[20] SCHALLENBERG RUDINGER A M, LENZ H, POLSAKIEWIEZ M, et al. A survey of PPR proteins identifies DYW domains like those of land plant RNA editing factors in diverse eukaryotes [J]. RNA Biology, 2014,10(9):1 549-1 556.
[21] BARKAN A, SMALL I. Pentatricopeptide repeat proteins in plants [J]. Annual Review of Plant Biology, 2014,65(1):415-442.
[22] SAHA D, PRASAD A M, SRINIVASAN R. Pentatricopeptide repeat proteins and their emerging roles in plants [J]. Plant Physiology and Biochemistry, 2007,45(8):521-534.
[23] SCHMITZ-LINNEWEBER C, WILLIAMS CARRIE R E, WILLIAMS-VOELER P M, et al. A pentatricopeptide repeat protein facilitates the trans-splicing of the maize chloroplast rps12 pre-mRNA [J]. Plant Cell, 2006,18(10):2 650-2 663.
[24] RUDINGER M, POLSAKIEWICZ M, KNOOP V. Organellar RNA editing and plant-specific extensions of pentatricopeptide repeat proteins in Jungermanniid but not in Marchantiid liverworts [J]. Molecular Biology and Evolution, 2008,25(7):1 405-1 414.
[25] SALONE V, RUDINGER M, POLSAKIEWICZ M, et al. A hypothesis on the identification of the editing enzyme in plant organelles [J]. FEBS Letters, 2007,581(22):4 132-4 138.
[26] SHIKANAI T. RNA editing in plant organelles: machinery, physiological function and evolution [J]. Cellular and Molecular Life Sciences, 2006,63(6):698-708.
[27] YIN P, LI Q, YAN C, et al. Structural basis for the modular recognition of single-stranded RNA by PPR proteins [J]. Nature, 2013,504:168-171.
[28] EMANUELSSON O, NIELSEN H, VON H. Predicting subcellular localization of proteins based on their N-terminal amino acid sequence [J]. Journal of Molecular Biology, 2000,300(4):1 005-1 016.
[29] HAILI N, PLANCHARD N, ARNAL N, et al. The MTL1 pentatricopeptide repeat protein is required for both translation and splicing of the mitochondrial NADH dehydrogenase subunit7 mRNA inArabidopsis[J]. Plant Physiology, 2015,170(1):354-366.
[30] CAO Z, YU Q, SUN Y, et al. A point mutation in the pentatricopeptide repeat motif of the AtECB2 protein causes delayed chloroplast development [J]. Journal of Integrative Plant Biology, 2011,53(4):258-269.
[31] DING Y, LIU N, TANG Z, et al.Arabidopsisglutamine-rich protein23 is essential for early embryogenesis and encodes a novel nuclear PPR motif protein that interacts with RNA polymerase Ⅱ subunit Ⅲ [J]. Plant Cell, 2006,18(4):815-830.
[32] HAMMANI K, GOBERT A, HLEIBIEH K, et al. AnArabidopsisdual-localized pentatricopeptide repeat protein interacts with nuclear proteins involved in gene expression regulation [J]. Plant Cell, 2012,23(2):730-740.
[33] CHATEIGNER-BOUTIN A L, FRANCS-SMALL C C D, DELANNOY E, et al. OTP70 is a pentatricopeptide repeat protein of the E subgroup involved in splicing of the plastid transcript rpoC1 [J]. The Plant Journal, 2011,65(4):532-542.
[34] WEIBENBERGER S, SOLL J, CARRIE C. The PPR protein SLOW GROWTH 4 is involved in editing of nad4 and affects the splicing of nad2 intron 1 [J]. Plant Molecular Biology, 2017,93(4-5):355-368.
[35] LEE K, HAN J H, PARK Y, et al. The mitochondrial pentatricopeptide repeat protein PPR19 is involved in the stabilization of NADH dehydrogenase 1 transcripts and is crucial for mitochondrial function andArabidopsisthalianadevelopment [J]. New Phytologist, 2017,215(1):202-216.
[36] MARIENFELD J, UNSELD M, BRENNICKE A. The mitochondrial genome ofArabidopsisis composed of both native and immigrant information [J]. Trends in Plant Science, 1999,4(12):495-502.
[37] HANDA H. The complete nucleotide sequence and RNA editing content of the mitochondrial genome of rapeseed (BrassicanapusL.) comparative analysis of the mitochondrial genomes of rapeseed andArabidopsisthaliana[J]. Nucleic Acids Research, 2003,31(20):5 907-5 916.
[38] HAZLE T, BONEN L. Comparative analysis of sequences preceding protein-coding mitochondrial genes in flowering plants [J]. Molecular Biology and Evolution, 2007,24(5):1 101-1 112.
[39] KUBO T, NISHIZAWA S, SUGAWARA A. The complete nucleotide sequence of the mitochondrial genome of sugar beet (BetavulgarisL.) reveals a novel gene for tRNA (Cys) (GCA) [J]. Nucleic Acids Research, 2000,28(13):2 571-2 576.
[40] NOTSU Y, MASOOD S, NISHIKAWA T, et al. The complete sequence of the rice (OryzasativaL.) mitochondrial genome: frequent DNA sequence acquisition and loss during the evolution of flowering plants [J]. Molecular Genetics and Genomics, 2002,268(4):434-445.
[41] 李玉秋,赵洪锟,谭化,等.八种植物线粒体基因组结构特征分析与比较[J].生物技术通报,2011(10):156-162.
[42] ANDERSSON S, KARLBERG O, CANBACK B, et al. On the origin of mitochondria: a genomics perspective [J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2003,358:165-179.
[43] MILLAR A, WHELAN J, SMALL I. Recent surprises in protein targeting to mitochondria and plastids [J]. Current Opinion in Plant Biology, 2006,9(6):610-615.
[44] MILLARA A H, HEAZLEWOOD J L, KRISTENSEN B K, et al. The plant mitochondrial proteome [J]. Trends in Plant Science, 2005,10(1):36-43.
[45] HAMMANI K, GIEGE P. RNA metabolism in plant mitochondria [J]. Trends in Plant Science, 2014,19(6):380-389.
[46] NICHOLLS T J, RORBACH J, MINCZUK M. Mitochondria: mitochondrial RNA metabolism and human disease [J]. The International Journal of Biochemistry & Cell Biology, 2013,45(4):845-849.
[47] BONEN L. Cis-and trans-splicing of group Ⅱ introns in plant mitochondria [J]. Mitochondrion, 2008,8(1):26-34.
[48] LONGEVIALLE A F D, SMALL I D, LURIN C. Nuclearly encoded splicing factors implicated in RNA splicing in higher plant organelles [J]. Molecular Plant, 2010,3(4):691-705.
[49] BROWN G G, FRANCS SMALL D F C, OSTERSETZER-BIRAN O. Group Ⅱ intron splicing factors in plant mitochondria [J]. Frontiers in Plant Science, 2014,5:35.
[50] CHENG S, GUTMANN B, ZHONG X, et al. Redefining the structural motifs that determine RNA binding and RNA editing by pentatricopeptide repeat proteins in land plants [J]. The Plant Journal, 2016,85(4):532-547.
[51] SCHMITZLINNEWEBER C, SMALL I. Pentatricopeptide repeat proteins: a socket set for organelle gene expression [J]. Trends in Plant Science, 2008,13(12):663-670.
[52] LONGEVIALLE A, MEYER E, ANDRES C, et al. The pentatricopeptide repeat gene OTP43 is required for trans-splicing of the mitochondrial nad1 intron1 inArabidopsisthaliana[J]. Plant Cell, 2007,19(10):3 256-3 265.
[53] LIU Y, HE J, CHEN Z, et al. ABA overly-sensitive 5 (ABO5), encoding a pentatricopeptide repeat protein required for cis-splicing of mitochondrial nad2 intron 3, is involved in the abscisic acid response inArabidopsis[J]. The Plant Journal, 2010,63(5):749-765.
[54] COLAS D F C, FALCON D L A, LI Y, et al. The pentatricopeptide repeat proteins TANG2 and ORGANELLE TRANSCRIPT PROCESSING439 are involved in the splicing of the multipartite nad5 transcript encoding a subunit of mitochondrial complex I [J]. Plant Physiology, 2014,165(4):1 409-1 416.
[55] KOPRIVOVA A, FRANCS-SMALL C C D , GALDER G, et al. Identification of a pentatricopeptide repeat protein implicated in splicing of intron 1 of mitochondrial nad7 transcripts [J]. Journal of Biological Chemistry, 2010,285(42):32 192-32 199.
[56] HSIEH W, LIAO J, CHANG C, et al. The SLOW GROWTH3 pentatricopeptide repeat protein is required for the splicing of mitochondrial NADH dehydrogenase subunit7 intron 2 inArabidopsis[J]. Plant Physiology, 2015,168(2):490-501.
[57] HAILI N, ARNAL N, QUADRADO M, et al. The pentatricopeptide repeat MTSF1 protein stabilizes the nad4 mRNA inArabidopsismitochondria [J]. Nucleic Acids Research, 2013,41(13):6 650-6 663.
[58] DONIWA Y, UEDA M, UETA M, et al. The involvement of a PPR protein of the P subfamily in partial RNA editing of anArabidopsismitochondrial transcript [J]. Gene, 2010,454(1-2):39-46.
[59]CHATEIGNER-BOUTIN A L, FRANCS-SMALL C C D, FUJII S, et al. The E domains of pentatricopeptide repeat proteins from different organelles are not functionally equivalent for RNA editing [J]. The Plant Journal, 2013,74(6):935-945.
[60] BOUSSARDON C, AVON A, KINDGREN P, et al. The cytidine deaminase signature HxE(x)n CxxC of DYW1 binds zinc and is necessary for RNA editing of ndhD-1 [J]. New Phytologist, 2014,203(4):1 090-1 095.
[61] BREHME N, BAYER-CSASZAR E, GLASS F, et al. The DYW subgroup PPR protein MEF35 targets RNA editing sites in the mitochondrial rpl16,nad4 and cob mRNAs inArabidopsisthaliana[J]. Plos One, 2015,10(10):e140680.
[62] LIU Z, DONG F, WANGX, et al. A pentatricopeptide repeat protein restores nap cytoplasmic male sterility inBrassicanapus[J]. Journal of Experimental Botany, 2017,68(15):4 115-4 123.
[63] SATO S, NAKAMURA Y, KANEKO T. Complete structure of the chloroplast genome ofArabidopsisthaliana[J]. DNA Research, 1999,6(5):283-290.
[64] ZHANG H, CUI Y, HUANG C, et al. PPR protein PDM1/SEL1 is involved in RNA editing and splicing of plastid genes inArabidopsisthaliana[J]. Photosynthesis Research, 2015,126(2-3):311-321.
[65] WU W, LIU S, RUWE H, et al. SOT1, a pentatricopeptide repeat protein with a small MutS-related domain, is required for correct processing of plastid 23S-4.5S rRNA precursors inArabidopsisthaliana[J]. The Plant Journal, 2016,85(5):607-621.
[66] HEIN A, POLSAKIEWICZ M, KNOOP V. Frequent chloroplast RNA editing in early-branching flowering plants: pilot studies on angiosperm-wide coexistence of editing sites and their nuclear specificity factors [J]. BMC Evolutionary Biology, 2016,16(1):23.
[67]RAMOS VEGA M, GUEVARA-GARCIA A, LIAMAS E, et al. Functional analysis of theArabidopsisthalianaCHLOROPLAST BIOGENESIS 19 pentatricopeptide repeat editing protein [J]. New Phytologist, 2015,208(2):430-441.
[68] DE LONGEVIALLE A F, HENDRICKSON L, TAYLOR N L, et al. The pentatricopeptide repeat gene OTP51 with two LAGLIDADG motifs is required for the cis-splicing of plastid ycf3 intron2 inArabidopsisthaliana[J]. The Plant Journal, 2008,56(1):157-168.
[69] NAKAMURA T, MEIERHOFF K, WESTHOFF P, et al. RNA-binding properties of HCF152, anArabidopsisPPR protein involved in the processing of chloroplast RNA [J]. European Journal of Biochemistry, 2003,270(20):4 070-4 081.
[70] ANDRES-COLAS N, ZHU Q, TAKENAKA M, et al. Multiple PPR protein interactions are involved in the RNA editing system inArabidopsismitochondria and plastids [J]. Proceedings of the National Academy of Sciences, 2017,114(33):8 883-8 888.
[71] MIZUKI T, ZEHRMANN A, VERBITSKIY D, et al. Multiple organellar RNA editing factor (MORF) family proteins are required for RNA editing in mitochondria and plastids of plants [J]. Proceedings of the National Academy of Sciences, 2012,109(13):5 104-5 109.
[72] 姜上川.ABA信号转导关键调节子SOAR1调控拟南芥发育与抗逆反应[D].北京:清华大学,2015.
[73] KLEFFMANN T, RUSSENBERGER D, VON ZYCHLINSKI A, et al. TheArabidopsisthalianachloroplast proteome reveals pathway abundance and novel protein functions [J]. Current Biology, 2004,14(5):354-362.
[74] BARKAN A, WALKER M, NOLASCO M. A nuclear mutation in maize blocks the processing and translation of several chloroplast mRNA and provides evidence for the differential translation of alternative mRNA forms [J]. Embo Journal, 1994,13:3 170-3 181.
[75] ZHOU W, KARCHER D, FISCHER A, et al. Multiple RNA processing defects and impaired chloroplast function in plants deficient in the organellar protein-only RNase P enzyme [J]. PLoS ONE, 2015,10(3):e120533.
[76] ICHINOSE M, SUGITA M. RNA editing and its molecular mechanism in plant organelles [J]. Genes, 2017,8(1):5.
[77] VOGEL J, BORNER T, HESS W R. Comparative analysis of splicing of the complete set of chloroplast group Ⅱ introns in three higher plant mutants [J]. Nucleic Acids Research, 1999,27(19):3 866-3 874.
[78] BARKAN A, KLIPCAN L, OSTERSETZER O, et al. The CRM domain: an RNA binding module derived from an ancient ribosome-associated protein [J]. RNA, 2006,13(1):55-64.
[79] RUWE H, WANG G, GUSEWSKI S, et al. Systematic analysis of plant mitochondrial and chloroplast small RNAs suggests organelle-specific mRNA stabilization mechanisms [J]. Nucleic Acids Research, 2016,44(15):7 406-7 417.
[80] KHROUCHTCHOVA A, MONDE R A, BARKAN A. A short PPR protein required for the splicing of specific group Ⅱ introns in angiosperm chloroplasts [J]. RNA, 2012,18(6):1 197-1 209.
[81] WANG Y, REN Y, ZHOU K, et al. WHITE STRIPE LEAF4 encodes a novel P-Type PPR protein required for chloroplast biogenesis during early leaf development [J]. Frontiers in Plant Science, 2017,8:1 116.
[82] HUANG C, YU Q, LI Z, et al. Porphobilinogen deaminase HEMC interacts with the PPR-protein AtECB2 for chloroplast RNA editing [J]. The Plant Journal, 2017,92(4):546-556.
[83] YAN J, ZHANG Q, GUAN Z, et al. MORF9 increases the RNA-binding activity of PLS-type pentatricopeptide repeat protein in plastid RNA editing [J]. Nature Plants, 2017,3:17 037.
[84] OKUDA K, SHOKI H, ARAI M, et al. Quantitative analysis of motifs contributing to the interaction between PLS-subfamily members and their target RNA sequences in plastid RNA editing [J]. The Plant Journal, 2014,80(5):870-882.
[85] YAGI Y, TACHIKAWA M, NOGUCHI H, et al. Pentatricopeptide repeat proteins involved in plant organellar RNA editing [J]. RNA Biology, 2014,10(9):1 419-1 425.
[86] HAAG S, SCHINDLER M, BERNDT L, et al. Crystal structures of theArabidopsisthalianaorganellar RNA editing factors MORF1 and MORF9 [J]. Nucleic Acids Research, 2017,45(8):4 915-4 928.
[87] DU L, ZHANG J, QU S, et al. The pentratricopeptide repeat protein pigment-defective mutant2 is involved in the regulation of chloroplast development and chloroplast gene expression inArabidopsis[J]. Plant and Cell Physiology, 2017,58(4):747-759.
[88] BAYER-CSASZAR E, HAAG S, JORG A, et al. The conserved domain in MORF proteins has distinct affinities to the PPR and E elements in PPR RNA editing factors [J]. Biochimica et Biophysica Acta (BBA)-Gene Regulatory Mechanisms, 2017,1860(8):813-828.

