利用CRISPR/Cas9系统定向编辑水稻SD1基因
胡雪娇 杨佳 程灿 周继华 牛付安 王新其 张美良 曹黎明,* 储黄伟,*
农作物的驯化是一个野生植物品种不断变异加上人工选择的过程[1]。到了近代,人们利用物理、化学等手段诱导农作物的遗传物质发生变异,再从诱变群体中选择符合人们某种要求的突变个体,进而培育成新的品种。在过去几十年里,用这种诱变育种方法获得了大量有价值的种质资源[2]。然而,诱变育种存在着变异的位点随机,有益突变频率较低以及变异方向和性质难以控制等问题。随着功能基因组学以及基因编辑技术的发展,使得定向编辑特定靶基因,从而快速获得具有预期性状的农作物新品种这一目标成为可能。CRISPR/Cas9是2013年开始发展起来的一种基因编辑技术[3-6],与锌指核酸内切酶(zinc finger endonucleases,ZFNs)[7]和转录激活因子样效应物核酸酶(transcription activatorlike effector nucleases,TALENs)[8]等基因编辑技术相比,CRISPR/Cas9技术由于设计使用简单、灵活,费用较低等优点,成为最受关注、使用最为广泛的基因编辑技术[9]。利用CRISPR/Cas9技术已经在水稻[10-12]、玉米[13]、小麦[11,14]、高粱[12]、大豆[15-17]、番茄[18]等作物中实现了基因定点编辑。最近的研究发现使用CRISPR/Cas9技术在转基因水稻的T0代植株中,就可以获得纯合的靶基因突变植株,并且编辑后的基因可以稳定遗传到下一代[19]。
水稻SD1基因,被誉为“绿色革命基因”,编码赤霉素合成途径中一个关键的酶——赤霉素20-氧化酶,功能缺失突变的等位基因sd1会使得水稻植株获得半矮秆的表型,并显著提高产量[20-22]。我国水稻矮秆育种中普遍利用的籼稻矮源矮仔占、矮脚南特和低脚乌尖等品种,其矮生性均由sd1控制[23]。例如,低脚乌尖的SD1基因发生了382 bp的缺失[20]。20世纪50年代至60年代初开始的矮秆育种主要是在籼稻品种中进行的,中国选育的代表性矮秆品种矮脚南特和矮仔占,以及随后菲律宾、韩国和美国等国相继育成的IR8、统一和Calrose 76等矮秆高产品种都是籼稻品种[23]。Asano等[24]通过比较半矮秆的粳稻日本晴和高秆的籼稻Kasalath发现,日本晴的SD1基因中存在两个SNP位点,导致SD1编码的赤霉素20-氧化酶在第100和340位的两个氨基酸分别为谷氨酸和谷氨酰胺,而Kasalath的SD1蛋白在第100和340位氨基酸分别是甘氨酸和精氨酸,并且这两个功能性的SNP位点在所有粳稻品种中都存在,而几乎所有的籼稻地方品种和野生稻的SD1序列都与Kasalath的SD1一致。因此,人类在驯化粳稻品种的过程中,已经将突变的矮秆基因sd1筛选并固定下来。
上海市农业科学院在利用“籼粳架桥”技术[25]创制恢复系的过程中,获得一些各方面性状优良,但株型偏高的恢复系。申繁17和申繁24从以粳稻恢复系ZR213为母本、CR-3为父本的重组自交系后代中选育而来。ZR213、CR-3以及申繁17和申繁24的株高都相对偏高。本研究以申繁17和申繁24为材料,经测序发现这两个品种的SD1基因序列和籼稻的SD1等位基因一致,用CRISPR/Cas9技术对这两个品种的SD1基因进行基因编辑,以期快速获得不含转基因序列的矮秆恢复系材料。
1 材料与方法
1.1 植物材料、载体和菌株
转基因受体材料为上海市农业科学院作物育种栽培研究所选育的粳稻恢复系申繁17和申繁24。本研究所用的CRISPR/Cas9中间载体psgR-Cas9-Os由中国科学院上海植物逆境生物学研究中心朱健康教授提供[10]。大肠杆菌Top10、农杆菌EHA105和pCAMBIA1300载体由本实验室保存。
1.2 主要试剂
限制性内切酶BbsⅠ、T4 DNA连接酶购自NEB公司;Taq酶、氨苄青霉素、卡那霉素、LB培养基等其他试剂购自上海生工生物工程有限公司。
1.3 引物
本研究所用的引物序列见表1。引物由上海生工生物工程有限公司合成。
1.4 申繁17和申繁24的SD1基因型检测
以申繁17和申繁24的基因组DNA为模板,以引物EXON1-F和EXON1-R扩增SD1第1外显子,以引物EXON3-F和EXON3-R扩增SD1第3外显子,然后将PCR扩增产物测序,测序结果与日本晴和Kasalath的SD1序列进行比对。
1.5 CRISPR/Cas9靶位点选择
使用 CRISPR Primer Designer软件[25]进行CRISPR/Cas9靶位点选择。然后通过水稻基因组BLAST分析,确保靶位点的特异性。
1.6 CRISPR/Cas9表达载体的构建
根据靶位点的序列,合成两条引物Oligo-F和Oligo-R,每条引物(10µmol/L)各取1µL,加入8µL退火缓冲液(TE+50 mmol/L NaCl),以0.1℃/s的速率从95℃降到16℃,获得靶位点接头。中间载体psgR-Cas9-Os用BbsⅠ酶切,回收载体片段,然后以1∶3的摩尔数比与靶位点接头混合,用T4连接酶链接,转化大肠杆菌Top10,用引物为M13-F和Oligo-R进行菌落PCR鉴定,并测序验证。获得的中间载体psgR-Cas9-Os-SD1用EcoRⅠ和HindⅢ双酶切后,回收大约5.6 kb长的片段,并用EcoRⅠ和HindⅢ双酶切载体pCAMBIA1300,回收载体片段,将回收的片段与载体连接,转化大肠杆菌Top10,同样用引物M13-F和Oligo-R进行菌落PCR鉴定,并测序验证,获得双元载体CRISPR-SD1。

表1 本研究所用的引物Table 1.Primers used in this research.
1.7 阳性转基因植株的获得及验证
获得的载体CRISPR-SD1用农杆菌EHA105介导的方法转化申繁17和申繁24的愈伤组织,用潮霉素筛选获得T0代植株。取叶片提取基因组DNA,用CRISPR-SD1载体特异性引物M13-F和Oligo-R进行PCR扩增,扩增产物的大小为253 bp,能扩增出目的片段的植株为转基因阳性植株。
1.8 靶位点序列的检测
取叶片提取基因组DNA,以引物SEQ1-F和SEQ1-R进行PCR扩增,扩增产物连入pMD18-T载体,转化大肠杆菌Top10。随机挑取10个阳性克隆进行测序,测序结果与野生型的SD1基因编码区序列比对,如果10个克隆的序列全部是突变的,则为纯合突变体,如果10个克隆的序列既有突变的,也有野生型的,则为杂合子。
2 结果与分析
2.1 申繁17和申繁24的SD1基因序列的分析及靶位点设计
为了分析ZR213、CR-3、申繁17和申繁24中SD1基因的基因型,对其SD1基因的第1和第3外显子进行测序,发现ZR213、CR-3、申繁17和申繁24中SD1基因型与籼稻Kasalath的SD1等位基因型是一致的(图1)。表明在“籼粳架桥”创制恢复系的过程中,将籼稻的SD1等位基因导入到了申繁17和申繁24中,导致这两个品种株高过高。
为了构建编辑SD1基因的CRISPR/Cas9表达载体,将SD1基因cDNA序列输入CRISPR Primer Designer软件,找到了7个CRISPR/Cas9靶位点(表2),用BLAST分析这些靶位点序列在水稻基因组中的特异性,最后选用靶位点1(Target 1)作为SD1基因编辑的靶位点。
2.2 CRISPR/Cas9基因编辑载体构建
根据靶位点的序列,合成两条引物Oligo-F和Oligo-R(表1)。将Oligo-F和Oligo-R混合复性后获得的靶位点片段与用BbsⅠ酶切后的psgR-Cas9-Os载体连接。连接产物转化大肠杆菌后进行菌落PCR鉴定,鉴定所用引物为M13-F和Oligo-R,在阳性克隆中可以鉴定出253 bp的条带。共鉴定了8个克隆,其中有5个克隆正确(图2),选取1号克隆进行测序验证,并将最终获得的载体命名为psgR-Cas9-Os-SD1。
将载体psgR-Cas9-Os-SD1用EcoRⅠ和HindⅢ双酶切,回收5.6 kb的片段,然后与经EcoRⅠ和HindⅢ双酶切回收的pCAMBIA1300载体连接,转化大肠杆菌后,再次用引物M13-F和Oligo-R进行菌落PCR鉴定,共鉴定了8个克隆,其中7个克隆为阳性克隆(图2),最后选取1号克隆进行测序验证正确,将获得的CRISPR/Cas9基因编辑载体命名为CRISPR-SD1。

图1 水稻SD1氨基酸序列的比对Fig.1.Comparison of rice SD1 amino acid sequence.

表2 水稻SD1基因中的CRISPR/Cas9靶位点Table 2.Putative CRISPR/Cas9 target sites in rice SD1 gene.
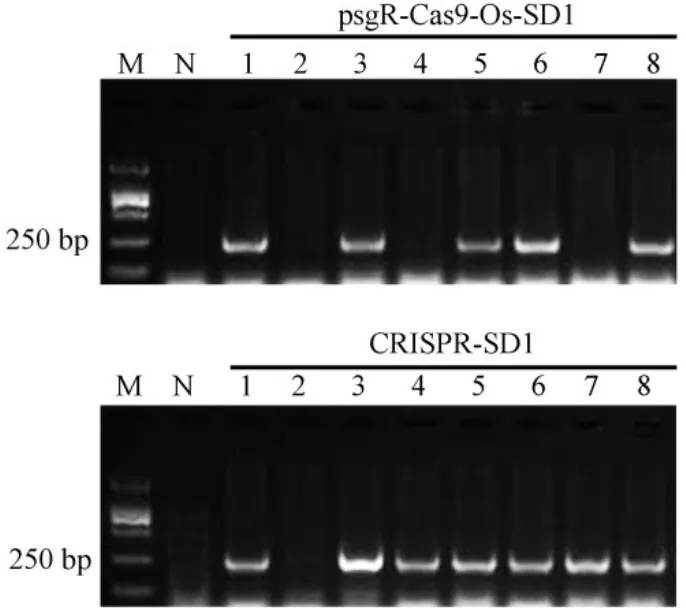
图2 阳性克隆的菌落PCR筛选Fig.2.Screening positive clone by colony PCR.
2.3 农杆菌介导的遗传转化及转基因植株靶位点突变类型分析
CRISPR-SD1载体用农杆菌介导的方法转化水稻恢复系申繁17和申繁24的愈伤组织,用潮霉素筛选获得转基因苗。申繁17和申繁24分别获得了10株和18株T0代转基因苗。用载体特异的引物M13-F和Oligo-R进行PCR鉴定,在10株申繁17的转基因苗中,有3株阳性苗,而在18株申繁24的转基因苗中,有5株阳性苗。
为了分析这8株阳性转基因苗中的SD1靶位点序列是否已经被编辑,用引物SEQ1-F和SEQ1-R在转基因苗中扩增包含SD1基因靶位点的DNA片段,进行测序。结果发现在这8株转基因苗中,靶位点序列都发生了不同类型的突变。其中有4个株系插入1个碱基,还有另外4个株系发生了不同数目碱基的缺失突变(图3)。

图3 T0代转基因株系中SD1基因突变类型的分析Fig.3.Mutation type in SD1 gene of T0transgenic lines.
在8个突变株系中除了繁17-1和繁24-8两个株系的突变分别导致缺失5和23个氨基酸外,其他的突变株系的突变都导致了移码突变使得翻译提前终止。在T1突变体中发现繁17-1和繁24-8的株高与其野生型相比没有显著的变化。而其他突变株系的株高与野生型相比显著降低,且同一个品种不同突变株系之间株高的差异不显著。
2.4 不含转基因的矮秆突变体的获得
为了获得不含转基因(T-DNA)序列的矮秆申繁17和申繁14株系,选取2个纯合的转基因株系繁17-4和繁24-5开展进一步研究。这2个转基因株系分别种植24株T1代植株,在苗期分别取单株叶片,提取DNA,用转基因载体的特异性引物M13-F和Oligo-R,以及潮霉素和Cas9基因特异的引物进行PCR鉴定,在繁17-4和繁24-5的T1代群体中,分别鉴定出了10株和8株不含转基因载体序列的单株(图4)。同时,对不含转基因载体序列单株的靶基因位点进行测序,结果显示T1代中的靶位点突变和上一代是一致的,说明用CRISPR/Cas9基因编辑获得的突变是可以稳定遗传的。在此,分别将获得的不含转基因载体片段的繁17-4和繁24-5株系命名为矮繁17和矮繁24。在T2代,测量了矮繁17和矮繁24株高、有效穗数、穗长、千粒重、每穗粒数和结实率等农艺性状,发现与它们各自的野生型申繁17和申繁24相比,除了株高出现显著下降外(下降约25%),而其他农艺性状均没有出现显著的改变(图 5)。

图4.T1代中不含转基因载体序列植株的PCR筛选Fig.4.PCR screening of transgenic-free plants in T1generation.

图5 野生型与T2代sd1突变体的农艺性状(n=20)Fig.5.Agronomic traits of wild type and T2-generation sd1 mutants(n=20).
3 讨论
水稻矮秆品种的选育和应用是水稻育种领域的一个重大的突破[23],过去一般利用与矮源品种进行杂交,并结合表型或分子标记辅助选择的方法进行矮秆品种选育,费时费力。现代分子生物技术育种的兴起为矮秆育种提供了一些比较快捷高效的手段,可以直接抑制或者敲除矮秆基因,而不改变基因组中其他基因的序列和表达水平,从而可以获得预期的株高表型,而对其他的性状不会有影响。Qiao等[26]利用RNAi技术抑制半矮秆基因SD1的表达,获得了半矮秆的水稻品种。然而RNAi技术本身存在一些缺点,限制了RNAi技术在作物分子育种领域的应用。比如RNAi技术常常存在脱靶的问题,一些和靶基因序列相近的基因表达也会受到抑制;RNAi技术通过降解靶基因的mRNA进行基因干涉,降低其表达水平,而非完全敲除靶基因的功能[27]。基因编辑技术,特别是CRISPR/Cas9技术的发展,使得RNAi技术在功能基因组学和作物分子育种领域的应用正迅速地被取代。
Zhang等[19]发现利用CRISPR/Cas9技术对水稻进行基因编辑,在T0代转基因植株中即可获得纯合的基因突变植株,并且这些突变可以稳定遗传给下一代。由于转基因过程中,如果转基因是单拷贝插入的情况下,根据孟德尔遗传定律,在T1转基因的群体中,就会出现1/4的植株不含转基因序列。因此,从理论上讲用CRISPR/Cas9技术,在第2代就可以获得不含转基因的突变体。
本研究利用CRISPR/Cas9技术对粳稻恢复系申繁17和申繁24进行SD1基因编辑,发现在获得的8个转基因株系中,有5个株系在T0代就获得了纯合的sd1突变。选取其中2个纯合的转基因株系繁17-4和繁24-5分别种植成T1群体,筛选出了不含转基因序列的个体,并且这些个体中SD1的突变方式与T0一致,说明CRISPR/Cas9技术编辑后的突变位点可以稳定地遗传给下一代。株高测定结果表明,CRISPR/Cas9技术编辑后获得的半矮秆品种的株高与其野生型相比大约降低25%。本研究结果表明CRISPR/Cas9技术是一种非常有效的水稻矮秆育种工具。
[1]Doebley J F,Gaut B S,Smith B D.The molecular genetics of crop domestication.Cell,2006,127(7):1309-1321.
[2]Liu Q,Xu X,Ren X,Fu H,Wu D,Shu Q.Generation and characterization of low phytic acid germplasm in rice(Oryza sativaL.).Theor Appl Genet,2007,114(5):803-814.
[3]Cho S W,Kim S,Kim J M,Kim J S.Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease.Nat Biotechnol,2013,31(3):230-232.
[4]Cong L,Ran F A,Cox D,Lin S,Barretto R,Habib N,Hsu P D,Wu X,Jiang W,Marraffini L A,Zhang F.Multiplex genome engineering using CRISPR/Cas systems.Science,2013,339(6121):819-23.
[5]Jinek M,East A,Cheng A,Lin S,Ma E,Doudna J.RNA-programmed genome editing in human cells.Elife,2013,2:e00471.
[6]Mali P,Aach J,Stranges P B,Esvelt K M,Moosburner M,Kosuri S,Yang L,Church G M.CAS9 transcriptional activators for target specificity screening and paired nickases forcooperative genome engineering.Nat Biotechnol,2013,31(9):833-838.
[7]Mani M,Kandavelou K,Dy F J,Durai S,Chandrasegaran S.Design,engineering,and characterization of zinc finger nucleases.Biochem Biophys Res Commun,2005,335(2):447-457.
[8]Bogdanove A J,Voytas D F.TAL effectors:Customizable proteins for DNA targeting.Science,2011,333(6051):1843-1846.
[9]Samanta M K,Dey A,Gayen S.CRISPR/Cas9:An advanced tool for editing plant genomes.Transgenic Res,2016,25(5):561-573.
[10]Mao Y,Zhang H,Xu N,Zhang B,Gou F,Zhu J K.Application of the CRISPR-Cas system for efficient genome engineering in plants.Mol Plant,2013,6(6):2008-2011.
[11]Shan Q,Wang Y,Li J,Zhang Y,Chen K,Liang Z,Zhang K,Liu J,Xi J J,Qiu J L,Gao C.Targeted genome modification of crop plants using a CRISPR-Cas system.Nat Biotechnol,2013,31(8):686-688.
[12]Jiang W Z,Zhou H B,Bi H H,Fromm M,Yang B,Weeks D P.Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification inArabidopsis,tobacco,sorghum and rice.Nucleic Acids Res,2013,41(20):e188.
[13]Liang Z,Zhang K,Chen K,Gao C.Targeted mutagenesis inZea maysusing TALENs and the CRISPR/Cas system.J Genet Genom,2014,41(2):63-68.
[14]Wang Y,Cheng X,Shan Q,Zhang Y,Liu J,Gao C,Qiu J L.Simultaneousediting of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew.Nat Biotechnol,2014,32(9):947-951.
[15]Jacobs T B,LaFayette P R,Schmitz R J,Parrott W A.Targeted genome modifications in soybean with CRISPR/Cas9.BMC Biotechnol,2015,15:16.
[16]Li Z,Liu Z B,Xing A,Moon B P,Koellhoffer J P,Huang L,Ward R T,Clifton E,Falco S C,Cigan A M.Cas9-Guide RNA directed genome editing in soybean.Plant Physiol,2015,169(2):960-970.
[17]Sun X,Hu Z,Chen R,Jiang Q,Song G,Zhang H,Xi Y.Targeted mutagenesis in soybean using the CRISPR-Cas9 system.Sci Rep,2015,5:10342.
[18]Brooks C,Nekrasov V,Lippman Z B,Van Eck J.Efficient gene editing in tomato in the first generation using the clustered regularly interspaced short palindromic repeats/CRISPR-associated9 system.Plant Physiol,2014,166(3):1292-1297.
[19]Zhang H,Zhang J,Wei P,Zhang B,Gou F,Feng Z,Mao Y,Yang L,Zhang H,Xu N,Zhu J K.The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation.Plant Biotechnol J,2014,12(6):797-807.
[20]Monna L,Kitazawa N,Yoshino R,Suzuki J,Masuda H,Maehara Y,Tanji M,Sato M,Nasu S,Minobe Y.Positional cloning of rice semidwarfing gene,sd-1:Rice“Green Revolution Gene”encodes a mutant enzyme involved in gibberellin synthesis.DNA Res,2002,9(1):11-17.
[21]Spielmeyer W,Ellis M H,Chandler P M.Semidwarf(sd-1), “green revolution”rice,contains a defective gibberellin 20-oxidase gene.Proc Natl Acad Sci USA,2002,99(13):9043-9048.
[22]Sasaki A,Ashikari M,Ueguchi-Tanaka M,Itoh H,Nishimura A,Swapan D,Ishiyama K,Saito T,Kobayashi M,Khush G S,Kitano H,Matsuoka M.Green revolution:A mutant gibberellin-synthesis gene in rice.Nature,2002,416(6882):701-702.
[23]谷福林,翟虎渠,万建民,张红生.水稻矮秆性状研究及矮源育种利用.江苏农业学报,2003,19(1):48-54.Gu F L,Zhai H Q,Wan L M,Zhang H S.Study on Inheritance of dwarf character and its utilization in rice(Oryza sativaL.)breeding.Jiangsu J Agric Sci,2003,19(1):48-54.(in Chinese with English abstract)
[24]Asano K,Yamasaki M,Takuno S,Miura K,Katagiri S,Ito T,Doi K,Wu J,Ebana K,Matsumoto T,Innan H,Kitano H,Ashikari M,Matsuoka M.Artificial selection fora green revolution gene during japonica rice domestication.Proc Natl Acad Sci USA,2011,108(27):11034-11039.
[25]Yan M,Zhou S R,Xue H W.CRISPR Primer Designer:Design primers for knockout and chromosome imaging CRISPR-Cas system.J Integr Plant Biol,2015,57(7):613-617.
[26]Qiao F,Zhao K J.The Influence of RNAi targeting ofOsGA20ox2gene on plant height in rice.Plant Mol Biol Rep,2011,29(4):952.
[27]Harrison M M,Jenkins B V,O’Connor-Giles K M,Wildonger J.A CRISPR view of development.Genes Dev,2014,28(17):1859-1872.

