鸭源鸡杆菌flfA基因的克隆及功能区域分析
王坤芃,彭志锋,刘盼盼,王继洋,王 艳,王川庆,杨 霞*
(1.河南农业大学 牧医工程学院,河南 郑州 450002; 2.河南牧业经济学院 动物医学院,河南 郑州 450002)
鸭源鸡杆菌(Gallibacteriumanatis,G.anatis)是巴氏杆菌科鸡杆菌属的一个代表性菌种[1-2]。G.anatis为条件性致病菌,常寄生于鸡上呼吸道和下生殖道,主要引起鸡输卵管炎和腹膜炎等,导致蛋鸡产蛋数量和质量下降、死亡率增加,给养禽业带来巨大经济损失[3-6]。滥用抗生素使得G.anatis多重耐药严重[7-9],目前尚无有效预防措施。为了预防G.anatis感染,探究其致病机制及研发新型疫苗是当前的研究热点。
菌毛是细菌表面的一种毛发样的蛋白结构,其在细菌粘附宿主过程中发挥重要作用,而且还是多种细菌的重要毒力因子[10-11]。Bager等[12]研究发现,G.anatis12565/12缺失flfA基因后,对鸡只的致病力显著下降。G.anatis可粘附于鸡口咽原代上皮细胞[13-14],菌毛在其中发挥的作用还不得而知。探明菌毛的作用不仅对G.anatis致病机制的研究具有重要意义,而且可为新型疫苗的研发提供候选方向。为此,采用PCR方法检测了18株G.anatis的菌毛基因flfA,并对其进行克隆及功能区域分析,以期为G.anatis防控及新型疫苗的研发提供参考。
1 材料和方法
1.1 菌株
PDS-RZ-1-SLG等18株G.anatis由河南农业大学传染病教研室保存[3];pMD18-T载体购于宝生物工程(大连)有限公司。18株参试G.anatis信息具体见表1。
1.2 主要试剂
绵羊血琼脂平板购自郑州安图生物工程有限公司;蛋白胨、酵母提取物购自北京奥博星生物技术有限责任公司;DL2000 DNA Marker、TaqDNA聚合酶、限制性内切酶NcoⅠ和XhoⅠ、T4 DNA连接酶等购自宝生物工程(大连)有限公司;DNA胶回收试剂盒、DNA产物纯化试剂盒、质粒DNA小量抽提试剂盒购自生工生物工程(上海)股份有限公司。

表1 18株参试G.anatis菌株信息
1.3 引物设计
参考G.anatisUMN179的flfA基因序列,使用Primer Premier 5.0设计1对扩增flfA全基因的特异性引物,上游引物flfA-F:5′-TAATTCTGGAGGAGTATTTAAAAAATG-3′,下游引物flfA-R:5′-AATCTGCCCGATAAGTGGAAAAAG-3′,预期片段大小为620 bp左右。
1.4 菌株培养
取-70 ℃保存的18株G.anatis甘油菌分别划线接种于绵羊血琼脂平板,37 ℃恒温箱中培养18 h,分别挑单菌落接种于含5%血清的BHI液体培养基中,37 ℃、200 r/min过夜培养18 h。
1.5 细菌基因组提取
采用煮沸法粗提细菌基因组:分别取1 mL的供试菌株过夜培养物,8 000 r/min离心5 min;弃去上清液,用100 μL ddH2O重悬菌体,沸水煮10 min,再冰浴5 min,10 000 r/min离心5 min;取上清于新的EP管内,-20 ℃保存备用。
1.6 flfA基因的克隆及序列分析
PCR扩增采用50 μL反应体系。反应条件为:95 ℃ 5 min;94 ℃ 30 s,50 ℃ 30 s,72 ℃ 40 s,共35个循环;最后72 ℃延伸10 min。并将参考菌株UMN179扩增产物设置为阳性对照。PCR扩增产物用1%琼脂糖凝胶电泳检测。将胶回收纯化的PCR产物连接到pMD18-T载体上,再转化到DH5α感受态细胞,将PCR和双酶切鉴定为阳性的质粒进行测序鉴定。
将18株G.anatisflfA基因核苷酸序列及UMN179的flfA核苷酸序列进行比对,通过MEGA 5.2软件绘制系统发育进化树并对其进行遗传进化分析。
1.7 FlfA氨基酸序列同源性分析
从18株G.anatis中挑选ZZ-HL-1-SLG、YZ-11-XZ、SHAN-XY-4-SLG、ZZ-XY-1-SLG和PDS-RZ-1-SLG 5株进行FlfA氨基酸序列比对分析,使用MEGA 5.05软件对5株G.anatis进行遗传进化分析。
1.8 FlfA理化性质分析
使用在线软件Protparam(http://www.expasy.org/tools/protparam.html)分别分析FlfA的分子质量、等电点及氨基酸组成等。使用 Proscale(http://web.expasy.org/protscale/)在线软件分析FlfA的亲水性与疏水性。
1.9 FlfA蛋白B细胞抗原表位的预测
使用DNAStar软件中的MegAlign程序对flfA基因推导氨基酸序列进行同源性比对分析,使用Protean程序预测FlfA可能存在的线性B细胞优势抗原表位。
2 结果与分析
2.1 flfA基因PCR扩增结果
分别以18株G.anatis分离株的基因组为模板,扩增flfA全基因。扩增产物经1%凝胶电泳检测,在约620 bp处均有1条特异条带(图1)。测序结果显示,18株G.anatis均具有F17样菌毛结构蛋白编码基因。18株G.anatisflfA基因核苷酸序列登录号分别为KX268327.1—KX268344.1。
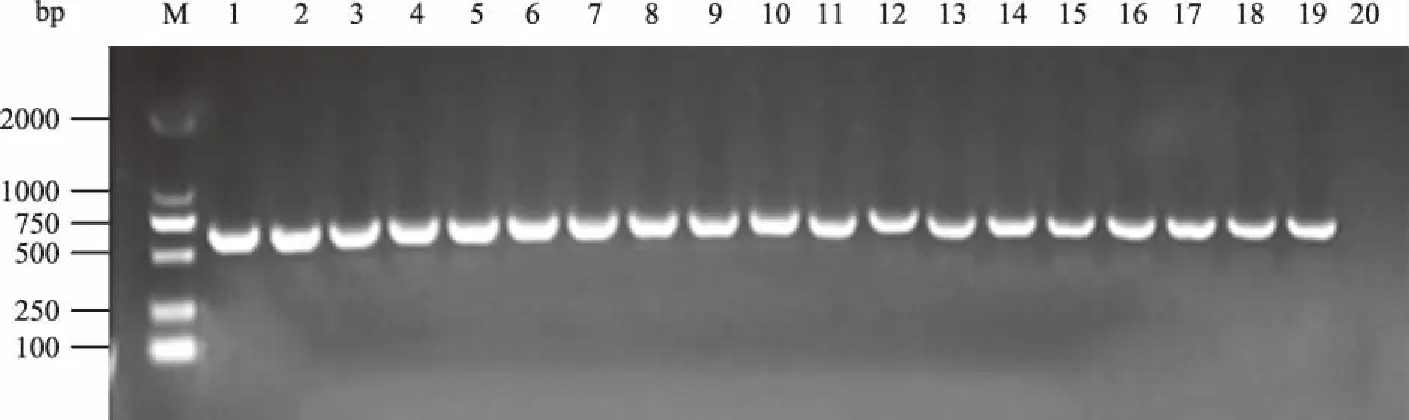
M:DL2000 DNA Marker;1—18:国内18株G.anatis分离株的扩增结果;19:阳性对照;20:空白对照
2.2 基于flfA核苷酸序列的G.anatis系统进化分析
基于flfA核苷酸序列,分析18株G.anatis分离株系统进化关系,并分析其核苷酸同源性。结果显示,18株G.anatis分离株flfA基因核苷酸同源性为70.2%~99.8%。不同地区的18株G.anatisflfA核苷酸序列与UMN179的核苷酸序列对比,同源性为34.6%~69.0%。从构建的进化树可以看出,18株国内分离株与参考菌株UMN179分布于3个基因进化群。第一大分支上有10株G.anatis,其中UMN179与SHAN-XY-1-QG在此分支上,其同源性为100%;第二大分支上只有SHAN-XY-4-SLG和ZZ-XY-1-SLG,两者同源性为100%;第三大分支包括PDS-RZ-1-SLG在内的7株G.anatis,其中LH-BJT-11-SLG、XX-HJ-4-SLG、YQ-2-5-SLG和PDS-RZ-1-SLG的同源性为100%(图2)。
2.3 FlfA氨基酸序列同源性分析
选取ZZ-HL-1-SLG、YZ-11-XZ、SHAN-XY-4-SLG、ZZ-XY-1-SLG和PDS-RZ-1-SLG等5株G.anatis分离株进行flfA编码氨基酸序列分析。5株国内分离株间氨基酸序列同源性为63.1%~98.8%,与参考株UMN179的FIfA氨基酸同源性为13.1%~58.6%。FlfA氨基酸存在6个高变区,分别是17、25—30、123、125、164—165位氨基酸及169—170位氨基酸(图3)。
2.4 FlfA的理化性质
通过使用在线软件对G.anatisPDS-RZ-1-SLG菌毛蛋白FIfA的理化性质进行分析,结果表明,FlfA由190个氨基酸残基组成,等电点为6.72,其理论分子质量为19.79 ku;FlfA由19种氨基酸组成,含量最高的氨基酸残基有A(14.2%)、T(13.7%)、L(10.0%),含量较低的氨基酸残基有R(1.1%)、C(1.1%)、H(1.1%)。FIfA理论半衰期为30 h,不稳定系数为12.42,属于稳定性蛋白;其主要疏水性部位是21—27、42—50、98—106、109—120、137—170位氨基酸,主要亲水性部位是1—20、32—37、92—98、121—129、172—181位氨基酸。
2.5 FlfA线性B细胞抗原表位预测
通过预测FlfA蛋白不同同系物的B细胞抗原表位可得,主要线性B细胞抗原表位有6~8个,分别位于23—35、50—60、65—75、85—95、100—110、115—125、140—160、175—185位氨基酸。这些抗原表位均处于较保守的区域(图4)。

▭:从flfA基因群的不同分支中选取的进行氨基酸序列比对和B细胞抗原表位预测的菌株序列图2 基于G.anatis flfA全基因序列的系统进化分析

图3 G.anatis FlfA氨基酸序列的6个高变区
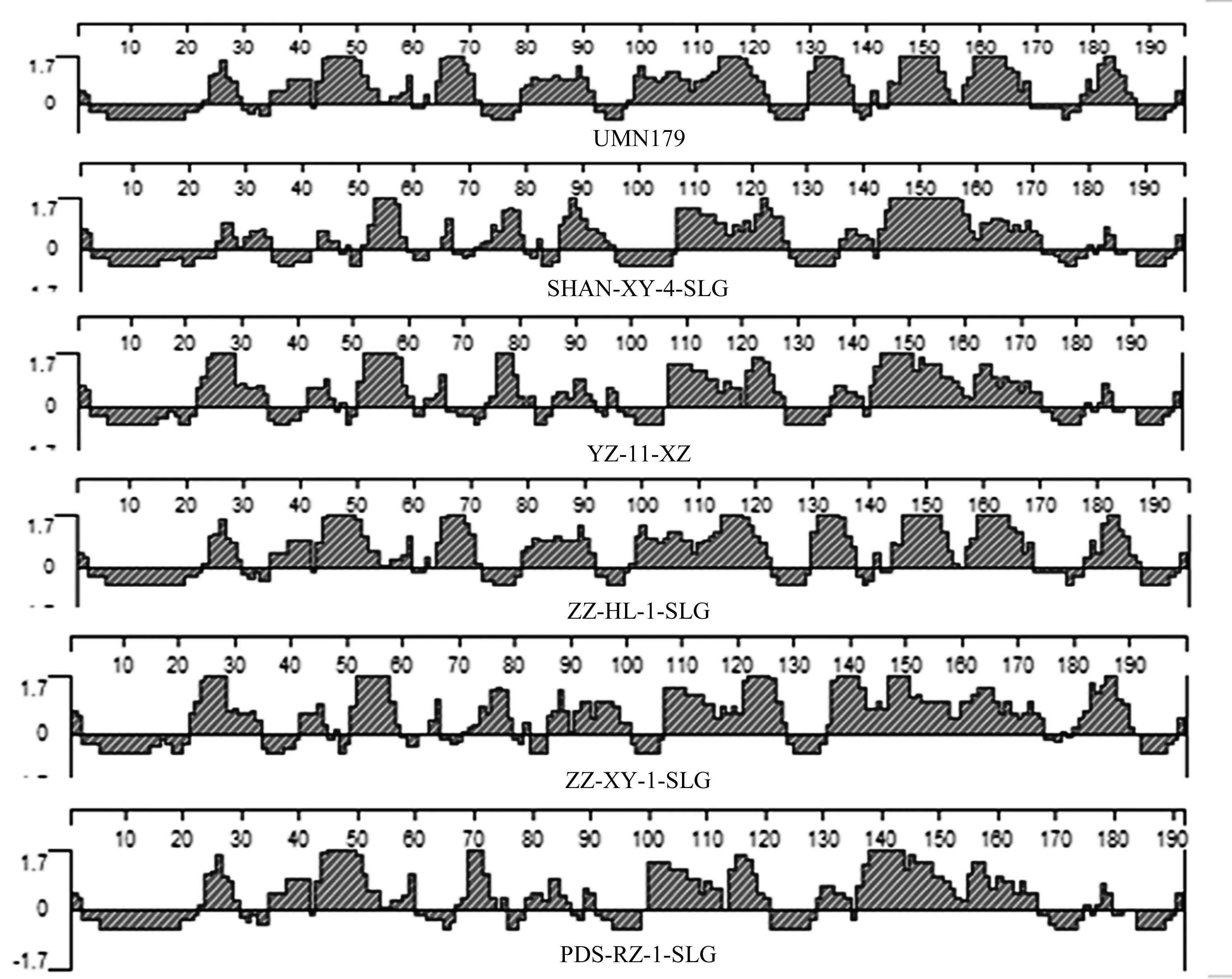
图4 G.anatis FlfA蛋白的B细胞抗原表位预测
3 结论与讨论
菌毛是大多数革兰氏阴性菌及少数革兰氏阳性菌菌体表面的丝状蛋白附着物。G.anatis菌毛蛋白具有抗原性,与其致病性有关。为深入了解G.anatisFlfA功能,利用生物信息学技术进行分析,为进一步研究其致病机制及防控措施提供基础。
菌毛在病原菌识别、粘附宿主细胞过程中发挥着重要作用,能使病原菌粘附于宿主细胞表面,如粘附于呼吸道、消化道和泌尿生殖道黏膜上皮细胞[15]。F17样菌毛能够结合宿主细胞的N-乙酰氨基葡萄糖(Glc-NAc)[10]。F17样菌毛形成需要基因簇的表达,编码F17样菌毛的基因簇由4个基因构成,分别为结构蛋白编码基因flfA(大小为5 070 bp)、导向蛋白编码基因flfC、周质伴侣蛋白编码基因flfD和粘附素编码基因flfG[12]。FlfA在FlfD和FlfC的辅助下分泌到胞外,并完成折叠、组装,以此构成了菌毛的主干;FlfG位于菌毛的顶部,主要作用是识别与结合受体[16]。在G.anatis基因组中鉴定到F17菌毛的基因簇[17]。本研究结果显示,G.anatis分离株普遍含有F17样菌毛结构蛋白编码基因flfA,提示F17样菌毛可能在该菌致病过程中发挥重要的作用。
本研究中,18株G.anatis分离株的flfA基因同源性为70.2%~99.8%;系统发育进化树分析显示,18株G.anatis分布于三大分支。YZ-14-QG、ZK-XC-7-QG、LH-BJT-6-SLG和UMN179等构成第一分支;SHAN-XY-4-SLG和ZZ-XY-1-SLG等构成第二分支;YZ-11-XZ、ZZ-HL-2-GZ和 PDS-RZ-1-SLG等构成第三分支;在第一分支中YZ-14-QG、ZZ-HL-1-SLG、XX-HJ-6-SLG与SHAN-XY-1-GZ的flfA核苷酸同源性为100%;第三分支中LH-BJT-11-SLG、XX-HJ-4-SLG、YQ-2-5-SLG和PDS-RZ-1-SLG的flfA核苷酸同源性为100%。SHAN-XY-1-QG株与UMN179株在同一分支且亲缘关系较近,与PDS-RZ-1-SLG亲缘关系相对较远。不同省份的菌株在三大分支上均有分布,以上结果提示,G.anatisflfA的保守性与菌株分离地域无关。对5株不同的flfA基因推导的氨基酸序列分析发现,氨基酸同源性为63.1%~98.8%,氨基酸序列变异区发生在同一位置,存在6个高变区,引起突变的主要原因是缺失,但是并未改变FlfA氨基酸编码序列,因此保证了其功能的稳定性。
Lucio等[13]发现,flfA基因编码的菌毛蛋白在G.anatis粘附黏膜上皮细胞及生物被膜形成中发挥重要作用,是其重要毒力因子之一,并且具有良好的免疫原性[12]。因此,菌毛蛋白在疫苗研制和诊断试剂开发等方面具有重要意义,而抗原表位在免疫识别和结合过程中发挥着重要作用,可直接影响疫苗的免疫效果。动物免疫重组菌毛蛋白后可产生有效的保护性抗体,因此菌毛蛋白是G.anatis亚单位疫苗的潜在候选抗原[18-19]。本研究对G.anatisFlfA线性B细胞抗原表位进行预测发现,FlfA蛋白具有6~8个优势抗原表位,且分布于序列保守区。由此推测,不同的FlfA蛋白间存在着较强的交叉免疫反应,可作为疫苗候选抗原。
本研究成功克隆出G.anatis中国分离株flfA基因,并预测其分子生物学特征,其编码蛋白是位于病原菌表面且保守性较高的蛋白质。FlfA蛋白优势抗原表位均位于序列保守区,表明该蛋白质可作为疫苗的候选蛋白之一。
参考文献:
[1] Christensen H,Bisgaard M,Bojesen A M,etal.Genetic relationships among avian isolates classified asPasteurellahaemolytica,ActinobacillussalpingitidisorPasteurellaanatiswith proposal ofGallibacteriumanatisgen.nov.,comb.nov.and description of additional genomospecies withinGallibacteriumgen.nov [J].International Journal of Systematic and Evolutionary Microbiology,2003,53(1):275-287.
[2] Bisgaard M,Korczak B M,Busse H J,etal.Classification of the taxon 2 and taxon 3 complex of Bisgaard withinGallibacteriumand description ofGallibacteriummelopsittacisp.nov.,Gallibacteriumtrehalosifermentanssp.nov.andGallibacteriumsalpingitidissp.nov [J].International Journal of Systematic and Evolutionary Microbiology,2009,59(4):735-744.
[3] 王川庆,陈陆,杨霞,等.蛋鸡群卡氏杆菌感染情况的初步研究[J].河南农业科学,2008(3):97-100.
[4] Neubauer C,DE Souza-Pilz M,Bojesen A M,etal.Tissue distribution of haemolyticGallibacteriumanatisisolates in laying birds with reproductive disorders [J].Avian Pathology,2009,38(1):1-7.
[5] Paudel S,Liebhart D,Hess M,etal.Pathogenesis ofGallibacteriumanatisin a natural infection model fulfils Koch’s postulates:1.Folliculitis and drop in egg production are the predominant effects in specific pathogen free layers [J].Avian Pathology,2014,43(5):443-449.
[6] Singh S V,Singh B R,Sinha D K,etal.Gallibacteriumanatis:An emerging pathogen of poultry birds and domiciled birds [J].Journal of Veterinary Science & Technology,2016,7(3):324.
[7] 彭志锋,高冬生,刘红英,等.鸭源鸡杆菌整合子及其与耐药性的相关性分析[J].畜牧兽医学报,2016,47(8):1676-1681.
[8] 高冬生.株鸭源鸡杆菌部分耐药基因及其与耐药性关系的研究[D].郑州:河南农业大学,2011.
[9] Bojesen A M,Vazquez M E,Bager R J,etal.Antimicrobial susceptibility and tetracycline resistance determinant genotyping ofGallibacteriumanatis[J].Veterinary Microbiology,2011,148(1):105-110.
[10] Liu C C,Ou S C,Tan D H,etal.The fimbrial protein is a virulence factor and potential vaccine antigen of avibacterium paragallinarum [J].Avian Diseases,2016,60(3):649-655.
[11] Maier B,Wong G C.How bacteria use type Ⅳ pili machinery on surfaces[J].Trends Microbiol,2015,23(12):775-788.
[12] Bager R J,Nesta B,Pors S E,etal.The fimbrial protein FlfA fromGallibacteriumanatisis a virulence factor and vaccine candidate [J].Infection and Immunity,2013,81(6):1964-1973.
[13] Lucio M L S,Vaca S,Vzquez C,etal.Adhesion ofGallibacteriumanatisto chicken oropharyngeal epithelial cells and the identification of putative fimbriae [J].Advances in Microbiology,2012,2(4):505-510.
[14] Vaca S,Monroy E,Rojas L,etal.Adherence ofGallibacteriumanatisto inert surfaces [J].J Anim Vet Adv,2011,10(13):1688-1693.
[15] Spaulding C N,Hultgren S J.Adhesive pili in UTI pathogenesis and drug development [J].Pathogens,2016,5(1):30.
[16] Wilkie I,Harper M,Boyce J,etal.Pasteurella multocida:Diseases and pathogenesis[M].Berlin:Springer Publishing Company,2012:1-22.
[17] Bager R J,Kudirkiene E,da Piedade I,etal.In silico prediction ofGallibacteriumanatispan-immunogens [J].Veterinary Research,2014,45(1):80.
[18] Novotny L A,Jurcisek J A,Ward M O,etal.Antibodies against the majority subunit of type Ⅳ Pili disperse nontypeableHaemophilusinfluenzaebiofilms in a LuxS-dependent manner and confer therapeutic resolution of experimental otitis media [J].Mol Microbiol,2015,96(2):276-292.
[19] Okay S,Ozcengiz E,Gursel I,etal.Immunogenicity and protective efficacy of the recombinantPasteurellalipoproteinE and outer membrane protein H fromPasteurellamultocidaA:3 in mice [J].Res Vet Sci,2012,93(3):1261-1265.

