食管鳞癌患者血浆lncRNA TUC338表达变化及其临床意义
孙开颜,赵新炜,杨丽君,董翠翠,董树岭,尹惠卿,明亮,贺付成
(1 郑州大学第一附属医院,郑州450052;2 天津儿童医院)
食管癌的主要组织学类型为鳞癌和腺癌,其中90%以上为鳞癌[1]。据统计,食管鳞癌(ESCC)早期诊治者5年生存率可超过90%[2~4]。但目前临床上缺少早期诊断敏感、特异的无创性诊断技术或指标。长链非编码RNA(lncRNA)是一种没有编码功能、长度大于200 nt的功能性RNA分子,其不编码蛋白质,而是以RNA的形式在表观遗传调控、基因转录调节、转录后加工等方面调控基因表达[5]。在体内,lncRNA可参与多种生物学过程的调控,在细胞分化、胚胎与组织发育等多种生命活动中发挥重要作用[6~8]。近年研究发现,肿瘤患者血浆lncRNA水平特异性变化与病情进展有关,可能作为癌基因或抑癌基因参与肿瘤的发生、发展[9~11]。推测lncRNA有可能成为一种ESCC早期诊断的非侵入性肿瘤生物标志物。本研究观察了ESCC患者血浆lncRNA TUC338表达变化,并分析lncRNA TUC338早期诊断ESCC的潜在价值,旨在为寻找新的肿瘤生物标志物提供理论依据。
1 资料与方法
1.1 临床资料 选择2016年7月~2017年1月郑州大学第一附属医院收治的ESCC患者116例(ESCC组)。所有患者经消化内镜活检或术后组织病理检查明确诊断,术前未接受任何抗肿瘤治疗。排除伴发器质性或系统性疾病,如糖尿病、高血压等,以及合并其他肿瘤者。其中,男56例、女60例,年龄45~84(65.76±7.83)岁;肿瘤直径:≤3 cm 54例,>3 cm 62例;肿瘤部位:胸上段41例,胸中段39例,胸下段36例;组织分化程度:高中分化65例,低未分化51例;TNM分期:Ⅰ、Ⅱ期53例,Ⅲ、Ⅳ期63例;有淋巴结转移59例,无淋巴结转移57例。同期另选在本院体检的健康者39例(对照组),男21例、女18例,年龄44~85(65.62±7.24)岁。两组性别、年龄具有可比性。本研究经郑州大学第一附属医院医学伦理委员会批准,患者均知情同意。
1.2 血浆和食管组织lncRNA TUC338表达检测 ①样本收集与处理:ESCC组于术前、对照组于体检日采集空腹静脉血2 mL,置于EDTA-K2抗凝管中,静置20 min,3 000 r/min离心10 min,小心将上层血浆转移至新的EP管中,12 000 r/min离心10 min,将上层血浆再次转移至新的EP管中,-80 ℃保存备用。另选ESCC组织及其配对的癌旁正常组织(距癌组织边缘>5 cm),置于RNA保存液中,-80 ℃保存备用。②血浆和食管组织总RNA提取:取上述血浆250 μL,加入TRIzol LS 750 μL,充分混匀,按照TRIzol LS试剂盒说明提取总RNA;取癌组织及其配对的癌旁正常组织各30 mg,液氮下研磨成粉末,然后加入TRIzol 1 mL,充分混匀,按照TRIzol试剂盒说明提取总RNA。经NanoDrop 2000C紫外分光光度计检测,OD260/OD280为1.8~2.0,表明提取的总RNA纯度合格,调整总RNA浓度至300 ng/μL进行后续实验。③逆转录反应:将总RNA按PrimeScriptTMRT reagent Kit with gDNA Eraser试剂盒说明逆转录为cDNA,所得cDNA -20 ℃保存。④实时荧光定量PCR:lncRNA TUC338及其内参GAPDH引物由上海生工生物工程有限公司设计合成。引物序列:lncRNA TUC338上游引物5′-TCCACAGGACAGGTACAGCA-3′,下游引物5′-GCCTTGGAGACTGAACATCC-3′;GAPDH上游引物5′-GGAGCGAGATCCCTCCAAAAT-3′,下游引物5′-GGCTGTTGTCATACTTCTCATGG-3′。取cDNA 2 μL,按SYBR®Premix Ex TaqTMⅡ试剂盒说明进行PCR扩增。PCR反应体系共20 μL:SYBR Premix Ex Taq Ⅱ(Tli RNaseH Plus,2×)10 μL,PCR Forward Primer 0.8 μL,PCR Reverse Primer 0.8 μL,DNA 模板2 μL,dH2O 6.4 μL;反应条件:95 ℃预变性30 s,95 ℃变性5 s、58 ℃退火34 s共40个循环。以GAPDH为内参,采用2-ΔΔCt法计算lncRNA TUC338相对表达量。实验重复3次,取平均值。
1.3 相关性分析 随机选择46例ESCC患者,采用Spearman秩相关分析法分析血浆与肿瘤组织lncRNA TUC338表达的关系。分析血浆lncRNA TUC338表达与ESCC患者临床病理参数的关系。
1.4 血浆lncRNA TUC338表达对ESCC的诊断效能分析 绘制血浆lncRNA TUC338表达诊断ESCC的受试者工作特征(ROC)曲线,计算曲线下面积。以cut off值为截断值,计算血浆lncRNA TUC338表达诊断ESCC的敏感性及特异性。

2 结果
2.1 两组血浆lncRNA TUC338表达比较 ESCC组及对照组血浆lncRNA TUC338相对表达量分别为2.60±0.83、1.03±0.32,二者比较P<0.01。
2.2 ESCC组织及癌旁正常组织lncRNA TUC338表达比较 ESCC组织及癌旁正常组织lncRNA TUC338相对表达量分别为2.47±0.69、1.02±0.29,二者比较P<0.01。
2.3 ESCC患者血浆与肿瘤组织lncRNA TUC338表达的关系 Spearman秩相关分析显示,ESCC患者血浆与肿瘤组织lncRNA TUC338表达呈正相关关系(r=0.643,P<0.01)。
2.4 血浆lncRNA TUC338表达与ESCC患者临床病理参数的关系 见表1。
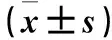
表1 血浆lncRNA TUC338表达与ESCC患者临床病理参数的关系
2.5 血浆lncRNA TUC338表达对ESCC的诊断效能 ROC曲线分析显示,血浆lncRNA TUC338表达诊断ESCC的曲线下面积为0.684(95%CI: 0.567~0.780,P<0.01),其cut off值为1.415,此时其诊断ESCC的敏感性为63.4%、特异性为78.1%。
3 讨论
研究发现,lncRNA在血液中稳定性较高,可通过包被在外泌体、微泡等微粒中免于被核糖核酸酶降解,而且在室温下放置24 h以上甚至反复冻融亦相当稳定[12]。与miRNA一样,lncRNA亦能存在于肿瘤患者尿液、唾液和血液中[13],如lncRNA HOTTIP-005在胰腺癌患者血浆中异常表达,可作为胰腺癌诊断和患者预后判断的潜在生物标志物[14];lncRNA-HEIH在肝细胞癌患者血浆中高表达,其表达变化与肝细胞癌病情进展有关,可用于肝癌的早期诊断以及患者预后监测[15]。因此,血浆中某些核酸分子水平变化有助于肿瘤的诊断[16]。但血浆lncRNA能否作为ESCC早期诊断的生物标志物尚不十分清楚。
人TUC338序列全长590 bp,基因位于12号染色体。多聚胞嘧啶结合蛋白2基因独立转录形成UC338转录片段,TUC338为克隆该转录片段而来。Braconi等[17]研究发现,TUC338在人肝细胞癌中表达显著增加,沉默TUC338表达可抑制肝癌细胞生长。牟丽莎等[18]研究发现,应用RNAi技术下调TUC338表达可使膀胱癌细胞凋亡增加,推测TUC338可作为膀胱癌药物治疗潜在的作用靶点。Ouyang等[19]研究发现,抑制TUC338表达后,CAL-27和SCC-9两种舌鳞癌细胞增殖均受到抑制,细胞凋亡增加,说明TUC338可参与舌鳞癌的发生、发展。本研究结果显示,ESCC组血浆lncRNA TUC338相对表达量明显高于对照组;ESCC组织lncRNA TUC338相对表达量明显高于癌旁正常组织。提示lncRNA TUC338可能在ESCC发生、发展过程中具有癌基因作用。
本研究还发现,ESCC患者血浆与肿瘤组织lncRNA TUC338表达呈正相关关系,说明血浆lncRNA TUC338表达可反映肿瘤组织lncRNA TUC338表达。进一步研究发现,血浆lncRNA TUC338表达与ESCC组织TNM分期有关。肿瘤恶性程度越高,血浆lncRNA TUC338表达越高,提示血浆lncRNA TUC338可能参与ESCC的恶性转化。本研究ESCC有淋巴结转移者血浆lncRNA TUC338表达高于无淋巴结转移者,提示血浆lncRNA TUC338可能参与ESCC的侵袭和转移。本研究ROC曲线分析显示,血浆lncRNA TUC338诊断ESCC的敏感性为63.4%、特异性为78.1%,提示血浆lncRNA TUC338有可能作为诊断ESCC的潜在生物标志物。但由于本研究样本量较少,其结论还有待于进一步研究证实。
综上所述,ESCC患者血浆LncRNA TUC338高表达,其表达变化与肿瘤TNM分期、淋巴结转移有关;血浆LncRNA TUC338有可能作为诊断ESCC的潜在生物标志物。
参考文献:
[1] Rustgi AK, El-Serag HB. Esophageal carcinoma[J]. N Engl J Med, 2014,371(26):2499-2509.
[2] Hong L, Han Y, Zhang H, et al. Prognosis-related microRNAs in esophageal cancer[J]. Expert Opin Biol Ther, 2014,14(4):483-489.
[3] Zhang HL, Liu RL, Shi YT, et al. Analysis of the survival in patients after surgical resection of thoracic esophageal cancer[J]. Zhonghua Zhong Liu Za Zhi, 2009,31(7):541-545.
[4] Pennathur A, Gibson MK, Jobe BA, et al. Oesophageal carcinoma[J]. Lancet, 2013,381(9864):400-412.
[5] Shen Z, Li Q, Deng H, et al. Long non-coding RNA profiling in laryngeal squamous cell carcinoma and its clinical significance: potential biomarkers for LSCC[J]. PLoS One, 2014,9(9):e108237.
[6] Ge Y, Yan X, Jin Y, et al. MiRNA-192 [corrected] and miRNA-204 Directly Suppress lncRNA HOTTIP and Interrupt GLS1-Mediated Glutaminolysis in Hepatocellular Carcinoma[J]. PLoS Genet, 2015,11(12):e1005726.
[7] Sahu A, Singhal U, Chinnaiyan AM. Long noncoding RNAs in cancer: from function to translation[J]. Trends Cancer, 2015,1(2):93-109.
[8] Mehta SL, Kim T, Vemuganti R. Long noncoding RNA fosDT promotes ischemic brain injury by interacting with REST-associated chromatin-modifying proteins[J]. J Neurosci, 2015,35(50):16443-16449.
[9] Tong YS, Wang XW, Zhou XL, et al. Identification of the long non-coding RNA POU3F3 in plasma as a novel biomarker for diagnosis of esophageal squamous cell carcinoma[J]. Mol Cancer, 2015(14):3.
[10] Rivas A, Burzio V, Landerer E, et al. Determination of the differential expression of mitochondrial long non-coding RNAs as a noninvasive diagnosis of bladder cancer[J]. BMC Urol, 2012(12):37.
[11] Shao Y, Ye M, Jiang X, et al. Gastric juice long noncoding RNA used as a tumor marker for screening gastric cancer[J]. Cancer, 2014,120(21):3320-3328.
[12] Huang X, Yuan T, Tschannen M, et al. Characterization of human plasma-derived exosomal RNAs by deep sequencing[J]. BMC Genomics, 2013(14):319.
[13] Hauptman N, Glavac D. Long non-coding RNA in cancer[J]. Int J Mol Sci, 2013,14(3):4655-4669.
[14] Wang Y, Li Z, Zheng S, et al. Expression profile of long non-coding RNAs in pancreatic cancer and their clinical significance as biomarkers[J]. Oncotarget, 2015,6(34):35684-35698.
[15] 杨哲锋,李娟,马志元,等.血浆LncRNA-HEIH表达水平与肝癌发病风险的关联分析[J]. 现代肿瘤医学,2015,23(1):80-84.
[16] Wang J, Zhang KY, Liu SM, et al. Tumor-associated circulating microRNAs as biomarkers of cancer[J]. Molecules, 2014,19(2):1912-1938.
[17] Braconi C, Valeri N, Kogure T, et al. Expression and functional role of a transcribed noncoding RNA with an ultraconserved element in hepatocellular carcinoma[J]. Proc Natl Acad Sci U S A, 2011,108(2):786-791.
[18] 牟丽莎,朱书,蔡志明.TUC338在膀胱癌中的表达及作用机制研究[J].深圳中西医结合杂志,2016,26(20):1-3.
[19] Ouyang KX, Zou R, Liang J, et al. TUC338 overexpression leads to enhanced proliferation and reduced apoptosis in tongue squamous cell carcinoma cells in vitro[J]. J Oral Maxillofac Surg, 2017,75(2):423-428.

