功能化纳米金目视检测铅
刘 欢,白 拖,曹婉婷,徐梦妮,黄 婷*
(1.西安石油大学 化学化工学院,陕西 西安 710065;2.安康学院 化学化工学院,陕西 安康 725000)
铅难以被微生物自然降解,微量的铅会随着食物链慢慢在人体及动物体内累积,当铅累积至一定量时,便会对人体的九大系统造成一定程度的损害。当前,铅污染已对人类生存环境造成了严重的威胁,所以,控制铅污染是十分必要的。目前,检测铅含量的方法如光谱、质谱、色谱和电化学分析法已经开发出来[1-4],这些方法虽然检测灵敏度高、选择性强、准确度高,但大多依赖大型仪器设备、耗时、费用高、不便携带,并需要训练有素的专业技术人员对复杂样品进行预处理,不利于现场监测铅含量。因此,人们对研究一个具有高灵敏度、高选择性、低费用、能实时分析,以及需要少量的样品预处理就可以检测铅的方法充满兴趣。
研究表明,脱氧核酶DNA分子对许多特定的化学反应具有高催化活性。1994年首次报道“8-17”脱氧核酶对铅具有高特异性[5],反应机理主要是当出现Pb2+时,酶链将底物链剪切成两段[6-8]。同时,基于“8-17”脱氧核酶检测Pb2+的各种生物传感器已经被设计出来,其检出限范围是皮摩尔水平到微摩尔的水平[9-10]。然而,大多数生物传感器因其操作的复杂性不适合现场测试,陆和[11]研究出一个方便又实用的涂有“8-17”脱氧核酶和纳米金的试纸来检测Pb2+,检出限为0.5μmol/L,但是,它在一些特定条件下不够敏感,不能满足检测需求。因此,仍然需要进一步优化和改进以获得较低的检出限。
Mirkin等人首次提出生物条形码检测技术[10],主要基于功能化纳米金与特定的探针,可以识别具体目标分析物和大量的寡核苷酸链。这些作为代理靶标具有相同序列的寡核苷酸链被称为条形码,它们很容易被其相应识别代理捕获,从而有效地增强检测灵敏度[12-13]。同时,大量条形码的存在可以大大降低了特定探针的使用。通过使用生物条形码检测技术检测DNA,使其检测灵敏度达到纳摩尔级,并具有选择性高、准确性强、简单和成本低的优点,可以对Pb2+现场检测[14]。
1 实验部分
1.1 试剂和仪器
链酶亲和素(SA),上海生物源叶有限公司;塑料粘合底板、NC(硝酸纤维)膜、结合垫、吸水垫、样品垫,上海杰一生物有限公司;铅(Pb(NO3)2),天津化学试剂厂;17E酶链、17DS地物分解链、条形码DNA、捕获DNA,生工生物工程(上海)有限公司;牛血清蛋白(BSA)、三羟甲基氨基甲烷(Tris)、磷酸盐缓冲液 (PBS)、盐酸胍 (Gu-HCl)、琥珀4-(N-马来酰亚胺甲基)环己烷-1-羧酸(SMCC),Sigma-Aldrich公司;纳米金 (AuNPs),南京吉仓纳米材料有限公司。
铅标准储备液的配制(100 nmol/L):准确称取0.0033g硝酸铅(分析纯),加入少量水溶解,定容于100 mL容量瓶中,得到浓度为100nmol/L的铅标准溶液,再分别配置浓度为50 nmol/L、20 nmol/L、10 nmol/L、5 nmol/L、0 nmol/L的铅标准溶液。
将1mg链霉亲和素溶解于1mL水中,得到浓度为1mg/mL的链霉亲和素溶液。
1.2 实验方法
1.2.1 试纸条组成材料的选择
1.2.1.1 样品垫的选择。样品垫的作用:(1)吸收样品,可使样品均匀混合并迁移到结合垫和NC膜;(2)以防检测时测试样品浸渍超重;(3)过滤样品中的颗粒物、悬浮物、有色杂质等;(4)可被化学物质浸润,以实现对样品的功能化改性。选择样品垫时,应注意样品垫不能与所测试的材料发生任何反应,所以样品垫通常由对水有大的亲和能力的惰性材料制成。本实验选用GL-b01样品垫。
1.2.1.2 结合垫的选择。结合垫材料需满足以下条件:(1)对水有大的亲和能力,可以吸收纳米金探针,在其有效的质量保证期内,当样品通过结合垫时,使纳米金探针获得有效释放。结合垫应该完全可逆吸附或者释放纳米金探针;(2)流动性好,确保样本可以均匀地迁移到NC膜;(3)不允许与样品反应,也不能与任何测试对象发生反应。本实验选用Ahlatrom 9864金垫。
1.2.1.3 反应膜的选择。反应膜须具以下特性:(1)高蛋白吸附功能使得测试线顺利形成;(2)非特异性结合尽量达到最小,以便读取测试结果。本实验选用NC膜作为反应膜材料。
1.2.1.4 吸收垫的选择。测试条的尾端就是吸收垫,它的作用是吸收反应膜上过剩的样品。添加吸收垫后,样品可流动到反应膜末端,从而对其进行分析。选用合适的材料充当吸收垫至关重要,应选择吸水性好且吸水容量大的亲水性材料充当吸收垫。本实验选用H-5072吸收垫。
1.2.2 测试条的制备
将25mm长的硝酸纤维(NC)膜贴在塑料粘合底板上,再将15mm长的吸收垫放在一边,长约15mm的结合垫放在另一边,最后,将长约15mm的样品垫粘贴在结合垫上面。所有的搭接接口有2mm的重叠,测试条的宽度为4mm。将浓度为1mg/mL的链霉亲和素和浓度为100μ mol/L的捕获DNA-BSA轭合物分别涂在控制区域(线C)和测试区域(检测线T),将纳米金结合物涂在结合垫上。然后在37℃的温度下,干燥测试条4小时,再将其保存在干燥器中。
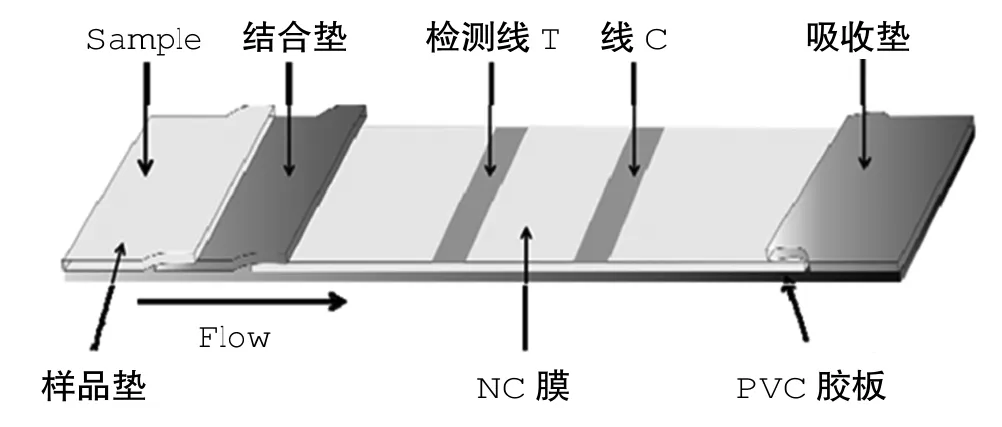
图1 测试条结构图
1.2.3 测试条的检测原理
在纤维膜上将涂入的已知抗体或者抗原包裹,待测试样品加入后,样品中的抗原或者抗体在纤维膜的毛细作用下迁移,与纤维膜上的抗体或者抗原发生反应,进而与纳米金标记物发生反应,形成检测红线,达到了检测目的。
1.2.4 铅的测定方法
将测试条的样品垫浸入样品溶液中约2 min,直到液体已经迁移到硝酸纤维(NC)膜并且完全水化结合垫,然后将测试条立即水平放置让其继续流动。约6 min后,可以直观地看到测试结果。即不含铅Pb2+的样品中,只在控制区观测到红色线;含Pb2+的样品中,控制区和测试区均出现红色线。
1.2.5 样品处理
取样检测汉江水,用孔径为0.45μm的纤维素膜过滤,将某浓度的铅标准液添加到水试样里,调节pH值=7,备用。
2 结果与讨论
2.1 检测结果和机理
图2为铅离子测试条的结构及测量原理示意图。如图2 A所示,在结合垫上发现金纳米共轭物。“17DS-17E”双链,就是“8-17”脱氧核酶,它包含一条酶链17E和一条分裂底物链17DS,对Pb2+有特异性。“17DS-17E”的双链都是DNA,腺嘌呤核糖核苷作为底物链17DS的一个分裂点。当Pb2+存在时,酶链17E在底物链17DS腺嘌呤核糖核苷处催化水解17DS,使17DS分裂为两个片段。
在不考虑信息素的迹浓度挥发的理想状态下,信息素的迹浓度增加和有效信息增加计算方式见表1,其中I表示蚂蚁发现一处食物源并有效反馈的信息数值.
测试结果表明,当Pb2+不存在时,金纳米结合物不分裂,并且迁移至硝酸纤维膜,直至控制区。在控制区,经过特定的链霉亲和素与生物素相互作用产生的链霉亲和素可以捕获生物素化的轭合物,从而产生一个控制线(如图2 B)。当Pb2+存在时,17E酶链催化底物链17DS使其在腺嘌呤核糖核苷处裂开。因此,金纳米结合物被分裂成三个部分:含生物素的DNA片段、17E酶链、含有条形码DNA和残留17DS的纳米金结合物。被分裂的纳米金结合物将迁移越过控制区直到测试区,条形码DNA和互补的捕获DNA杂化,从而在测试区形成红色线。当然,未分裂的纳米金结合物将被链霉亲和素在控制区域捕获。因此,控制区红色线仍可观测到。也就是说,两个区域的观测线均变红,(如图2 C),则表示Pb2+存在。

图2 测试条的结构及测量原理示意图
2.2 灵敏度
本实验分别用0、5、10、20、50、100 nmol/L一系列标准浓度的Pb2+溶液,检测测试条的敏感性,测试结果如图3所示。当显色只出现在硝酸纤维膜的控制区时,Pb2+溶液浓度低于10 nmol/L;显色同时出现在硝酸纤维膜的控制区和测试区时,表明Pb2+浓度高于检测极限,测试结果视为正常。因此10 nmol/L被认为是探测极限。

图3 试纸的灵敏度分析
2.3 选择性
为研究测试条的选择性,对浓度均为0.1 mmol/L的其他二价金属离 子 Hg2+、Cu2+、Ni2+、Mn2+、Cd2+、Fe2+溶液,以及浓度为10 mmol/L的Mg2+、Ca2+溶液,用测试条逐个分析。结果如图4所示,硝酸纤维膜上只出现一个红色的控制线,说明这些离子对试纸条的检测不会造成干扰。

图4 干扰金属的测定
2.4 稳定性
将测试条长期储存在密封铝袋中。在储存期间,每月用10组测试条对Pb2+溶液和无Pb2+溶液进行测试,并统计分析这些测试条。结果显示,测试条在三个月内均没有失去活性和颜色强度,表明测试条足够稳定。
2.5 样品分析
为了验证测试条的有效性,采用加标法,配制不同浓度梯度的Pb2+加标汉江水溶液作为样品,浓度分别为0、5、10、20、50、100 nmol/L,用测试条检测。实验结果如下页表1所示,Pb2+溶液浓度为0、5 nmol/L时,仅控制区出线红色线,检测线未有色;而当Pb2+溶液浓度为10、20、50、100 nmol/L时,检测线和控制线均出现红色线。实验所测结果与对比结果一致,因而此测试条对Pb2+的检测结果准确可信。
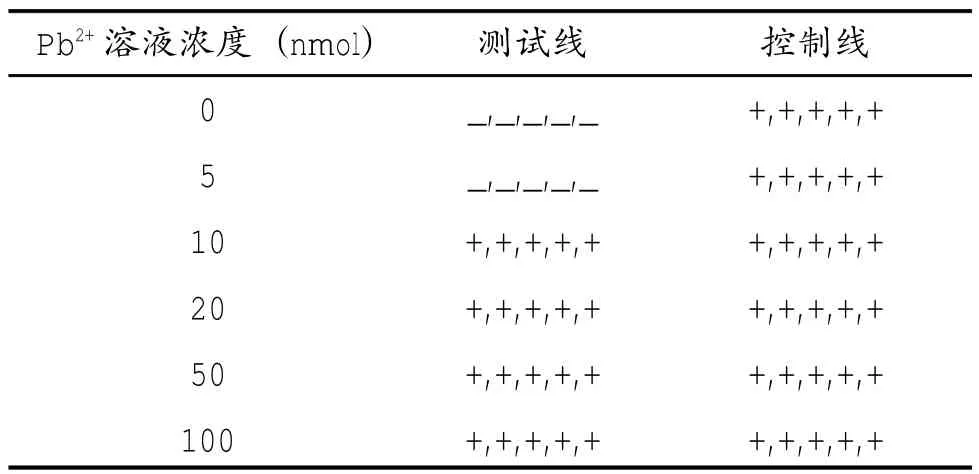
表1 试纸条检测加标实际水样结果(n=5)
3 结论
通过实验测试研究,成功开发出基于脱氧核酶和条形码DNA的功能化纳米金目视检测Pb2+的测试条。此方法检测Pb2+灵敏度高,由于DNA条形码的检出限可达10 nmol/L,以及“8-17”脱氧核酶对Pb2+的特异性,使其对Pb2+具有高选择性;测试条还具有足够稳定性,能长期使用,检测过程不需任何仪器。因此,该测试条可作为一种简单、快速、灵敏、选择性高和低成本的工具用于现场检测Pb2+含量。
参考文献:
[2]BARBASTE M,HALICZ L,GALY A,et al.Evaluation of the accuracy of the determination of lead isotope ratios in wine by ICP MS using quadrupole,multicollector magnetic sector and time-of-flight analyzers[J].Talanta,2001,54 (2):307–317.
[3]GÓRECKI T,PAWLISZYN J.Determination of tetraethyllead and inorganic lead in water by solid phase microextraction/gas chromatography[J].Analytical Chemistry,1996,68(17):3008-3014.
[4]NDLOVU T,AROTIBA O A,SAMPATH S,et al.Electrochemical detection and removal of lead in water using poly modified recompressed exfoliated graphite electrodes[J].JournalofApplied Electrochemistry,2011,41(12):1389-1396.
[5]LI J,LU Y.A highly sensitive and selective catalytic and biosensor for lead ions[J].Journal of the American Chemical Society,2000,122 (42) :10466-10467.
[6]LIU J,LU Y.A colorimetric lead biosensor using DNAzyme-directed assembly of gold nanoparticles[J].Journal ofthe American ChemicalSociety,2003,125(22):6642-6643.
[7]LIU J,LU Y.Accelerated color change of gold nanoparticles assembled by DNAzymes for simple and fast colorimetric Pb2+detection[J].Journal of the American Chemical Society,2004,126(39): 12298-12305.
[8]LIU J,LU Y.Optimization of a Pb2+directed gold nanoparticle/DNAzyme assembly and its application as a colorimetric biosensor for Pb2+[J].Chemistry of Materials, 2004, 16(17):3231-3238.
[9]NAM J M,THAXTON C S, MIRKIN C A.Nanoparticle-based bio-bar codes for the ultra sensitive detection of proteins[J].Science,2003,301 (5641) :1884-1886.
[10]NAM J M,STOEVA S I,MIRKIN C A.Bio-barcode-based DNA detection with PCR-like sensitivity[J].Journal of the American Chemical Society,2004,126(19): 5932-5933.
[11]MAZUMDAR D,LIU J,LU G,et al.Easy-to-use dipstick tests for detection of lead in paints using non-cross-linked gold nanoparticl-DNAzyme conjugates[J].Chemical Communications,2010,46 (9):1416-1418.
[12]KNECHT M R,SETHI M.Bio-inspired colorimetric detection of Hg2+and Pb2+heavy metal ions using Au nanoparticles[J].Analytical and Bioanalytical Chemistry, 2009, 394(1):33-46.
[13]LIN Y W,HUANG C C,CHANG H T.Gold nanoparticle probes for the detection of mercury,lead and copper ions[J].The Analyst,2011,136 (5):863-864.
[14]KIM H K,RASNIK I,LIU J,et al.Dissecting metal ion–dependent folding and catalysis of a single DNAzyme[J].Nature Chemical Biology,2007,3 (12):763-768.

