面向标准化数据整合的医学通用数据模型探析
随着科学技术的快速发展,全球科学数据呈爆发性的增长态势,科学研究进入数据密集型的大数据时代。而医学领域是大数据应用的重要领域之一,大数据的快速发展促进了数据驱动的精准医疗模式的发展,近年国内外都开展了大量基于医学大数据的精准医学研究项目[1]。2016年,我国将精准医学研究列入国家“十三五”科技发展重大专项,并上升为国家战略。但是由于各数据资源的产生单位不同、建设时期不同,采用的数据标准也不尽相同,导致在数据资源的整合方面存在很大难度。为了更有效地利用医学数据,需要针对数据资源的异构现象,建立统一的数据汇交标准,实现多源异构数据的整合。
国内外的学者致力于多源电子化医学数据的数据整合研究,衍生出许多标准模型。2006年,美国国立神经疾病和卒中研究所(National Institutes of Neurological Disorders and Stroke,NINDS)开展了卒中通用数据元素(Common Data Element,CDE)的编制工作,以实现转化医学研究、临床研究和人群研究等各类数据的整合共享[2]。也有研究者基于HL7临床文档架构(Clinical Document Architecture,CDA)标准提出了针对异构临床数据信息系统的集成方案[3-4]。CDA标准是HL7 V3的一部分,专门规定了临床文档内容的标准化,但是CDA只规范了文档内容表达,不涉及文档实例的打包和交换机制[5-6]。美国观察性医疗结果合作组织也建立了一套统一的框架——通用数据模型,帮助解决科学研究中数据结构和内容的标准化问题,该模型目前已广泛地应用于各类科学研究[7-8]。
为了更有效地研究医学数据标准化整合,本文深入探析了美国观察性医疗结果合作组织(Observational Medical Outcomes Partnership,OMOP)建立的通用数据模型(Common Data Model,CDM)的主要模块架构,梳理和总结了多源数据向CDM转换的流程、每一步的实现方法和主要思路,并介绍了当前模型的应用情况,以促进对模型的理解和数据规范化的实践。最后,结合我国精准医学大数据整合共享的实际需求探讨了模型应用中的关键问题并提出了相关建议,以期为促进我国精准医学大数据的集成整合和共享利用提供有益思路和方法。
1 OMOP CDM的整体架构
1.1 概述
OMOP创建于2008年,并不断发展为一个新的合作项目,即观察性健康数据科学和信息学(Observational Health Data Sciences and Informatics,OHDSI)[9]。OHDSI是一个涉及多类利益相关方的跨学科合作项目,致力于通过大规模的数据分析发挥观察性健康数据的更多价值,目前OHDSI研究网络已覆盖全球6亿多患者。OHDSI主要研究涉及开发、分析功能的软件工具,包括OMOP CDM和可应用于数据抽取-转换-加载(Extraction-Transformation-Loading,ETL)过程的工具等。
OMOP CDM定义了一种统一的数据标准,可以规范多源异构的观察性数据的格式和内容,如图1所示。
数据集1、数据集2和数据集3是来自不同数据源、具有不同数据结构的3个数据集。通过对这3个数据集进行数据的抽取、转换、加载,将数据集转换到OMOP CDM中,最后可形成统一的标准化数据结构,支持在该数据结构的基础上进行后续的数据分析并得到分析结果。
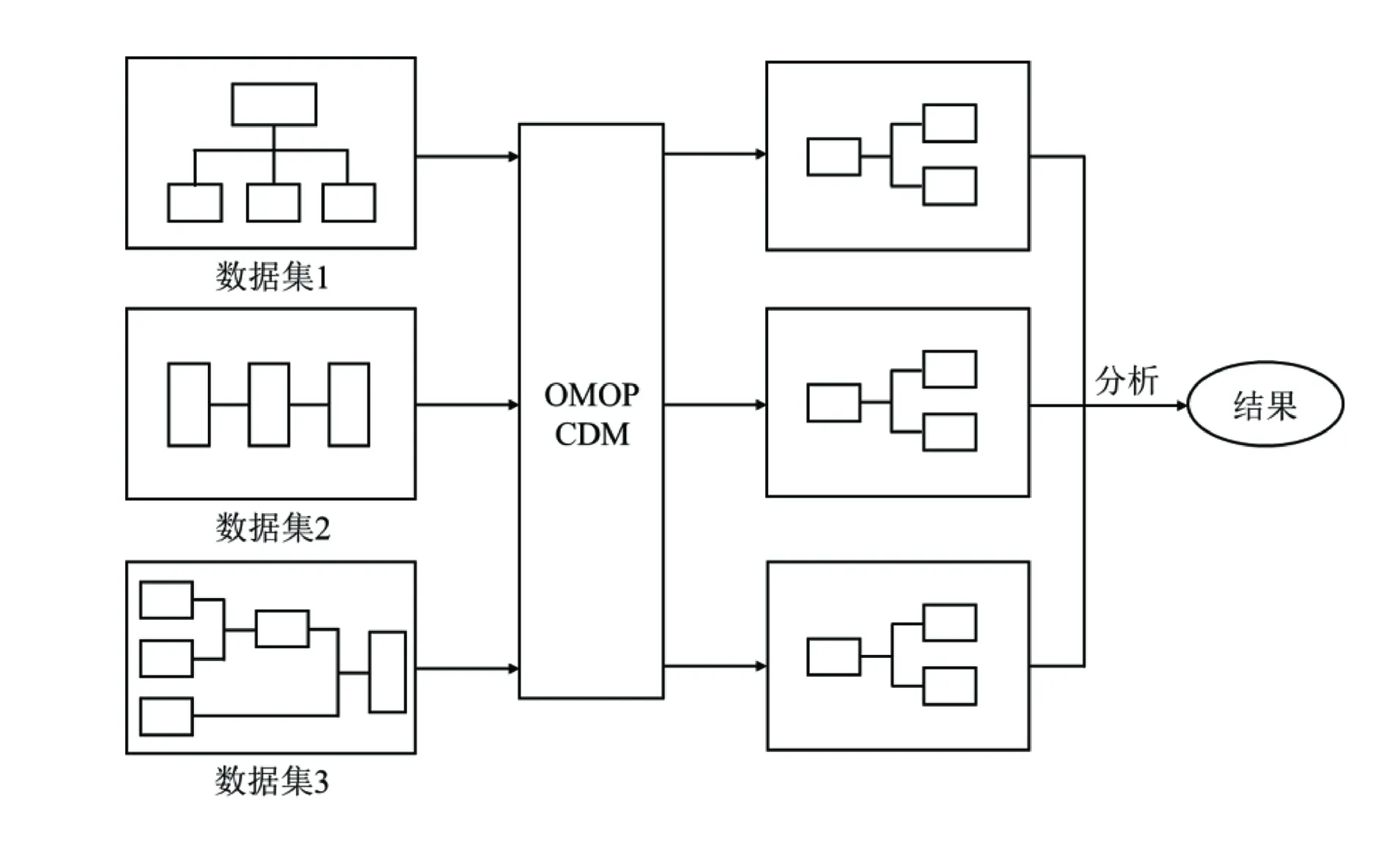
图1 将不同结构的数据集转换为OMOP CDM
1.2 OMOP CDM的主要模块
经不断改进,OMOP CDM目前已更新到6.0版本[10],包括标准化词汇表、标准化元数据表、标准化临床数据表、标准化健康系统数据表、标准化健康经济数据表、标准化派生元素表、结果架构表等7大模块39个域表。
标准化词汇表包含了不同的标准术语以及源数据编码与标准术语的映射信息(表1),标准化元数据表储存了从源数据中派生的元数据的相关信息(表2),标准化临床数据表包含了每个受试者在有效观察期内的纵向临床数据以及相应的人口统计学信息(表3),标准化健康系统数据表描述了负责管理患者医疗保健事项的医疗保健提供者和医疗场所的相关信息(表4),标准化健康经济数据表包含了医疗保健的成本信息(表5),标准化派生元素表包含了从CDM的其他域表中获得的患者临床数据的相关信息(非源数据获得)(表6)。结果架构表是CDM 6.0版本的新模块,目前包含“队列”和“队列定义”两个域表(表7)。
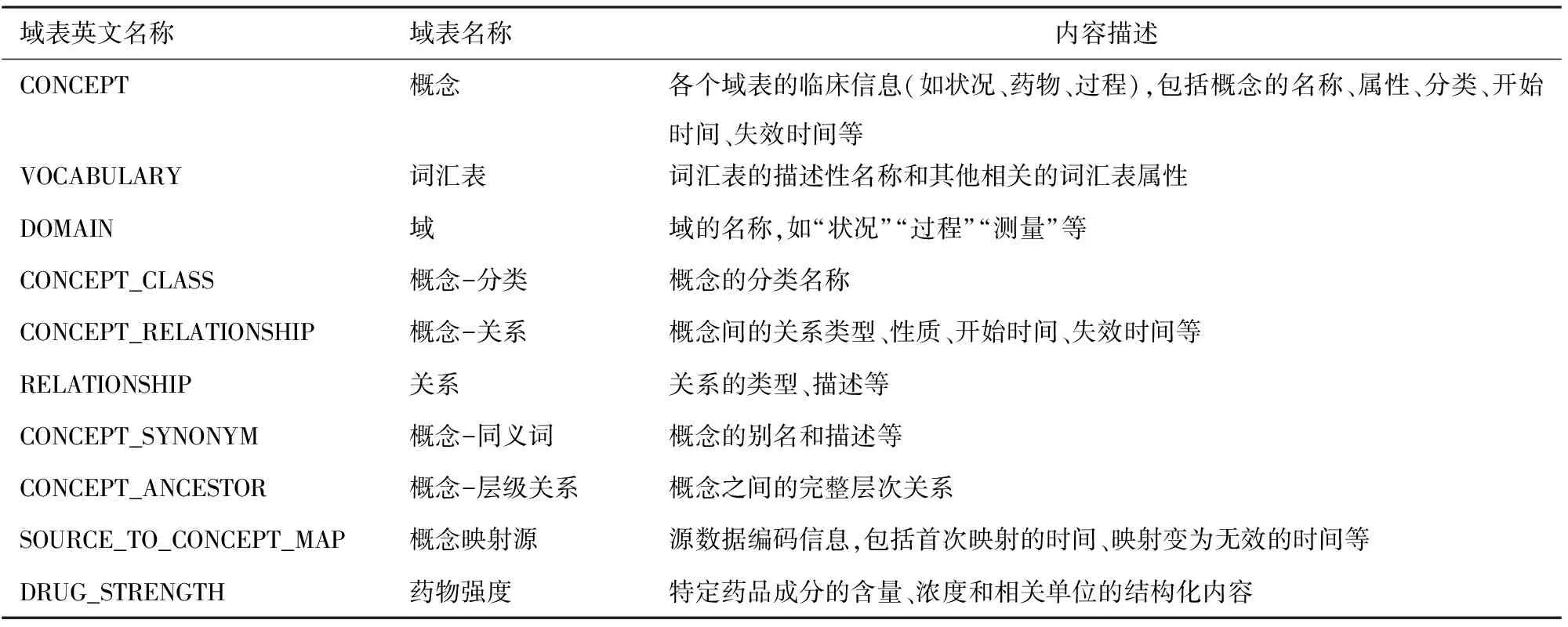
表1 标准化词汇表

表2 标准化元数据表
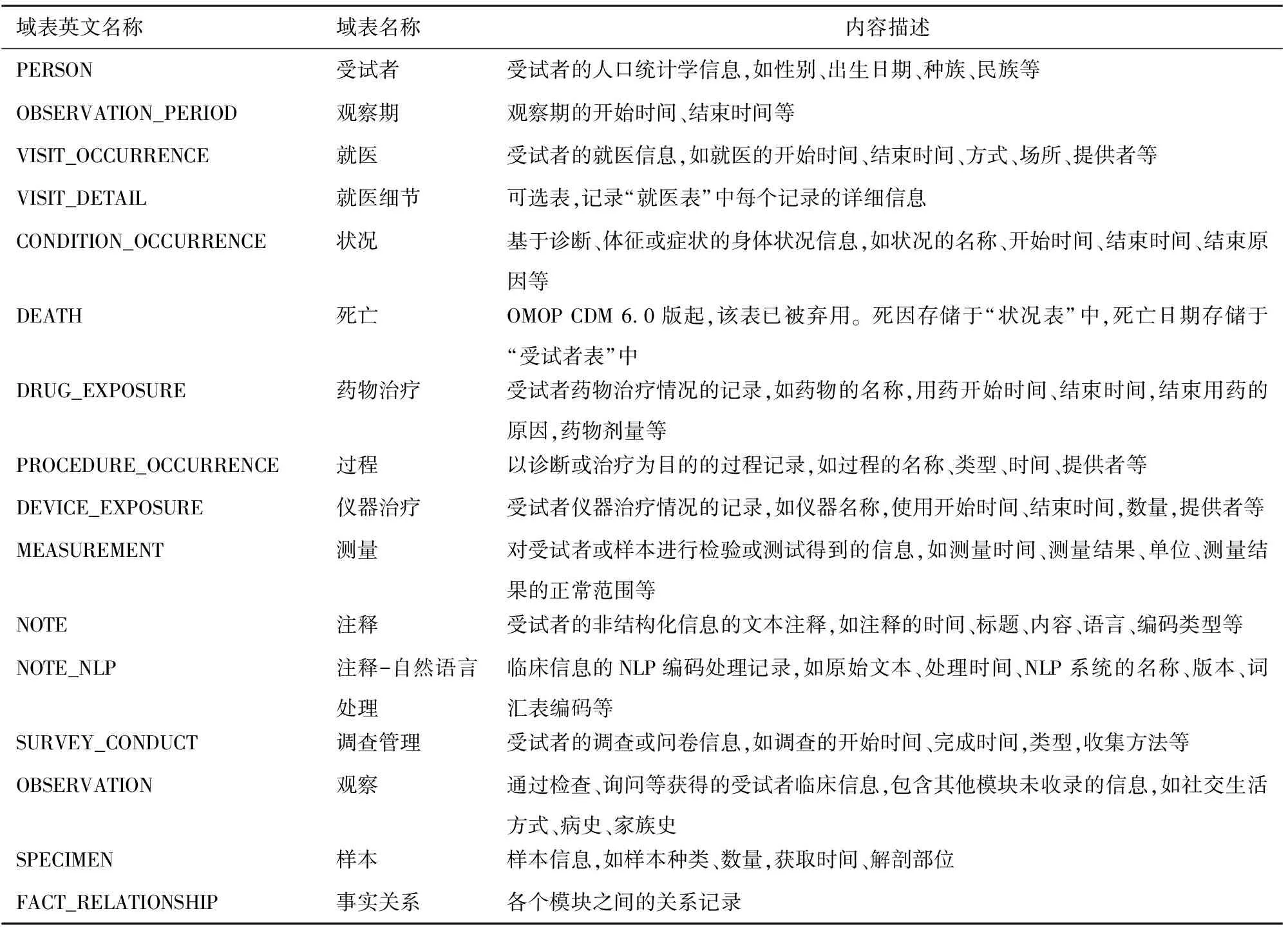
表3 标准化临床数据表

表4 标准化健康系统数据表

表5 标准化健康经济数据表

表6 标准化派生元素表

表7 结果架构表
2 源数据到OMOP CDM的转换方法
2.1 整体转换流程
将源数据转换为OMOP CDM需要经过ETL过程,即对源数据进行数据抽取、转换、加载等一系列操作,使源数据在语法和语义上与目标CDM的结构和术语协调一致[11]。本文对模型的具体转换流程进行梳理和总结,绘制了源数据到OMOP CDM的转换流程图(图2),通过4步处理将多源异构的源数据转换为统一的标准数据结构,便于数据综合利用与分析。
如图2所示,整个ETL过程可分为源数据分析、数据表与字段映射、标准术语映射、ETL实现4个部分。源数据分析是分析各个源数据表的内容和结构,了解源数据的信息记录方式;数据表与字段映射主要是进行整体映射的需求设计,首先建立源数据表与对应CDM域表的映射关系,然后进一步建立源数据字段与对应CDM字段的映射关系;标准术语映射是建立源数据编码到OMOP标准术语的映射,在进行源数据编码的提取时,应按不同域表(即状况、观察、测量、药物治疗等)进行源数据编码的分解提取,并与对应领域的标准术语建立映射;ETL实现是通过输入源数据和术语映射表,根据映射逻辑将源数据逐一映射到每一个需要的CDM域表中,从而将源数据转换为OMOP CDM的标准格式。
由于多源数据结构、类型的复杂性、规模的差异和标准的差异性,在将源数据转换为OMOP CDM的整体过程中,OHDSI在源数据分析、数据表与字段映射以及标准术语映射3个阶段分别提供了WhiteRabbit、Rabbit-In-a-Hat和Usagi工具,辅助研究人员进行转换过程的基本数据分析和转换规则设计。最后的ETL实现阶段较为复杂,OHDSI没有提供集成的ETL实现工具,需要研究人员根据数据的实际情况通过个性化定制实现。目前多使用Java、C#等语言开发ETL生成器以完成最终源数据的标准转换。
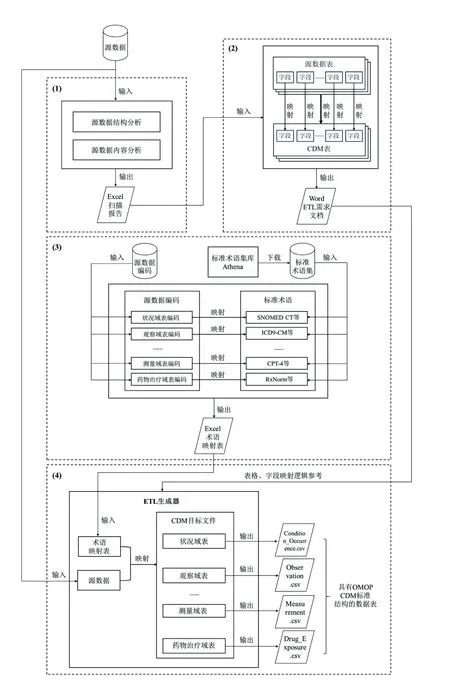
图2 源数据到OMOP CDM的转换流程
2.2 具体转换步骤
2.2.1 源数据分析
首先通过源数据结构和内容分析了解数据集的结构特征。以一份调查问卷数据集为例,调查问卷数据集包含个人信息、人口社会学特征、生活方式、疾病状况、体格检查5个模块(表8),分别对应Personal information.csv、Demographics.csv、Lifestyle.csv、Disease status.csv、Physical examination.csv 5个数据表。
将调查问卷数据集的5个数据表导入WhiteRabbit工具,进行数据扫描,生成并导出名为“ScanReport”的Excel扫描报告(图3)。
该扫描报告包含各个数据表的信息,每个数据表的字段信息,各个字段不同值的列表以及各个值的出现频率。数据表和字段信息可以帮助了解数据结构,数据值和值的出现频率可以帮助识别具体信息的记录方式(如性别的编码方式是“m”和“f”还是“1”和“2”等)。

表8 调查问卷数据集与OMOP CDM的匹配
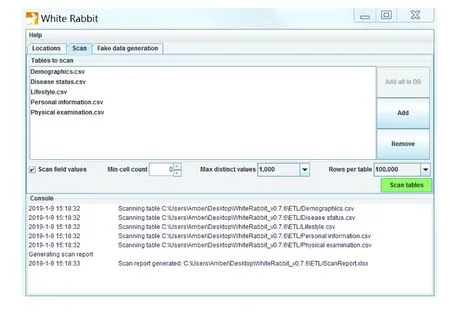
图3 使用WhiteRabbit进行调查问卷数据集的扫描报告
2.2.2 数据表与字段映射
将得到的ScanReport报告导入Rabbit-In-a-Hat工具,进行数据映射的需求设计。Rabbit-In-a-Hat是一个可视化的操作工具,支持研究人员手动建立各个源数据表、字段与CDM域表、字段的映射关系,并支持添加相关的转换逻辑和注释。值得注意的是,Rabbit-In-a-Hat允许研究人员选择不同版本的CDM架构文件,研究人员既可以使用OMOP提供的CDM架构文件,也可以根据研究需求创建自定义版本的CDM架构文件。本次转换选择6.0版本的OMOP CDM架构文件。
首先建立数据表的连接。一个源数据表可以映射到多个CDM域表,一个CDM域表也可以接收多个源数据表的映射。图4展示了Rabbit-In-a-Hat的可视化操作界面,其中个人信息模块的Personal information.csv数据表可映射到CDM的Person域表,人口社会学特征模块的Demographics.csv数据表可映射到CDM的Person域表,疾病状况模块的Disease status.csv数据表可映射到CDM的Condition_occurrence、Drug_era、Care_site以及Cost域表,体格检查模块的Physical examination.csv数据表可映射到CDM的Measurement域表,生活方式模块的Lifestyle.csv数据表无可匹配映射的CDM域表。
然后进一步建立各字段到CDM字段的连接。由于数据集的独特性,源数据中可能存在不能映射到CDM中的表格或字段,CDM中也可能存在无法从源数据获取填充信息的表格或字段。如图5所示,以Personal information.csv数据表和Person域表为例,建立源字段与Person域表字段的连接,并标注相应的映射逻辑。完成全部表格和字段的连接建立和映射逻辑标注后,Rabbit-In-a-Hat可生成并导出构建ETL的需求设计文档。
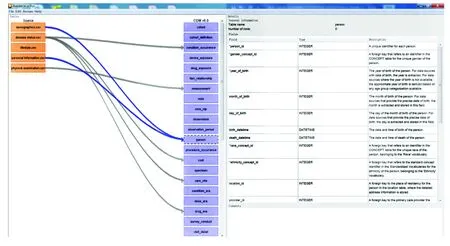
图4 Rabbit-In-a-Hat中各模块数据表与CDM域表之间的连接
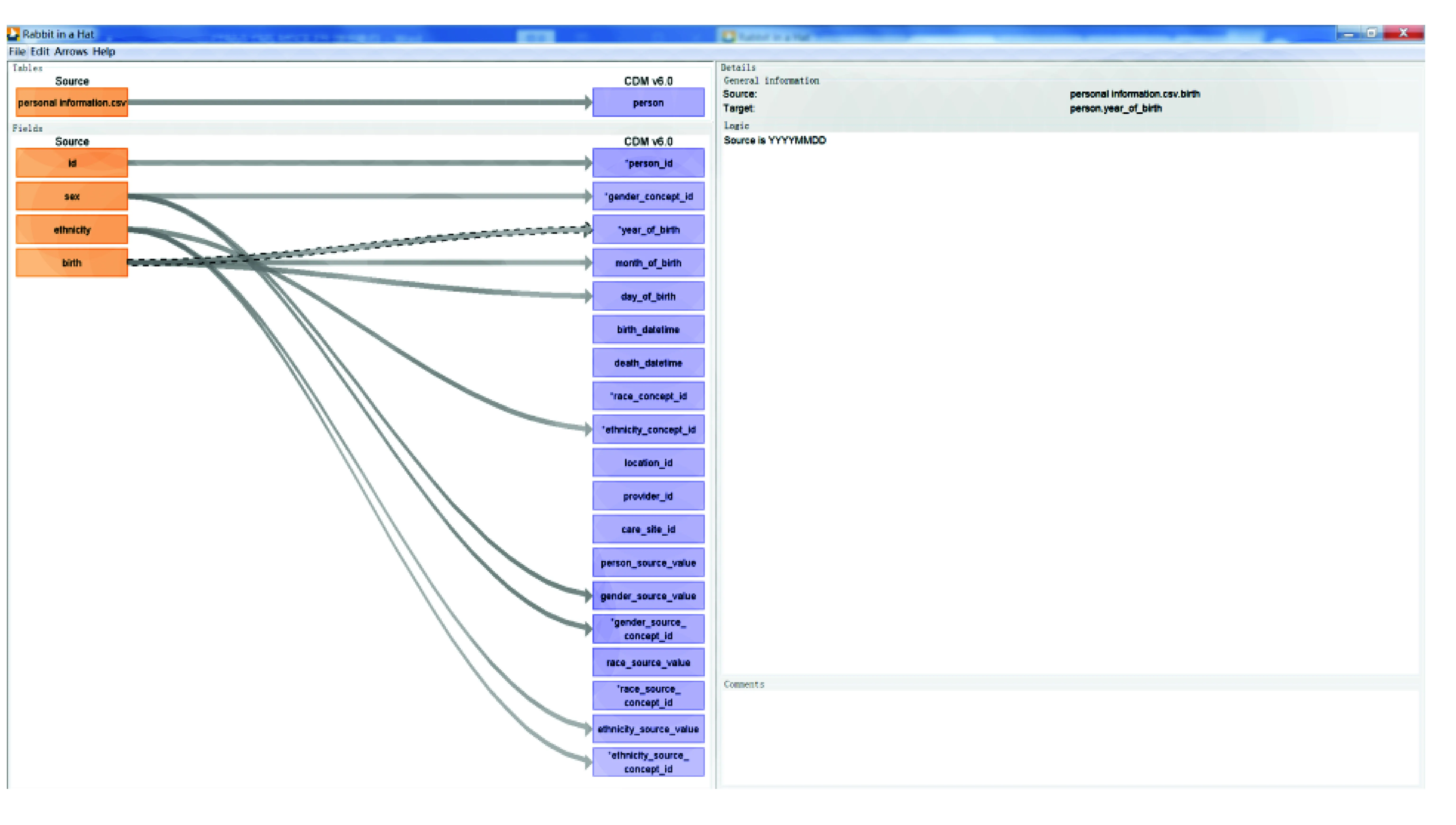
图5 Rabbit-In-a-Hat中数据字段与CDM字段的连接(以Person域表为例)
2.2.3 标准术语映射
由于源数据通常会使用与OMOP CDM不同的编码标准,所以需要进行从源数据编码到OMOP标准术语的映射。OMOP CDM使用多种标准术语集,且不同域表和标准术语之间并不是一一对应的关系。如RxNorm标准术语可应用于药物域表,ICD9-Procedure标准术语可应用于过程域表,而SNOMED标准术语则包含了所有医学领域的概念,可应用于多个域表的映射。不同域表与标准术语的对应情况如表9所示。
工具Usagi可辅助研究人员进行术语映射的构建。Usagi支持导入源数据编码文件和OMOP标准术语文件,通过术语相似度的方法自动的建立各个源编码到OMOP标准术语的建议映射,并获得每一个映射的匹配得分(通常是0~1的数值,1为自信匹配)。
如果建议映射不正确,Usagi允许研究人员进行概念的手动搜索,从而对映射进行修改。最后,研究人员对可以在ETL过程中应用的映射进行逐条批准,生成并导出包含源数据编码、源概念ID、源术语ID、映射后的目标概念ID、目标术语ID等字段信息术语映射表(图6)。
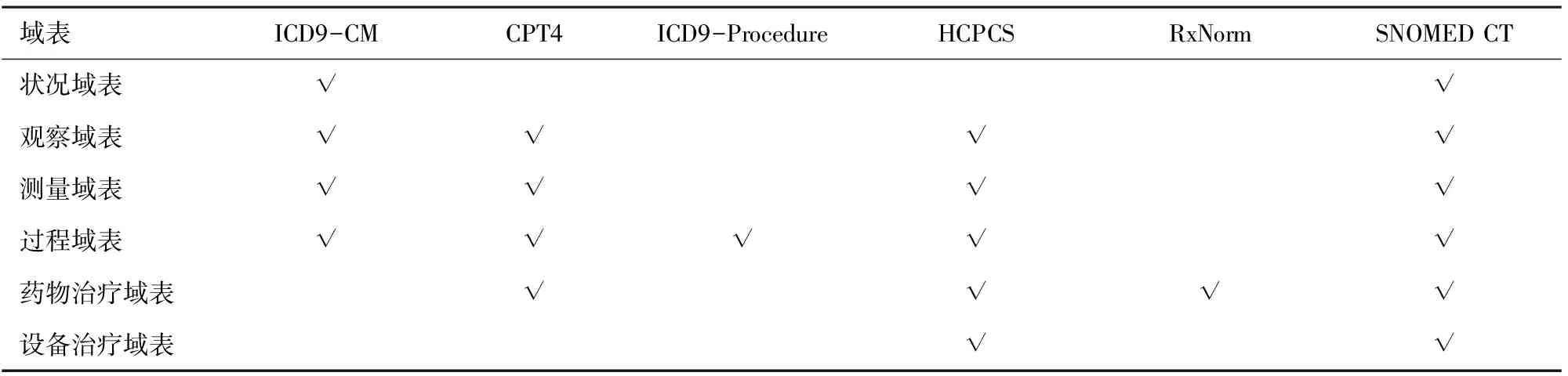
表9 不同域表与标准术语的对应情况
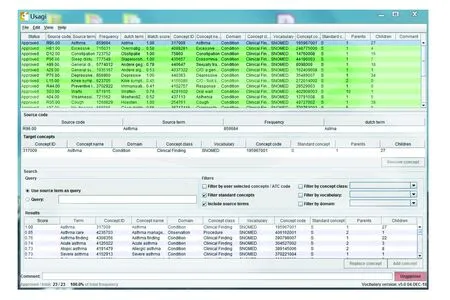
图6 使用Usagi进行源数据编码到标准术语的映射
2.2.4 ETL实现
完成数据表与字段映射和标准术语映射后,可进行最终的ETL实现。在数据表与字段映射时得到的ETL需求文档可作为实现ETL的整体数据映射规范,然后需要根据ETL需求文档中记录的表和字段的映射逻辑编写程序,构建需包含数据质量控制、各字段的标准编码格式转换、各字段的数据映射实现等多种功能的ETL生成器。
将源数据和通过Usagi得到的术语映射表输入到ETL生成器中时,首先应对质量较差、不能满足研究目的的源数据进行剔除,然后将需要转换编码格式的字段数据转换为标准术语的格式,并逐一完成Person域表、Condition_occurrence域表、Drug_era域表、Care_site域表、Cost域表以及Measurement域表中各字段与各模块数据字段之间的映射,转换为OMOP CDM标准结构的数据信息将分别存储在Person.csv、Condition_occurrence.csv、Drug_era.csv、Care_site.csv、Cost.csv以及Measurement.csv文件中。由于不同的源数据集通常具有不同的内容结构和编码规则,所以每个ETL生成器的程序也都具有一定的独特性,需要研究人员根据数据集的实际情况进行个性化的构建。
通过完整的ETL过程,将多源异构的科学数据统一转换为OMOP CDM的标准格式,可实现数据的整合,便于研究人员对数据进行综合分析。
3 OMOP CDM的应用
2008年美国食品药品管理局(Food and Drug Administration,FDA)开展了“哨兵行动(Sentinel Initiative)”,希望利用不同来源的电子医疗保健数据,实现产品安全性的实时连续监测,以加强相关产品上市后的安全性识别与分析,其中就包括了OMOP[12]。此后OMOP CDM逐渐应用到更多领域,如帮助实现多源医学数据的综合利用、解决医学数据整合中的数据标准问题、促进跨中心跨地区的科学研究合作等。
OMOP成立初期,与OMOP CDM相关的研究多集中在美国、欧洲等地。英国临床实践研究数据链(Clinical Practice Research Datalink,CPRD)[13]是一个初级护理数据库,记录了1 400万患者的人口统计信息、人体测量信息、生活方式信息、医疗诊断以及处方数据信息等,被认为是英国的人群代表。CPRD在国际上被广泛应用于流行病学研究,但是由于CPRD使用自身独有的Multilex编码标准,极大地限制了与其他数据库进行数据整合和相关研究的可能性。Matcho等人[14]将CPRD的数据转换到OMOP CDM,其转化的所有要素都被评估为高质量。研究者同时进行了验证工作,在原始CPRD数据和CPRD CDM数据中检查使用非甾体抗炎药和首次急性心肌梗死的风险,结果显示两项数据的患病率相等,证明CPRD可以准确地转换为OMOP CDM。Voss等人[15]也曾将6个不同来源的患者级数据库转换为OMOP CDM,探讨将不同观察健康数据库网络标准化到CDM和术语表中的优点与成本,研究评估了在标准化的转换过程中的信息丢失程度,结果显示转换为OMOP CDM 的信息损失最小,并且数据的标准化过程提高了数据质量和分析效率,促进了跨数据库的数据研究比较。
近年来,不仅欧美发达国家致力于应用OMOP CDM进行相关研究,亚洲地区的研究者也开始尝试将医学健康领域的数据转化到OMOP CDM中,开展了许多标准化的数据研究。韩国亚洲大学医学院的You Seng Chan等人[16]将韩国国民健康保险服务-国家样本队列(NHIS-NSC)数据库中113万受试者的数据转换为OMOP CDM,平均转化率达到了99.1%。该研究是亚洲国家将国家队列数据库转换为通用的OMOP CDM格式的第一次尝试,这也使NHIS-NSC成为了支持多方面医学研究的宝贵资源。北京大学的孙一鑫等人[17]为实现多源临床数据资源的整合共享,同样基于OMOP CDM 制定了呼吸系统疾病的专病队列数据标准。他们分析了各个来源的专病队列的数据特征,然后与OMOP CDM中的已有模块进行匹配,建立了基于OMOP CDM的呼吸队列通用数据标准,进行呼吸系统疾病数据的回顾性整合。
基于OMOP CDM可以将不同数据库的数据转换成通用格式,方便研究人员进行跨数据库的数据抽取、整合,有利于开展不同数据库的综合研究或对照研究。同时,结合OMOP CDM的标准结构,可建立不同特异性专病队列的数据标准,有助于日后开展长期随访和数据采集。
4 问题与建议
4.1 问题
我国启动了精准医学研究专项,项目需要汇集我国各地域的自然人群队列、乳腺癌、食管癌、胃癌、心血管疾病、脑血管疾病等多类型专病人群队列、罕见病人群队列等产出的精准医学大数据,亟待精准医学大数据规范和集成标准,促进数据存储、利用和共享。OMOP CDM为我国多来源、多结构化的精准医学大数据的整合、利用提供了宝贵的思路和方法,值得研究者探索和借鉴。由于我国精准医学大数据从疾病类型、数据类型、语种、术语标准化程度等各方面均与国外的数据存在显著差异,因此CDM模型的具体应用可能存在以下问题。
一是我国的精准医学大数据包括组学数据、影像数据、病理数据、体检数据、随访数据等多类型数据。OMOP CDM的现有架构包括患者、状况、观察、测量、药物治疗、随访等数据,虽然覆盖了其中一些数据类型,但范围并不全面,不能很好地满足我国精准医学数据的整合需求。二是国外的医学术语和编码标准相对于国内发展快、应用较为广泛,而国内医学术语和编码标准研发和应用还不完善。OMOP CDM使用的标准术语均为外文标准,国内医学数据中虽然有些直接使用英文术语和编码,但仍有部分需要进行中文标准转换的数据和很多缺乏标准描述的数据,这些数据无法很好地实现OMOP CDM的映射。三是OMOP CDM的相关工具目前仅支持进行英文数据的转换,不支持非英文数据的转换,缺乏本地化映射、转换等处理工具支持。
4.2 建议
针对上述问题,OMOP CDM的本地化应用中应注意开展以下3个方面的工作。
4.2.1 扩展OMOP CDM构建数据标准化模型
OMOP CDM最初多应用于药物和器械安全性的相关研究。随着OMOP CDM的应用领域逐渐扩大,涉及了流行病学、神经学、药学、消化科学等多方面研究,最新版的OMOP CDM也包含了针对临床数据、健康系统数据、经济学数据、队列数据等多类型数据的标准模块。但在实际应用中,由于多源数据的复杂性,各类型的医学数据并不能完全与OMOP CDM包含的模块相匹配,研究人员应详细分析需要标准化整合的多源数据的类型、结构、变量、变量赋值、单位、标准和编码等,根据实际需求和提取数据共性特征构建本地化的通用数据模型。
4.2.2 采用和建立适用的医学术语和编码标准
OMOP CDM使用的标准术语和编码包括药物标准RxNorm、临床标准SNOMED CT、手术标准ICD9-CM等,标准化术语的应用更有助于数据的标准化和互操作。我国术语和编码标准化的建设一直落后于国际水平,由于语种、标准适用性和应用性问题,国内医学数据在标准方面的突出问题主要是缺乏标准规范的应用、国际标准本地化问题[18]、适合我国医学数据标准的制定不足等。种种原因导致基于OMOP CDM进行数据转换时,无法基于已有工具开展标准的映射,单纯基于不同语种的术语翻译会影响映射准确性。因此,除需要在数据创建时促进医学术语和编码标准的应用外,还迫切需要建立适用于我国研究现状的医学术语和编码标准以及建立多语种对照,以适应我国医学数据集成整合和与国际多源医学数据的集成整合。
4.2.3 研发本地化数据标准化模型转换工具
目前与OMOP CDM相关的研究工作多集中于美国、欧洲等地的数据库,OHDSI提供的可应用于ETL过程的转换工具也都只能进行英文数据的转换。2016年,OHDSI在我国建立分部,通过利用数据科学和信息学方法,促进我国健康医疗数据的集成整合的研究。但是想要有效推进OMOP CDM模型更广泛地应用,有待更多研究者结合我国的实际情况,对现有的OMOP CDM以及相关研究方法和工具进行拓展和本地化,建立适用于中文的医学数据标准化转换工具,开展中文医学数据的整合和集成实践。
5 结语
我国在医学数据的整合方面尚未形成统一的数据模型与标准,OMOP CDM为多源异构的医学数据整合提供了思路和方法,值得借鉴学习。因此,本文对OMOP CDM支持多源数据转换的总体流程和具体步骤进行了系统地分析和总结,梳理了存在的问题并进行分析和提出了建议。目前,我国对OMOP CDM的研究尚处于探索阶段,将模型运用到我国精准医学大数据的汇交整合中还存在一些问题和挑战,今后应注重CDM的本土化研究,将现有模型与我国数据整合的实际情况相结合,建立和完善我国医学数据整合的方法和标准。

