基于毛细管电泳的片段分析和克隆测序在脊髓小脑共济失调动态突变检测中的应用研究
陈园园 郝莹 张瑾 张鑫 谢坤铭 丁铭 顾卫红
三核苷酸重复序列疾病(TRD)系致病基因内三核苷酸重复序列拷贝数在细胞减数分裂过程中发生动态突变,异常扩展导致的一组遗传性疾病。正常人携带的三核苷酸重复序列在一定范围内存在多态性,并在代间传递过程中保持稳定。当重复序列拷贝数超过一定阈值时呈现出不稳定性,部分呈现出逐代扩展趋势。目前已发现30余种由三核苷酸异常扩展导致的疾病[1⁃3],相关重复序列包括胞嘧啶⁃腺嘌呤⁃鸟嘌呤(CAG)、胞嘧啶⁃胞嘧啶⁃鸟嘌呤(CCG)、鸟嘌呤⁃腺嘌呤⁃腺嘌呤(GAA)、胞嘧啶⁃胸腺嘧啶⁃鸟嘌呤(CTG)和鸟嘌呤⁃胞嘧啶⁃鸟嘌呤(GCG),主要位于基因编码区、内含子区和非翻译区。脊髓小脑共济失调(SCA)有9种亚型属于三核苷酸重复序列疾病,即SCA1型、SCA2型、SCA3型、SCA6型、SCA7型、SCA8型、SCA12型、SCA17型和齿状核红核苍白球路易体萎缩(DRPLA),三核苷酸重复扩展次数通常<200次;此外,亨廷顿病(HD,致病基因为IT15基因)、类亨廷顿病2型(致病基因为JPH3基因)、脊髓延髓肌萎缩症(SBMA,致病基因为SBMA基因)等也属于三核苷酸重复序列疾病。
三核苷酸重复序列动态突变的检测可以采用基于毛细管电泳的片段分析、克隆测序、直接测序和蛋白印迹法等技术,其中,蛋白印迹法操作繁琐,已较少用于常规基因检测。目前的常规检测方法是基于毛细管电泳的片段分析。本研究采用基于毛细管电泳的片段分析和克隆测序检测14例脊髓小脑共济失调患者(包括3例SCA2型、2例SCA7型、7例SCA8型和2例SCA17型)致病基因三核苷酸重复序列,比较两种方法的准确性和稳定性。
对象与方法
一、研究对象
研究对象为2005年8月-2016年5月中日友好医院运动障碍与神经遗传病研究中心收集的临床拟诊为脊髓小脑共济失调家系先证者,选取其中的SCA2型3例、SCA7型2例、SCA8型7例和SCA17型2例共14例,均为汉族,男性10例,女性4例;年龄10~58岁,平均(33.42±13.62)岁。本研究经中日友好医院道德伦理委员会审核批准,所有受试者及其家属均知情同意并签署知情同意书。
二、研究方法
1.样本采集 所有受试者空腹采集外周静脉血各5 ml,以质量分数为3.8%的枸橼酸钠进行抗凝,采用标准酚氯仿DNA提取法提取基因组DNA。
2.三核苷酸重复序列突变分析 采用聚合酶链反应(PCR)、琼脂糖凝胶电泳和基于毛细管电泳的片段分析进行脊髓小脑共济失调致病基因动态突变分析。(1)PCR反应:在Ensembl数据库中检索SCA2、SCA7、SCA8和SCA17基因重复序列,基于重复片段两侧序列采用Primer 3.0软件设计引物序列并进行同源比对,由北京赛百盛基因技术有限公司合成,SCA2基因正向引物(F引物)序列:5'⁃TTTGGTAGCAACGGCAAC⁃3',反向引物(R引物)序列:5'⁃CGGGCTTGCGGACATTGG⁃3';SCA7 基因正向引 物 (F引 物 ) 序 列 :5'⁃GGAGTCGAAAGCGAAAGCTA⁃3',反向引物(R 引物)序列:5'⁃AACCCACAGATTCCACGACT⁃3';SCA8基 因 正 向 引 物(F引 物 ) 序 列 :5'⁃GGTCCTTCATGTTAGAAAACCTGGCT⁃3',反向引物(R 引 物 ) 序 列 :5'⁃TTTGAGAAAGGCTTGTGAGGACTGAGAATG ⁃3';SCA17基因正向引物(F引物)序列:5'⁃GGGACGTTGACTGCTGAAC⁃3',反向引物(R引物)序列:5⁃TTCTTCTTGCTTTCCACAGG⁃3'。荧光引物序列由上海普迈生物科技有限公司合成,SCA7、SCA8和SCA17基因正向引物经D4荧光标记,SCA2基因引物采用荧光标记M13末端加尾法标记。SCA2基因正向引物(F引物)序列:5'⁃TTTGGTAGCAACGGCAAC⁃3',反向引物(R 引物 +M13引 物 ) 序 列 :5'⁃CACGACGTTGTAAAACGACCGGGCTTGCGGAATT⁃GG⁃3';SCA7基因正向引物(F荧光引物)序列:5'⁃TTTTTTGTTACATTGTAGGAGCG⁃3',反向引物(R引物)序列:5'⁃AACCCACAGATTCCACGACT⁃3';SCA8基因正向引物(F荧光引物)序列:5'⁃GGTCCTTCATGTTAGAAAACCTGGCT⁃3',反向引物(R 引 物 ) 序 列 :5'⁃TTTGAGAAAGGCTTGTGAGGACTGAGAATG ⁃3';SCA17基因正向引物(F荧光引物)序列:5'⁃GGGACGTTGACTGCTGAAC⁃3',反向引物(R引物)序列:5⁃TTCTTCTTGCTTTCCACAGG⁃3'。PCR 反应体系共 25 μl,依次加入 dNTPs 2.50 mmol,2× GC 缓冲液Ⅰ12.50 μl,SCA2、SCA7、SCA8 和 SCA17 基因正向引物和反向引物各5 pmol,模板DNA 100 ng,Taq酶1 U,加灭菌去离子水补充至25 μl。SCA2基因PCR反应条件为95℃预变性5 min,以94℃ 30 s、58℃ 30 s、72℃ 45 s循环35次,72℃延伸10 min;SCA7基因PCR反应条件为94℃预变性5 min,94℃ 50 s、60℃ 30 s、72℃ 1 min,每个循环减1℃共循环15次,94 ℃ 50 s、54 ℃ 30 s、72 ℃ 1 min,共循环29次,72℃延伸10 min;SCA8基因PCR反应条件为94℃预变性5 min,94℃ 45 s、69℃ 30 s、72℃ 2 min,每个循环减0.50℃共循环20次,94℃45 s、60 ℃ 30 s、72 ℃ 2 min,共循环10次,72 ℃延伸10 min;SCA17基因PCR反应条件为95℃预变性5 min,94 ℃ 30 s、58 ℃ 30 s、72 ℃ 45 s,循环35次,72 ℃延伸10 min。(2)琼脂糖凝胶电泳:取5 μl PCR扩增产物进行质量分数为3%的琼脂糖凝胶电泳,电压100 V,电泳50 min,对于出现2条电泳条带的样品进行基于毛细管电泳的片段分析。(3)基于毛细管电泳的片段分析:采用美国Beckman Coulter公司生产的CEQ⁃8000核酸分析仪对PCR扩增产物进行片段分析,包括 20 μl甲酰胺,以 0.25 μl CEQ DNA Size Standard Kit⁃600 片段作为标准内标,1 μl PCR扩增产物混匀后上样;预设程序进行电泳分离,分离条件为毛细管温度达50℃时,90℃变性120 s,电压2 kV下注入样本30 s、电压4.80 kV下电泳70 min,以预设分析参数进行片段分析。
3.三核苷酸重复序列拷贝数计算 在Ensembl数据库中检索SCA2、SCA7、SCA8和SCA17基因及其包含的三核苷酸重复序列,在CEQ⁃8000核酸分析仪Analysis Parameters窗口STR Locus Tag界面设置重复次数与片段长度的对应关系,以SCA17基因为例:正向引物经D4荧光标记,扩增出287 bp片段,此片段包含38次CAG重复序列,因此设置CAG重复序列与片段长度的关系时,以287 bp对应38次CAG重复序列为标准进行设置。采用美国Beckman Coulter公司生产的GenomelabTMGeXP Genetic Analysis System软件,以GS⁃600 LIZ片段为相对分子质量标记,计算样本扩增片段长度,并自动生成扩增片段包含的重复序列。
4.动态突变的克隆测序 将14例样本送检宝生物工程(大连)有限公司进行克隆测序,将PCR扩增产物分别切胶回收后,克隆至TaKaRa pMD®18⁃T载体,选取阳性克隆进行DNA测序,计算CAG重复序列。
结 果
一、SCA2基因CAG重复序列分析
对3例 SCA2型患者(编号:A15⁃71、A15⁃25和A16⁃9)的样本进行基于毛细管电泳的片段分析和克隆测序(表1)。SCA2正常等位基因CAG重复序列为14~32次,异常等位基因CAG重复序列为33~77次。基于毛细管电泳的片段分析显示,3例样本扩展片段CAG重复序列分别为31、30和32次;毛细管电泳峰形特征为,大多数正常重复序列为单一锐利主峰,其右侧有一矮峰,而扩展重复序列为簇状低矮爪形峰,CAG重复序列的计数以中间高峰为准(图1)。对3例样本扩展片段CAG重复序列分别选取3个不同菌落进行克隆测序,其结果显示,CAG重复序列分别为37/40/40、37/38/39和38/39/40次;每例样本的不同克隆测序结果CAG重复序列存在差异,且同一例样本的不同克隆测序结果亦存在碱基差异。
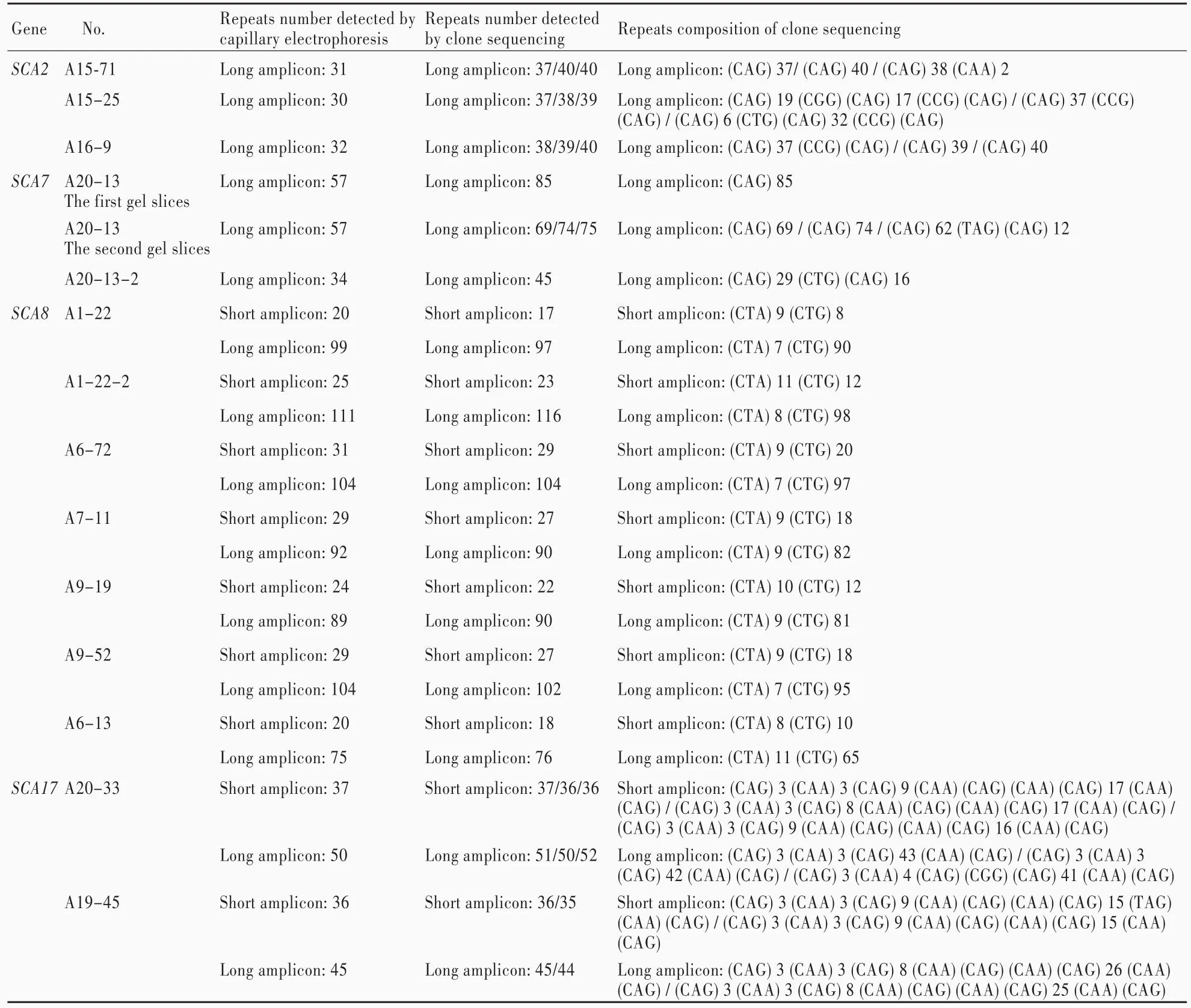
表1 SCA2、SCA7、SCA8和SCA17基因基于毛细管电泳的片段分析和克隆测序结果Table 1. Results of expanded repeats numbers of SCA2,SCA7,SCA8 and SCA17 genes detected by capillary electrophoresis and clone sequencing
二、SCA7基因CAG重复序列分析
对2例SCA7型患者(编号:A20⁃13和 A20⁃13⁃2)的样本进行基于毛细管电泳的片段分析和克隆测序(表1)。SCA7正常等位基因CAG重复序列为4~27次,异常等位基因CAG重复序列为37~200次。基于毛细管电泳的片段分析显示,2例样本扩展片段CAG重复序列分别为57和34次。对A20⁃13样本扩展重复序列进行2次切胶回收克隆测序,第1次获得的CAG重复序列为85次,第2次选取3个不同菌落进行克隆测序,CAG重复序列分别为69、74和75次;对A20⁃13⁃2样本扩展重复序列进行的克隆测序,CAG重复序列为45次。
三、SCA8基因CTA/CTG重复序列分析
对7例SCA8型患者的样本进行基于毛细管电泳的片段分析和克隆测序(表1)。SCA8正常等位基因重复序列为15~37次,异常等位基因重复序列>100次,某些个体携带中间重复等位基因。基于毛细管电泳的片段分析显示,7例样本扩展重复序列分别为99、111、104、92、89、104和75次。克隆测序显示,7例样本扩展重复序列分别为97、116、104、90、90、102和76次。

图1 SCA2基因毛细管电泳峰形图显示,正常片段呈单一锐利主峰(黑箭头所示),扩展重复序列呈簇状低矮爪形峰(红箭头所示)Figure 1 Capillary electrophoresis of SCA2 gene. Normal amplicon showed a single sharp main peak(black arrow indicates)and expanded repeats showed a cluster of low claw⁃shaped peak(red arrow indicates).
四、SCA17基因CAG重复序列分析
对2例SCA17型患者(编号:A20⁃33和A19⁃45)的样本进行基于毛细管电泳的片段分析和克隆测序(表1)。SCA17正常等位基因CAG重复序列25~42次,异常等位基因CAG重复序列为45~66次。基于毛细管电泳的片段分析显示,2例样本短片段/扩展片段CAG重复序列分别为37/50和36/45次。克隆测序显示,A20⁃33样本3个菌落短片段重复序列为37/36/36次、扩展片段重复序列为51/50/52次,A19⁃45样本2个菌落短片段重复序列为36/35次、扩展片段重复序列为45/44次。
讨 论
三核苷酸重复序列动态突变是一种特殊的突变方式,多种疾病表现为患病后代的异常重复序列进一步扩展,导致发病年龄提前、疾病进展加快,严重影响患者生活质量和婚育。我们研究团队多年来致力于对此类突变患者进行常规检测,包括基于毛细管电泳的片段分析和克隆测序,积累较为丰富的临床经验。
基于毛细管电泳的片段分析有以下几方面特点:(1)稳定性佳,如本研究SCA7基因A20⁃13样本,我们研究团队曾对多个样本进行重复分析,结果完全一致。(2)存在问题,相关基因CAG重复区及其两侧GC含量升高,导致PCR扩增产物迁移行为不同于碱基随机组成的片段,毛细管电泳采用变性后的单链DNA,可以在电泳时产生“发卡”形二级结构,形成局部不完全碱基互补的双链DNA,导致DNA链变短,如果以GC含量相对均衡的内参照物作为对照,判读的单链DNA长度小于 实 际 长 度[4⁃6]。 本 研 究 SCA2 和 SCA7 基 因CAG重复序列基于毛细管电泳的片段分析结果均小于克隆测序结果。陈朴等[4]的研究显示,SCA2和SCA7基因PCR扩增产物的高迁移率与其重复序列两侧GC含量升高密切相关,究其原因可能是由于侧翼GC含量升高可以增加单链DNA形成更加复杂二级结构的机会,从而使DNA链进一步缩短,对其迁移率产生更加显著的影响。
克隆测序有以下几方面特点:(1)其优点是可视性佳,直观。(2)其缺点是步骤繁琐;切胶时难以完全分开短片段和长片段;克隆过程中DNA复制忠实性较低,结果不稳定。本研究对3例SCA2基因样本行多个克隆测序,结果均不同,且出现多种碱基突变;对SCA7基因同一样本的扩展重复序列进行2次切胶克隆测序,结果亦存在明显差异。克隆测序结果的不稳定性可能与大肠杆菌中序列异常重组有关,使三核苷酸产生不稳定性扩增[7⁃9]。转入大肠杆菌的重组双链 DNA 在 CTG⁃CAG处发生DNA双链断裂,产生2个缺口,遂在断裂缺口处以其他双链DNA为模板,在大肠杆菌复制和修复功能作用下经组合形成新的双链DNA。与断裂前相比,重新组合的双链DNA CAG重复序列扩展。在基因重组过程中,CAG重复序列越多、越易形成“发卡”形结构以稳定 CAG 序列[7,10⁃13]。
采用基于毛细管电泳的片段分析和克隆测序检测SCA2和SCA7基因扩展重复序列存在显著差异,而SCA8和SCA17基因扩展重复序列差异不显著,究其原因,可能是由于SCA2和SCA7基因CAG重复序列较单纯,在代间传递过程中易扩展[14⁃15];SCA8和SCA17基因重复序列不单纯,尤其是SCA17基因,重复序列包含多个胞嘧啶⁃腺嘌呤⁃腺嘌呤(CAA)序列,可能对重复序列具有稳定作用[16⁃17]。
通过长期的大量实验和数据积累,我们认为,基于毛细管电泳的片段分析是检测基因动态突变的稳定、可靠方法,在判读重复序列时存在的差异是可以预知的,而且方法便于操作,适用于常规基因检测。与此同时,我们结合家系患者的临床表型特点和代间传递规律,对基于毛细管电泳的片段分析结果进行判读,以保证基因检测结果的准确性。
近年来,基因测序技术发展迅速,但是目前应用较为广泛的二代基因测序由于读长所限,难以对三核苷酸重复序列动态突变进行检测。2013年,Loomis等[18]采用单分子实时测序(SMRT)首次获得脆性X染色体综合征(FRAX)基因三核苷酸重复序列的全长信息,为三核苷酸重复序列动态突变的检测提供新的技术。
[1]Orr HT,Zoghbi HY.Trinucleotide repeat disorders[J].Annu Rev Neurosci,2007,30:575⁃621.
[2]Ellegren H.Heterogeneous mutation processes in human microsatellite DNA sequences[J].Nat Genet,2000,24:400 ⁃402.
[3]Bauer PO, Nukina N. The pathogenic mechanisms of polyglutamine diseases and current therapeutic strategies[J].J Neurochem,2009,110:1737⁃1765.
[4]Chen P,Ma MY,Shang HF,Su D,Zhang SZ,Yang Y.Copy number variation of trinucleotide repeat in dynamic mutation sites of autosomal dominant cerebellar ataxias related genes[J].Zhonghua Yi Xue Yi Chuan Xue Za Zhi,2009,26:626⁃633[.陈朴,马明义,商慧芳,苏丹,张思仲,杨元.常染色体显性小脑性共济失调致病基因动态突变位点三核苷酸重复变异的研究[J].中华医学遗传学杂志,2009,26:626⁃633.]
[5]Nakagawa T,Ueda M,Baba Y.Separation of triplet repeat DNA by capillary electrophoresis and the conformational analysis by atomic force microscope[J].Nucleic Acids Symp Ser,2000,44:67⁃68.
[6]Kiba Y,Baba Y.Unusual capillary electrophoretic behavior of triplet repeat DNA[J].J Biochem Biophys Methods,1999,41:143⁃151.
[7]Zheng QJ,Gan SR,Wang N,Wu ZY.The reliability of cloning⁃sequencing to detect the number of trinucleotide repeats[J].Zhonghua Shen Jing Ke Za Zhi,2010,43:659⁃663[.郑巧娟,甘世锐,王柠,吴志英.克隆测序方法检测三核苷酸重复次数的可靠性[J].中华神经科杂志,2010,43:659⁃663.]
[8]Jakupciak JP,Wells RD.Gene conversion (recombination)mediates expansions of CTG·CAG repeats[J].J Biol Chem,2000,275:40003⁃40013.
[9]Jakupciak JP,Wells RD.Genetic instabilities of triplet repeat sequences by recombination[J].IUBMB Life,2000,50:355⁃359.
[10]Wells RD,Parniewski P,Pluciennik A.Small slipped register genetic instabilities in Escherichia coli in triplet repeat sequences associated with hereditary neurological diseases[J].J Biol Chem,1998,273:19532⁃19541.
[11]Usdin K,Woodford KJ.CGG repeats associated with DNA instability and chromosome fragility form structures that block DNA synthesis in vitro[J].Nucleic Acid Res,1995,23:4202 ⁃4209.
[12]Mitas M,Yu A,Dill J,Haworth IS.The trinucleotide repeat sequence d(CGG)15 forms a heat⁃stable hairpin containing Gsyn.Ganti base pairs[J].Biochemistry,1995,34:12803 ⁃12811.
[13]Gacy AM,Goellner G,Juranic N,Macura S,McMurray CT.Trinucleotide repeats thatexpand in human disease form hairpin structures in vitro[J].Cell,1995,81:533⁃540.
[14]Wang CR,Jiang H.Progress study on the mechanism of CAG repeats dynamic mutation in polyQ disease[J].Zhongguo Xian Dai Shen Jing Ji Bing Za Zhi,2012,12:363⁃366.[王 春 荣, 江宏.多聚谷氨酰胺病CAG重复序列动态突变机制研究进展[J].中国现代神经疾病杂志,2012,12:363⁃366.]
[15]Ramos EM,Martins S,Alonso I,Emmel VE,Saraiva⁃Pereira ML,Jardim LB,Coutinho P,Sequeiros J,Silveira I.Common origin of pure and interrupted repeat expansions in spinocerebellar ataxia type 2(SCA2)[J].Am J Med Genet B Neuropsychiatr Genet,2010,153B:524⁃531.
[16]Gao R,Matsuura T,Coolbaugh M,Zühlke C,Nakamura K,Rasmussen A,Siciliano MJ,Ashizawa T,Lin X.Instability of expanded CAG/CAA repeats in spinocerebellar ataxia type 17[J].Eur J Hum Genet,2008,16:215⁃222.
[17]Tomiuk J,Bachmann L,Bauer C,Rolfs A,Schöls L,Roos C,Zischler H,Schuler MM,Bruntner S,Riess O,Bauer P.Repeat expansion in spinocerebellar ataxia type 17 alleles of the TATA⁃box binding protein gene:an evolutionary approach[J].Eur J Hum Genet,2007,15:81⁃87.
[18]Loomis EW,Eid JS,Peluso P,Yin J,Hickey L,Rank D,McCalmon S,Hagerman RJ,Tassone F,Hagerman PJ.Sequencing the unsequenceable:expanded CGG⁃repeat alleles of the fragile X gene[J].Genome Res,2013,23:121⁃128.

