系统性红斑狼疮小鼠肠道微生物与抗dsDNA抗体水平的相关性
邹桂香,段新旺*,牛海涛
(1. 南昌大学第二附属医院 风湿免疫科,南昌 330006; 2. 中国医学科学院医学实验动物研究所,北京协和医学院比较医学中心,北京 100021)
系统性红斑狼疮(systemic lupus erythematosus,SLE,简称狼疮),是以抗核抗体为代表的多种致病性自身抗体增加、免疫复合物异常沉积及免疫细胞功能失调而引起的一种慢性多器官损害的自身免疫性疾病。作为一种具有代表性的系统性的自身免疫性结缔组织病,发病具有明显的性别差异,男女比例大体上为1∶9[1]。虽然国际上对狼疮的研究很多,但确切的发病机制仍不清楚[2]。研究普遍认为其发病机制与基因遗传、免疫失调、激素及环境等多种因素相关[3]。狼疮是一种多基因易感性疾病,有明显的家系聚集性。相关研究中报道,狼疮患者中同卵双胞胎共同患病率为24%~57%,远高于异卵双胞胎共同患病率的2%[4-5]。所以同卵双胞胎中有一方发病,另一方的不发病的概率也有40%以上,环境因素可能起到了重要作用。
系统性红斑狼疮小鼠模型B6.NZMSle1/2/3(TC小鼠)是一种是由C57BL/6 J小鼠与狼疮NZM2410小鼠杂交后建立的、同时携带三种狼疮易感基因(Sle1/Sle2/Sle3)的纯合遗传性自发狼疮小鼠,该模型可模拟人类狼疮的自身免疫特征,其发病主要特征表现为抗dsDNA阳性[6]。然而我们的研究发现虽然TC小鼠基因型完全相同,但有很少一部分雌鼠不发病,表现为抗dsDNA阴性,该部分雌鼠不发病的原因至今未明。相关研究提示,基因遗传因素虽然在狼疮的发病中发挥重要作用,但环境因素也与狼疮的发生发展密切相关[7]。我们推测肠道微生物作为环境因素的重要组成部分,与TC小鼠的dsDNA水平相关。相关报道,狼疮发病的危险因素包括:使用抗生素、寄生虫药物或胃肠道疾病相关药物等[8]。上述危险因素提示肠道微生物菌群结构与狼疮发病高度相关。
肠道微生物菌群参与了人体的代谢、营养和免疫调节功能[9-10]。通过与相邻的肠道黏膜环境直接相互作用,肠道微生物菌群可影响肠道黏膜的通透性,进而参与局部和全身的免疫炎症活动[11]。越来越多的证据表明,胃肠道中的微生物菌群影响啮齿动物模型中自身免疫疾病的发生发展[12]。
本研究通过检测同周龄TC雌鼠的抗dsDNA抗体,比较dsDNA阳性组与dsDNA阴性组的肠道微生物菌群结构差异,探讨狼疮TC小鼠肠道微生物菌群结构与抗dsDNA抗体的关系。
1 材料与方法
1.1 材料
1.1.1 实验动物
TC小鼠为纯合狼疮小鼠(B6.NZMSle1/2/3),由美国佛罗里达大学Morel Laurence教授实验室赠送并在中国医学科学院医学实验动物研究所SPF3级动物房饲养及繁殖【SYXK(京)2015-0035】,在饲养及繁殖期间,使用小鼠标准饲料和洁净饮用水(由中国医学科学院医学实验动物研究所提供)。饲养环境:相对湿度40%~70%,温度20~ 26℃,昼夜交替各半。所有实验操作符合实验动物伦理要求(伦理审批号:ILAS— FG-2015-016)。
1.1.2 试剂材料
ELISA法检测抗dsDNA抗体实验的相关试剂:包被抗原dsDNA,购于Sigma公司;标记抗体Goat Anti-Mouse IgG,购于Southern Biotech公司;酶底物显色剂Phosphatase substrate, 购于Sigma公司。小鼠粪便DNA提取试剂盒,购于天根生化科技(北京)有限公司。
1.2 方法
1.2.1 抗dsDNA抗体检测
30周龄TC雌性小鼠35只,体重25~30 g,脸颊采血后离心,取血清。ELISA检测血清中抗dsDNA抗体,具体操作步骤参考文献[13]。
1.2.2 小鼠粪便收集
无菌环境下收集TC小鼠的新鲜粪便,每份标本粪便均不少于3粒,快速置于灭菌离心管中,-80℃保存。
1.2.3 DNA提取和测序
按照说明书,使用粪便DNA提取试剂盒对小鼠粪便样本中的微生物总DNA进行提取;利用琼脂糖凝胶电泳检测DNA的纯度和浓度,将基因组DNA样品稀释至1 ng/μL作为模板,针对细菌的16S rRNA基因高变区V3~V4区进行PCR扩增。PCR产物检验合格后,通过混样、建库及文库质控,将检测合格的文库进行高通量测序。
1.3 数据分析与统计学方析
将原始序列进行碱基去除过滤,再去除嵌合体序列得到最终有效的序列。根据最终有效序列的相似性对微生物菌群进行聚类,相似性为97%的序列聚类成为OTUs(operational taxonomic units)。采用OTUs聚类分析及物种注释、多样性指数分析及组间差异物种分析等对两组TC小鼠微生物菌群进行生物学信息分析。
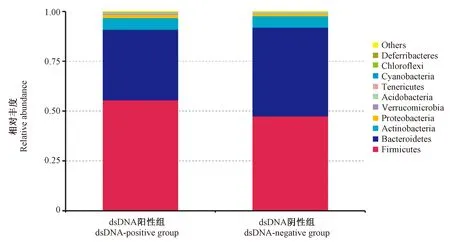
注:不同颜色代表不同的菌门。图2 组间门水平相对丰度结构对比图Note. Different color represents different phyla.Fig.2 Relative abundance of the gut microbiota between groups at phylum level
微生物菌群数据采用Qiime软件计算Shannon,Simpson指数及UniFrac距离并采用R软件(Version 2.15.3)进行Alpha多样性指数及Beta多样性指数组间差异分析,进行t检验,及作图。血清检测抗体数据采用GraphPad Prism 6软件进行t检验。所有数据均以P<0.05为差异有显著性的标准。
2 结果
2.1 抗dsDNA抗体检测
TC小鼠的dsDNA检测结果如图1所示:同龄TC小鼠多数为dsDNA阳性(dsDNA-pos.),然而很少部分TC小鼠为dsDNA阴性(dsDNA-neg.)。
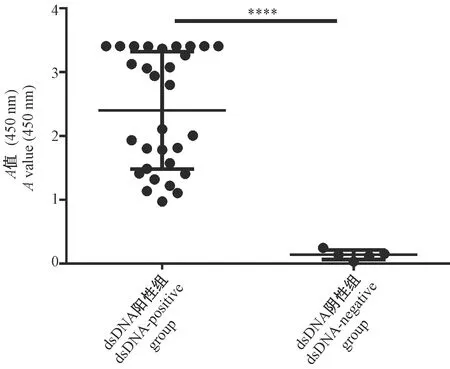
注:dsDNA阳性的TC小鼠(n=30); dsDNA阴性的TC小鼠(n=5);****P<0.0001。图1 TC小鼠dsDNA浓度Note. dsDNA-positive lupus TC mice (n=30); dsDNA-negative lupus TC mice (n=5).****P<0.0001.Fig.1 dsDNA concentration in the TC mice
2.2 肠道微生物菌群生物学信息分析
2.2.1 肠道微生物物种丰度分析
为了解两组样本的物种丰度含量,根据不同分类水平的物种注释及丰度信息,选取两组在门水平上平均丰度排名前10的物种,根据其在每组中的丰度信息,得到门水平上物种相对丰度柱形图,可直观查看每组样本在门水平上相对丰度较高的物种及其比例。两组组间门水平相对丰度信息如图2所示:dsDNA阳性组TC小鼠Firmicutes(厚壁菌门)的物种丰度含量高于dsDNA阴性组,dsDNA阳性组TC小鼠Bacteroidetes(拟杆菌门)的物种丰度含量低于dsDNA阴性组,提示厚壁菌门与拟杆菌门和TC小鼠dsDNA水平有一定相关性。
2.2.2 肠道微生物菌群多样性比较分析
肠道微生物菌群多样性可用Alpha多样性指数和Beta多样性指数进行分析。通过Alpha多样性指数分析,本研究未发现dsDNA阳性与dsDNA阴性组TC小鼠组间物种多样性均值差异有显著性。
Beta多样性是对微生物群落构成的比较分析,评估不同组样本微生物群落的差异。如图3所示:利用OTUs的丰度信息构建UniFrac距离(Unweighted UniFrac,Beta多样性指标之一),通过t检验,dsDNA阳性组的物种群落多样性显著低于dsDNA阴性组TC小鼠(P<0.05)。提示TC小鼠的微生物菌群多样性与抗dsDNA抗体水平相关。

图3 Beta多样性组间差异箱形图Fig.3 Box graph of the β-diversity of group differences
2.2.3 肠道微生物菌群结构组间差异物种分析
通过在门、纲、目、科、属水平上对两组测序样本进行比较分析,发现dsDNA阳性组与dsDNA阴性组TC小鼠分别在门水平的Chloroflexi(绿弯菌门);纲水平的Betaproteobacteria(β变形菌纲)、Deltaproteobacteria(δ变形菌纲)和Ktedonobacteria(纤线杆菌纲);目水平的Burkholderiales(伯克氏菌目)、Desulfovibrionales(脱硫弧菌目)和Ktedonobacterales(纤线杆菌目);科水平的Alcaligenaceae(产碱菌科)和Desulfovibrionaceae(脱硫弧菌科);属水平Parasutterella(副萨特氏菌属)、Desulfovibrio(脱硫弧菌属)上差异均有显著性(图4),提示TC小鼠的肠道微生物菌群结构可能与抗dsDNA抗体水平相关。
3 讨论
3.1 肠道微生物菌群多样性与系统性红斑狼疮的关系
相关研究分析,肠道微生物菌群整体多样性降低将增加自身免疫性疾病的风险[14]。本研究发现dsDNA阳性的TC小鼠和dsDNA阴性的TC小鼠之间在Beta多样性指数中差异有显著性,且dsDNA阳性组TC小鼠的Unweighted UniFrac数值显著低于dsDNA阴性组,说明dsDNA阳性组的肠道微生物菌群多样性指数降低可能与系统性红斑狼疮的发病相关,提示肠道微生物菌群多样性降低与狼疮抗dsDNA抗体水平相关。
3.2 肠道微生物菌群结构与系统性红斑狼疮的关系
本研究肠道微生物菌群的门、纲、目、科、属不同物种分类水平中,既有益生菌如乳杆菌,也有与宿主共存的机会致病菌如大肠杆菌,还有致病菌如金葡菌等。且dsDNA阳性组与阴性组TC小鼠在肠道微生物菌群的各个分类水平差异均有显著性,提示肠道微生物菌群结构与狼疮抗dsDNA抗体水平相关。
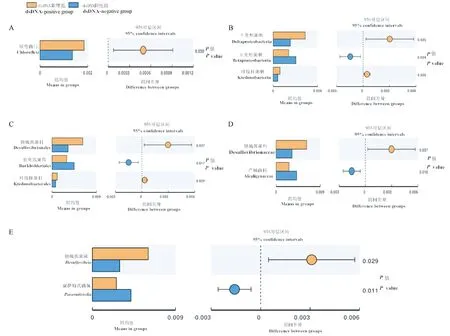
注:橙色表示dsDNA阳性组TC小鼠,蓝色表示dsDNA阴性组TC小鼠。A图,左侧表示绿弯菌门水平;B图,左侧表示β变形菌纲、δ变形菌纲和纤线杆菌纲水平;C图,左侧表示伯克氏菌目、脱硫弧菌目和纤线杆菌目水平;D图,左侧表示产碱菌科和脱硫弧菌科水平;E图,左侧表示副萨特氏菌属和脱硫弧菌属水平;所有A、B、C、D、E图,右侧均为该图左侧物种在两组组间差异的统计学分析结果P值,P<0.05为差异具有显著性。图4 门、纲、目、科、属水平组间差异物种对比图Note. Orange indicates dsDNA positive TC mice and blue indicates dsDNA negative TC mice. The left side of Panel A shows the level of Chloroflexi. The left side of Panel B shows the levels of Betaproteobacteria, Deltaproteobacteria, and Ktedonobacteria. The left side of Panel C shows the levels of Burkholderiales and Desulfovibrionales. The left side of Panel D shows the levels of Alcaligenaceae and Desulfovibrionaceae. The left side of Panel E shows levels of Parasutterella and Desulfovibrio. All of the right side of Panels A, B, C, D, E are P value of statistical analysis between the two groups. Significant difference was shown as P<0.05.Fig.4 Comparison chart of differential species of gut microbiota between the groups at phylum, class, order, family, and genus levels
肠道微生物菌群中厚壁菌门和拟杆菌门在本研究中两组占90%以上,属于优势菌群。厚壁菌门和拟杆菌门的比例被称为F/B比率,可作为评估肠道微生物菌群失调的生物标志物[15],且F/B比率被认为与人类肠道微生物菌群成分有显著的相关性[16]。有文献报道,狼疮患者,其肠道微生物菌群F/B比率低于健康人[17]。但有另外文献报道,高血压和肥胖患者肠道微生物菌群F/B比率高于健康人[18-19]。尽管肠道微生物菌群F/B比率被认为是肠道微生物菌群紊乱的标志,但F/B比率的升高或降低与疾病的相关性仍存在很大争议。本研究dsDNA阳性组TC小鼠的F/B比率高于dsDNA阴性组,与人类狼疮报道的F/B比率低于健康人群差异较大,可能与TC小鼠的狼疮易感基因相关,有待进一步深入研究。
相关研究发现,狼疮小鼠病情的严重程度与肠道中乳酸杆菌的消耗和毛螺菌的增加有一定相关性[20]。本研究中发现Lachnospiraceae_NK4A136_group(毛螺菌)在dsDNA阳性组TC小鼠含量高于dsDNA阴性组, 提示毛螺菌的增加与抗dsDNA水平正相关。Proteobacteria(变形菌门)在两组中含量在1.2%~1.6%左右,在该菌门下,dsDNA阳性组与dsDNA阴性组TC小鼠在属水平副萨特氏菌属和脱硫弧菌属差异具有显著性。变形菌门大多数都是致病菌,虽然在肠道微生物菌群中内含量较少,但是本研究中两种差异具有显著性的菌属均属于变形菌门,提示变形菌门在狼疮的发生发展中发挥了一定作用。本研究的结果为进一步深入研究肠道微生物菌群与狼疮发病机制的关系提供了依据。
参考文献(References)
[1] Alarcón-Segovia D, Alarcón-Riquelme ME, Cardiel MH, et al. Familial aggregation of systemic lupus erythematosus, rheumatoid arthritis, and other autoimmune diseases in 1,177 lupus patients from the GLADEL cohort [J]. Arthritis Rheum, 2005, 52(4):1138-1147.
[2] Crispín JC, Liossis SN, Kis-Toth K, et al. Pathogenesis of human systemic lupus erythematosus: recent advances [J]. Trends Mol Med, 2010, 16(2): 47-57.
[3] Tsokos GC. Systemic lupus erythematosus [J]. N Engl J Med, 2011, 365(22): 2110-2121.
[4] Deapen D, Escalante A, Weinrib L, et al. A revised estimate of twin concordance in systemic lupus erythematosus [J]. Arthritis Rheum, 1992, 35(3): 311-318.
[5] Block SR, Winfield JB, Lockshin MD, et al. Studies of twins with systemic lupus erythematosus. A review of the literature and presentation of 12 additional sets [J]. Am J Med, 1975, 59(4): 533-552.
[6] Morel L, Croker BP, Blenman KR, et al. Genetic reconstitution of systemic lupus erythematosus immunopathology with polycongenic murine strains [J]. Proc Natl Acad Sci U S A, 2000, 97(12): 6670-6675.
[7] Luppi P, Rossiello MR, Faas S, et al. Genetic background and environment contribute synergistically to the onset of autoimmune diseases [J]. J Mol Med (Berl), 1995, 73(8): 381-393.
[8] 叶冬青,胡以松,李向培,等. 系统性红斑狼疮发病的环境因素及其与几种趋化因子基因的交互作用 [J]. 中华流行病学杂志, 2004,25(11): 949-953.
Ye DQ, Hu YS, Li XP, et al. Influences of environmental factors and interaction of several chemokines gene-environmental on systemic lupus erythematosus [J]. Chin J Epidemiol, 2004, 25(11): 949-953.
[9] McLean MH, Dieguez D Jr, Miller LM, et al. Does the microbiota play a role in the pathogenesis of autoimmune diseases? [J]. Gut, 2015, 64(2): 332-341.
[10] Sathyabama S, Khan N, Agrewala JN. Friendly pathogens: prevent or provoke autoimmunity [J]. Crit Rev Microbiol, 2014, 40(3): 273-280.
[11] Cox AJ, West NP, Cripps AW. Obesity, inflammation, and the gut microbiota [J]. Lancet Diabetes Endocrinol, 2015, 3(3): 207-215.
[12] Vieira SM, Pagovich OE, Kriegel MA. Diet, microbiota and autoimmune diseases [J]. Lupus, 2014, 23(6): 518-526.
[13] Niu H, Sobel ES, Morel L. Defective B-cell response to T-dependent immunization in lupus-prone mice [J]. Eur J Immunol, 2008, 38(11): 3028-3040.
[14] Petersen C, Round JL. Defining dysbiosis and its influence on host immunity and disease [J]. Cell Microbiol, 2014, 16(7): 1024-1033.
[15] Mariat D, Firmesse O, Levenez F, et al. The Firmicutes / Bacteroidetes ratio of the human microbiota changes with age [J]. BMC Microbiol, 2009, 9: 123.
[16] Ley RE, Turnbaugh PJ, Klein S, et al. Microbial ecology: human gut microbes associated with obesity [J]. Nature, 2006, 444(7122): 1022-1023.
[17] Hevia A, Milani C, López P, et al. Intestinal dysbiosis associated with systemic lupus erythematosus [J]. MBio, 2014, 5(5):e01548-14.
[18] Yang T, Santisteban MM, Rodriguez V, et al. Gut dysbiosis is linked to hypertension [J]. Hypertension, 2015, 65(6): 1331-1340.
[19] Sanz Y, Moya-Pérez A. Microbiota, inflammation and obesity [J]. Adv Exp Med Biol, 2014, 817: 291-317.
[20] Zhang H, Liao X, Sparks JB, et al. Dynamics of gut microbiota in autoimmune lupus [J]. Appl Environ Microbiol, 2014, 80(24): 7551-7560.

