组蛋白赖氨酸甲基转移酶DOT1L抑制剂分子水平高通量筛选模型的建立
徐 威,苏明波,周宇波
(1.上海大学生命科学学院,上海200444;2.国家新药筛选中心,上海201203)
组蛋白的共价修饰主要发生在赖氨酸(K)和精氨酸(R)残基上,包括甲基化、乙酰化、磷酸化、泛素化等,是一种重要的表观遗传调控作用机制.赖氨酸的甲基化和去甲基化在组蛋白修饰中尤为常见,主要由组蛋白去甲基化酶(histone dememethylase,HDM)和组蛋白甲基转移酶(histone methyltransferase,HMT)共同调节细胞内组蛋白的甲基化水平[1].大量研究表明:组蛋白的共价修饰作用参与众多基因的表达调控,促进细胞生长发育,调节机体新陈代谢,参与相关疾病肿瘤的发生和发展过程,在表型和基因型之间承载着重要的桥梁作用[2].
类端粒沉默干扰体1(disruptor of telomeric silencing 1-like,DOT1L)是一种作用在组蛋白H3第79位赖氨酸(H3K79)上的甲基转移酶,是目前所知在酵母菌和哺乳动物体内唯一作用于该位点的甲基转移酶[3].DOT1L不含大部分甲基转移酶所共有的在进化上极端保守的SET结构域.除了调控某些基因的转录激活程序以外,DOT1L还参与DNA的修复、细胞的生长分化以及细胞周期的调控等[4].在体内条件下,DOT1L的敲除将导致小鼠胚胎致死.DOT1L在小鼠胚胎干细胞的生长发育过程、心脑血管的发生和发展过程以及胚胎软骨组织的形成中都发挥着重要作用[5].大量研究结果表明:由DOT1L催化调控的H3K79位点单,双甲基化程度的异常与重组型混合系白血病(mixed lineage leukemia,MLL)的发生与发展密切相关[6].在重组型MLL细胞中,染色质的异常转位导致MLL基因与其融合伴侣(AF4,AF9或AF10)发生融合,使DOT1L异常聚集,促进了H3K79位点的高度甲基化,导致诸如HOXA9和MEIS1的癌基因过量表达,促进了细胞的恶性增殖[7].重组型MLL可以发展成急性髓性白血病(acute myelocytic leukemia,AML)或者急性淋巴细胞白血病(acute lymphoblastic leukemia,ALL),这表明DOT1L是潜在的白血病治疗靶点[8].基于计算机水平的辅助测试新技术的出现和发展,极大地提高了靶点药物筛选的时效性和可靠性[9].
目前在研的DOT1L抑制剂有3种:①2011年Epizyme公司推出了第一种DOT1L抑制剂EPZ004777,它是底物SAM的竞争性抑制剂,分子水平IC50约为0.4 nmol/L,选择性较好,能显著抑制重组型MLL细胞系和MLL-AF4,MLL-AF9型小鼠造血细胞的增殖[10];②2013年Epizyme公司推出了第二种DOT1L抑制剂EPZ-5676,其对DOT1L靶点有着更为强烈的抑制效果,分子水平IC50为80 pmol/L,选择性较好,在抑制MLL-AF4重组型细胞MV-4-11的IC50为3.5 nmol/L,对H3K79位点双甲基化程度抑制的IC50为3 nmol/L,目前处于临床一期研究阶段[11];③2012年加拿大多伦多大学结构基因组学会推出了另一种DOT1L小分子抑制剂SGC0946,分子水平IC50为300 pmol/L,对H3K79位点双甲基化程度抑制的IC50为8.8 nmol/L,选择性较好,对HOXA9和MEIS1基因的抑制效果更为显著[12].
1 材料与方法
1.1 材料
SAM缓冲液:5 mmol/L硫酸,10%乙醇溶液.反应缓冲液:50 mmol/L三羟甲基氨基甲烷盐酸,pH=8.0,150 mmol/L氯化钠,3 mmol/L氯化镁,0.1%牛血清白蛋白.高盐缓冲液:50 mmol/L三羟甲基氨基甲烷盐酸,pH=7.4,1 mmol/L氯化钠,0.1%Tween-20,0.3%多聚赖氨酸.检测缓冲液:25 mmol/L羟乙基哌嗪乙硫磺酸,pH=7.4,0.01%Tween-20.
1.2 实验方法
本实验采用PE公司提供的AlphaLISA DOT1L Histone H3 Lysine-N-methyltransferase Assay试剂盒检测H3K79位点甲基转移酶DOT1L的活性,DOT1L小分子抑制剂高通量筛
选(high throughout screening,HTS)模型反应原理如图1所示.

表1 实验试剂Table 1 Experiment reagents
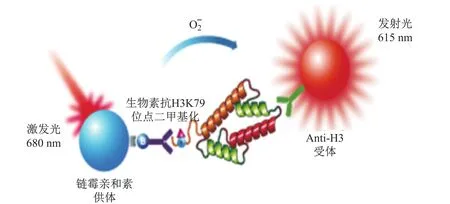
图1 DOT1L小分子抑制剂高通量筛选模型反应原理Fig.1 Reaction mechanism of DOT1L small molecule inhibitors HTS model
由图1可以看出,组蛋白核小体H3K79位点能够在组蛋白甲基转移酶DOT1L的催化作用下,由低甲基化程度向高甲基化程度转化.AlphaLISA能够检测核小体上甲基化修饰的H3K79位点的二甲基化(H3K79me2)程度.通过甲基化抗体将受体珠和供体珠连接在一起,当发生甲基化时,给予680 nm波长的激发光,供体珠能够产生高能态的单线氧.高能态的单线氧能够进一步激发受体珠发射出520~620 nm波长的荧光,通过检测荧光强度来反映甲基化的H3K79me2含量.
2 结果
2.1 最佳反应酶浓度和反应时间的确定
AlphaLISA活性检测是一种基于终点法的测活方法,需要保证检测时DOT1L甲基转移酶的酶活始终保持在初速度反应水平,即在整个活性检测过程中DOT1L的酶活始终保持在线性范围内.
为了探索最佳反应酶浓度和反应时间,本工作设计了如下反应实验体系.在White Opaque OptiPlateTM-384孔板中依次加入1µL 10倍缓冲液,2µL终浓度依次为100,25,12.5,3.125,1.562 5,0 nmol/L的DOT1L酶.2µL 20µmol/L的S-腺苷甲硫氨酸(S-adenosyl methionine,SAM)和0.150 ng/µL寡聚核小体混合物,反应时长为180,120,60,30,0 min.达到反应时间后,用高盐溶液终止反应,15 min后加入受体珠和抗体的混合液,60 min后加入供体珠,30 min后进行Evision检测.实验结果如图2所示.

图2 反应体系中最佳DOT1L酶浓度和最佳反应时间的检测Fig.2 Detection of the best enzyme concentration and reaction time
由图2可以看出,随着酶浓度的升高和反应时间的延长,接收到的信号值逐渐增强.但是,在高浓度DOT1L酶的催化作用下,整条曲线并非呈线性正相关.综合考虑节约成本、反应体系的稳定性和实验的可操作性,最终选定5 nmol/L的DOT1L酶浓度,反应时长180 min.
2.2 寡聚核小体最佳浓度的确定
寡聚核小体是DOT1L高通量筛选体系建立实验中重要的底物之一,组蛋白甲基转移酶DOT1L能够将核小体组蛋白H3第79位的赖氨酸甲基化,实验依赖于完整的核小体结构.为探索最佳寡聚核小体反应浓度,选择终浓度为0.150,0.125,0.100,0.075,0.050,0.025 ng/µL的寡聚核小体,5 nmol/L的DOT1L酶浓度,20µmol/L的SAM,反应时长180 min,其他反应条件不变.实验结果如图3所示.
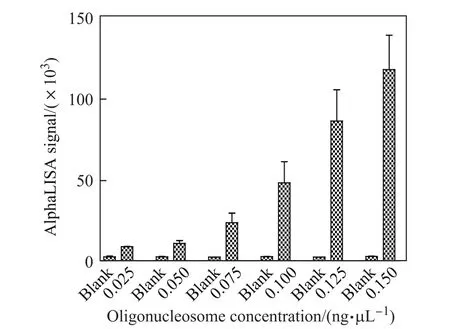
图3 反应体系中底物核小体最佳浓度的检测Fig.3 Detection of the best oligonucleosome concentration
由图3可以看出:在一定浓度范围内,接收到的信号值随寡聚核小体浓度的升高而增大.实验本底维持在一个相对较低的水平,证明了实验的稳定性.综合考虑高通量筛选的需求和节约成本的原则,最终选定寡聚核小体的终浓度为0.150 ng/µL.
2.3 底物SAM反应Km值的测定和最佳浓度的确定
组蛋白去甲基转移酶DOT1L在适宜条件下可催化H3K79的甲基化水平升高反应,该反应需要一个甲基的供体SAM.在底物SAM存在的条件下,DOT1L将SAM上的甲基转移到H3K79位的赖氨酸上,完成催化反应.为检测SAM反应的Km值,依次选择终浓度为0.1,0.3,0.9,2.7,8.1,24.3,72.9和218.7µmol/L的SAM,5 nmol/L的DOT1L酶浓度,0.150 ng/µL寡聚核小体,反应时长180 min,其他反应条件不变.实验结果如图4所示.

图4 反应体系中底物SAM最佳浓度的检测Fig.4 Detection of the best SAM concentration
由图4可以看出,在一定范围内,接收到的信号值随SAM浓度的升高而增大,但达到一定浓度后,底物SAM的浓度不再成为体系中的限制因素,故信号值不再增大.最终检测SAM在反应体系中的Km值为13.46µmol/L,选定SAM的终浓度为20µmol/L.
2.4 多聚赖氨酸浓度的确定
多聚赖氨酸(poly-L-lysine,PL)是一种由20~30个赖氨酸残基聚合而成的多聚蛋白复合体,具有优良的抑菌防腐和食品保鲜功能.赖氨酸是人体必需氨基酸之一,是一种极性带正电荷的碱性氨基酸.大量赖氨酸通过肽键复合到一起,为反应体系提供了极强的极性环境,有利于反应信号的稳定和放大.PL最佳浓度检测实验结果如图5所示.
利用AlphaLISA方法检测DOT1L的酶活,需要保证实验组和对照组的差异足够大,形成的窗口足够宽,为进一步进行小分子抑制剂的筛选打下基础.由图5可以看出,PL的存在极大地提高了反应的窗口值,在最高为0.300 00%的PL浓度条件下,实验组信号值可以达到空白组信号值的13.2倍,并且随着PL浓度的降低而减小.根据实验结果,最终确定0.030 00%的PL浓度为最佳反应浓度.
2.5 阳性抑制剂EPZ-5676抑制效果的确定
通过前期实验已经基本确定了反应体系中各成分的最佳浓度与最佳反应时间,10倍缓冲液,终浓度为5 nmol/L的DOT1L酶,20µmol/L的SAM,0.150 ng/µL的寡聚核小体,0.030 00%的PL高盐溶液,反应时间3 h,体系的稳定性和可适用性需要进一步以阳性化合物作验证.EPZ-5676是一种国际公认的选择性较好的DOT1L二代抑制剂,是底物SAM的竞争型抑制剂,目前处于临床一期研究价段.以EPZ-5676为阳性化合物,检测其在分子水平的IC50值,结果如图6所示.
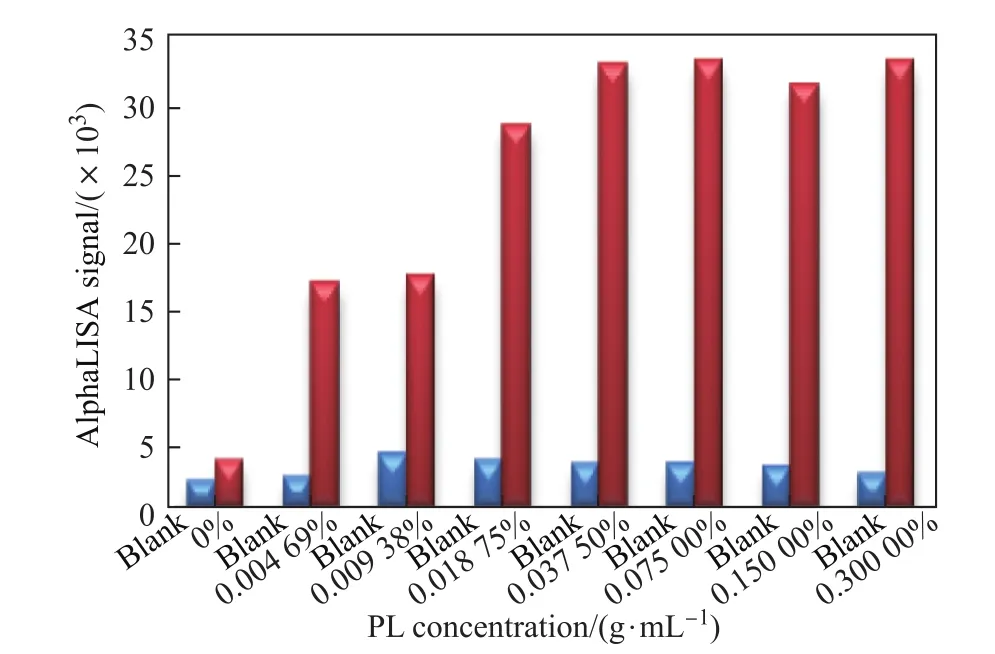
图5 反应体系中PL最佳浓度的检测Fig.5 Detection of the best PL concentration

图6 阳性化合物EPZ-5676的IC50检测Fig.6 Detection of the IC50 of positive compound EPZ-5676
利用优化后的DOT1L测活体系,检测不同浓度条件下EPZ-5676对DOT1L甲基转移酶的抑制作用(见图6).结果显示,阳性抑制剂EPZ-5676对DOT1L甲基转移酶的抑制IC50为0.48 nmol/L,与文献[11]报道基本一致,说明已成功建立组蛋白甲基转移酶DOT1L高通量筛选模型.
2.6 筛选系统的稳定性和可操作性
高通量筛选实验需要比较阳性对照组、未知样品组以及阴性对照组的检测结果,需要筛选数以万计的未知样品,耗费大量金钱和实验材料.故在高通量筛选实验开始之前,需要进行一些小样本容量的检测实验,以确定所建立的实验体系是否适用于后续的大规模筛选体系.
为了保证在机器加样条件下高通量筛选的稳定进行,本工作优化了加样过程,以确定是否适合进行高通量筛选.并考察了高通量筛选体系中Bravo加样系统以及酶活性检测过程中可能引起的系统变异程度.Z′因子是关于信号区间和变异程度的组合,是衡量高通量筛选稳定性与可靠性的重要指标,能综合反映信噪比和测量偏差的大小.一般认为,Z′>0.5的筛选体系适合进行高通量筛选.依据Z′因子计算公式,对所建立的DOT1L抑制剂筛选体系的Z′因子进行检测,结果为0.54(见图7),符合高通量筛选的质量控制要求,表明可以开展高通量筛选实验.

图7 Z′因子的测定Fig.7 Detection of the Z′factor
3 讨论
分子水平药物筛选过程是新药开发与后续体内评价的关键步骤,是一个通过应用系统化的实验手段从数以万计的候选化合物中筛选出对某一特定靶点具有较高抑制活性的化合物的过程.这一过程要求实验方法灵敏度高、特异性强、稳定性好、自动化程度高,同时要尽可能地降低成本.近年来随着能量共振转移技术的发展与成熟,多功能AlphaLISA法已逐渐成为被广泛应用的药物筛选分析法.经过充分的标记特异性和交叉反应验证后的AlphaLISA法,可广泛应用于表观遗传中特定的甲基化、乙酰基化修饰的快速检测.该方法稳定性好,特异性强,反应过程无需洗涤[13].AlphaLISA DOT1L Histone H3 Lysine-N-Methyltransferase Assay方法将完整的寡聚核小体结构作为底物,通过生物素链接的特异性识别H3K79位二甲基化程度的抗体,识别特定的表观遗传标记,链霉亲和素供体另一端携带的供体珠在接收到特定波长的激发光后将发射出高能态的单线氧.高能态单线氧被锚定在组蛋白H3C末端的受体珠接收后,释放出另一特定波长的反射光,通过接收这一反射光的强度达到检测组蛋白H3第79位赖氨酸二甲基化程度产物的目的.整个过程依赖于完整的空间核小体构象结构,但是也有一定缺陷.首先,这种检测方式不能排除荧光化合物的干扰;其次,如果化合物的作用方式并非在于抑制了酶的活性,而在于干扰了供体抗体与H3K79二甲基化位点的结合,或者干扰了受体抗体与H3的结合,都会导致假阳性的产生.在后续实验中应尽量避免这一结果带来的影响.
以高通量筛选为基础的先导化合物发现技术是近年来迅速发展并被不断优化的药物筛选分析新方法.高通量筛选以其快速、灵敏、规模大、成本低、稳定性好、操作简单、化合物用量少等特点而普遍受到药物研发者的青睐.本工作结合组蛋白甲基转移酶DOT1L小分子抑制剂自身的研发特点,通过建立以能量共振转移技术为基础的高通量筛选模型,发掘具有新颖结构的DOT1L小分子抑制剂,增强对于DOT1L靶点本身的认识,在理解和阐述新的相关机制的同时,发现潜在的候选化合物.另一方面,通过与组蛋白甲基转移酶家族其他抑制剂筛选模型进行比较分析,进一步评价化合物的选择性,筛选出抑制效果好、针对性强的DOT1L抑制剂,为后续临床实验打下坚实的基础.
4 结束语
本实验通过建立以能量信号共振转移为基础的组蛋白甲基转移酶DOT1L高通量筛选模型,进一步优化反应体系,确定最终的最佳反应DOT1L酶浓度为5 nmol/L,最佳底物SAM浓度为20µmol/L,最佳寡聚核小体浓度为0.150 ng/µL,PL浓度为0.030 00%,最佳反应时长为3 h.利用优化体系对DOT1L阳性抑制剂EPZ-5676进行IC50检测,得到IC50值为0.48 nmol/L,与已有文献报道相一致,说明EPZ-5676可以作为DOT1L小分子抑制剂高通量筛选模型的阳性化合物.为了确保机器自动加样的稳定性和准确性,本工作对所建立的筛选模型的Z′因子进行检测,结果为0.54,达到了高通量筛选模型的建立标准,表明已成功建立组蛋白甲基转移酶DOT1L小分子抑制剂高通量筛选模型.
参考文献:
[1]BANNISTER A J,KOUZARIDES T.Regulation of chromatin by histone modif i cations[J].Cell Research,2011,21(3):381-395.
[2]PORTELA A,ESTELLER M.Epigenetic modif i cations and human disease[J].Nat Biotech,2010,28(10):1057-1068.
[3]WONG M,POLLY P,LIU T.The histone methyltransferase DOT1L:regulatory functions and a cancer therapy target[J].American Journal of Cancer Research,2015,5(9):2823-2837.
[4]MIN J,FENG Q,LI Z,et al.Structure of the catalytic domain of human DOT1L,a non-SET domain nucleosomal histone methyltransferase[J].Cell,2003,112(5):711-723.
[5]WEN L,FU L,GUO X,et al.Histone methyltransferase DOT1L plays a role in postembryonic development in Xenopus tropicalis[J].FASEB Journal:Oきcial Publication of the Federation of American Societies for Experimental Biology,2015,29(2):385-393.
[6]STEIN E M,TALLMAN M S.Mixed lineage rearranged leukemia:pathogenesis and targeting DOT1L[J].Current Opinion in Hematology,2015,22(2):92-96.
[7]LIU W,DENG L,SONG Y,et al.DOT1L inhibition sensitizes MLL-rearranged AML to chemotherapy[J].PLoS ONE,2014,9(5):e98270.
[8]CHEN C W,ARMSTRONG S A.Targeting DOT1L and HOX gene expression in MLL-rearranged leukemia and beyond[J].Experimental Hematology,2015,43(8):673-684.
[9]朱晓华,冯玉田,章玉鉴.基于组件技术的虚拟仪器开发方法的研究[J].上海大学学报(自然科学版),1999,5(4):357-361.
[10]DAIGLE S R,OLHAVA E J,THERKELSEN C A,et al.Selective killing of mixed lineage leukemia cells by a potent small-molecule DOT1L inhibitor[J].Cancer Cell,2011,20(1):53-65.
[11]DAIGLE S R,OLHAVA E J,THERKELSEN C A,et al.Potent inhibition of DOT1L as treatment of MLL-fusion leukemia[J].Blood,2013,122(6):1017-1025.
[12]YU W,CHORY E J,WERNIMONT A K,et al.Catalytic site remodelling of the DOT1L methyltransferase by selective inhibitors[J].Nature Communications,2012,3:1288.
[13]PETERS C D,JESPERSEN B,NORREGAARD R.AlphaLISA versus ELISA-based detection of interleukin 18 in healthy subjects and patients with end-stage renal disease[J].Scandinavian Journal of Clinical and Laboratory Investigation,2012,72(8):583-592.

