转Bt Cry1Ac基因抗虫棉花外源基因漂移研究
李海强,李号宾,王冬梅,汪 飞,丁瑞丰,潘洪生,阿克旦·吾外士,刘 建
(新疆农业科学院植物保护研究所/农业部西北荒漠绿洲作物有害生物综合治理重点实验室/农业部库尔勒作物有害生物综合观测实验站,乌鲁木齐 830091)
0 引 言
【研究意义】棉花是我国重要的经济作物,从20世纪90年代起,已有不同品种的转Bt基因抗虫棉进行了商业化种植[1-4]。转基因作物的大面积种植,在带来巨大效益的同时,也面临着转基因作物生态安全问题[5-6]。其中,外源基因向非转基因植物逃逸的生态风险,以及转基因作物花粉的基因漂移产生的生态后果,是转基因作物生态风险评估及其生态影响监测的主要内容[7-8]。棉花是常异花授粉,花朵较大,易造成外媒传粉的发生。虽然棉花自身的传播距离十分有限,但如果借助昆虫和风力的传播就可使得漂移距离明显提高。基因漂移是生物进化的一个重要过程和非常普遍的自然现象,转基因植物向野生的相同种群漂移导致的影响仍不明确。【前人研究进展】影响基因漂移的因子有很多,包括植物种类(Johnson,2006;Bonnett, 2008)、花粉的产量(Alexis ,2011)、气候因子(王天宇,2001)、传粉昆虫(Allen,2005)、转基因作物种植面积(Scheffer,1995)等[9-14]。转基因植物花粉传播距离和基因漂移率是目前基因漂移研究的主要内容。【本研究切入点】新疆棉区是我国重要的棉花生产基地,随着转基因棉花的引进和大面积种植,对其生态安全性的影响研究也显得非常迫切。新疆气候与土壤条件与内地相比有极大的差异,转基因抗虫棉的种植和研究均晚于内地,对转BtCry1Ac基因棉花外源基因漂移规律的研究鲜有报道。研究转BtCry1Ac基因棉花外源基因漂移规律。【拟解决的关键问题】2014~2015年在新疆吐鲁番市研究了转Bt基因棉花外源基因Cry1Ac,从其种植小区向周围常规棉花的漂移情况,为转Bt基因抗虫棉花基因流的风险评估提供理论依据。
1 材料与方法
1.1 材 料
研究采用的转Bt基因抗虫棉为GK19和SGK321,分别含有单价的BtCry1Ac基因和双价的BtCry1Ac+ CpTI基因,两个品种同时也含有抗卡纳霉素标记基因。常规棉为吐鲁番地区主要栽培品种岱棉80。转基因棉花材料由中国农业科学院植物保护研究所提供。
1.2 方 法
1.2.1基因漂移试验田间种植和取样
转Bt基因抗虫棉SGK321 和GK19外源基因Cry1Ac的漂移试验,于2014年分别在吐鲁番市原种场(E89.3387,N42.8695)和吐鲁番市二堡乡(E89.5000,N42.9135)进行。吐鲁番市原种场试验地面积约12 hm2(180亩),为近似平行四边形地块,其周围1 km之内没有其他棉田。在试验地中部种植面积为5 m×5 m=25 m2的转基因抗虫棉SGK321小区一个,其余均种植当地主栽品种岱棉80。吐鲁番市二堡乡试验地面积约10 hm2(150亩),近似长方形地块,周围1 km之内没有其他棉田。在试验地中部种植面积为5 m×5 m=25 m2的转Bt基因抗虫棉GK19小区一个,其余均种植当地主栽品种岱棉80。棉花栽培和田间管理方法均按照当地常规棉花生产技术管理。棉花成熟收获时,在抗虫棉小区东、南、西、北4个方向上距小区边缘1、3、5、7、10、15、20、25、30、40、50、70、90和120 m处,分别采摘岱棉80吐絮的棉铃45个,单铃编号分开保存,分开轧花,轧花后得到的种子也按编号的单铃分开保存。
1.2.2外源基因检测
2015年4月,将2010年单铃收获的种子种植于库尔勒市托布力其乡大田。每一个棉铃的种子种植一行,插牌标记。在棉花生长到2~3片真叶时,用1%的卡那霉素溶液涂抹每株棉花上的一张叶片,第5 d观察叶片变色情况,并根据变色情况初步判断植株中是否含有外源基因。如果叶片上涂抹过卡那霉素的地方变黄甚至产生枯斑,则说明不会含外源基因;如果不变色,则说明可能含有外源基因[7]。采集涂抹后未变色的棉株上的叶片,采用改良的CTAB法提取棉叶总DNA,选用特异性引物对样品内的Cry1Ac外源基因进行PCR定性检测。上下游引物分别为5'GAAGGATTGAGCAATCTCTAC 3'和5'AATCAGCCTAGTAAGGTCGT 3'。PCR反应体系:2×TaqPCR Master Mix(北京英俊生物科技有限公司)10 μL,上下游引物各0.5 μL(上海生工生物技术有限公司合成),DNA模板2 μL,双蒸水7 μL[4]。如果棉叶总DNA中含有Cry1Ac外源基因,则说明发育出该棉株的种子中含有Cry1Ac外源基因,进而说明产生该种子的棉铃也含有Cry1Ac外源基因。
1.3 数据处理
根据每株棉花Cry1Ac外源基因检测结果,计算1.2.1试验步骤中每个取样点的Cry1Ac外源基因漂移情况。外源基因漂移主要研究种子外源基因漂移,1.2.1试验步骤中每个取样点可以得到含有Cry1Ac外源基因的种子数、不含Cry1Ac外源基因的种子数、种子外源基因漂移率等数据。
种子外源基因漂移率%=取样点上含有Cry1Ac外源基因的种子数/取样点上取到的总种子数×100。
根据1.2.1试验步骤中各个取样点的距离、方向、外源基因漂移数据,采用SPSS Statistics 17.0软件,建立Cry1Ac外源基因漂移的二项分类Logistic回归预测模型,分析取样距离、取样方向与外源基因漂移之间的关系。预测变量为取样距离和取样方向。取样距离为连续区间变量。取样方向为无序分类变量,采用Indicator哑变量编码方法,将每一个方向转化为以北方为参照的3个哑变量,即方向(1)、方向(2)、方向(3),东方的三个哑变量为(1,0,0)、南方的三个哑变量为(0,1,0)、西方的三个哑变量为(0,0,1),北方的三个哑变量为(0,0,0)。应变量为外源基因漂移情况,为二项分类数据,1代表发生Cry1Ac基因漂移,0代表未发生Cry1Ac基因漂移。由于检测的种子数很多,将数据按频数表的形式输入,频数为应变量结果相同的铃数或种子数。
2 结果与分析
2.1 不同方向和不同距离上的外源基因Cry1Ac漂移率
2.1.1不同距离上的种子漂移率
研究发现在距离转基因棉花SGK321小区边缘1、3、5、7、10、15、20、25、30、40、50、70、90、120 m均能检测到都含有外源基因Cry1Ac种子,种子漂移率范围为0~5%,最小值为东向20 m,南向30 m,西向90 m和北向90 m,取值均为0。最大值为南向1 m处,达到了5%。而南、西和北向120 m处由于试验地原因,没有采集到基因漂移的种子。图1
研究表明,距离转基因棉花GK19小区边缘1、3、5、7、10、15、20、25、30、40、50、70、90、120 m均能检测到都含有外源基因Cry1Ac种子。取值范围为0~10.09%,其中东向1、3、5、15 m,南向15 m,西向7 m,北向1、3、7、25、90 m取值均为0。西向距离转基因棉花GK19小区边缘40 m处漂移率为最高达到了10.09%。 图2

图1SGK321的Cry1Ac基因在东(E)、南(S)、西(E)、北(N)四个方向不同距离种子漂移率
Fig.1The seed frequency of transgenic Cry1Ac cotton SGK321 in different direction and distance

图2GK19的Cry1Ac基因在东(E)、南(S)、西(E)、北(N)四个方向不同距离种子漂移率
Fig.2The seed frequency of transgenic Cry1Ac cotton GK19 in different direction and distance
2.2 外源基因Cry1Ac漂移Logisitc回归预测
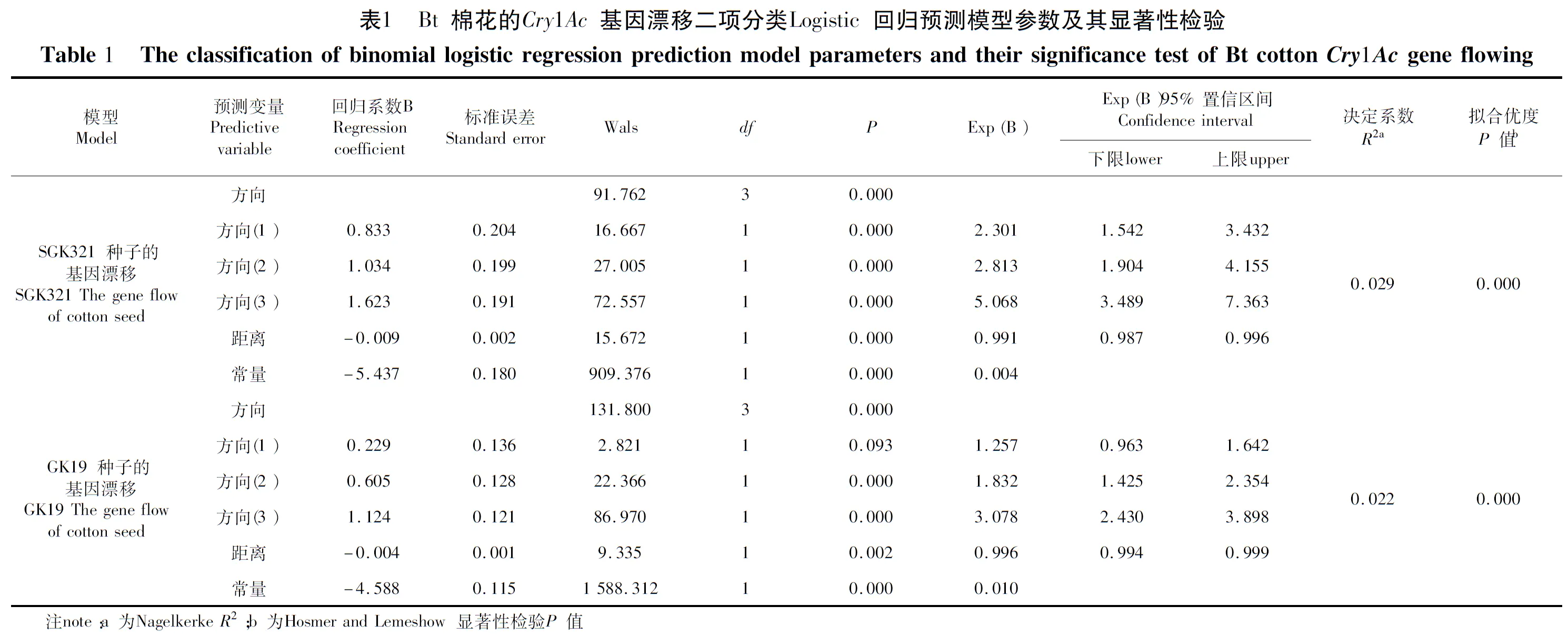
建立的用距离和方向预测SGK321、GK19种子的外源基因漂移四个Logistic回归模型的参数及其显著性检测。表1
研究表明,从种子外源基因漂移来看,方向对SGK321种子的外源基因漂移有显著影响,向东方、南方、西方的漂移显著多于向北方的漂移,向东方、南方、西方漂移的优势分别是向北方漂移的2.301、2.813、5.068倍。距离对SGK321种子的外源基因漂移有显著影响,同一方向上距离每增加1 m,漂移优势下降为1 m前漂移优势的0.991倍。
方向对GK19种子的外源基因漂移有显著影响,向南方、西方的漂移显著多于向北方的漂移,向南方、西方漂移的优势分别是向北方漂移的1.832、3.078倍。距离对GK19种子的外源基因漂移有显著影响,同一方向上距离每增加1 m,漂移优势下降为1 m前漂移优势的0.996倍。
两个模型的Nagelkerke决定系数都很小,小于0.03,说明两个模型的拟合很不理想。两个模型的Hosmer-Lemeshow拟合优度检测,Logistic回归预测模型的拟合优度较差(P﹤0.05),说明基因漂移的观察结果和模型拟合结果差异较大。表明影响外源基因漂移的因子,并不是距离和方向共同作用的。表1
3 讨 论
研究表明,在自然条件下,转Bt基因抗虫棉SGK321和GK19的外源基因Cry1Ac可以从这两种棉花的小区向周围的常规棉花漂移,使周围常规棉花产生含有Cry1Ac基因的种子。这与国内外相关报道的研究结果一致,只是各地得到的漂移距离不同。沈法富等[15](2001)认为Bt基因流最远可达72 m,王长永[16]等(2007)、贺娟[17]等(2013)、张宝红等[18]、连丽君等[19]认为转基因棉的花粉最远的传播距离分别为25、36、50、100 m。Heuberger等[20]和Van Deynze等[21]分别证实在750 m 和1 625 m 处可以发现转基因棉的外源基因Cry1Ac。研究表明棉花种子的漂移率,在距离转基因棉花小区边缘1~120 m均能检测到含有外源基因Cry1Ac。
不同的研究者得出的外源基因漂移距离不同的原因主要是研究方法差异较大。虽然棉花自身的传播距离十分有限,但如果借助昆虫和风力的传播就可使得漂移距离明显提高。贺娟等[17](2013)利用温室内人工创造风力和释放传粉昆虫蜜蜂的条件下研究了外源基因Cry1Ac漂移的距离。研究表明,风力处理的样本,外源基因最远漂移距离为25.6 m,蜜蜂处理的最远漂移距离为36 m。朱家林等(2013)在温室中创造人工定向风和非定向风条件研究了转基因棉花外源基因Cry1Ac漂移率,结果表明,在定向风处理中外源基因漂移率最远距离为25.6 m,非定向风处理中外源基因漂移率最远距离为36 m。Heuberger[20], Van Deynze[13], Llewellyn[21],Smith[22]均认为访花昆虫,如蜜蜂等对外源基因的漂移影响显著。Richards(2005)[23]认为昆虫携带的花粉可以使得,依靠昆虫传粉的棉花的基因漂移率具有一定的波动性。王长永[16](2007)在自然条件下,利用花粉粒染色法研究了Bt外源基因漂移率,结果表明,外源基因最远漂移距离为25 m。Van Deynze(2005)年在自然条件下对转基因抗除草剂基因漂移进行了研究,发现外源基因最远漂移距离为1 625 m。
影响转基因棉花漂移率的因素较多。研究由于条件限制没有记录气候因子,比如风、温度、光照等因子,导致试验结果没能够反应出气候因子对外源基因漂移带来的影响。记录气候因子,同时合理的利用统计的方法研究自然条件下转基因棉花的漂移率显得更为准确和合理。另外,检测方法的灵敏度也会影响杂交后代的转基因检测结果。试验由于检测样本数量太大,苗期通过卡那霉素的涂抹初步的鉴定了部分棉苗,随后的棉花材料进行分子析检测。卡那霉素的检测并不是完全可靠的,可能会漏掉部分阳性基因。因此,所有的检测均应该通过卡那霉素检测结合PCR检测更能准确的检测外源基因漂移。另外,研究对于传粉昆虫的背景研究较少,棉花花期传粉昆虫的种类以及昆虫是否会随风迁移等问题仍不清楚。
基因漂移是自然界客观存在的事实,影响转基因棉花基因漂移的因素非常复杂,风向、传粉昆虫,都是不可忽视的重要因子,需要综合考虑多因子对外源基因漂移带来的影响。因此,准确的研究特定的转基因植物基因流监测, 需根据具体目标、监测要求以及资源情况, 选用一种或综合使用几种方法进行监测。
4 结 论
研究表明在距离转基因棉花小区边缘1~120 m均能检测到含有外源基因Cry1Ac。SGK321种子漂移率最大漂移率是向南方向距离转基因棉花小区边缘1 m处,漂移率为5%;GK19最大漂移率是向西方向达到了10.09%。以方向因素和距离因素建立Cry1Ac基因漂移Logistic回归预测模型,方向和距离对种子外源基因漂移有一定影响,但影响外源基因漂移的因子并不是方向和距离联合决定的,用这两个因子并不能准确的预测外源基因漂移率。
参考文献(References)
[1] 陆宴辉.Bt 棉花害虫综合治理研究前沿[J]. 应用昆虫学报,2012, 49(4): 809-819.
LU Yan-hui. (2012). Advance in insect pest management in Bt cotton worldwide [J].ChineseJournalofAppliedEntomology, 49(4): 809-819. (in Chinese)
[2] 刘晨曦,吴孔明. 转基因棉花的研发现状与发展策略[J].植物保护,2011, 37(6): 11-17.
LIU Chen-xi, WU Kong-ming. (2011). Current progress in research and development of transgenic cotton and a strategic prospect for China [J].PlantProtection, 37(6): 11-17. (in Chinese)
[3] 李燕娥,吴霞,张林水,等.我国与山西省转基因棉花发展状况与趋势[J].山西农业科学,2011, 39(1): 6-8.
LI Yan-e, WU Xia, ZHANG Lin-shui, et al. (2011). Progress and Trends of Transgenic cotton in Shanxi and China [J].JournalofShanxiAgriculturalSciences, 39(1): 6-8. (in Chinese)
[4] 朱家林,贺娟,牛建群,等.风向因素对转基因抗虫棉花基因漂移效率的影响[J].生态学报, 2013, 33(21): 6 803-6 812.
ZHU Jia-lin, HE Juan, NIU Jian-qun, et al. (2013). The influence of wind direction on pollen-mediated gene flow in transgenic insect resistant cotton [J].ActaEcologicaSinica, 33(21): 6,803-6,812. (in Chinese)
[5] 王根平,杜文明,夏兰琴.植物安全转基因技术研究现状与展望[J].中国农业科学,2014, 47(5): 823-843.
WANG Gen-ping, DU Wen-ming, XIA Lan-qin. (2014). Current Status of Transgenic Technologies for Safety Consideration in Plants and Future Perspectives [J].ScientiaAgriculturaSinica, 47(5): 823-843. (in Chinese)
[6] 牟文雅,贾艺凡,赵宗潮,等.转基因作物外源基因漂移风险及其控制技术研究进展[J].生态学杂志,2016, 5(1): 243-249.
MOU Wen-ya, JIA Yi-fan, ZHAO Zong-chao, et al. (2016). Advanced on ecological risk of exogenous transgene flow and corresponding control techniques for transgenic crops [J].ChineseJournalofEcology, 5(1): 243-249. (in Chinese)
[7] 叶鹏盛, 曾华兰,李琼芳,等.利用卡那霉素间接鉴定转基因抗虫棉的研究[J].四川农业大学学报,2003, 21(2): 129-131.
YE Peng-sheng, ZENG Hua-lan, LI Qiong-fang, et al. (2003). Study on the Identification of Transgenic Cotton by Using Kanamycin as an Indirect Marker [J].JournalofSichuanAgriculturalUniversity, 21(2): 129-131. (in Chinese)
[8] 焦悦, 梁晋刚,翟永. 转基因作物安全评价研究进展[J].作物杂志,2017, 27(6): 681-687.
JIAO Yue, LIANG Jin-gang, ZHAI Yong. (2017). Progress in Safety Assessment of Genetically Modified Crops [J].Crops,27(6): 681-687. (in Chinese)
[9] Johnson, P. G., Larson, S. R., Anderton, A. L., Patterson, J. T., Cattani, D. J., & Nelson, E. K. (2006). Pollen-mediated gene flow from kentucky bluegrass under cultivated field conditions.CropScience, 46(5):1,990-1,997.
[10] Bonnett, G. D., Nowak, E., Olivares-Villegas, J. J., Berding, N., Morgan, T., & Aitken, K. S. (2008). Identifying the risks of transgene escape from sugarcane crops to related species, with particular reference to saccharum spontaneum, in australia.TropicalPlantBiology, 1(1):58-71.
[11] Alexis M, Benjamin L, Bruno A, et al. (2011). Modeling diurnal and seasonal patterns of maize pollen emission in relation to meteo-rological factors [J].AgriculturalandForestMeteorology, 151(1): 11-21.
[12] 王天宇,赵治海,闫洪波.谷子抗除草剂基因从栽培种向其近缘野生种漂移的研究[J].作物学报,2001, 27(6): 681-687.
WANG Tian-yu, ZHAO Ye-hai, YAN Hong-bo, et al. (2001). Gene Flow from Cultivated Herbicide-resistant Foxtail Millet to its Wild Relatives: a Basis for Risk Assessment of the Release of Transgenic Millet [J].ActaAgronomicaSinica, 27(6): 681-687. (in Chinese)
[13] Van Deynze, A. E., Sundstrom, F. J., & Bradford, K. J. (2005). Pollen-mediated gene flow in california cotton depends on pollinator activity.CropScience, 45(4): 1,565-1,570.
[14] Scheffler, J. A., Parkinson, R., & Dale, P. J. (1995). Evaluating the effectiveness of isolation distances for field plots of oilseed rape (brassica napus) using a herbicide-resistance transgene as a selec
Tablemarker.PlantBreeding, 114(4): 317-321.
[15] 沈法富,于元杰,张学坤,等.转基因棉花的Bt基因流[J].遗传学报,2001, 28(6): 562-567.
SHEN Fa-fu, YU Yuan-jie, ZHANG Xue-kun, et al. (2001). Bt Gene Flow of Transgenic Cotton [J].ActaGeneticaSinica, 28(6): 562-567. (in Chinese)
[16] 王长永,刘燕,周骏,等.花粉介导的转Bt基因棉花田间基因流监测[J].应用生态学报,2007, 18(4): 801-806.
WANG chang-yong, LIU Yan, ZHOU Jun, et al. (2007) Monitoring of pollen-mediated gene flow from transgenic Bt cotton [J].ChineseJournalofAppliedEcology, 18(4): 801-806. (in Chinese)
[17] 贺娟,朱威龙,朱家林,等. 风、蜜蜂因素对转Cry1Ac基因棉花花粉介导的基因漂移的影响[J].棉花学报,2013, 25(5): 453-458.
HE Juan, ZHU Wei-long, ZHU Jia-lin, et al. (2013). The Influences of Wind and Bee on Pollen-mediated Gene Flow from TransgenicCry1AcCotton [J].CottonScience, 25(5): 453-458. (in Chinese)
[18] 张宝红,郭腾龙.转基因棉花基因花粉散布频率及距离的研究[J].应用与环境生物学报,2000, 6(1): 39-42.
ZHANG Bao-hong, GUO Teng-long. (2000). Frequency and distance of pollen dispersal from transgenic cotton [J].ChineseJournalofApplied&EnvironmentalBiology, 6(1): 39-42. (in Chinese)
[19] 连丽君,李莹,王娟,等. 转 betA/als基因棉花生存竞争力和基因漂流的调查[J].山东大学学报(理学版),2009, 44(5): 20-27.
LIAN Li-jun, LI Ying, WANG Juan, et al. (2009) Investigations on survival competitiveness and gene flow of transgenic cotton with betA /als gene in the field [J].JournalofShandongUniversity(NaturalScience) , 44(5): 20-27. (in Chinese)
[20] Heuberger, S., Ellerskirk, C., Tabashnik, B. E., & Carrière, Y. (2010). Pollen- and seed-mediated transgene flow in commercial cotton seed production fields.PlosOne, 5(11): e14128.
[21] Llewellyn, D., Tyson, C., Constable, G., Duggan, B., Beale, S., & Steel, P. (2007). Containment of regulated genetically modified cotton in the field.AgricultureEcosystems&Environment, 121(4): 419-429.
[22] Mallory‐Smith, C., & Zapiola, M. (2008). Gene flow from glyphosate‐resistant crops.PestManagementScience, 64(4): 428.
[23] Richards, J. S., Stanley, J. N., & Gregg, P. C. (2005). Viability of cotton and canola pollen on the proboscis of helicoverpa armigera : implications for spread of transgenes and pollination ecology.EcologicalEntomology, 30(3):327-333.

