黑龙江省2016年4起胃肠炎暴发中诺如病毒分子特征分析
许军 石鑫 舒畅 冷焱 曹博 孙巍
150030 哈尔滨,黑龙江省疾病预防控制中心(许军、石鑫、舒畅、曹博、孙巍);158100 鸡西市疾病预防控制中心(冷焱)
诺如病毒(Norovirus, NoV)属于杯状病毒科,是引起暴发和散发性急性胃肠炎的主要致病原。诺如病毒传播力强,可通过水、食物、气溶胶和接触等多种途径传播,主要发生在医院、学校、幼托机构、养老院等相对密闭的场所,主要表现为呕吐、腹泻等[1]。2016年黑龙江省共报告4起急性胃肠炎暴发疫情,发病211例,无死亡病例。本研究对相关样本进行病毒核酸检测和序列分析,以了解这4起暴发疫情的病原分子特征,为今后此类疾病监测和疫情处置提供参考数据。
1 材料与方法
1.1标本采集由疫情发生地的疾病预防控制中心进行现场流行病学调查,采集现症患者的肛拭子或粪便标本,低温送至黑龙江省疾病预防控制中心,-70 ℃保存待检。
1.2病毒核酸提取用无菌生理盐水将粪便标本制成10%悬液,振荡每个标本1 min,置离心机2 370×g离心5 min。取上清液200 μl,采用德国Qiagen公司的RNeasy Mini Kit试剂盒进行病毒核酸提取,提取方法按照说明书进行。

表1 2016年黑龙江省4起胃肠炎暴发疫情概况
1.3荧光定量RT-PCR、RT-PCR及序列测定使用江苏硕世NoV GI/GII荧光定量PCR检测试剂盒检测NoV GI、GII。选取NoV阳性标本,采用引物P290/P289扩增病毒的聚合酶区(RdRp)片段[2];采用引物G1SKF/G1SKR、COG2F/G2SKR分别扩增GI、GII部分衣壳蛋白(VP1)片段[3];采用引物MON432/G1SKR、MON431/G2SKR分别扩增包括GI、GII的ORF1/2重叠区基因序列[4],进行重组分析。扩增PCR产物经1.5%琼脂糖凝胶电泳鉴定,送至赛默飞世尔科技公司进行双向序列测定。
1.4序列拼接和进化分析序列拼接使用DNAStar软件中的Seqman程序。从GenBank下载NoV GI、GII各基因型参考株序列,使用Mega6.0软件的Clustal W进行序列比对,采用邻接法构建进化树,bootstrap重复检验1 000次。重组分析采用SimPlot3.5.1软件进行。
2 结果
2.1疫情概况和特征2016年黑龙江省共报告4起胃肠炎暴发疫情(表1),其中2016年10月1起,11月2起,12月1起。疫情发生在学校、幼托机构,其中学校2起,幼托机构2起。共发病211例,无死亡病例。其中男性患者97例,女性患者114例,男女性别比为1∶1.2。病例最小年龄为3岁,最大为48岁。不同年龄段中,≤5岁共28例,占13.27%;6~18岁年龄组共127例,占60.19%;≥19岁共56例,占26.54%。病例病程短、病情轻,主要临床表现为呕吐(90.5%)、腹痛(71.1%)、发热(57.8%)和腹泻(53.6%)(表2)。
2.2序列测定及系统进化分析随机选取每起疫情的阳性标本3~4份扩增NoV部分RdRp和VP1片段,进行序列测定分析,构建进化树,如图1、2和表3。
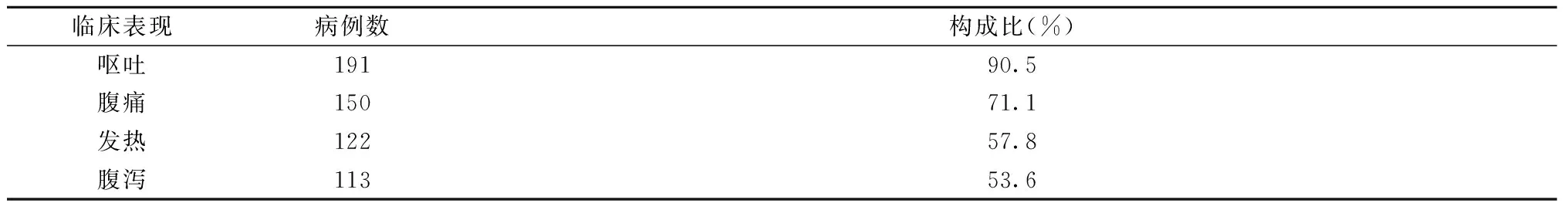
表2 2016年黑龙江省4起胃肠炎暴发疫情病例临床特征
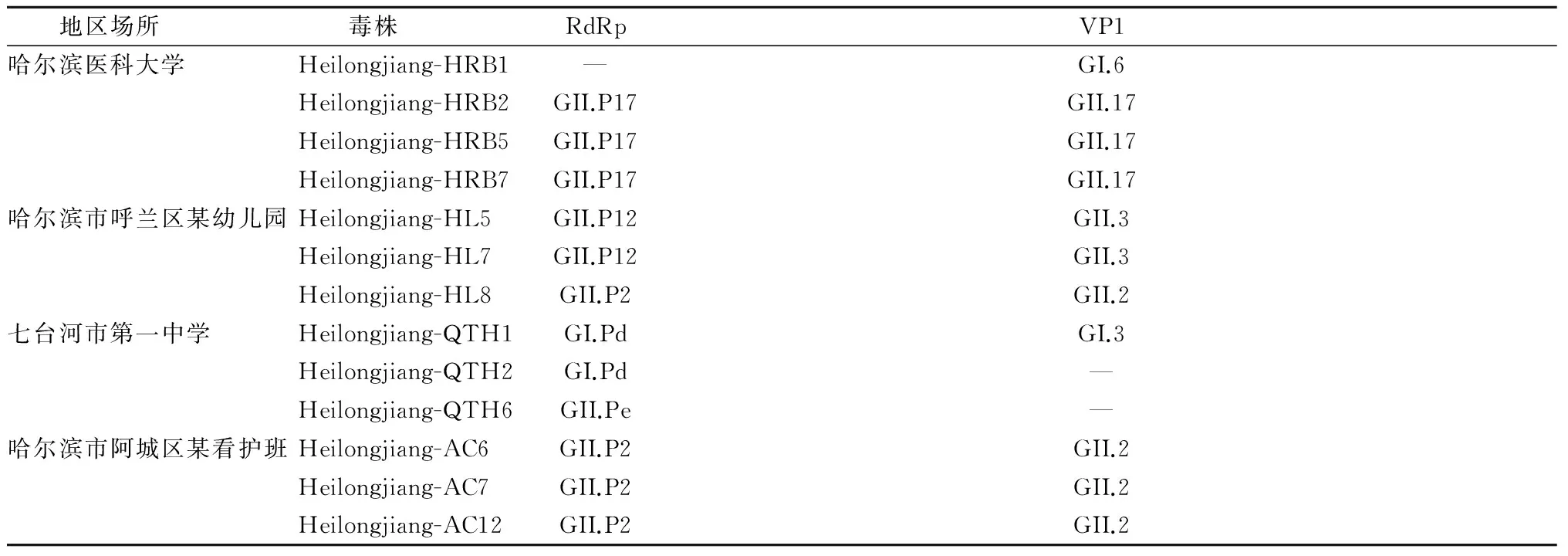
表3 2016年黑龙江省4起胃肠炎暴发疫情诺如病毒分型结果
注:“-”表示未测出序列
Note: “-”indicates undetected sequence
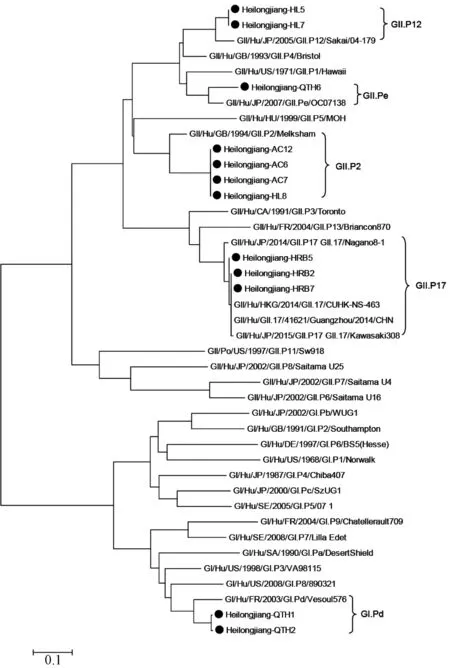
“●”表示2016年黑龙江省4起暴发疫情阳性标本图1 NoV RdRp区核苷酸序列进化分析“●”indicates stains of 4 outbreaks of gastroenteritis in Heilongjiang, 2016Fig.1 Phylogenetic tree based on partial RdRp gene sequences
2.3重组分析RdRp区和VP1区分型结果不一致的阳性株为疑似重组型。通过序列分析发现本研究存在疑似重组型:GII.P12/GII.3型、GI.Pd/GI.3型,重组位点需要进一步通过重组分析软件确定。对疑似重组型用引物MON432/G1SKR、MON431/G2SKR分别扩增包括NoV GI、GII的ORF1/2重叠区基因序列,用SimPlot3.5.1软件对序列进行重组鉴定(图3)。选取样本Heilongjiang-HL7、Heilongjiang-QTH1分别作为GII.P12/GII.3、GI.Pd/GI.3重组型的代表株进行重组分析。结果显示Heilongjiang-HL7在RdRp区与GII.12/Sakai/04-179(AB220922)参考株具有高度同源性,在VP1区与GII.12/Sakai/04-179同源性降低,与GII.3/Toronto(U02030)参考株的同源性迅速增高。Heilongjiang-QTH1在RdRp区与参考株GI.Pd/Vesoul576(EF529738)具有高度同源性,在VP1区与GI.Pd/Vesoul576同源性降低,与参考株GI.3/VA98115(AY038598)同源性迅速增高。

● 表示2016年黑龙江省4起暴发疫情阳性标本图2 NoV VP1区核苷酸序列进化分析“●”indicates stains of 4 outbreaks of gastroenteritis in Heilongjiang, 2016Fig.2 Phylogenetic tree based on partial VP1 gene sequence
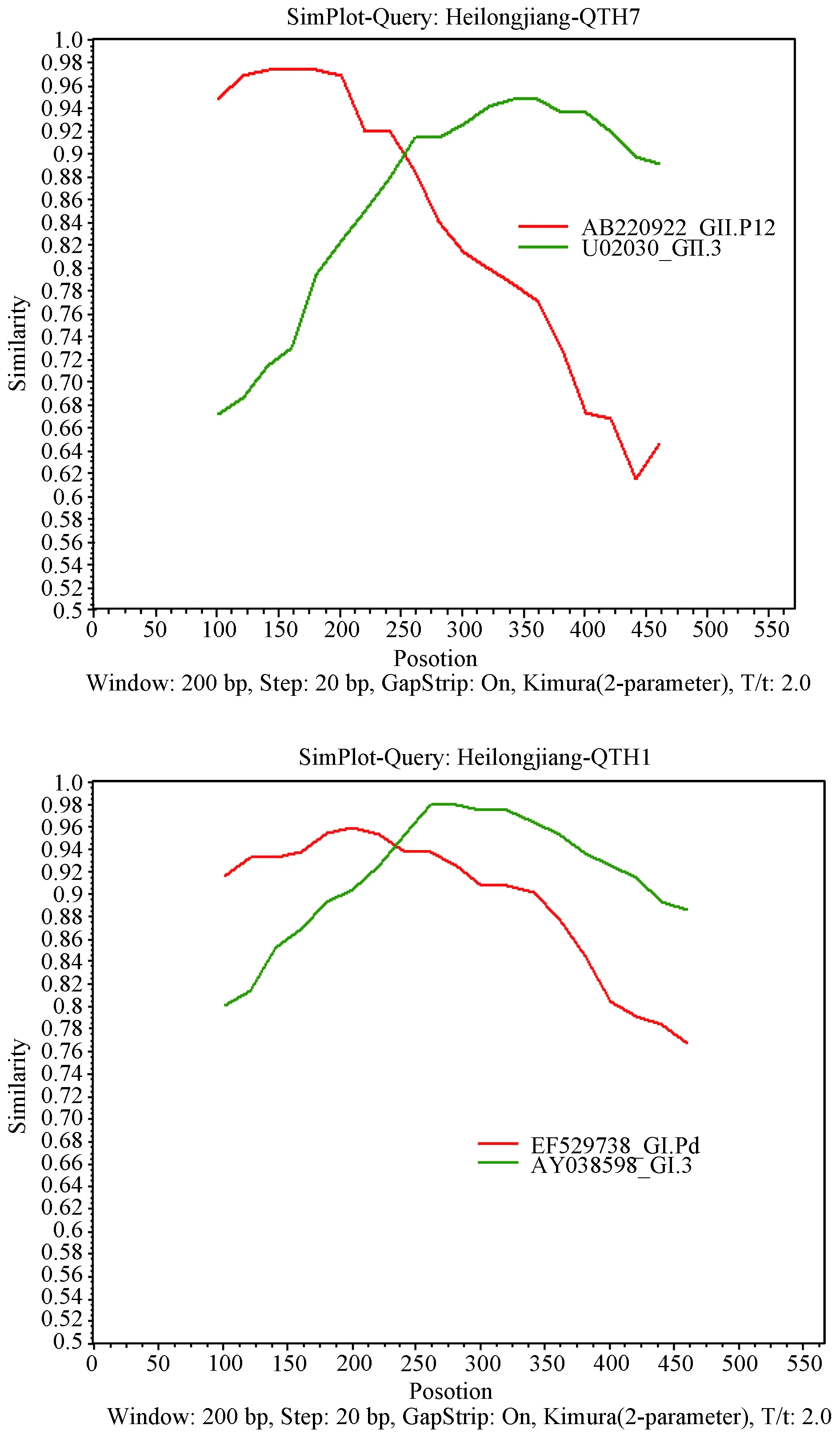
图3 重组株RdRp和VP1区基因序列Simplot分析Fig.3 Simplot analysis of partial polymerase and capsid gene sequence of the strains
3 讨论
本研究在2016年黑龙江省4起急性胃肠炎暴发疫情样本中均检出NoV ,NoV 阳性率为34.3%(35/102),这4起暴发疫情均发生在冬季。提示NoV 已成为我省急性胃肠炎暴发的重要病原。托幼机构和学校是NoV 胃肠炎暴发的重要场所,主要症状表现为呕吐。
NoV 为单股正链RNA病毒,分为3个开放阅读框(ORFs)。ORF1编码非结构蛋白,包括RNA依赖的RNA聚合酶(RdRp),ORF2编码主要结构蛋白(VP1),ORF3编码次要结构蛋白(VP2)。根据基因特征,NoV 被分为6个基因群(Genogroup,GI-GVI),感染人类的主要为GI、GII和GIV,GI和GII可进一步分为9个和22个基因型[5]。NoV 易发生变异,主要通过突变和重组两个机制发生变异,这使NoV 的基因和抗原性具有丰富的多样性。通常报道的重组主要是NoV RdRp区和VP1区属于不同的基因型,重组位点一般位于ORFl/ORF2重叠区[6]。本研究选择了较为保守的部分RdRp区和VP1区进行测序分析。结果显示哈尔滨市南岗区高校疫情由GII.17型、GI.6型引起;呼兰区幼托机构疫情由GII.2型、GII.P12/GII.3型引起;七台河市中学疫情由GII.Pe、GI.Pd/GI.3型引起;阿城区幼托机构疫情由GII.2引起。
有研究显示,在引起胃肠炎暴发的NoV 中,GII一直处于优势地位,其中GII.4型一直是引起人类急性胃肠炎暴发的主要基因型,其次为GII.3型。GII.3型常以基因型间重组的形式出现,如GII.Pa/GII.3、GII.Pb/GII.3、GII.P12/GII.3[7]。但在2014年底出现了一种新的GII.17变异株,并在中国(广州[8]、北京[9])和日本[10]引起暴发流行。监测数据显示2015年我国NoV 相关急性胃肠炎主要是由GII.17新变异株引起。本研究根据部分RdRp区和VP1区基因序列构建的遗传进化树显示,在哈尔滨市南岗区高校疫情检测到的GII.17型与2014—2015年日本、2014年中国(广州、香港)分离株亲缘关系接近,可能为同一遗传进化来源。新GII.17型在主要结构蛋白VP1的P2区上发生了一系列的氨基酸突变,这些位点包括病毒与宿主细胞上受体的结合位点及病毒抗原性决定域等。这些氨基酸突变很可能是导致病毒突然暴发流行的主要原因[11]。
在黑龙江省2016年11、12月的2起疫情中均检测到GII.2型NoV ,并且在其中1起疫情中还检测到GII.P12/GII.3重组型。这2起疫情的GII.2型NoV 核苷酸序列高度同源,提示NoV 在黑龙江省流行过程中不同基因型持续的共同进化和循环。我省对2012—2015年病毒性腹泻监测结果显示:480例腹泻样本中GII.P12/GII.3重组型检出3例、GII.4_Sydney2012型检出3例[12]。2016年我国NoV 暴发以GII.2型为主,GII.2型是我省首次在急性胃肠炎暴发病例中检出该基因型病毒。我国2014—2015年在安徽省[13]、广东省[14]均发生GII.2型NoV 引起的急性胃肠炎暴发疫情。此外还在1起疫情中检测到GI.Pd/GI.3重组型。我国由GI.Pd/GI.3重组型NoV 引起的急性胃肠炎暴发疫情在以往的文献中报道较少,也是黑龙江省首次检测到的GI重组型。依据目前文献澳大利亚维多利亚州2002—2010年69起胃肠炎暴发疫情由GI引起,其中有5起是由GI.Pd/GI.3重组型NoV 引起[15]。重组可使病毒的抗原决定基因发生改变,病毒逃避宿主的免疫应答,引起暴发或流行。黑龙江省至今还未见关于GII.2型、GI.6型、GI.Pd/GI.3重组型NoV 暴发疫情和散发病例的报道,这提示NoV 在黑龙江省存在多个基因型的流行,因此需要加强监测,弄清来源,关注其发展,为疫情处置和科学防控提供参考数据。
利益冲突无
[1]廖巧红, 冉陆, 靳淼, 等. 诺如病毒感染暴发调查和预防控制技术指南(2015版)[J]. 中华预防医学杂志, 2016, 50(1): 7-16. DOI: 10.3760/cma.j.issn.0253-9624.2016.01.003.
[2]Jiang X, Huang PW, Zhong WM, et al. Design and evaluation of a primer pair that detects both Norwalk-and Sapporo-like caliciviruses by RT-PCR[J]. J Virol Methods, 1999, 83(1-2): 145-154. DOI:10.1016/S0166-0934(99)00114-7.
[3]Kojima S, Kageyama T, Fukushi S, et al. Genogroup-specific PCR primers for detection of Norwalk-like viruses[J]. J Virol Methods, 2002, 100(1-2):107-114. DOI: 10.1016/s0166-0934 (01)00404-9.
[4]Cannon JL, Barclay L, Collins NR, et al. Genetic and epidemiologic trends of norovirus outbreaks in the United States from 2013 to 2016 demonstrated emergence of novelGII.4 recombinant viruses[J]. J Clin Microbiol, 2017, 55(7): 2208-2221. DOI: 10.1128/JCM.00455-17.
[5]Vinjé J. Advances in laboratory methods for detection and typing of norovirus[J]. J Clin Microbiol, 2015, 53(2): 373-381. DOI: 10.1128/JCM.01535-14.
[6]Bull RA, Hansman GS, Clancy LE, et al. Norovirus recombination in ORF1/ORF2 overlap[J]. Emerg Infect Dis, 2005, 11(7): 1079-1085. DOI: 10.3201/eid1107.041273.
[7]Mahar JE, Bok K, Green KY, et al. The importance of intergenic recombination in norovirus GII.3 evolution[J]. J Virol, 2013, 87(7): 3687-3698. DOI: 10.1128/JVI.03056-12.
[8]Lu J, Sun L, Fang L, et al. Gastroenteritis outbreaks caused by Norovirus GII.17, Guangdong province, China, 2014-2015[J]. Emerg Infect Dis, 2015, 21(7): 1240-1242. DOI:10.3201/eid2107.150226.
[9]Gao Z, Liu B, Huo D, et al. Increased norovirus activity was associated with a novel norovirus GII.17 variant in Beijing, China during winter 2014-2015[J]. BMC Infect Dis, 2015, 15: 574. DOI: 10.1186/s12879-015-1315-z.
[10]Matsushima Y, Ishikawa M, Shimizu T, et al. Genetic analyses of GII.17 norovirus strains in diarrheal disease outbreaks from December 2014 to March 2015 in Japan reveal a novel polymerase sequence and amino acid substitutions in the capsid region[J]. Euro Surveill, 2015, 20(26): pii21173. DOI: 10.2807/1560-7917.ES2015.20.26.21173
[11]Lu J, Fang L, Zheng H, et al. The evolution and transmission of epidemic GII.17 noroviruses[J]. J Infect Dis, 2016, 214(4): 556-564. DOI: 10.1093/infdis/jiw208.
[12]许军, 石鑫, 舒畅, 等. 黑龙江省2012-2015年诺如病毒感染的分子流行病学研究[J]. 中国公共卫生管理, 2016, 32(5): 569-572. DOI: 10.19568/j.cnki.23-1318.2016.05.001.
[13]史永林, 孔翔羽, 靳淼, 等. 安徽省诺如病毒胃肠炎暴发的分子病原学特征研究[J]. 中华实验和临床病毒学杂志, 2015, 29(4): 310-312. DOI: 10.3760/cma.j.issn.1003-9279.2015. 04.007.
[14]谢才文, 唐 薇, 杜春生, 等. 广州市某所特殊教育学校诺如病毒急性胃肠炎暴发疫情分析[J]. 疾病监测与控制杂志, 2017, 11(1): 30-32.
[15]Bruggink LD, Oluwatoyin O, Sameer R, et al. Molecular and epidemiological features of gastroenteritis outbreaks involving genogroup I norovirus in Victoria, Australia, 2002-2010[J]. J Med Virol, 2012, 84(9): 1437-1448. DOI: 10.1002/jmv.23342.

