食品用酶毕赤酵母表达载体的构建
刘文山,刘立辉,傅荣昭
邦泰生物工程(深圳)有限公司,广东 深圳 518102
毕赤酵母由于其强大的分泌表达能力,已经成为最常用的真核表达系统,利用毕赤酵母表达各种的蛋白和酶类已广泛应用于医药、工业和食品等领域[1]。此外,毕赤酵母已被美国食品药品管理局(FDA)认定为 GRAS(GenerallyRecog⁃nized as Safe)微生物,毕赤酵母表达的乳糖酶也在国内被批准用于食品行业。可以预期,越来越多的食品用酶将可以通过毕赤酵母生产。
对于食品用酶而言,其安全评价至关重要,不仅要求生产菌株的安全性,而且还要求其不能含有非食品级菌种来源的功能性DNA片段[2]。然而,目前所使用的绝大多数毕赤酵母表达载体最终均将大肠杆菌来源的抗性标记和大肠杆菌复制区整合到了酵母基因组上,这对于食品安全而言是不利的。pAO815和pPink系列载体虽然在酵母中不用抗性筛选,但也含有大肠杆菌的氨苄青霉素或卡那霉素抗性标签及大肠杆菌复制区,并最终整合到酵母基因组上。因此,我们尝试开发一种产生高拷贝但不会将这些细菌来源的DNA片段整合到酵母基因组上的质粒。
1 材料和方法
1.1 材料
大肠杆菌DH5α用于克隆,采用LB培养基于37℃培养;毕赤酵母菌株GS115用于提取基因组扩增ADE2基因;毕赤酵母GS115Δade2由本公司保存,作为蛋白表达宿主,在YPD培养基中于30℃培养;DNA聚合酶为南京诺唯赞生物科技有限公司的Phanta高保真酶;质粒提取试剂盒和凝胶回收试剂盒均来源于天根公司;蛋白marker为Thermo的PageRuler;DNA marker、限制性内切酶和T4DNA连接酶均来源于TaKaRa公司;引物及序列见表1。
1.2 pMA的构建
pMA构建流程如图1所示。分别PCR扩增如下片段:①片段1:AOX1启动子的5'端(BglⅡ和SacⅠ之间的部分,209 bp),以pPIC9k为模板,采用引物1和2扩增(扩增条件:95℃预变性3 min;95℃变性30 s,55℃退火30 s,72℃延伸30 s,循环30次;72℃延伸5 min);②片段2:Amp抗性标签和pUC复制区(1762 bp),以pPIC9K为模板,采用引物3和4扩增(扩增条件:95℃预变性3 min;95℃变性30 s,55℃退火30 s,72℃延伸1 min,循环30次;72℃延伸5 min);③片段3:AOX1启动子的SacⅠ位点到AOX1终止子(1414 bp),以pPIC9k为模板,采用引物5和6扩增(扩增条件:95℃预变性3 min;95℃变性30 s,55℃退火30 s,72℃延伸1 min,循环30次;72℃延伸5 min;④片段4:ADE2筛选标记(1927 bp,3'端加入BglⅡ位点),以GS115基因组为模板,采用引物7和8扩增(扩增条件:95℃预变性5 min;95℃变性30 s,55℃退火30 s,72℃延伸1 min,循环30次;72℃延伸5 min)。

表1 PCR引物列表
用凝胶回收试剂盒回收上述片段,各取1 μL混合后作为模板,进行重叠延伸PCR扩增(扩增条件:95℃预变性3 min;95℃变性30 s,55℃退火30 s,72℃延伸3 min,循环30次;72℃延伸5 min)。将扩增得到的片段用BglⅡ酶切,回收后用T4DNA连接酶自连,转化大肠杆菌DH5α,对AOX1启动子到AOX1终止子部分测序正确后保存,将该载体命名为pMA。
1.3 表达载体pMA-AOLAC的构建
以pETAOLAC(含有来源于米曲霉的β-半乳糖甘酶表达基因,GenBank登录号为E12172,两端分别为EcoRⅠ和NotⅠ位点)为模板,用EcoRⅠ和NotⅠ酶切后插入pMA的EcoRⅠ和NotⅠ位点,利用引物9和10进行PCR验证。将构建的质粒命名为pMA-AOLAC。
1.4 重组酵母的构建
酵母转化采用电转化法[3],电转条件为0.2 mm电转杯,1500 V电压。电击完成后涂布MD平板(1.34 g/L YNB,2%葡萄糖,0.4 mg/L生物素,50 mg/L组氨酸),30℃培养2~3 d,挑取白色转化子接种到新的MD平板上,并用引物9和10进行菌落PCR验证。
1.5 蛋白表达及检测
重组酵母接种于BMGY培养基(10 g/L酵母提取物,20 g/L胰蛋白胨,100 mmol/L磷酸钾溶液,1%甘油,1.34 g/L YNB,0.4 mg/L生物素)中,30℃培养至D600nm为2~6,3000 r/min离心5 min,将菌体重悬到BMMY培养基(将BMGY中的甘油替换为0.5%甲醇)中于30℃培养,每24 h补加一次0.5%甲醇,96 h后取上清进行SDS-PAGE。β-半乳糖甘酶活性测定采用文献报道的方法[4],一个单位定义为每分钟产生1 μmol邻硝基苯酚所需酶量。
2 结果
2.1 表达载体pMA的构建
载体的构建如图1,通过重叠延伸PCR将AmpR+ori片段插入AOX1启动子的SacⅠ位点(片段1和片段2之间),并将ADE2表达框插入AOX1终止子的3'端。各片段PCR回收后琼脂糖凝胶电泳如图2A所示。将最终PCR产物用BglⅡ酶切,连接后得到的载体命名为pMA。
2.2 β-半乳糖苷酶重组表达菌株的构建及表达分析
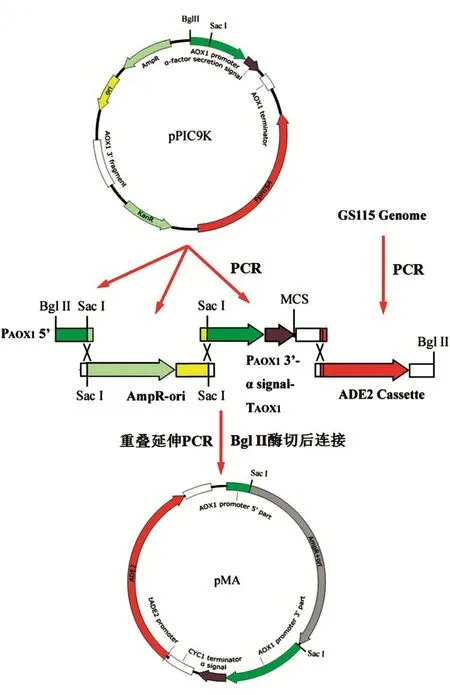
图1 载体pMA的构建
将来源于米曲霉的β-半乳糖苷酶编码基因插入pMA的EcoRⅠ和NotⅠ位点,将构建的载体命名为pMA-AOLAC。该载体经SacⅠ酶切后切除AmpR+ori片段,回收AOX1启动子3'端-α信号肽-AOLAC-AOX1终止子-ADE2表达框-AOX1启动 子 5'端(图 2B),用 于 转 化 毕 赤 酵 母GS115Δade2。挑取白色转化子进行PCR验证(图3A)后保存到甘油管中。挑取2株阳性克隆进行发酵测试,结果如图3B,可见明显的蛋白表达条带,经测定发酵液最高酶活为380 U/mL。
3 讨论
毕赤酵母常用载体如pPIC系列载体一般采用G418或博来霉素等抗性标记进行高拷贝筛选,有些载体还带有一个大肠杆菌的氨苄青霉素或卡那霉素的抗性标记。这些抗性标记如氨苄青霉素、卡那霉素、博来霉素、潮霉素等的抗性基因大多来源于大肠杆菌转座子[5-7],最终构建的重组菌株均将这些标记基因及大肠杆菌复制区整合到了酵母基因组上。对于表达食品用酶来说,这些片段通常是不允许的。此外,这些载体包含过多的元件使载体偏大,不利于载体构建和整合。这些基因还可能转移到环境中的其他微生物中,从而造成危害。
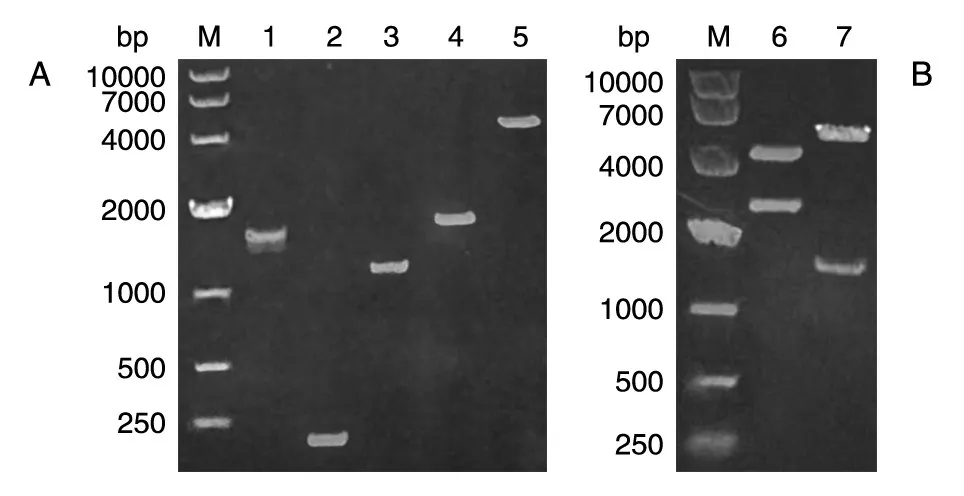
图2 pMA各片段的PCR扩增(A)及验证(B)M:DNA marker;1~5:分别为片段1、2、3、4和1~4重叠PCR产物;6:pMA的双酶切验证;7:pMA的SacⅠ线性化
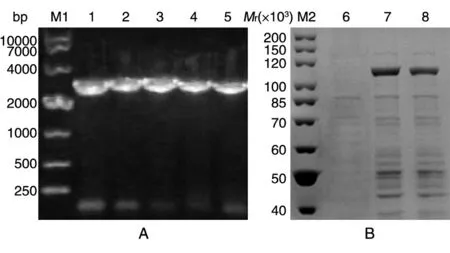
图3 重组酵母菌株的菌落PCR验证(A)及发酵液的SDS-PAGE检测(B)M1:DNA marker;M2:蛋白 marker;1~5:不同的转化子;6:GS115Δade2/pMA;7,8:GS115Δade2/pMA-AOLAC的1、2号转化子
为了使大肠杆菌来源的片段不整合到酵母基因组,我们以pPIC9K为模板,将氨苄青霉素抗性基因AmpR和大肠杆菌复制区ori插入AOX1启动子的SacⅠ位点。这样在进行基因组整合时,用SacⅠ酶切载体即可将该AmpR+ori片段切除,将该片段以外的部分整合到AOX1位点。为了避免使用G418抗性标记筛选而达到高拷贝的整合,我们采取pPINK载体的思路,将G418抗性基因替换为ADE2表达框。ADE2表达框来源于毕赤酵母基因组,包含截短ADE2启动子的部分序列、ADE2编码基因及其终止子。其中截短的启动子仅具有微弱的转录活性,从而必须产生多拷贝才能满足ADE2缺陷菌株对该酶的需求[8]。最终,除了目标基因以外,单次整合到基因组上的片段仅3.6 kb,远小于pPIC系列载体。除了将AmpR+ori片段插入AOX1中间,还可以将其插入ADE2的中间,最终将目标片段整合到基因组的ADE2位点。我们后续还会考虑将甲醇诱导的AOX1启动子替换为GAP等组成型启动子。为了验证载体pMA的实用性,我们利用该载体表达来源于米曲霉的β-半乳糖苷酶,结果达到了较高的表达水平。后续我们将利用该系统尝试表达不同的食品用酶,以降低一些食品用酶的生产成本。
[1] 朱泰承,李寅.毕赤酵母表达系统发展概况及趋势[J].生物工程学报,2015,31(6):929-938.
[2] 路福平,刘逸寒,薄嘉鑫.食品酶工程关键技术及其安全性评价[J].中国食品学报,2011,11(9):188-193.
[3] Cregg J M,Tolstorukov I,Kusari A,et al.Expression in the yeast Pichia pastoris[J].Methods Enzymol,2009, 463:169-189.
[4] Sheridan P P,Brenchley J E.Characterization of a salt-tolerant family 42 β-galactosidase from a psychro⁃philic antarctic Planococcus isolate[J].ApplEnviron Microbiol,2000,66(6):2438-2444.
[5] Hedges R W,Jacob A E.Transposition of ampicillin resistancefrom RP4 tootherreplicons[J].MolGen Genet,1974,132(1):31-40.
[6] Oka A,Sugisaki H,Takanami M.Nucleotide sequence ofthe kanamycin resistance transposon Tn903[J]J Mol Biol,1981,147(2):217-226.
[7] Blot M,Meyer J,Arber W.Bleomycin-resistance gene derived from the transposon Tn5 confers selective ad⁃vantage to Escherichia coli K-12[J].Proc Natl Acad Sci USA,1991,88(20):9112-9116.
[8] Du M,Battles M B,Nett J H.A color-based stable multi-copy integrant selection system for Pichia pasto⁃ris using the attenuated ADE1 and ADE2 genes as auxotrophic markers[J].Bioeng Bugs,2012,3(1):32-37.

