基因组选择在我国种猪育种中应用的探讨
周 磊,杨华威,赵祖凯,杨红杰,刘剑锋*
(1.中国农业大学动物科技学院,北京 100193;2.武汉天种畜牧有限责任公司,湖北武汉 430344;3.全国畜牧总站,北京 100125)
基因组选择(Genomic Selection,GS)是目前动植物育种领域革命性的、最前沿的技术,该技术于2001年由挪威科学家Meuwissen等[1]首次提出。高密度单核苷酸多态性(Single Nucleotide Polymorphisms,SNP)全基因组芯片的应用使GS技术快速、高效地应用到动植物的育种中成为现实。GS技术可以大幅度提高育种值估计的准确性,缩短世代间隔,并减少群体的近交水平增长速度。2008年,科学家们研发了针对奶牛的含5万个SNP标记的全基因组芯片[2],使GS技术首先在奶牛的遗传评估中得以成功应用。目前,GS技术已在动植物的遗传评估中广泛应用[3-5],在人类的疾病预测和复杂性状分析中也有研究[6-8]。
我国是世界第一大生猪生产国和消费大国,2016年我国猪肉产量占全球猪肉总产量的48.5%。在支撑我国养猪业发展的众多因素中,种猪性能起着极其重要的作用。我国商品种猪(杜洛克、大约克和长白猪)和商品猪的生产性能仍与欧美发达国家存在较大差距,我国母猪年产断奶仔猪数(PSY)目前仅为16~18头,而欧美发达国家可达25~30头,这主要是由于我国大部分种猪企业不能持之以恒地进行育种工作。GS技术可实现相比于常规育种30%以上的额外遗传进展,有利于迅速提升种猪的性能水平,目前已普遍应用于欧美国家的种猪企业,有望成为未来种猪遗传评估的主要技术。
1 GS技术发展背景
GS技术最早应用于奶牛的遗传评估[3,5]。GS技术可提高青年公母牛育种值的准确性,并减少后裔测定的成本和缩短群体的世代间隔,在奶牛育种中取得了巨大的成功。Garcia-Ruiz等[9]报道,通过持续7年的GS技术应用,美国荷斯坦奶牛中等遗传力性状(产奶性状)年遗传进展增速增加了50%~100%, 低遗传力性状(健康性状、繁殖性状)的年遗传进展增速则增加了300%~400%。由此,GS技术以惊人的速度迅速取代了基于系谱的常规评估,成为了目前美国奶牛最主要的遗传评估技术。截止到2015年,美国进行SNP芯片检测的荷斯坦牛总数已达100万头。丹麦、荷兰和加拿大等其他奶业发达国家均在奶牛遗传评估中广泛地应用了GS技术。我国于2010年启动在荷斯坦牛中应用GS技术,并通过GS技术迅速地提高了北京地区荷斯坦奶牛的生产水平。除了奶牛以外,GS技术也被广泛应用于猪、蛋鸡、水产动物、玉米、水稻和小麦等动植物品种的育种研究和应用。
Ramos等[10]研发了首款猪的全基因组SNP芯片,该款芯片使GS技术应用于猪的育种生产成为可能。随后,PIC、丹育国际(Danbred)、海波尔(Hypor)和加裕(Genesus)等众多国际猪育种公司先后将GS技术应用于各自的遗传评估策略中。以PIC公司为例,其2013年启动了GS技术,是世界上第一个启动GS技术的种猪公司。2014—2017年,每头PIC商品猪由于GS技术额外增加的收益从4.4元/头逐年增加到17.6元/头。根据PIC公司报道,对于不同的性状,GS技术评估的准确度比基于系谱有不同幅度的提高。对于中等遗传力性状,如背膘厚,GS技术的准确度提高约30%;对于低遗传力性状,如产仔数和死亡率,GS技术的准确度提高60%~140%[11]。据推断,通过应用GS技术,PIC的遗传进展平均增速增加了35%以上。目前,PIC已启动了利用全基因组序列进行GS的研究应用项目。加拿大加裕公司通过应用GS技术实现平均日采食量和背膘厚的育种值准确性分别提高了55%和39%。
2 GS技术应用前景分析
本研究通过简单的模拟分析,估算一个基础母猪存栏为1 000头的纯种(单个品种)种猪场,通过实施5年GS技术可实现的经济效益。分析中使用的主要群体和生产参数见表1。

表1 单品种纯种群体基因组选择模拟分析参数
基于上述参数设计,经过5年GS方案实施,该场应用GS技术的年利润与常规评估收益如图1所示。在实施GS方案后的1~2年,GS评估的效益优势并不明显,但从第3年开始GS比常规评估具有越来越大的优势。在该GS方案中,每年需检测约2 000头测定猪(候选群)的SNP芯片,每年的芯片检测成本约为74万元。第2~5年GS技术比常规评估额外增加分别77万、169万、277万、404万元的收益。5年GS方案的总投入约为370万元,5年GS方案比常规评估累计增加收入约927万元(假设GS的遗传进展为常规评估的2倍)。此分析中,尚未考虑GS在扩繁群和商品群带来的额外效益。由此可见,通过实施GS技术,仅该纯种场亦可实现额外的经济效益。GS技术更适合于具有纯种核心群、扩繁群和商品群的集团养殖公司,集团公司整个繁育体系均能利用GS技术获得更多的额外收益。
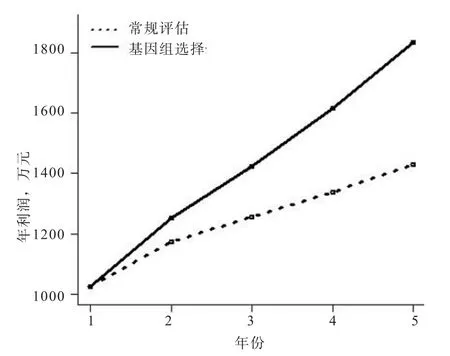
图1 某模拟纯种场5年实施GS技术的收益估算
GS技术应用于猪的育种主要有以下几方面优势:
1)大幅度提高繁殖力和肉质等低遗传力性状的育种值估计准确性。低遗传力性状常规评估的育种值准确性低,GS技术通过更加准确地估计个体之间的亲缘关系可大幅度提高低遗传力性状的育种值准确性[9,12]。肉质类性状需要对猪进行屠宰测定,测定成本昂贵,GS技术可以在保证一定的评估准确性的条件下适当地减少肉质性状的测定量。
2)一定程度地提高产量和屠体等中、高遗传力性状的育种值估计准确性,并可适当减少难以测定性状(如饲料转化率和胴体瘦肉率等)的测定量。基因组选择依赖于参考群与候选群之间的亲缘关系,因此每年依然需要对各性状进行一定数量的性能测定(用于扩充参考群),以保证参考群与最新的候选群存在稳定的遗传联系。
3)GS技术有利于应用于杂种优势的预测。在猪的“纯种选育”-“纯种扩繁”-“杂交生产”的三级金字塔繁育体系中,纯种群、扩繁群和商品群通常分布于不同的公司或猪场,不同级别间群体缺少系谱记录。基于系谱的常规评估难以有效地预测杂种优势,且难以利用杂种信息对纯种核心群进行评估。GS技术可通过芯片信息估计核心群与杂种后代的基因组亲缘关系(Genomic Relationships),进而实现更加准确的核心群评估和杂种优势预测[12]。这有利于更加充分地利用杂种后代的性能测定记录,加速选育提高核心群纯种个体后代的杂交性能[13]。
4)GS技术可提高多品种、多群体和多性状评估的准确性。多品种或多群体之间由于缺乏共同祖先,一般无法通过系谱构建亲缘关系矩阵。GS技术可利用基因组标记信息构建多品种、多群体的基因组亲缘关系矩阵,进而实现更大的参考群体,并实现准确性更高的多品种、多群体的遗传评估[14]。然而,针对没有遗传联系的多群体、多品种的联合评估,目前尚无特别有效的模型报道,因此需要进行更多理论和方法的研究探索。
5)基因组信息可用于控制选配、减少群体近交。由于基因组亲缘关系比基于系谱的亲缘关系更加准确地揭示了个体之间的实际亲缘关系,进而利用基因组亲缘关系可以更加准确、有效地控制选配,减少群体近交水平增加的速度。
6)GS技术可应用于加速疾病抵抗力和健康度选育的遗传进展。众多的疾病遗传机制复杂,基于个别遗传标记的分子标记辅助选择(Marker Assisted Selection,MAS)通常无法应用于抗病育种。GS技术可间接地捕获影响疾病性状的众多微效多基因,加速群体健康度和疾病抵抗力的遗传进展。如何利用基因组信息进行抗病育种亟需更多的研究和探讨。
3 GS技术在我国猪育种中的应用现状
由于我国种猪企业数量多、规模小,GS技术的应用起步较慢。广东温氏食品集团2011年在我国率先启动猪的GS技术[15]。目前为止,GS技术仅在我国少数的种猪公司展开,且进展相对比较缓慢。
GS技术在我国种猪企业应用的限制因素:① 缺乏长期积累的大量且准确的表型数据,如达100 kg校正日龄、达100 kg校正背膘厚和饲料转化率等性状的表型数据;②肉质、抗病和胴体类性状难以测定或测定成本昂贵;缺乏有效表型数据,导致难以建立GS技术所必须的一定规模的参考群体。③ 市场行情波动剧烈,影响育种公司进行长期稳定的育种工作和加大育种投入的决心。④ 缺乏可实施GS技术的易操作和应用的评估软件系统。如开福(KFNets)和GPS,均只能进行常规的基于系谱的遗传评估,尚未能实施利用基因组信息的GS技术。
4 GS技术在我国的应用策略
为实现GS技术应用效果的最优化,我国的种猪企业可采取以下步骤:
1)建立表型数据库:包括收集生长性能测定数据(达100 kg日龄、达100 kg背膘厚、眼肌面积和饲料转化率等)、繁殖数据(产仔间隔、仔猪死亡率、出生重等)和肉质数据等。低遗传力性状通过GS技术能够取得与常规评估相比更高的准确性[9,16]。GS技术可加速低遗传力性状(如产仔数、肌内脂肪含量等)的遗传进展。由此,我国种猪企业应加强表型数据的持续收集,尤其是低遗传力性状(肉质类、繁殖类等)数据。
2)构建大规模参考群:参考群规模越大,GS技术评估准确性越高[2]。参考群应包括本品种核心群的所有家系,涵盖所有核心群生产公猪和生产母猪。单个品种参考群的规模需达到1 000头或以上。参考群需保证含有一定比例的母猪[12],进而保证繁殖力性状具有一定规模的参考群。参考群个体同时需要有准确的表型数据。对参考群进行SNP芯片检测,目前可用的芯片有Illumina公司的PorcineSNP60 60K芯片、纽勤公司的PorcineSNP80 70K芯片和Affymetrix公司的Axiom PorcineHD 70K芯片等(表2)。江西农业大学最新研究的“中芯1号”,囊括多肋、肉色、系水力和肉质等重要性状因果基因紧密关联位点,也适合于我国商业化猪种的芯片测定。中国农业大学最新研发的CAUporcine55K 也适合于我国商业化品种芯片检测。该版芯片专门性的针对我国商品化猪种和地方品种设计。
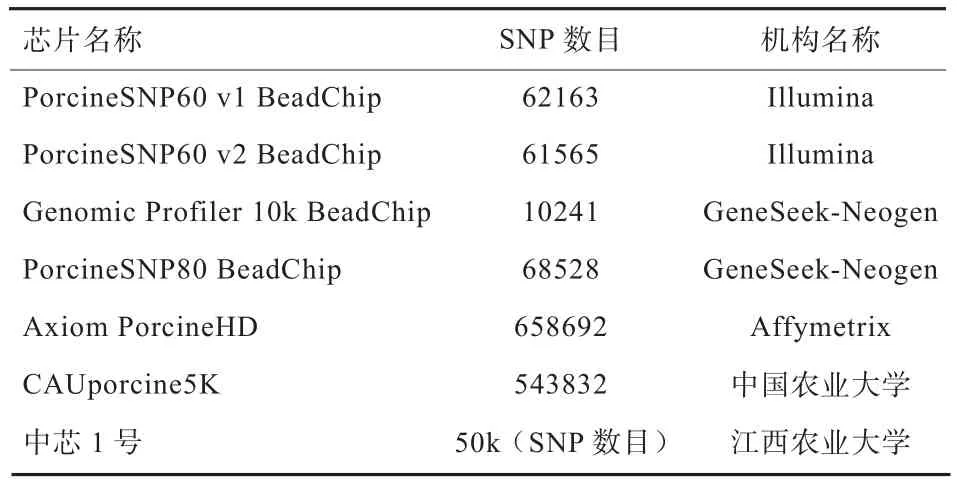
表2 目前可应用的猪全基因组芯片
3)候选群芯片检测:在拟进行生长性能测定的种猪中,挑选一定比例的(10%~50%)种猪进行芯片检测(构建候选群)。候选群的SNP芯片检测可以采取多种不同的策略。候选群可在种猪性能测定前(哺乳阶段)进行芯片基因型测定,该方案可用于筛选基因组育种值(GEBV)高的种猪进行饲料转化率的性能测定,并为后续测定种猪的选留提供参考信息。候选群也可在种猪性能测定结束后(达100 kg或120 kg)进行芯片检测,该方案可应用准确性更高的GEBV对测定种猪进行筛选。候选群采用的芯片版本需与参考群版本尽量保持一致,这有利于保证SNP标记在参考群和候选群具有类似的多态性,同时便于利用候选群体扩充参考群的规模。随着对候选群持续地进行芯片测定,参考群的规模亦随之逐渐增加。
4)GS技术评估和选留:根据候选群和参考群的数据(表型数据和基因型数据),应用适当的统计模型进行GS技术评估,并根据GEBV对候选群进行选留。应用GS技术的种猪选育流程的主要内容如图2所示。GS技术评估需要高配置的计算机运算系统和高性能计算平台,这一步我国育种企业可通过与研究高校或机构的“产、学、研、推”合作实现。
5 建议和展望
综合比较我国及国际的种猪企业,我国种猪企业需加速GS技术的应用。鉴于此,提出以下几点建议:①进一步加强性能测定和表型数据的收集。一定规模的表型数据是构建基因组选择参考群的基本条件之一。没有准确的表型和一定规模的参考群,GS技术难以取得理想的经济效益。② 推进区域性基因组联合评估联盟的发展。以区域性的种猪企业(国家核心场)为成员单位,构建由3~5家企业参与的区域性育种联盟。以联盟为平台,建立核心群之间的遗传联系,并实施联合基因组选择评估方案,最终实现准确性更高的遗传评估和更快的群体遗传进展。同时,联盟内企业分摊GS技术成本,可减轻单个企业的育种成本投入。③ 应用“单步法”或“一步法”GS技术[17-18]。该技术可同时对群体中检测芯片和不检测芯片的个体进行评估,最大程度利用基因组信息和系谱信息,实现全群最大的遗传进展。
GS技术已经在奶牛上广泛应用,并彻底地改变了荷斯坦牛的育种工作流程。猪的育种是基于多品种杂交的繁育体系。多品种基因组选择目前依然更多地处于研究阶段[19-20]。因此,GS技术在猪育种中的应用尚不及其在奶牛育种中的应用普遍。随着多品种GS方法的研究不断深入,基于GS技术的猪育种技术策略将不断地完善和发展,并为群体的性能改良提升带来新的突破。目前,大数据时代全基因组测序技术不断推陈出新,测序成本不断降低,众多研究机构已在进行基于测序数据的GS2.0研究。GS技术和测序技术必然将猪育种带入一个新的基因组时代[21]。
[1] Meuwissen T H, Hayes B J, Goddard M E. Prediction of total genetic value using genome-wide dense marker maps[J].Genetics, 2001, 157(4): 1819-1829.
[2] Matukumalli L K, Lawley C T, Schnabel R D,et al.Development and characterization of a high density SNP genotyping assay for cattle[J]. PLoS One, 2009, 4(4): e5350.
[3] Hayes B J, Bowman P J, Chamberlain A J,et al. Invited review:genomic selection in dairy cattle: progress and challenges[J]. J Dairy Sci, 2009, 92: 433-443.
[4] Su G, Guldbrandsten B, Gregersen V R,et al. Preliminary investigation on reliability of genomic estimated breeding values in the Danish Holstein population[J]. J Dairy Sci, 2010,93, 1175-1183.
[5] Van Raden P M, Van Tassell C P, Wiggans G R,et al. Invited review: reliability of genomic predictions for North American Holstein bulls[J]. J Dairy Sci, 2009, 92: 16-24.
[6] Campos G D L, Vazquez A I, Fernando R,et al. Prediction of complex human traits using the genomic best linear unbiased predictor[J]. PLoS Genet, 2013, 9(7): 523-528.
[7] Abraham G, Tye-Din J A, Bhalala O G,et al. Accurate and robust genomic prediction of celiac disease using statistical learning[J]. PLoS Genet, 2014, 10(2): 163-170.
[8] Spiliopoplou A, Nagy R, Bermingham L M,et al. Genomic prediction of complex human traits: relatedness, trait architecture and predictive meta-models[J]. Hum Mol Genet,2015, 24(14): 4167.
[9] Garcia-Ruiz A, Cole J B, Van Raden P M,et al. Changes in genetic selection differentials and generation intervals in US Holstein dairy cattle as a result of genomic selection[J]. Proc Natl Acad Sci, 2016, 113: E3995-E4004.
[10] Ramos A M, Croojmans R A, Affara N A,et al. Design of a high density SNP genotyping assay in the pig using SNPs identified and characterized by next generation sequencing technology[J].PLoS One, 2009, 4(8): e6524.
[11] 王晨, 秦珂, 薛明, 等. 全基因组选择在猪育种中的应用[J].畜牧兽医学报, 2016(1): 1-9.
[12] Samore A B, Fontanesi L. Genomic selection in pigs: state of the art and perspectives[J]. Ital J Anim Sci, 2016, 15(2): 211-232.
[13] Dekker J C. Marker-assisted selection for commercial crossbred performance[J]. J Anim Sci, 2007, 85(9): 2104.
[14] Verpneze R, Bastiaansen J W M, Lopes M S. Multi-population genomic selection in pigs[C]. San Diego, CA, USA: Proceedings of the 23rd International Plant Animal Genome, 2015:351.
[15] 王青来, 李亚兰. 全国首例全基因组选育特级种猪在粤诞生[J]. 农业知识, 2014(2): 25-25.
[16] Lillehammer M, Meuwissen T H, Sonesson A K. Genomic selection for maternal traits in pigs[J]. J Anim Sci, 2011,89(12): 3908-3916.
[17] Kang H, Zhou L, Mrode R,et al. Incorporating single-step strategy into random regression model to enhance genomic prediction of longitudinal trait[J]. Heredity, 2017, 119(6): 459-467.
[18] Chrsitensen O F, Madsen P, Nielsen B,et al. Single-step methods for genomic evaluation in pigs[J]. Animal, 2012,6(10): 1565-1571.
[19] Zhou L, Lund M S, Wang Y,et al. Genomic predictions across Nordic Holstein and Nordic Red using the genomic best linear unbiased prediction model with different genomic relationship matrices[J]. J Anim Breed Genet, 2014, 131(4): 249-257.
[20] Erbe M, Hayes B J, Matukumalli L K,et al. Improving accuracy of genomic predictions within and between dairy cattle breeds with imputed high-density single nucleotide polymorphism panels[J]. J Dairy Sci, 2012, 95(7): 4114-4129.
[21] 陈瑶生 , 王 健, 刘小红 , 等 . 中国生猪产业新趋势 [J]. 中国畜牧杂志, 2015, 51 (2): 8-19.

