基于非目标1H NMR指纹图谱技术验证中国葡萄酒原产地
樊双喜,钟其顶,黄占斌,杨彤辉
1 (中国矿业大学(北京) 化学与环境工程学院,北京,100083) 2(中国食品发酵工业研究院,北京,100015) 3(全国食品发酵标准化中心,北京,100015)
葡萄酒的品质主要取决于酿酒葡萄,由于葡萄种植具有比较明显的区域性特征,葡萄产地的气候和土壤等生态环境同样也会影响葡萄酒质量[1]。地理标志是目前国际上通行的对酒类产品进行管理的办法,用于突出某一葡萄酒产地的特点,防止假冒伪劣葡萄酒产品。经过多年的发展,我国对昌黎、沙城以及昌吉实施了原产地域产品/地理标志产品保护[2]。然而,据报道,在中国葡萄酒行业存在许多利益驱动的虚假产地葡萄酒欺诈事件,例如通过旧瓶装新酒、仿造酒标等手段冒充具有高价值产区的葡萄酒[3],严重损害了中国葡萄酒市场的声誉。因此,迫切需要开发能够准确验证葡萄酒原产地的技术[4]。
目前用于原产地葡萄酒分析的方法较多,例如气相色谱(GC)与质谱(MS)联用[5-6]、高效液相色谱法(HPLC)[7-8]、电感耦合等离子体质谱仪(inductively coupled plasma mass spectrometry,ICP-MS)[9-11]、近红外线(near infra-red,NIR)和中红外线(middle infra-red,MIR)光谱[12-14]、稳定同位素[15-18]、核磁共振光谱(nuclear magnetic resonance,NMR)[19],以及以上不同方法的组合[20-23]。利用葡萄酒特征组分(香气、类黄酮、有机酸、氨基酸、多酚、无机元素、同位素等)和多元统计分析技术结合,能够实现葡萄酒产地、品种和年份的判别。然而,特定特征组分检测技术在当代的食品安全与质量检测中具有一定的局限性,比如食品中蓄意掺假物质的手段层出不穷,尤其对于某些未知化学结构的添加物的样品,无法通过对特定目标化合物的定向检测达到区分的目的[24]。相对于目标检测技术而言,非目标检测技术经过多年的发展,已经在食品及原料的溯源分析、掺假检测等相关领域进行了比较深入的研究。非目标检测技术是以样品的整体化学或生物学信息为基础,通过分析化学、细胞和分子生物学等技术手段与多元统计学相结合来综合评判与分析食品特征[25],其中多元统计分析手段主要包括主成分分析(principal component analysis, PCA)、线性判别(linear discrimination analysis, LDA)、聚类分析(cluster analysis, CA)、偏最小二乘法 ( partial least squares, PLS)、正交偏最小二乘法判别分析(orthogonal partial least squares discriminant analysis, OPLS-DA)、K临近算法(k-nearest neighbors, KNN)、神经网络(artificial neural networks, ANN)等。
非目标一维核磁共振氢谱(1H NMR)技术作为一种快速、简单、无损检测方法,在葡萄酒产地验证以及特征分析领域显得越来越重要[19,26]。本研究首次采用非目标指纹图谱(1H NMR)结合PCA和LDA多元统计分析手段,初步建立了沙城、昌黎和昌吉三大特色原产地葡萄酒的验证模型,探讨了以非目标1H NMR技术实现葡萄酒原产地溯源的可行性。
1 材料与方法
1.1 材料
本实验所有葡萄酒样品均由企业提供。2010~2015年,从沙城、昌黎和昌吉葡萄酒原产地不同葡萄酒企业共收集224个葡萄酒样品,其中沙城101个(赤霞珠26个、玫瑰蜜9个、蛇龙珠15个、白玉霓 29个、龙眼 21个、霞多丽1个),昌黎60个(赤霞珠49个、玫瑰蜜2个、蛇龙珠9个),昌吉63个(赤霞珠44个、霞多丽6个、蛇龙珠9个、雷司令2个、味儿多和西拉各1个)。所有葡萄酒样品均存放在4 ℃冰箱避光冷藏。葡萄酒样品均为单一葡萄品种,没有混合其他葡萄品种。所有收集的葡萄酒样品均符合沙城、昌黎和昌吉葡萄酒地理标志产品国家标准的规定[2]。
1.2 仪器与试剂
叠氮化钠(超级纯,NaN3, Merck, Germany);3-(三甲基硅基)氘代丙酸钠(TSP)和KH2PO4(98%,Merck, Germany);重水(D2O,98%,Merck, Germany);NaOH和HCl(99%,Sigma-Aldrich,USA);核磁共振波谱仪(400 MHz,Bruker,Germany);pH计 (SevenCompactTMS210,Mettler-Toledo, Switzerland);涡流振荡器(Biosan Ltd, Latvia);5 mm 带帽NMR测试管(Wilmad Labglas Inc, USA)。
1.3 样品前处理
采用重水(D2O),叠氮化钠(NaN3),0.1% 3-(三甲基硅基)氘代丙酸钠(TSP),1 mol/L KH2PO4配制磷酸盐缓冲液。采用H3PO4或KH2PO4准确调节磷酸盐缓冲液的pH值至2.0。100 μL磷酸盐缓冲液加入900 μL葡萄酒,采用涡流振荡器振荡30 s 混合均匀,然后用2 mol/L 的NaOH或HCl准确调整混合物的pH值至3.0±0.02。准确取600 μL的混合物转移到5 mm NMR测试管,测试管之后采用一维核磁共振氢谱(1H NMR)技术进行检测。
1.4 1H NMR实验流程及仪器参数设置
一维核磁共振氢谱(1H NMR)检测一般实验流程包括调谐和匹配、温度控制、锁定、匀场、选择序列、参数设置以及最后样品检测。其中调谐和匹配、温度控制、锁定及匀场过程均可实现高度自动化,操作简单。仪器实验条件设置:检测温度(300±0.2) K。采用5 mm1H/D 探头,1H NMR的共振频率为400.27 MHz,Bruker Top Spin 2.1软件用于1H NMR图谱数据的采集, 检测信号时间3.984 6 s,延迟时间4.0 s,每次自由感应衰减扫描次数为16,空扫描次数为4,谱宽15.1 ppm,采样点数为65 536 (64 K),谱线加宽因子为0.3 Hz。以3-(三甲基硅基)氘代丙酸钠(TSP,δ=0)作为内标,化学位移的单位(ppm)。标准的脉冲序列(NOESYGPPS) 用于乙醇 (δ=1.2 ppm和3.6 ppm)和水(δ=4.8 ppm)的信号抑制,有效地减弱了水峰和乙醇峰对检测的干扰。样品检测前,需要等待5 min用来平衡温度。
1.5 多元统计分析
采用MestReNova 9.1软件(Mestrelab Research S.L., MestReNova (Mnova) NMR,USA) 首先对1H NMR图谱进行傅里叶变换,基线矫正、调整相位。然后对1H NMR图谱峰化学位移偏移进行校正对齐,最后对图谱进行分段积分,其值作为之后多元统计分析的输入变量。为了便于数据处理,同一产地葡萄酒样品分为一组。多元统计分析包括无监督主成分分析(PCA)和有监督线性判别分析(LDA)。
PCA作为多元探索性数据降维分析的第一步,根据解释1H NMR数据集内95%方差来确定主成分矩阵。主成分得分矩阵作为LDA的输入变量,由于主成分之间相互正交,因此可以避免1H NMR数据集里的初始变量之间高度共线性问题。LDA可以实现因变量组间差异的最大化,组内样品距离的最小化, 提高模型的有效性和解析能力[27]。PCA和 LDA数据分析均由R软件3.2.3版本[28]处理完成。PCA和LDA分析采用的R软件具体的数据包分别是mdatools[29]和 mass[30]。PCA和LDA分析之前,所有数据由R软件采用帕累托(Pareto)方法进行标准化。应用帕累托方法即每个变量除以其标准差(SD)的平方根,这有利于降低数据矩阵中变量具有很大的数值的相对重要性而不增加基线噪声[31-32]。
随后采用重复双随机交叉验证[33]和留一交叉验证方法[34],对建立的PCA/LDA模型的有效性进行验证,均由R软件编程完成。
2 结果与分析
2.1 1H NMR数据质量控制
所有224个葡萄酒样品采用核磁共振一维氢谱(1H NMR)进行测定。不同产地之间葡萄酒样品记录的氢谱看起来非常相似。由于采用标准的脉冲序列(NOESYGPPS)用于水(4.8 ppm)以及乙醇(1.2 ppm, 3.6 ppm)信号压制,葡萄酒中微量成分的信号强度得到了明显提高。将所有葡萄酒样品的核磁共振氢谱叠加,发现一些氢谱出现了基线较大的偏移,可能是由于水和乙醇的抑制不当,pH调节不准确,系统误差或其他原因造成[35]。因此,需要对这些葡萄酒样品进行重复测定,以保证数据质量的可靠性。
2.2 1H NMR葡萄酒主要代谢物的归属
在保证图谱数据质量的基础上,对1H NMR图谱的主要葡萄酒代谢物/成分(氨基酸、糖、有机酸、乙醇)进行了归属。通过葡萄酒加入相应的化学物质标品可以进一步验证主要代谢物的归属是否正确。本研究提供了一个典型中国红葡萄酒1H NMR图谱,并且在图谱上给出了葡萄酒主要代谢物/成分的共振信号(见图1)。
2.3 1H NMR图谱峰化学位移对齐以及数据降维
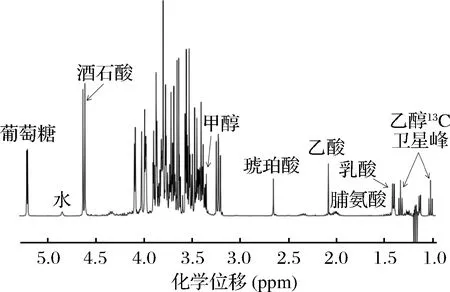
图1 一个典型的标有主要葡萄酒代谢物的中国红葡萄酒1H NMR图谱(其中果糖、葡萄糖和甘油信号出现在3~4.2 ppm高度重叠区域)Fig.1 One typical 1H NMR spectrum of a Chinese red wine with given resonance signals of the major wine metabolites/ ingredients (highly overlapped signals of fructose,glucose as well as glycerol occur in the range of 3.0-4.2 ppm)
由于样品pH值微小的变化会造成1H NMR图谱峰化学位移偏移[31],本研究葡萄酒氢谱一些典型峰信号出现的化学位移偏移如图2所示。

图2 葡萄酒样品一维核磁共振叠加氢谱化学位移在(1.95~2.05)ppm及(3.73~3.76)ppm区域对齐前后[(A)-(B)、(C)-(D)]的比较Fig.2 Comparison of raw and aligned 1H NMR spectra of wine samples: Raw (A) and aligned (C)1H NMR spectra in the region (1.95-2.05) ppm; Raw (B) and aligned (D) 1H NMR spectra in the region 3.73-3.76 ppm
适当的峰化学位移偏移校正对齐对随后的多元变量分析至关重要。化学位移偏移会引起化学计量学模型样本错误的分组。为了解决这一问题,至今为止已经报道了多种峰化学位移校正对齐方法[35]。MestReNova采用的是分段对齐的方法,该方法能够允许用户采用交互式页面,灵活的选择峰化学位移校正对齐的区域。每段的平均图谱作为参考图谱进行对齐,依次进行下去,直到得到满意的1H NMR图谱。图2-A和图2-B采用分段对齐后的图谱分别如图2-C和图2-D所示。由图2可知,分段对齐方法较好的解决了1H NMR图谱峰化学位移的偏移问题。在MestReNova完成1H NMR图谱整个峰化学位移对齐以及分段积分之后,整个1H NMR图谱被细分为多个区域,每个区域所有点的积分值累加起来可以抽象的代表整个原始图谱[36],这些代表性的区域积分值作为接下来建模的输入变量,从而实现对每1H NMR图谱约60 000个高共线性数据点的有效降维。
选取了0.01, 0.015, 0.02, 0.025, 0.03, 0.035 和0.04 ppm作为每个分段积分的区域宽度取值,结果表明0.02 ppm能够保持足够的数据分辨率和最大限度地减少图谱信息的损失。由于1H NMR中的水和乙醇信号的区域与本研究调查的问题无关,所以这些区域被排除在外。分段积分的有效区域是在0.8~9.5 ppm(其中不包含被特定脉冲序列压制极度变形的水峰δ(H2O)=4.65~5.10 ppm) 和乙醇峰(δ(CH3)=1.05~1.21 ppm,δ(CH2)=3.60~3.72 ppm),分段积分后能够获得大约400个分段积分值,这些分段积分值作为之后多元统计分析的输入变量。
2.4 变质葡萄酒的识别
在化学计量学数据分析之前,对所有的葡萄酒样品进行是否变质筛查。葡萄酒样品会由于不恰当的储存条件以及超过葡萄酒本身的货架期而导致变质或腐败[36]。通过叠加所有葡萄酒样品的核磁共振氢谱,结果发现一些葡萄酒样品图谱在2.07 ppm处的乙酸信号峰的绝对积分值明显高于其他大多数葡萄酒样品。因此,对这些葡萄酒样品需要仔细检查以发现潜在的变质。根据GB15037—2006规定,葡萄酒的挥发性酸的最大值为1.2 g/L。基于外部标准的PULCON(pulse-length-based concentration)[37]方法对所有葡萄酒样品中的乙酸含量进行定量。24个葡萄酒样品(10个赤霞珠,2个玫瑰蜜,9个蛇龙珠,3个霞多丽)乙酸含量已经超过1.2 g/L,因此在做进一步的统计分析之前需要排除这些样品。
然而GODELMANN等[37]直接删除乙酸信号峰建模,在本研究中则是保留了乙酸信号峰(乙酸含量低于1.2 g/L),与PAPOTTI等[38]有关于葡萄酒产地研究的1H NMR图谱前处理相一致。本研究很好地保持了非目标指纹图谱方法特点,而不是预先忽略乙酸或其他腐败参数对结果的影响。尽管乙酸与葡萄产地几乎没有关联,并且在随后建立的产地PCA/LDA模型时发现,乙酸信号峰区域并没有对产地的区别能力有重要贡献。
2.5 探索性数据分析
主成分分析同时也称主分量分析,利用降维的思想,通过线性变换把多指标转为少数几个综合指标,且综合指标不相关,这些新的综合指标按照方差依次递减的顺序排列[39]。
将1H NMR谱图进行峰对齐、分段积分等数据预处理操作后,将红、白葡萄酒数据导入R数据分析软件中,通过主成分分析,将红葡萄酒和白葡萄酒进行区分。由主成分分析可得图3。

图3 沙城(SC)、昌黎(CL)和昌吉(CJ)葡萄酒样品主成分1(PC1)和主成分2(PC2)得分图Fig.3 The score plot of principal component 1 (PC1) and principal component 2 (PC2) for the wine samples of Shacheng, Changli and Changji
图3初步反映了昌吉、昌黎和沙城葡萄酒样品在PC1(Comp 1)和PC2(Comp 2)组成的二维坐标体系中的无监督的分组情况,同时也可以得到PCA模型前两个主成分解释数据的方差累积贡献率约为80%,PC1占总变量的71.28%。PC2占总变量的8.23%。同时根据Kaiser准则,方差累积贡献率超过80%,表明PCA模型具有显著的实际意义。通过对图3进一步观察发现,昌黎葡萄酒样品聚集的较为分散,昌吉和沙城两个产地的葡萄酒样品区域仍然会有较大程度的重叠,结果表明PCA模型无法实现对葡萄酒产地进行区分。
2.6 线性判别分析(LDA)
为了解决PCA无法区分来自昌黎、 沙城以及昌吉产地的葡萄酒样品,引入了LDA方法进行数据分析。由于1H NMR图谱原始数据既有对分类起作用的差异变量,同时又包含了大量对分类无作用的变量,变量之间也存在高度共线性问题[40]。为了解决这一难题,本研究采用PCA所得的解释1H NMR数据集内95.8%方差确定的相互独立的10个主成分,作为LDA的输入变量。通过PCA/LDA建立的线性判别函数能够使这样具有高维(多变量)、小样本数据分类可视化效果最大化。由PCA/LDA线性判别函数1和2得分图4可以看出,城葡萄酒样品组内的离散的程度减少,组间的区分更加清晰,表明PCA/LDA模型能够很好地对沙城、昌黎以及昌吉产地葡萄酒进行有效的区分。
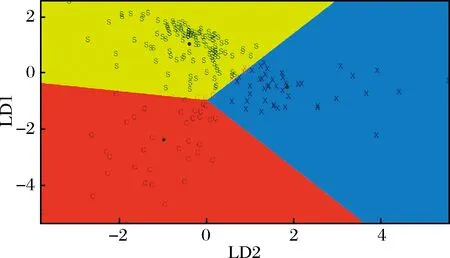
图4 PCA/LDA模型对昌黎(C)、昌吉(X)和沙城(S)葡萄酒样品线性判别函数(LD 1)和线性判别函数(LD 2)得分图Fig.4 The score plot of linear discriminant function 1 and linear discriminant function 2 of PCA/LDA for the wine samples of Changli (C), Changji (X) and Shacheng (S)
2.7 PCA/LDA模型有效性验证
在实际数据分析过程中PCA/LDA模型通常容易产生过拟合现象,从而得到较好的分类判别效果的假象。多元统计模型的验证对于任何指纹图谱的方法的评估和评价至关重要[41]。为此本研究分别采用了内部留一交叉验证方法和外部重复双随机交叉验证来评价PCA/LDA模型分类结果的可靠性。图5直观解释了两种验证方法的关键步骤。

图5 PCA/LDA模型验证方法Fig.5 The validation methods of PCA/LDA model
第一步采用内部留一交叉验证的方法评价PCA/LDA模型的性能。广泛运用的留一交叉验证方法的原理是:假设数据集有N个样本,随机选取N-1个样本作为训练样本,剩下的1个样本作为测试样本。这样每个样品将会被模型预测1次,因此得到的N个分类结果可以用来保守的衡量模型的分类性能。表1总结了采用内部交叉验证的PCA/LDA模型的昌黎、沙城和昌吉葡萄酒整个数据集分类结果。

表1 内部留一交叉验证的PCA/LDA模型对沙城、昌黎和昌吉葡萄酒整个数据集分类结果Table 1 Classification results of PCA/LDA models usinginternal leave one-out cross validation (LOOCV) forthe wines from Shacheng, Changli and Changji (entire data sets)
重复双随机交叉验证主要的原理是首先将原始完整数据集采用随机分层抽样方式(注:本研究随机分层重复抽样100次),将原始数据集分为两部分:(1)80%数据作为训练集用于建模;(2)20%数据作为外部验证集用于预测。由于对整个数据集随机外部验证集抽样100次,于是就得到了100个不同的外部验证集。对这100个外部验证集样本正确分类率的平均值、标准偏差、中位数以及中位数的绝对标准偏差进行统计分析(见表2),用这4项指标评估模型的有效性。正确分类率的平均值及中位数这2个指标数值越接近1、标准偏差以及中位数绝对标准偏差值越小,表明建立的模型正确分类能力越强。从表2可以看出,模型外部验证集昌黎和昌吉葡萄酒样本正确分类率的平均值和中位数均大于0.7,沙城葡萄酒样本正确分类率的平均值和中位数均高于0.9,平均值及中位数相对应的标准偏差均较小,且3个产地正确分类率的平均值与中间值统计学上无显著性差异(p>0.05), 以上结果表明重复双随机交叉验证能够充分表明建立的PCA/LDA模型的有效性。
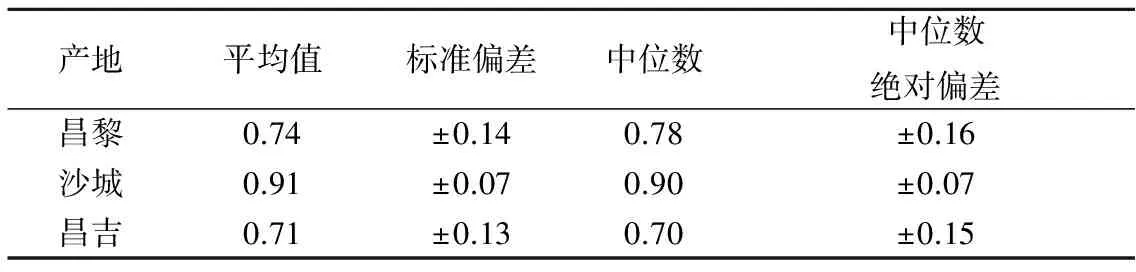
表2 重复双随机交叉验证外部验证集正确分类率的平均值、标准偏差、中位数以及中间值绝对偏差Table 2 The mean, the standard deviation (SD), themedian and the median absolute deviation (MAD) ofcorrect classification rate for the external validation set ofrepeated double random cross validation
3 结论
本文探讨了非目标1H NMR指纹图谱技术结合多元统计分析手段对昌黎、沙城和昌吉葡萄酒产地验证技术的可行性。研究结果表明,PCA/LDA模型能够实现葡萄酒产地比较好的区分,进一步表明1H NMR指纹图谱技术可以作为一项非常有效的验证葡萄酒产地真实性手段。建立的相关非目标1H NMR指纹图数据库能够推动我国葡萄酒产地真实性溯原制度的建立与完善,进一步保障葡萄酒市场稳定健康发展,维护消费者的合法权益。
然而,本研究依然存在一些缺陷,例如,昌黎、沙城和昌吉收集的大多数是赤霞珠葡萄酒样品;同时由于各大产地种植的葡萄品种及规模不一样,造成了每个葡萄酒产地收集到的葡萄酒样品品种及数量差别较大,这对建立产地识别模型提出了挑战。在未来的研究中,应该收集更多具有代表性的,如不同品种、不同年份、不同产地且来源真实可靠的葡萄酒样品,以保障建立一个更加真实可靠的非目标1H NMR指纹图数据库。
[1] SEN I,TOKATLI F.Authenticity of wines made with economically important grape varieties grown in Anatolia by their phenolic profiles[J].Food Control,2014,46(46):446-454.
[2] 地理标志产品保护规定[J].中国质量技术监督,2005(9):14-15.
[3] BALESTRINI P,GAMBLE P.Country-of-origin effects on Chinese wine consumers[J].British Food Journal,2006,108(5):396-412.
[4] 吴浩,靳保辉,陈波,等.葡萄酒产地溯源技术研究进展[J].食品科学,2014,35(21):306-314.
[5] 秦丽娜,倪元颖,梁方华,等.利用香气成分识别葡萄酒品种和产地的初步研究[J].酿酒科技,2008(2):40-44;46.
[6] 王方,王伟,张春娅,等.赤霞珠干红葡萄酒产地识别的研究[J].中外葡萄与葡萄酒,2008(1):4-7.
[7] GARCIA-PARILLA M C.Differentiation of wine vinegars based on phenolic composition[J].Journal of Agricultural and Food Chemistry,1997,45(9):3 487-3 492.
[8] 莫寅斌.贺兰山东麓地区葡萄酒指纹图谱的研究[D].杨凌:西北农林科技大学,2009.
[9] GEANA I,IORDACHE A,IONETE R,et al.Geographical origin identification of Romanian wines by ICP-MS elemental analysis[J]. Food Chemistry,2013,138(2-3):1 125-1 134.
[10] GREENOUGH J D,MALLORY-GREENOUGH L M,FRYER B J.Geology and wine 9:regional trace element fingerprinting of Canadian wines[J].Geoscience Canada,2005,32(3):129-137.
[11] COETZEE P P,VANHAECKE F.Classifying wine according to geographical origin via quadrupole-based ICP-mass spectrometry measurements of boron isotope ratios[J].Analytical and Bioanalytical Chemistry,2005,383(6):977-984.
[12] ARANA I,JAREN C,ARAZURI S.Maturity,variety and origin determination in white grapes (VitisviniferaL.) using near infrared reflectance technology[J].Journal of Near Infrared Spectroscopy,2005,13(1):349-357.
[13] PICQUE D,CATTENOZ T,CORRIEU G,et al.Discrimination of red wines according to their geographical origin and vintage year by the use of mid-infrared spectroscopy[J].Sciences Des Aliments,2005,25(3):207-220.
[14] FERN NDEZ K,AGOSIN E.Quantitative analysis of red wine tannins using Fourier-transform mid-infrared spectrometry[J].Journal of Agricultural and Food Chemistry,2007,55(18):7 294-7 300.
[15] CAMIN F,BONER M,BONTEMPO L,et al.Stable isotope techniques for verifying the declared geographical origin of food in legal cases[J].Trends in Food Science and Technology,2017,61:176-187.
[16] PAOLANARANJO R D D,BARONI M V,PODIO N S,et al.Fingerprints for main varieties of Argentinean wines:terroir differentiation by inorganic, organic, and stable isotopic analyses coupled to chemometrics[J].Journal of Agricultural and Food Chemistry, 2011,59(14):7 854-7 865.
[17] DUTRA S,ADAMI L,MARCON A,et al.Determination of the geographical origin of Brazilian wines by isotope and mineral analysis[J].Analytical and Bioanalytical Chemistry,2011,401(5):1 575-1 580.
[18] GEANA E I,POPESCU R,COSTINEL D,et al.Verifying the red wines adulteration through isotopic and chromatographic investigations coupled with multivariate statistic interpretation of the data[J].Food Control,2016,62:1-9.
[19] GODELMANN R,FANG F,HUMPFER E,et al.Targeted and non-targeted wine analysis by1H NMR spectroscopy combined with multivariate statistical analysis.Differentiation of important parameters:grape variety,geographical origin,year of vintage[J].Journal of Agricultural and Food Chemistry,2013,61(23):5 610-5 619.
[20] SELIH V S,SALA M,DRGAN V.Multi-element analysis of wines by ICP-MS and ICP-OES and their classification according to geographical origin in Slovenia[J].Food Chemistry,2014,153(153):414-423.
[21] 薛洁,蒋露.SNIF-NMR和IRMS技术在原产地葡萄酒鉴定中的应用[J].食品与发酵工业,2011,37(10):154-158.
[22] 蒋露,薛洁,林奇,等.SNIF-NMR和IRMS技术在葡萄酒质量评价中的初步研究[J].食品与发酵工业,2008,34(11):139-143.
[23] JIANG W,XUE J,LIU X,et al.The application of SNIF-NMR and IRMS combined with C, H and O isotopes for detecting the geographical origin of Chinese wines[J].International Journal of Food Science and Technology,2015,50(3):774-781.
[24] 俞良莉,陆维盈,刘洁,等.食品非目标性检测技术[J].食品科学技术学报,2016,34(6):1-6.
[25] 俞良莉,王硕,孙宝国.食品安全化学[M].上海:上海交通大学出版社,2014:283.
[26] DU Y Y,BAI G Y,ZHANG X,et al.Classification of wines based on combination of1H NMR spectroscopy and principal component analysis[J].Chinese Journal of Chemistry,2007,25(7):930-936.
[27] MARDIA K V, KENT J T, BIBBY J M.Multivariate analysis[M].San Diego:London Academic Press,1979.
[28] R Core Team.R:A language and environment for statistical computing.https://www.R-project.org/.
[29] KUCHERYAVSKIY S.mdatools: multivariate data analysis for chemometrics.https://CRAN.R-project.org/package=mdatools.
[30] VENABLES W,RIPLEY B,VENABLES W,et al.Modern Applied Statistics with S[M].4th ed. Berlin:Springer,2002.
[31] CRAIG A,CLOAREC O,HOLMES E,et al.Scaling and normalization effects in NMR spectroscopic metabonomic data sets[J]. Analytical Chemistry,2006,78(7):2 262-2 267.
[32] WINNING H,ROLDAN-MARIN E,DRAGSTED L O,et al.An exploratory NMR nutri-metabonomic investigation reveals dimethyl sulfone as a dietary biomarker for onion intake[J].Analyst,2009,134(11):2 344-2 351.
[33] FILZMOSER P,LIEBMANN B,VARMUZA K.Repeated double cross validation.Journal of Chemometrics[J].Journal of Chemometrics,2009,23(4):160-171.
[34] STONE, M.Cross-validatory choice and assessment of statistical predictions[J].Journal of the Royal Stastical Society.Series B (Methodological),1974,36(2): 111-147.
[35] VU T N,LAUKENS K.Getting your peaks in line:a review of alignment methods for NMR spectral data[J].Metabolites,2013,3(2): 259-276.
[36] ESSLINGER S,RIEDL J,FAUHL-HASSEK C.Potential and limitations of non-targeted fingerprinting for authentication of food in official control[J].Food Research International,2014,60(6):189-204.
[37] GODELMANN R,KOST C,PATZ C D,et al.Quantitation of compounds in wine using1H NMR spectroscopy:description of the method and collaborative study[J].Journal of AOAC International,2016,99(5):1 295-1 304.
[38] PAPOTTI G,BERTELLI D,GRAZIOSI R,et al.Application of one-and two-dimensional NMR spectroscopy for the characterization of protected designation of origin Lambrusco wines of Modena[J].Jounal of Agriculture and Food Chemistry,2013,61(8):1 741-1 746.
[39] 曾祥燕,赵良忠,孙文兵,等.基于PCA和BP神经网络的葡萄酒品质预测模型[J].食品与机械,2014,30(1):40-44.
[41] RIEDL J,ESSLINGER S,FAUHL-HASSEK C.Review of validation and reporting of non-targeted fingerprinting approaches for food authentication[J].Analytica Chimica Acta,2015,885:17-32.

