旱稻Tail-PCR技术主要影响因素的优化和验证
朱 昀,李朝炜,刘嗣凡,刘 颖,魏景芳
(河北科技大学,河北石家庄050018)
热不对称交错PCR(Thermal asymmetric interlaced PCR,Tail-PCR)是一种分离已知DNA序列旁侧未知序列的分子生物学技术。该方法通过不同特异引物与简并引物的组合,进行3轮PCR扩增,采取高温特异性扩增与低温随机扩增相间进行的特殊热循环程序,达到特异产物扩增的同时抑制由随机引物产生的非特异扩增的目的[1]。该方法简单易行,能够在较短的时间内获得目标片段,产物特异性高,结果较为稳定[2]。目前,采用此方法已成功对大豆、棉花、小麦、油菜、红豆杉、天山雪莲、拟南芥等植物已知序列的侧翼序列进行了分析[3-8]。
为了对已获得的大量转基因旱稻纯合体株系进行插入位点旁侧序列分析,本研究以转基因旱稻为材料,参考前人成功经验,结合河北科技大学生物技术实验室的实际情况,对Tail-PCR方法进行探索改进和优化,旨在为转基因旱稻的研究提供依据。
1 材料和方法
1.1 试验材料
转基因T3代旱稻由河北科技大学生物技术实验室通过农杆菌侵染法获得[9-11],通过Southern blot分析确定外源基因为单拷贝[12]。侵染所用质粒为改造的pCAMBIA3301。
1.2 试验方法
1.2.1 DNA 提取 采用改良的 CTAB 法[13]对旱稻基因组进行提取,略加改动。剪取转基因植株叶片0.2 g,放于2 mL装有小钢珠的离心管中,迅速置于液氮中冷冻2 min,在研磨机上进行研磨;叶片研成粉末后,向每个离心管中加入600 μL提取液[CTAB 3%(m/V),NaCl 1.4 mol/L,EDTA(pH 值 8.0)20 mmol/L,Tris-HCl(pH 值 8.0)100 mmol/L,β-巯基乙醇0.2%(m/V)],混匀后放于65℃水浴锅中水浴30 min,期间轻柔摇晃数次;将小钢珠从离心管中取出,12 000 r/min离心15 min;取上清于新的1.5 mL的离心管中,加等体积酚/氯仿/异戊醇,充分混匀;12 000 r/min离心15 min,吸取上清,加等体积氯仿/异戊醇,充分混匀;12 000 r/min离心10 min,吸取上清,加入-20℃提前预冷的异丙醇,使之与上清体积相等,轻柔地混合均匀,-20℃冰箱放置20 min;12 000 r/min离心 5 min,弃上清,75% 的乙醇洗涤1次,无水乙醇洗1次,室温晾干;待管壁无残留液体、DNA呈半透明状时,加50 μL TE溶解,-20℃保存。
1.2.2 引物设计 Tail-PCR的右臂第1条特异引物设计在pCAMBIA3301右边界以内,依次向外设计3条特异引物(R1、R2、R3),每条引物间隔50~150 bp。左臂的第 1条特异引物设计在 pCAMBIA3301左边界以内,依次向外设计3条特异引物(L1、L2、L3),每条引物间隔 50~150 bp。左右臂 6条特异引物退火温度在60~65℃,G+C含量在40%~60%。简并引物采用 Liu等[14]引物,共 5条(LAD1、LAD2、LAD3、LAD4、AC)。各引物序列见表1。

表1 引物序列
1.2.3 Tail-PCR扩增 将特异引物与简并引物配对进行3轮扩增。第1轮左臂侧翼序列分析的引物组合方式分别为 L1+LAD1、L1+LAD2、L1+LAD3、L1+LAD4,右臂侧翼序列分析的引物组合方式为R1+LAD1、R1+LAD2、R1+LAD3、R1+LAD4。第2 轮左臂引物组合方式为L2+AC,右臂引物组合方式为R2+AC。第3轮左臂引物组合方式为L3+AC,右臂引物组合方式为R3+AC。反应的第1轮产物稀释40倍作为第2轮模板使用,第2轮产物稀释10倍作为第3轮PCR反应的模板。反应体系见表2,PCR反应程序见表3。
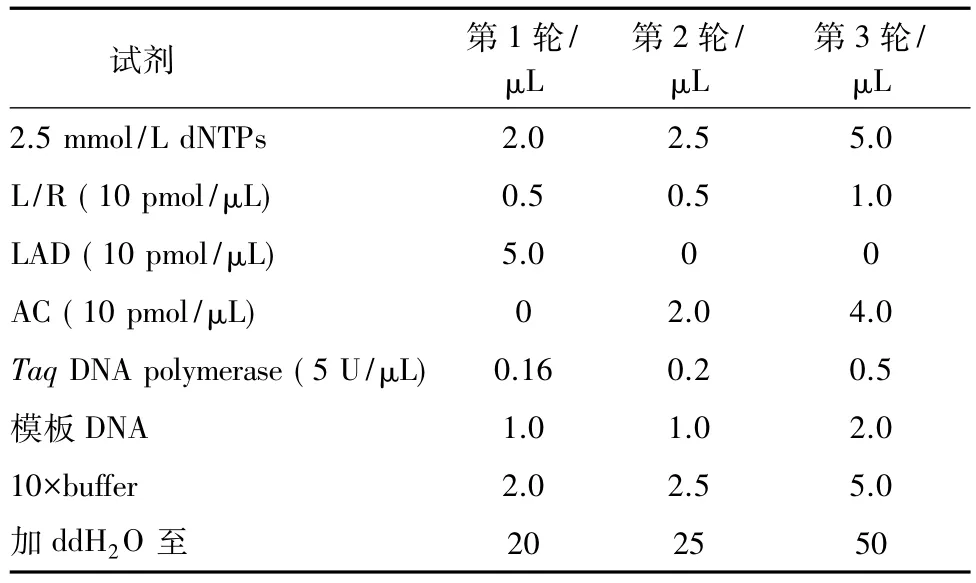
表2 Tail-PCR反应体系
1.2.4 琼脂糖凝胶电泳检测及分析 将Tail-PCR第2轮与第3轮反应产物进行琼脂糖凝胶电泳检测,由于3轮扩增的特异引物为巢式引物,所以第3轮扩增产物略小于第2轮。挑选在第2轮和第3轮扩增中大小关系适当并且大于500 bp的条带回收测序。将测序结果在NCBI上比对,分析插入位点旁侧序列。
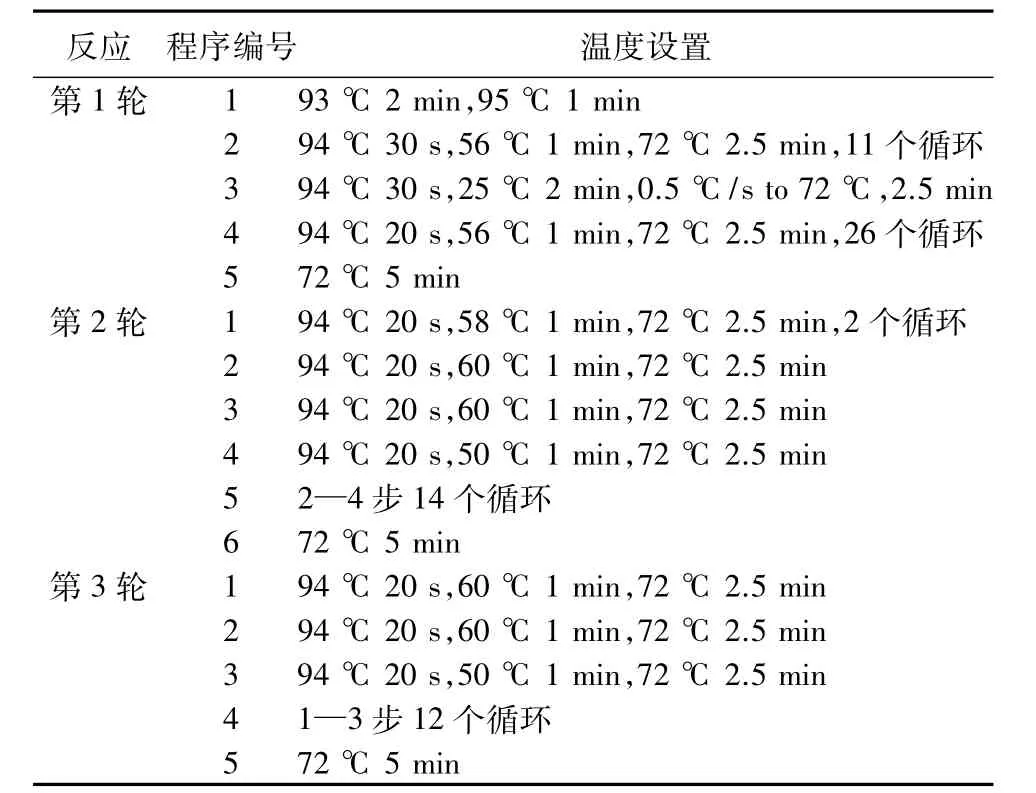
表3 Tail-PCR反应程序
2 结果与分析
2.1 DNA提取方式对扩增结果的影响
DNA质量的好坏直接影响Tail-PCR的结果。袁云香等[15]报道,改良的 SDS法提取的基因组DNA纯度及浓度较高,较之CTAB法、简易法及脲法效果更好。在试验中分别用SDS法、CTAB法和不经酚仿抽提处理的简易法对材料进行了DNA的提取。结果显示,SDS法与CTAB法获得的DNA作为模板,在Tail-PCR中均可进行良好扩增并得到清晰条带(图1-A,图1-B),而简易法获得的DNA由于纯度差、杂质多,在Tail-PCR中扩增大多出现弥散,不能很好地获得目的条带(图1-C)。
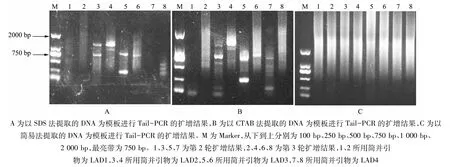
图1 不同DNA提取方法的Tail-PCR电泳结果
2.2 扩增产物稀释程度对后续扩增结果的影响
Tail-PCR需要设计3条特异引物并进行3轮扩增,第1轮扩增产物作为第2轮扩增的模板,第2轮扩增产物作为第3轮扩增的模板,经过3轮扩增得到逐渐富集的目的条带。由于每轮PCR均产生大量不同种产物,直接以这种产物作为模板会影响后续扩增的效果,所以在进行后2轮扩增时,需要对作为模板的前一轮扩增产物进行稀释,一般第2轮扩增会将第1轮扩增产物稀释40倍使用,第3轮扩增会将第2轮产物稀释10倍使用[14]。研究发现,当后2轮扩增产物出现弥散时,适当增大模板的稀释倍数,可以增加条带出现率,更好地得到可供分析的扩增结果(图2)。
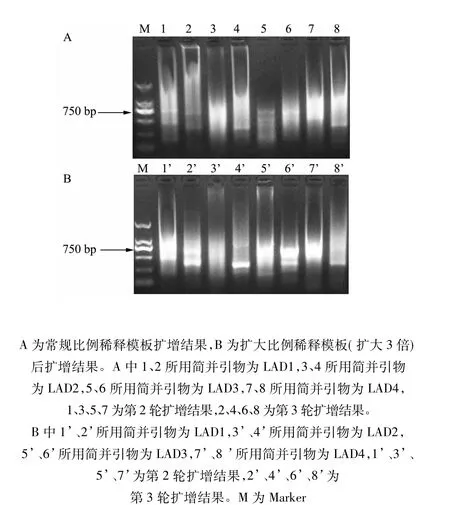
图2 以不同稀释倍数DNA为模板进行扩增比较
2.3 酶对扩增结果的影响
为了获得长片段扩增产物,经典的Tail-PCR的延伸时间一般为3 min,在本试验中,很少得到大于2 000 bp的片段,为了排除酶活力不够对扩增产物的影响,用LA Taq酶代替普通的Taq酶,结果发现,LA Taq酶并未获得更好的扩增结果(图3),在多个株系的试验中没有发现该酶能获得比普通酶更 长的扩增片段,反而增加了试验成本。
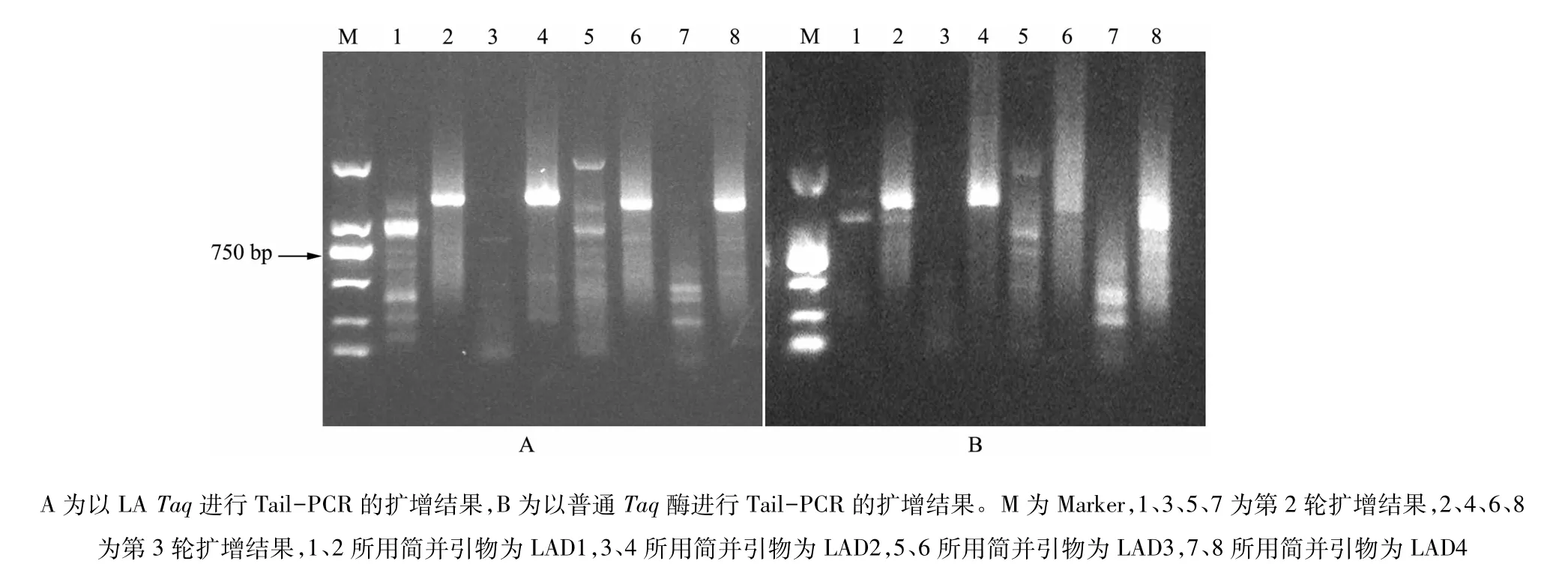
图3 不同的酶对扩增效果的影响
2.4 混合简并引物对扩增结果的影响
Liu等[14]认为,在第1轮扩增中将4种简并引物混合可以在当轮扩增中获得更多的条带,从而达到更好的扩增效果,尽管最终产物中会出现小一些的片段,但是非特异扩增仍能够有效滤去。此外,对于多株系的分析,混合简并引物的方法可以在短时间内对大量株系进行分析鉴定,从而提高效率。本试验中,混合简并引物扩增并没有产生比分开引物扩增更多的条带,而混合引物后产物在同一泳道进行电泳,不利于大小一致的带准确分离。因此,在本试验中,仍采用4种简并引物分别扩增的方法。
2.5 简并引物结合偏好性分析
利用已报道的4种LAD简并引物进行Tail-PCR扩增[14]。研究发现,简并引物与不同生物材料的DNA结合的偏好性不同。4种简并引物中,LAD2更易于与小麦基因组DNA结合。在选取的3个转基因小麦株系中,LAD2引物均可在Tail-PCR的第3轮检测中得到扩增结果,而LAD1在3个材料中均未获得扩增条带(图4-A,图4-B,图4-C)。针对旱稻材料,4种LAD引物与旱稻基因组DNA结合偏好性无明显差异。在随机选取的3个转基因旱稻株系中,LAD1、LAD2、LAD3、LAD4 四种简并引物均可与旱稻基因组结合并在3轮PCR中获得扩增结果(图 4-D,图 4-E,图 4-F)。
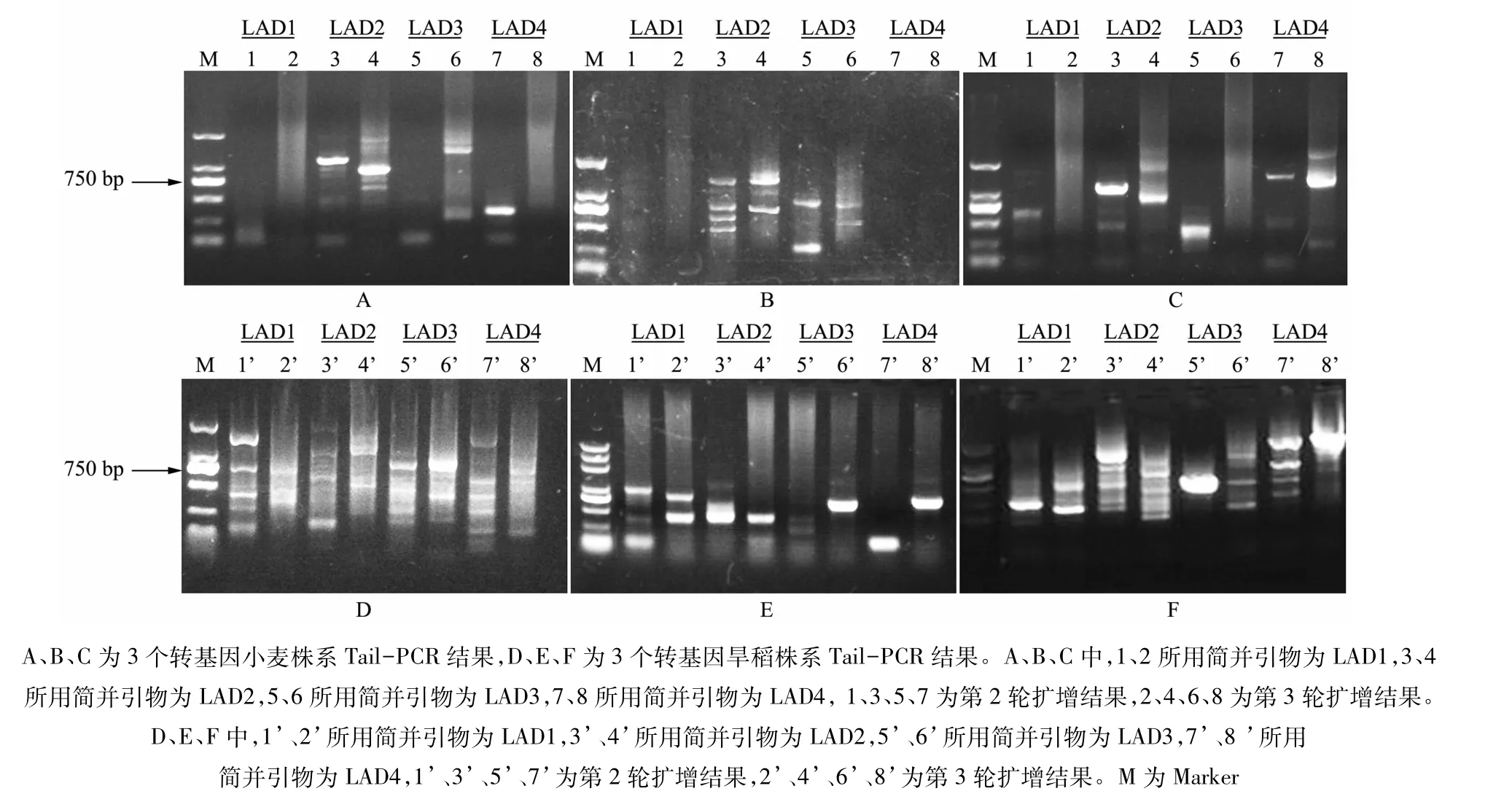
图4 4种简并引物与小麦、旱稻DNA结合偏好性比较
3 结论与讨论
Tail-PCR是一种用来分离与已知序列邻近的未知DNA序列的分子生物学技术,试验结果的好坏与模板质量、引物特异性、物种、已知序列特征、试验条件等诸多因素有关。本研究以转基因旱稻为材料,对Tail-PCR方法进行了分析优化,包括DNA样品质量、扩增产物稀释程度、酶的种类、简并引物组合方式、引物结合偏好性等。优化后的Tail-PCR技术结果稳定,重复性好,可以准确地获得转基因旱稻的插入位点的旁侧序列。
传统 Tail-PCR 延伸时间为 3 min[14],在本试验中,3轮扩增后电泳检测,很少出现大于2 000 bp的条带,换用LA酶后仍然如此。因此,为了缩短整个PCR的流程,将程序中的延伸时间都缩短到2.5 min。最终得到的片段虽然不长,但是已经可以准确地对外源基因的插入位点进行分析。
Tail-PCR中,需要设计3条特异引物,如果3条中的任意一条出现问题,会导致整个试验的失败。在进行特异引物的设计时,同时设计了4条引物,每条引物间距离控制在50~150 bp,退火温度均为60~65℃。进行PCR时,不同的特异引物组合会出现不同的扩增结果(即从4条中选3条扩增,可有多种选择),从而更易于得到不同转基因株系的侧翼序列。
Tail-PCR由于采用了简并引物,因此会出现非特异扩增,而非特异扩增由于是随机出现,所以一般不具备可重复性。试验中可以通过重复扩增来检验条带是否为特异结果。同时,由于过于短小的片段测序后得不到充分的信息,所以,在回收条带的选择上应不小于500 bp。
[1] Liu Y G,Whittier R F.Thermal asymmetric interlaced PCR:Automatable amplification and sequencing of insert end fragments from P1 and YAC clones for chromosome walking[J].Genomics,1995,28(3):457-463.
[2] 郑岑,张立平,唐忠辉,等.Tail-PCR技术及其在植物基因中的克隆[J].基因组学与应用生物学,2009,28(3):544-548.
[3] 仇艳光,田景汉,葛荣朝,等.Tail-PCR的改良及其在分离小麦基因启动子中的应用[J].生物工程学报,2008,24(4):695-699.
[4] 陈笑芸,汪小福,刘仁虎,等.不同Tail-PCR通用引物在转基因大豆、水稻、油菜侧翼序列分离中的应用评价[J].核农学报,2013,27(7):922-928.
[5] 林春燕,郭婧,赵广荣.热不对称交错PCR克隆bapt基因侧翼序列[J].化学工业与工程,2015,32(1):59-63.
[6] 张亚敏,杨晶,王爱英,等.天山雪莲rbcs基因启动子的克隆及序列分析[J].西北农业学报,2014,23(5):146-152.
[7] 梁成真,张锐,孙国清,等.优化Tail-PCR方法克隆棉花抗逆相关转录因子编码基因[J].棉花学报,2010,22(3):195-201.
[8] 田蕾,郭妍君,任丽,等.利用TAIL-PCR技术分析拟南芥At1g52910突变体插入位点的侧翼序列[J].中国野生植物资源,2016,35(4):23-25.
[9] 李朝炜,冯丹,翟会青,等.农杆菌介导的旱稻遗传转化主要影响因素[J].湖北农业科学,2014,53(15):3482-3487.
[10] 李朝炜,刘颖,朱昀,等.旱稻成熟胚愈伤组织诱导的影响因素[J].江苏农业科学,2015,43(2):52-54.
[11] 刘青,李朝炜,朱昀,等.抗逆转基因旱稻转化体系的优化[J].江苏农业科学,2015,43(11):42-45.
[12] 邢文岳,苏乐乐,李朝炜,等.地高辛标记对旱稻进行Southern杂交分析主要影响因素的优化和验证[J].江苏农业科学,2017,45(8):41-43.
[13] 陈昆松,李方,徐昌杰,等.改良CTAB法用于多年生植物组织基因组DNA大量提取[J].遗传,2004,26(4):529-531.
[14] Liu Y G,Chen Y L.High-efficiency thermal asymmetric interlaced PCR for amplification of unknown flanking sequences[J].BioTechniques,2007,43(5):649-656.
[15] 袁云香,孙丙耀.适于Tail-PCR模板的水稻基因组DNA提取方法的优化[J].安徽农业科学,2009,37(9):3964-3966.

