大麦黄花叶病抗性的遗传分析与QTL定位
费新茹,朱 娟,郭 红,吕 超,郭宝健,许如根
(1.扬州大学江苏省作物遗传生理国家重点实验室培育点/粮食作物现代产业技术协同创新中心/教育部植物功能基因组学重点实验室/扬州大学大麦研究所,江苏扬州 225009; 2.盐都区农业科学研究所,江苏盐城 224011)
大麦黄花叶病是东亚和欧洲等冬大麦区的主要病毒病害之一,由大麦黄花叶病毒(Barley yellow mosaic virus,BaYMV)和大麦温和花叶病毒(Barley mild mosaic virus,BaMMV)引起,携带病毒的禾谷多黏菌(Polymyxagraminis)可在土壤中存活10年以上[1]。化学方法很难防治大麦黄花叶病,发掘抗性种质、培育和利用抗病品种是防控该病害最经济有效的途径。关于大麦黄花叶病抗性遗传和基因定位的研究已有不少的报道。已定位分布于大麦7条染色体上的大麦黄花叶病抗性基因有18个, Rym14Hb、 Rym16Hb及 Rym17为显性基因,其余的均为隐性基因。不同来源的抗病基因对不同病毒株系的抗性不同,如中国地方品种木石港3号的两个抗性基因 Rym1和 Rym5,位于4HL上的 Rym1对欧洲现存的所有病毒株系均表现为完全抗性,对日本的毒株BaYMV-I、BaYMV-II、BaMMV-Kal和BaMMV-Nal表现为完全抗性,对日本另一毒株BaYMV-III表现为中抗[2]。位于3HL上的 Rym5对日本毒株BaYMV-I、BaYMV-II、BaYMV-IV、BaYMV-V以及BaMMV-Kal具有抗性,对BaYMV-III以及欧洲毒株BaMMV-SIL和BaMMV-Teik表现感病[3-4]。随着大麦黄花叶病病毒毒株的分化与变异,新类型毒株可能导致抗性品种抗性的下降或丧失。为确保大麦生产上应用品种对大麦黄花叶病的抗性,必须不断发掘新的大麦黄花叶病抗性基因并在育种中加以利用。
本研究以1个大麦黄花叶病抗感DH群体及其亲本为材料,对其大麦黄花叶病抗性基因进行遗传分析与QTL定位,分析扬饲麦1号大麦黄花叶病抗性基因的位置、效应及稳定性,以期发掘新的抗性基因,为大麦黄花叶病抗性基因的精细定位及分子标记辅助选择育种提供有利信息。
1 材料与方法
1.1 供试材料
本研究利用一套包含173个DH系的DH群体及其亲本对大麦黄花叶病的抗性位点进行QTL定位。该DH群体是以扬饲麦1号×Gairdner的F1经花药培养及染色体加倍技术构成的加倍单倍体,其母本扬饲麦1号是由扬州大学选育的抗大麦黄花叶病的六棱饲料大麦品种,父本Gairdner是由澳大利亚选育的感大麦黄花叶病的二棱啤酒大麦品种。该群体由澳大利亚塔斯马尼亚大学周美学教授提供。
1.2 田间种植
2014年和2015年秋季将试验材料播种于扬州大学大麦黄花叶病病圃。每份材料人工点播两行,行长1.2 m,行距0.2 m,每行点播40粒,三次重复。田间管理同大田栽培管理方法。
1.3 抗性鉴定
大麦黄花叶病病级调查标准参照黄培忠等的方法[5],稍作修改。大麦黄花叶病发病始期开始调查,每隔7 d调查1次,连续调查3期。每个株系连续调查10株,根据叶片病斑的有无及叶片黄化、萎缩程度,将大麦黄花叶病抗性分为以下4级:
1级:没有病斑,叶片呈正常绿色(图1A);
2级:叶色基本正常,叶脉周边出现不连续的黄花斑点(图1B);
3级:叶片黄化,黄花斑点连成线,但植株未矮化(图1C);
4级:叶片黄花斑点连成片,植株出现矮化或枯萎死亡现象(图1D)。

图1 大麦黄花叶病抗性级别
病情评价指标采用病情指数(Disease index,DI)来表示,病情指数公式:
DI=∑nx/4N×100
x代表每个调查植株的抗性级别(即1、2、3、4级),n代表各病级的植株数量,N代表所调查的总植株数。
1.4 亲本及DH群体的抗性差异分析
使用 EXCEL 2016对原始数据进行整理,利用 SPSS 16.0统计软件对双亲及DH群体进行抗性差异的统计分析。
1.5 DH群体遗传连锁图谱的构建
筛选亲本间多态性好的SSR标记,利用这些多态性标记对亲本及173个DH系进行基因型鉴定,与扬饲麦1号相同的带型记为0,与Gairdner相同的带型记为2,杂合或缺失记为1,利用JoinMap 4.0[6]软件构建DH群体的遗传连锁图谱。
1.6 QTL定位
利用两年共六个时期的数据,采用Windows QTL IciMapping 4.0[7]软件中的完备区间-加性模型(ICIM-ADD)定位方法,扫描步长为1 cM,以LOD值2.5作为阈值[8],QTL的命名遵循McCouch[9]的原则。
2 结果与分析
2.1 DH群体的遗传连锁图谱
对扬州大学农学院大麦研究所保存的1 008对SSR标记在亲本间进行多态性筛选,获得91对多态性好的SSR标记。利用这些多态性标记对亲本及173个DH系进行基因型鉴定(图2),利用JoinMap 4.0软件构建DH群体的遗传连锁图谱(图3),该图谱覆盖大麦全基因组1 161.99 cM, 91对SSR标记分布于大麦7条染色体上,标记间平均遗传距离为12.77 cM,平均每条染色体上的标记个数为13个。
P1:亲本扬饲麦1号;P2:亲本Gairdner;1~49:部分DH系。
P1:Yangsimai 1; P2:Gairdner; 1-49:Part of the DH lines.
图2引物Bmag0838对部分DH系的扩增条带
Fig.2AmplificationbandsofSSRmarkerBmag0838inDHpopulation
2.2 DH群体及其亲本的大麦黄花叶病病情指数
由表1可知,亲本及DH群体大麦黄花叶病病情指数平均值均以2016年略高,两年均以第二调查期病情指数最高。两年6个调查时期大麦黄花叶病病情指数在亲本间均存在极显著差异。从DH群体变异系数和变幅来看,黄花叶病病情指数在DH群体内存在广泛的变异,DH群体2015年三期黄花叶病病情指数离散程度均大于2016年同期离散程度,2015年第一期及2016年第三期DH群体大麦黄花叶病病情指数存在不同程度的超高亲或超低亲遗传现象。
由表2可知,黄花叶病病情指数在年份间、调查时期间、DH系间及基因型与调查时期互作间、基因型与年份互作间的差异均达到极显著水平,说明黄花叶病病情指数在DH群体各系间存在丰富的遗传变异,同时,年份间的气候条件及调查时期对黄花叶病病情指数有一定影响。
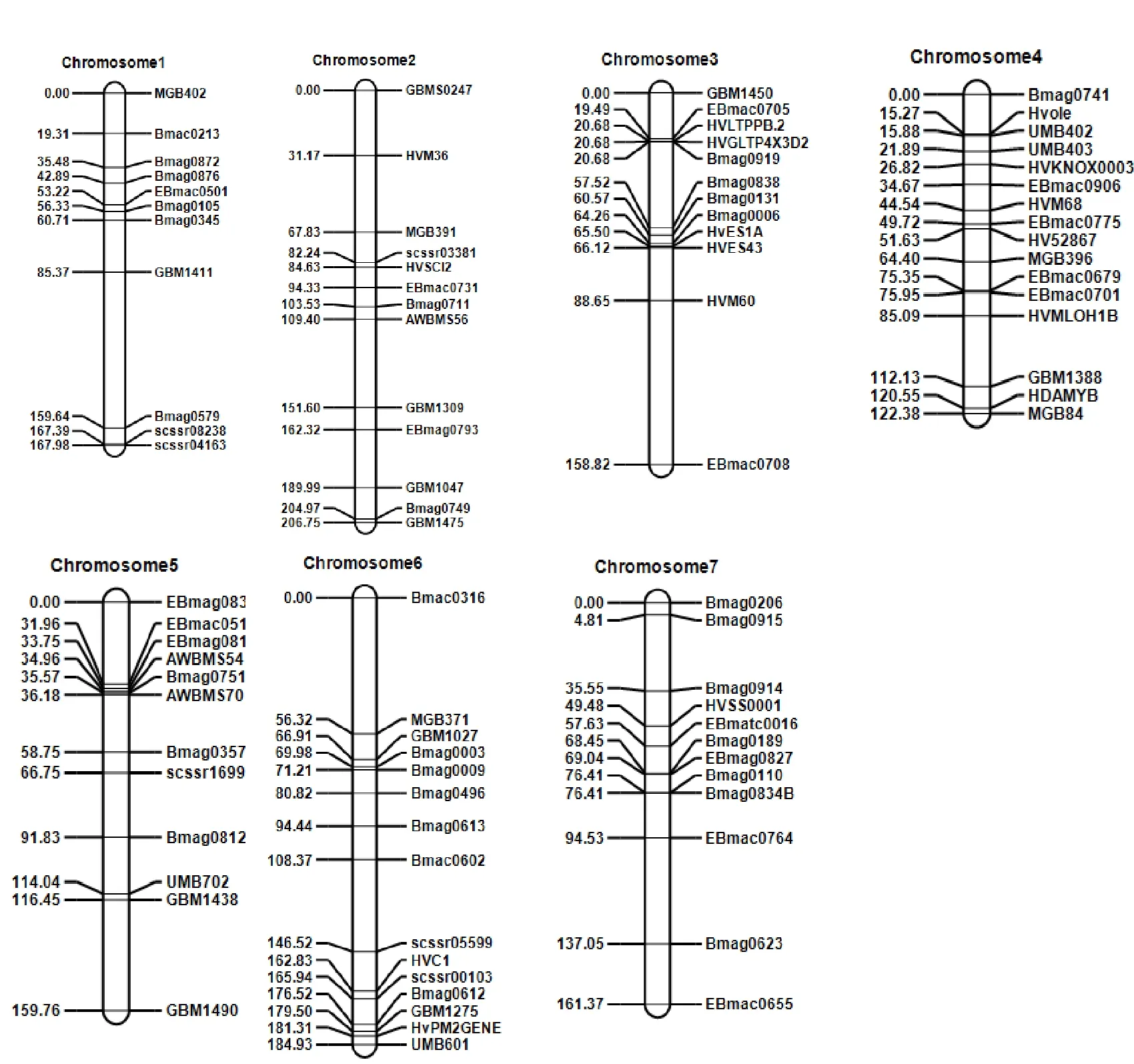
图3 大麦 DH 群体遗传连锁图谱
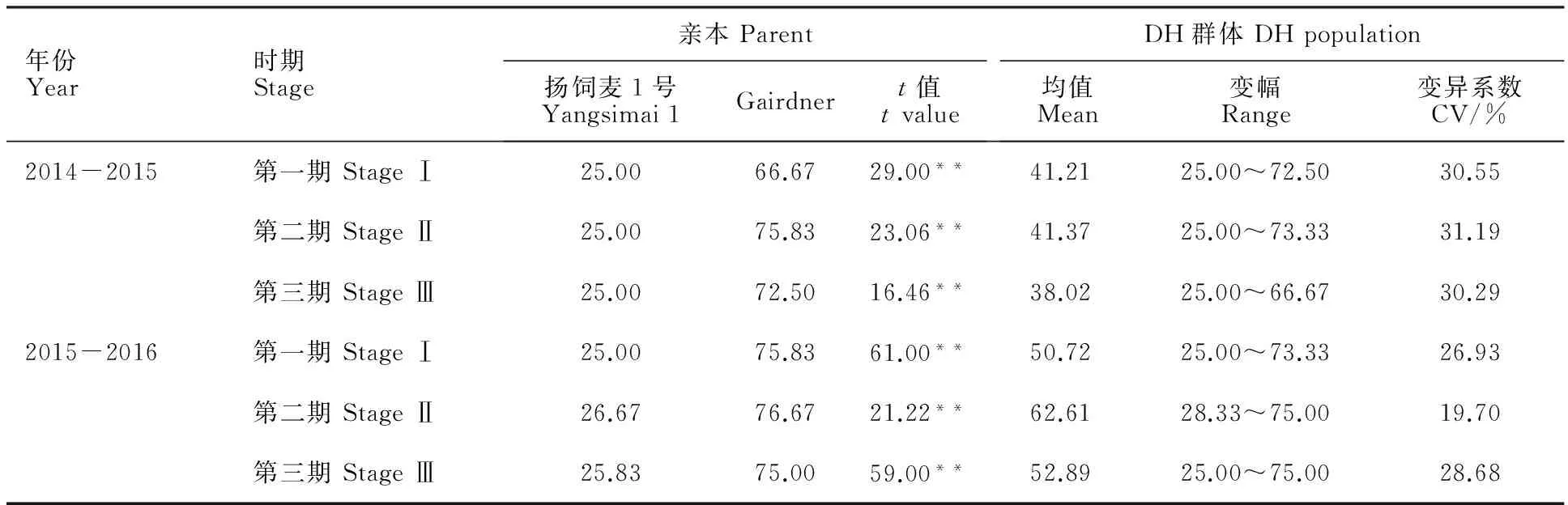
年份Year时期Stage亲本Parent扬饲麦1号Yangsimai1Gairdnert值tvalueDH群体DHpopulation均值Mean变幅Range变异系数CV/%2014-2015第一期StageⅠ25.0066.6729.00**41.2125.00~72.5030.55第二期StageⅡ25.0075.8323.06**41.3725.00~73.3331.19第三期StageⅢ25.0072.5016.46**38.0225.00~66.6730.292015-2016第一期StageⅠ25.0075.8361.00**50.7225.00~73.3326.93第二期StageⅡ26.6776.6721.22**62.6128.33~75.0019.70第三期StageⅢ25.8375.0059.00**52.8925.00~75.0028.68
*表示亲本在0.05水平差异显著;**表示亲本在0.01水平差异显著。下同。
* and ** mean significantly different between parents at 0.05 and 0.01 levels,respectively.The same in table 2.
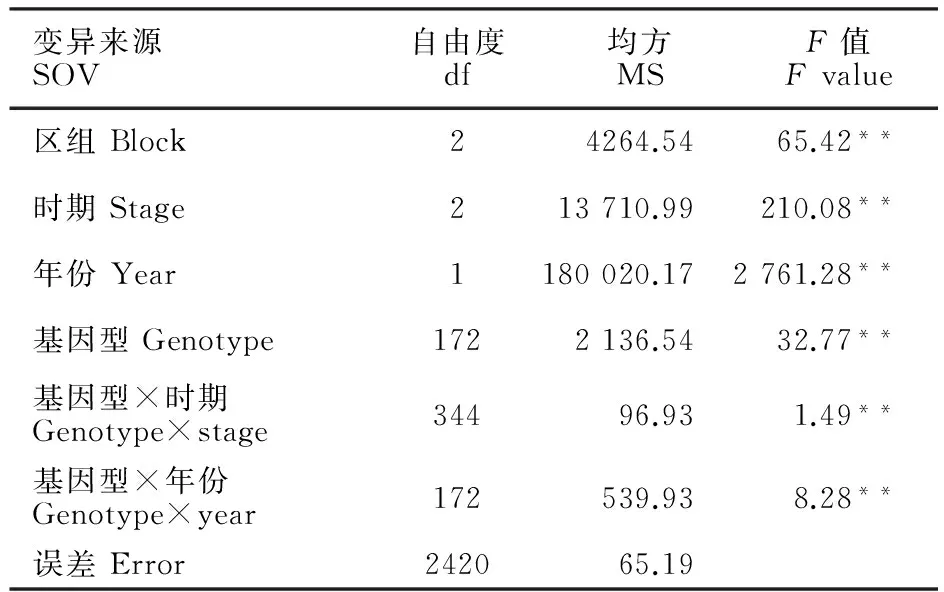
表2 不同调查时期DH群体病情指数的方差分析Table 2 ANOVA of DI in DH population at different survey stages
2.3 大麦黄花叶病抗性的QTL定位结果
采用Windows QTL IciMapping完备复合区间作图法,以大麦黄花叶病病情指数为指标,进行大麦黄花叶病抗性QTL定位,结果(表3)发现,两年6个调查时期共检测到9个与大麦黄花叶病抗性相关的QTLs,分布于1H、2H、4H、5H和7H上。位于1H上的 qRYM-1Ha在2015年第一期及2016年3个时期均被检测到,位于MGB402~Bmac0213标记区间内,可解释3.19%~38.98%的表型变异; qRYM-1Hb仅在2015年第一期和第二期被检测到,位于GBM1411~Bmag0579标记区间内,可解释的表型变异分别为10.39%和5.61%; qRYM-1Hc 除在2015年第一时期未被检测到外,在其余5个时期均被检测到,位于Bmag0345~GBM1411标记区间内,可解释3.17%~21.46%的表型变异。位于2H上的 qRYM-2Ha在两年间六个时期均被检测到,位于EBmag0793~GBM1047标记区间内,可解释的表型变异为5.61%~38.04%; qRYM-2Hb在 2015年第二、三期和2016年第一期被检测到,位于GBM1047~Bmag0749标记区间内,可解释的表型变异为11.40%~18.80%。位于4H上的 qRYM-4Ha在2015年第三期和2016年第一期被检测到,位于MGB396~ HVMLOH1B标记区间内,可解释的表型变异为3.21%~6.53%。位于5H上的 qRYM-5Ha,仅在2015年第一、二期被检测到,位于Bmag0812~GBM1438标记区间内,可解释的表型变异在5%以下。位于7H上的 qRYM-7Ha仅在2015年间第三期被检测到,位于EBmag0827~Bmag0110标记区间内,可解释的表型变异为4.21%; qRYM-7Hb仅在2016年第一、三期被检测到,位于Bmag0206~Bmag0915标记区间内,可解释的表型变异分别为4.18%和3.07%。从抗性QTLs的加性效应来看,除 qRYM-4Ha抗性QTLs来自感病亲本Gairdner外,其余所有抗性QTLs均来自于抗病亲本扬饲麦1号。
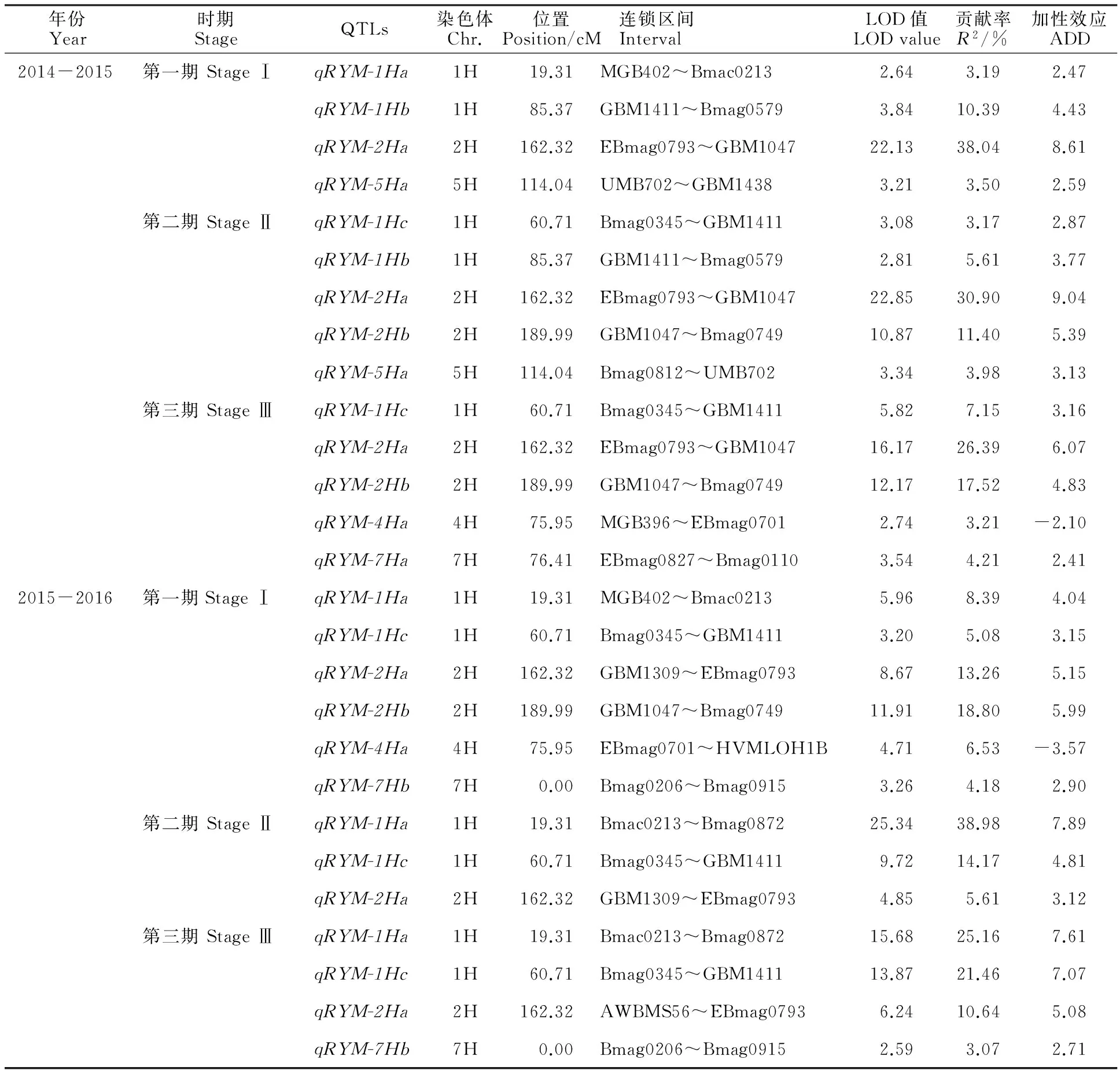
表3 大麦黄花叶病抗性QTLs在染色体上的位置及效应分析Table 3 Chromosome positions and effects analysis of resistance QTLs to barley yellow mosaic disease
3 讨 论
大麦黄花叶病主要通过病土、病根残体和病田流水并以土壤中的禾谷多黏菌为媒介进行传播,其发病严重度与大麦品种的抗病能力、大田土壤中病毒株系的种类与数量、气温以及土壤湿度等因素有关。大麦黄花叶病通过接种鉴定比较困难,成功率低,通常在黄花叶病病圃田中自然诱发。因此,环境对大麦黄花叶病鉴定的影响较大。土壤温度在10~16℃时最适宜病情发展,当温度高于20℃病害症状会有所减轻、逐渐隐症。多雨高湿环境适于禾谷多黏菌休眠孢子的萌发和游动孢子的侵染,有利于病害的发生。本研究中,2015年与2016年间黄花叶病病情指数的差异显著,且2016年各期黄花叶病病情指数均高于2015年黄花叶病病情指数,这可能是因为2015-2016年扬州大麦越冬期气温较高、雨水匀和,有利于禾谷多黏菌的游动与侵染传毒,从而导致返青后大麦黄花叶病发病较重。
大麦黄花叶病一般表现为多基因控制的数量性状,同时受环境及基因与环境互作的影响[10-11]。本研究两年共检测到9个抗大麦黄花叶病QTLs,其中,两年共同检测到的QTLs有5个,说明大麦黄花叶病的确是受多基因控制的性状,受到年份间环境及QTLs与环境互作效应的影响。
目前,已经有部分大麦黄花叶病抗性QTLs被定位,若干抗性基因被克隆且部分已应用于大麦品种改良中[2,3,4,12-26]。与已有关于大麦黄花叶病抗性基因定位的研究进行比较,发现本研究检测到的部分QTLs可能与已报道的基因是等位基因或是同一基因。如 qRYM-1Hc的位置与Yang等[27]利用抗病品种HHOR3365与感病品种Igri构建的DH群体定位到的位于标记k04311-1与k04452之间的 rym7相近,可能是 rym7的等位基因。 qRYM-2Ha和 qRYM-2Hb的位置与Ruge-Wehling等[24]从球茎大麦中定位到大麦黄花叶病抗性基因 Rym16Hb的位置相近,可能是 Rym16Hb的等位基因。位于1H上 qRYM-1Ha和位于4H上 qRYM-4Ha所在区域至今没有相关的QTL位点报道,很可能是新的抗大麦黄花叶病QTL位点,但该结论尚需进一步验证,今后将构建次级作图群体,从而对大麦黄花叶病抗性QTLs进行精细定位。
[1]CHEN J,SWABY A G,ADAMS M J,etal.Barley mild mosaic virus inside its fungal vector,Polymyxagraminis[J].AnnalsofAppliedBiology,2010,118(118):615.
[2]OKADA Y,KASHIWAZAKI S,KANATANI R,etal.Effects of barley yellow mosaic disease resistant gene rym1,on the infection by strains of barley yellow mosaic virus and barley mild mosaic virus [J].TheoreticalandAppliedGenetics,2003,106(2):181.
[3]KANYUKA K,MCGRANN G,ALHUDAIB K,etal.Biological and sequence analysis of a novel European isolate of barley mild mosaic virus that overcomes the barley rym5 resistance gene [J].ArchivesofVirology,2004,149(8):1469.
[4]HABEKUSS A,KUHNE T,KRAMER I,etal.Identification of barley mild mosaic virus isolates in Germany breaking rym5 resistance [J].JournalofPhytopathology,2008,156(1):36.
[5]黄培忠,朱睦元.大麦抗黄花叶病的遗传方式和育种策略[J].浙江大学学报,1989,16(3):310.
HUANG P Z,ZHU M Y.The mode of inheritance and breeding strategies of resistance to barley yellow mosaic virus in barley [J].JournalofZhejiangUniversity,1989,16(3):310.
[6]VANOOIJEN J W.JoinMap 4,software for the calculation of genetic linkage maps in experimental populations [M].Kyazma BV,Wageningen,Netherlands.2006.
[7]MENG L,LI H,ZHANG L,etal.QTL IciMapping:Integrated software for genetic linkage map construction and quantitative trait locus mapping in biparental populations [J].TheCropJournal,2015,3(3):269.
[8]MIYAZAKI C,OSANAI E,SAEKI K,etal.Mapping of quantitative trait loci conferring resistance to barley yellow mosaic virus in a chinese barley landrace mokusekko 3 [J].BreedingScience,2001,51(3):171.
[9]MCCOUCH S R,CHO Y G,YANO M,etal.Report on QTL nomenclature [J].RiceGenetNews,1997,11(14):11.
[10]俞志隆,徐阿炳,朱睦元,等.大麦黄花叶病发病率的双列分析[J].上海农业学报,1988,4(3):29.
YU Z L,XU A B,ZHU M Y,etal.Diallel analysis on incidence percentage of yellow mosaic disease in barley [J].ActaAgricultureShanghai,1988,4(3):29.
[11]俞志隆,徐阿炳,朱睦元,等.大麦黄花叶病严重度的遗传分析[J].遗传学报,1988(6):416.
YU Z L,XU A B,ZHU M Y,etal.Genetic analysis on the severity score of yellow mosaic disease in barley(HordeumvulgareL.) [J].JournalofGeneticsandGenomics,1988(6):416.
[12]UKAI Y,YAMASHITA A.Induced mutation for resistance to barley yellow mosaic virus [J].JapaneseJournalofBreeding,1980,30(2):125.
[13]PELLIO B,STRENG S,BAUER E,etal.High-resolution mapping of the Rym4/Rym5 locus conferring resistance to the barley yellow mosaic virus complex(BaMMV,BaYMV,BaYMV-2) in barley(Hordeumvulgaressp.vulgare L.) [J].TheoreticalandAppliedGenetics,2005,110(2):283.
[14]KUHNE T,SHI N,PROESELER G,etal.The ability of a bymovirus to overcome the rym4-mediated resistance in barley correlates with a codon change in theVPgcoding region on RNA1 [J].JournalofGeneralVirology,2003,84(Pt 10):2853.
[15]TAKAHASHI R,HAYASHI J,INOUYE T,etal.Studies on resistance to yellow mosaic disease in barley.I.Tests for varietal reactions and genetic analysis of resistance to the disease [J].BerOharaInstLandwBiol,OkayamaUniversity,1973,16:1.
[16]FURUSHO M,BABA T,YAMAGUCHI O,etal.Breeding of a new malting barley cultivar Houshun by the bulbosum method [J].BreedingScience,1999,49(4):281.
[17]KANYUKA K,DRUKA A,CALDWELL D G,etal.Evidence that the recessive bymovirus resistance locus rym4 in barley corresponds to the eukaryotic translation initiation factor 4E gene [J].MolecularPlantPathology,2005,6(4):449.
[18]KANYUKA K,WARD E,ADAMS M J.Polymyxagraminisand the cereal viruses it transmits:A research challenge [J].MolecularPlantPathology,2003,4(5):393.
[19]GRANER A,BAUER E,KELLERMANN A,etal.RFLP analysis of resistance to the barley yellow mosaic virus complex [J].Agronomie,1995,15(7-8):475.
[20]GRANER A,STRENG S,KELLERMANN A,etal.Molecular mapping of genes conferring resistance to soil-borne viruses in barley—An approach to promote understanding of host-pathogen interactions [J].JournalofPlantDiseases&Protection,1999,106(4):405.
[21]BAUER E,WEYEN J,SCHIEMANN A,etal.Molecular mapping of novel resistance genes against barley mild mosaic virus(BaMMV) [J].TheoreticalandAppliedGenetics,1997,95(8):1263.
[22]WERNER K,RONICKE S,GOUIS J L,etal.Mapping of a new BaMMV-resistance gene derived from the variety 'Taihoku A' [J].JournalofPlantDiseases&Protection,2003,110(3):304.
[23]RUGE B,LINZ A,PICKERING R,etal.Mapping of Rym14Hb,a gene introgressed fromHordeumbulbosumand conferring resistance to BaMMV and BaYMV in barley [J].TheoreticalandAppliedGenetics,2003,107(6):965.
[24]RUGE-WEHLING B,LINZ A,HABEKUSS A,etal.Mapping of Rym16Hb,the second soil-borne virus-resistance gene introgressed fromHordeumbulbosum[J].TheoreticalandAppliedGenetics,2006,113(5):867.
[25]LE GOUIS J,DEVAUX P,WERNER K,etal. Rym15 from the Japanese cultivar Chikurin Ibaraki 1 is a new barley mild mosaic virus(BaMMV) resistance gene mapped on chromosome 6H [J].TheoreticalandAppliedGenetics,2004,108(8):1521.
[26]KAI H,TAKATA K,TSUKAZAKI M,etal.Molecular mapping of Rym17,a dominant and rym18 a recessive barley yellow mosaic virus(BaYMV) resistance genes derived fromHordeumvulgareL [J].TheoreticalandAppliedGenetics,2012,124(3):577.
[27]YANG P,PEROVIC D,HABEKU A,etal.Gene-based high-density mapping of the gene rym7 conferring resistance to barley mild mosaic virus(BaMMV) [J].MolecularBreeding,2013,32(1):27.

