荞麦ARF基因家族的鉴定及生物信息学分析
李洪有,霍冬敖,蔡 芳,张晓娜,石桃雄,陈其皎,陈庆富
(贵州师范大学 荞麦产业技术研究中心, 贵州 贵阳 550001)
生长素信号在植物生长发育过程中起着至关重要的作用。生长素响应因子(auxin response factor, ARF)作为一类重要的转录因子,其在接受上游传来的生长素信号后,特异性结合在下游生长素响应基因启动子序列中的生长素响应元件(auxin response elements, AuxRE)TGTCTC上,激活或者抑制下游靶基因的表达,进而调控植物的生长发育[1-2]。研究表明,所有ARF蛋白都包含一个保守的N-端B3-型DNA结合结构域和一个具有激活或抑制作用的中心ARF功能域。此外,部分ARF蛋白还含有一个具有二聚作用的C-端Aux /IAA结构域[3]。
目前,拟南芥基因组中共有23个ARF转录因子基因被鉴定,研究表明它们调控了拟南芥多个生长发育过程。其中,AtARF1、AtARF2、AtARF3、AtARF4、AtARF5、AtARF7和AtARF19调控了叶发育,包括对叶长、叶形、叶扩增起始、微管发育、叶脉形成的调控[4-8]。AtARF2、AtARF3、AtARF4、AtARF5、AtARF7、AtARF8、AtARF10、AtARF16和AtARF19调控了根发育,包括对胚根形成、侧根形成和生长、根干细胞分化的调控[9-13]。AtARF1、AtARF2、AtARF6、AtARF8和AtARF17调控了花器官发育,包括对花粉壁形成、花器官衰老和脱落的调控[14-16]。此外,AtARF2调控了种子大小,AtARF8调控了角果的形态[17-18]。目前,ARF基因家族已在包括水稻、玉米、大豆、高粱、谷子、番茄等作物在内的超过30种植物中被鉴定[1-2,19-20]。
荞麦作为一种粮药兼用作物,具有极高的营养价值和医疗保健作用。目前,有关荞麦ARF转录因子家族的研究未见报道。鉴定荞麦ARF转录因子并对它们进行生物信息学分析,对进一步研究它们在荞麦生长发育过程中,特别是在种子发育过程中的作用具有重要意义。最近甜荞简化基因组测序的完成,为从全基因组水平上挖掘荞麦ARF家族基因提供了可能[21]。本研究利用BlastP比对程序鉴定了荞麦ARF基因家族,并对其家族成员的理化性质、亚细胞定位、保守结构域、进化关系、磷酸化位点等进行了预测分析,为进一步克隆荞麦ARF基因和鉴定它们的生物学功能提供了有用信息。
1 材料与方法
1.1 荞麦ARF转录因子的鉴定及序列分析
为了鉴定荞麦基因组中的ARF转录因子,以23个拟南芥ARF的蛋白序列作为查询序列对荞麦基因组数据库BGBD(Buckwheat Gennome Database,http://buckwheat.kazusa.or.jp/)[20]进行BlastP比对搜索,序列有E-value<-10确定为初步候选序列。将初步候选序列手动剔除重复序列后进行Pfam保守结构域(http://pfam. xfam. org/)分析[22],剔除不同时含B3和ARF结构域的蛋白序列,获得荞麦ARF转录因子家族成员序列。利用BioXM 2.6 软件(http://zhanglab.njau.edu.cn/)计算每个荞麦ARF蛋白的等电点和分子量。
1.2 荞麦ARF蛋白的亚细胞定位预测
利用WoLF PSORT(http://wolfpsort.org)在线数据库预测每个荞麦ARF蛋白的亚细胞定位情况[23]。
1.3 荞麦ARF蛋白保守结构域和保守基序(motif)分析
利用HMMER(https://www.ebi.ac.uk/Tools/hmmer/)在线数据库对每个荞麦ARF蛋白的保守结构进行分析[24]。利用MEMM(Multiple Expectation Maximization for Motif Elicitation, http://meme-suite.org/)在线数据库分析每个荞麦ARF蛋白的保守基序,基序长度设置为6~200个氨基酸,基序个数设置为10个[25]。
1.4 荞麦ARF蛋白序列磷酸化位点分析
利用NetPhos 3.1 Server(http://www.cbs.dtu.dk/services/NetPhos/)在线软件对每个荞麦ARF蛋白的氨基酸序列进行潜在磷酸化位点预测分析[26]。
1.5 荞麦ARF蛋白系统进化分析
利用ClustalX 1.83 对21个荞麦和23个拟南芥ARF蛋白序列进行多序列比对,随后利用MEGA 5.0 软件采用NJ(neighbor-joining)法构建系统发育树,重复计算值设为1 000次[27]。
2 结果与分析
2.1 荞麦ARF基因家族的鉴定
通过Blastp比对和Pfam分析,从甜荞基因组中共鉴定了21个非冗余的ARF基因,并将它们命名为FeARF1到FeARF21(表1)。21个FeARF基因均含有内含子,内含子数目为1到14个不等,其中内含子数目最多的是FeARF9和FeARF10(14个),最少的是FeARF15和FeARF18(1个)。21个FeARF蛋白长度为294(FeARF8)到1074(FeARF20)个氨基酸不等,蛋白分子量大小范围为33.46(FeARF8)到118.96 ku(FeARF8)不等。21个FeARF蛋白预测的理论等电点范围为5.38(FeARF10)到9.14(FeARF7),表明不同的FeARF蛋白可能在不同的微环境中发挥它们的功能(表1)。
2.2 荞麦ARF家族基因的亚细胞定位预测
利用WoLF PSORT对21个FeARF蛋白进行亚细胞定位预测,结果除FeARF8预测定位于叶绿体和细胞核,FeARF17预测定位于叶绿体外,其余FeARF蛋白均预测定位于细胞核,这符合它们作为转录因子调控下游基因表达。
2.3 荞麦ARF蛋白保守结构域和保守基序分析
利用HMMER在线数据库对每个荞麦ARF蛋白的保守结构进行分析,结果如图1所示,21个荞麦ARF蛋白均含有B3 DNA 结合结构域和ARF结构域。此外,FeARF1、FeARF2、FeARF10、FeARF12、FeARF14、FeARF19、FeARF20和FeARF21还含有Aux/IAA结构域(图1)。
利用MEME在线数据库对荞麦ARF蛋白进行保守基序分析,结果总共10个保守的蛋白基序被鉴定,每个基序长度在13~55氨基酸之间(表2)。在21个FeARF蛋白中,FeARF1、FeARF2、FeARF5、FeARF6、FeARF9、FeARF10、FeARF12、FeARF19、FeARF20和FeARF21包含了全部10个基序;FeARF3、FeARF4和FeARF13含有8个基序;FeARF14、FeARF15、FeARF16和FeARF17含有7个基序;FeARF7、FeARF11和FeARF18含有6个基序;FeARF8含有5个基序(图2)。利用SMART和Pfam在线数据库对10个基序进行注释发现,基序1、2代表B3 DNA结合结构域,基序5、7代表ARF结构域,基序4、9代表Aux/IAA结构域,而其他4个基序没有生物学功能被注释,其生物学功能有待进一步调查。
表1荞麦FeARF家族基因
Table1PredictedFeARFgenes in buckwheat
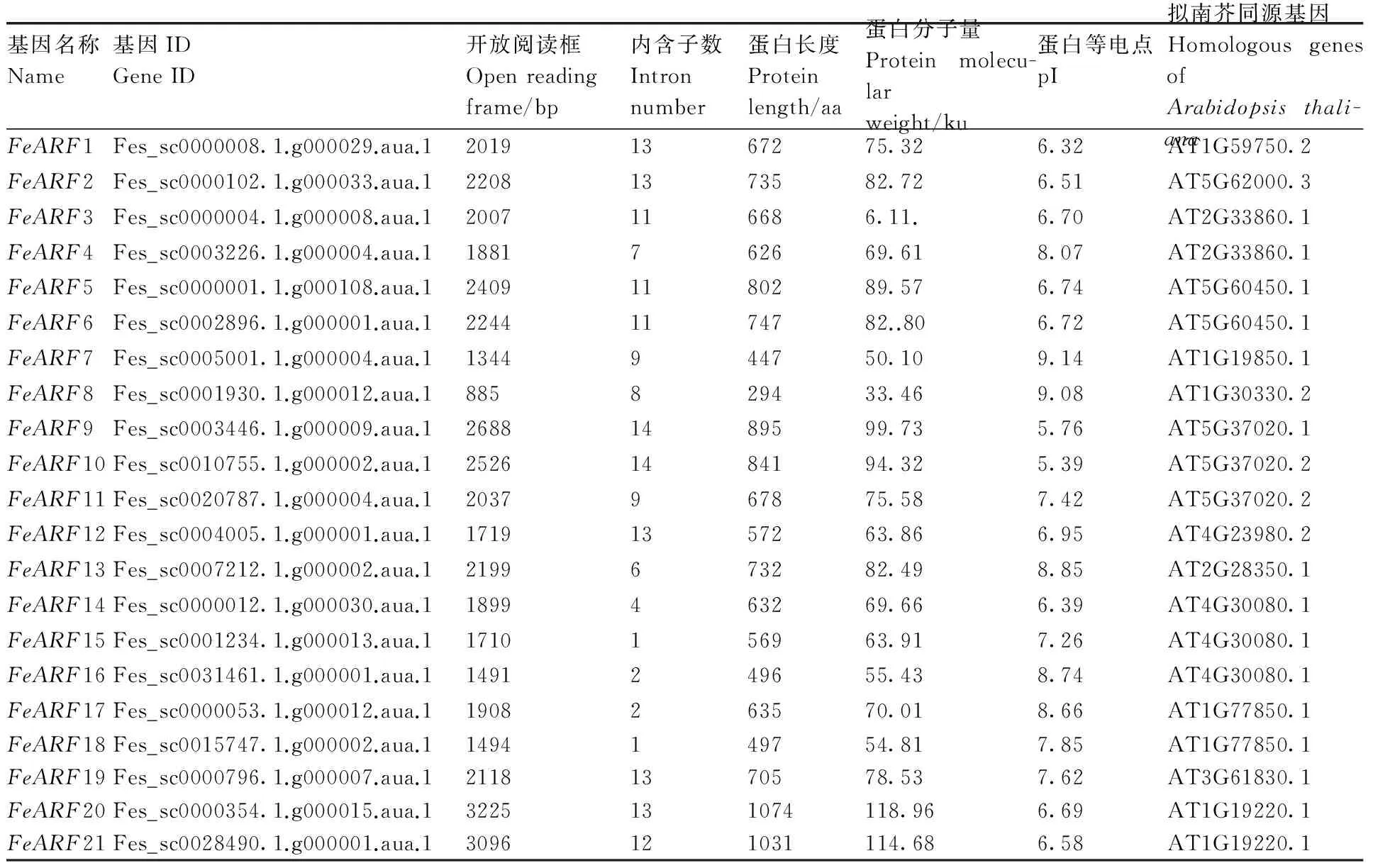
基因名称Name基因IDGeneID开放阅读框Openreadingframe/bp内含子数Intronnumber蛋白长度Proteinlength/aa蛋白分子量Proteinmolecu-larweight/ku蛋白等电点pI拟南芥同源基因HomologousgenesofArabidopsisthali-anaFeARF1Fes_sc0000008.1.g000029.aua.120191367275.326.32AT1G59750.2FeARF2Fes_sc0000102.1.g000033.aua.122081373582.726.51AT5G62000.3FeARF3Fes_sc0000004.1.g000008.aua.12007116686.11.6.70AT2G33860.1FeARF4Fes_sc0003226.1.g000004.aua.11881762669.618.07AT2G33860.1FeARF5Fes_sc0000001.1.g000108.aua.124091180289.576.74AT5G60450.1FeARF6Fes_sc0002896.1.g000001.aua.122441174782..806.72AT5G60450.1FeARF7Fes_sc0005001.1.g000004.aua.11344944750.109.14AT1G19850.1FeARF8Fes_sc0001930.1.g000012.aua.1885829433.469.08AT1G30330.2FeARF9Fes_sc0003446.1.g000009.aua.126881489599.735.76AT5G37020.1FeARF10Fes_sc0010755.1.g000002.aua.125261484194.325.39AT5G37020.2FeARF11Fes_sc0020787.1.g000004.aua.12037967875.587.42AT5G37020.2FeARF12Fes_sc0004005.1.g000001.aua.117191357263.866.95AT4G23980.2FeARF13Fes_sc0007212.1.g000002.aua.12199673282.498.85AT2G28350.1FeARF14Fes_sc0000012.1.g000030.aua.11899463269.666.39AT4G30080.1FeARF15Fes_sc0001234.1.g000013.aua.11710156963.917.26AT4G30080.1FeARF16Fes_sc0031461.1.g000001.aua.11491249655.438.74AT4G30080.1FeARF17Fes_sc0000053.1.g000012.aua.11908263570.018.66AT1G77850.1FeARF18Fes_sc0015747.1.g000002.aua.11494149754.817.85AT1G77850.1FeARF19Fes_sc0000796.1.g000007.aua.121181370578.537.62AT3G61830.1FeARF20Fes_sc0000354.1.g000015.aua.13225131074118.966.69AT1G19220.1FeARF21Fes_sc0028490.1.g000001.aua.13096121031114.686.58AT1G19220.1
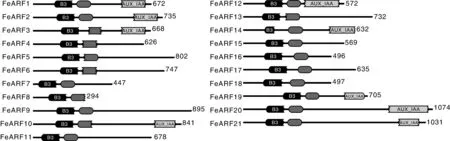
黑色代表B3 DNA结合结构域,深灰色代表ARF结构域,浅灰色代表AUX/IAA结构域。Black boxes represented B3 DNA binding domain, dark grey boxes represented ARF domain, light grey represented AUX/IAA domain.图1 荞麦ARF蛋白的结构域示意图Fig.1 The domain structure of ARF proteins in buckwheat
2.4 荞麦ARF家族成员的磷酸化修饰位点预测
磷酸化在蛋白激活和信号转导中发挥着至关重要的作用。利用NetPhos 2. 0 Server 软件对21个荞麦ARF家族蛋白的氨基酸序列进行潜在磷酸化位点预测。结果如表3所示,21个ARF蛋白的氨基酸序列均含有潜在的丝氨酸Ser、苏氨酸Thr和酪氨酸Tyr磷酸化位点,其中磷酸化位点数目最多是Ser,其次为Thr(FeARF20除外),最少的为Tyr。此外,在21个ARF蛋白中,FeARF20含有最多的磷酸化位点,共64个;FeARF8磷酸化位点数目最少,共20个。
2.5 荞麦ARF家族基因的系统发育分析
为了进一步理解荞麦ARF基因家族的进化关系,构建了拟南芥ARF基因家族(23个)和荞麦ARF基因家族(21个)的系统进化树。如图3所示,44个ARF蛋白可以分为Ⅰ、Ⅱ、Ⅲ三个亚家族,其中,Ⅰ亚家族可以进一步分为Ⅰa和Ⅰb(只含拟南芥ARF蛋白)两个家族,Ⅱ亚家族可以进一步分为Ⅱa和Ⅱb两个家族。系统进化树分析显示,除Ⅰb家族外,大多数荞麦ARF 蛋白与拟南芥ARF蛋白有“一一对应”关系。此外,通过系统进化树进一步发现荞麦基因组中存在4对旁系同源基因(FeARF3/FeARF4、FeARF5/FeARF6、FeARF14/FeARF16、FeARF17/FeARF18)。
表2荞麦FeARF蛋白保守基序
Table2Conserved motif of FeARF proteins

基序Motif氨基酸个数Numberofaminoacid氨基酸序列SequenceofaminoacidMotif155PAQELVAKDLHGNEWKFRHIYRGQPRRHLLTTGWSVFVSSKKLVAGDSVLFJRGEMotif241RQPPHSFCKTLTASDTSTHGGFSVPRRAAEKIFPPLDYSQQMotif340SSLNSELWHACAGPLVSJPPKGSRVVYFPQGHLEQASASTMotif441WQJVYTDDENDMMLVGDDPWZEFCNMVSKIKIYSSZEVQKMMotif521MHTRVSVGMRFKMRFETEESSMotif641ZLRLGIRRANRQQTVMPSSVJSSDSMHJGVLAAAAHAASTNMotif729PKILCRVVBVTLLADPETDEVYAQITLLPMotif821DPVRWPNSKWRSLQVRWDEPEMotif933AVGRAIDLSKLSSYDELLSELERLFGJEGZLEDMotif1013LZRQKRVSPWEIE
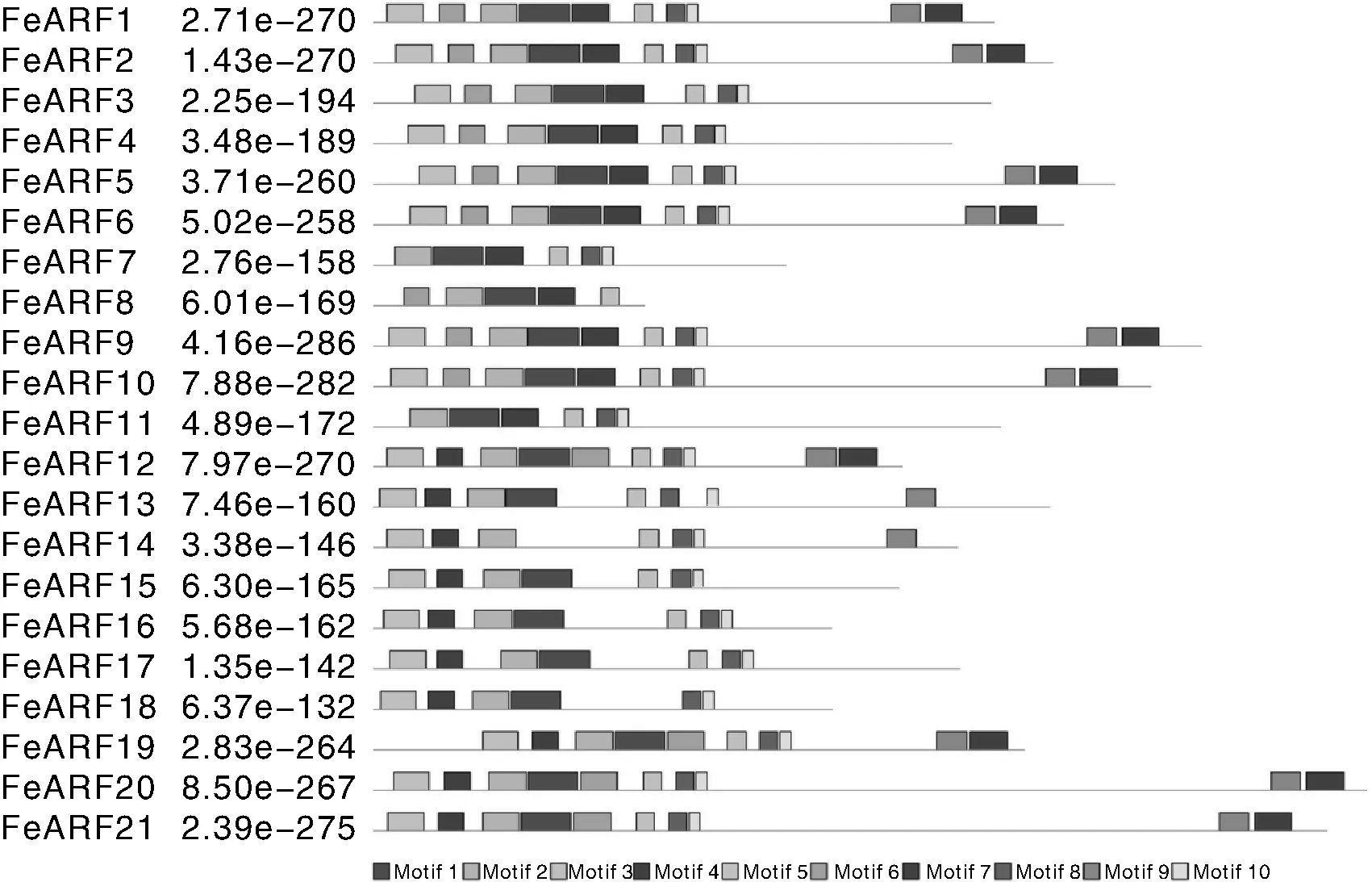
图2 荞麦ARF蛋白保守性基序分析Fig.2 Conserved motif analysis of buckwheat ARF proteins
表3荞麦ARF蛋白的磷酸化位点预测
Table3The predicted phosphorylation sites of ARF proteins from buckwheat
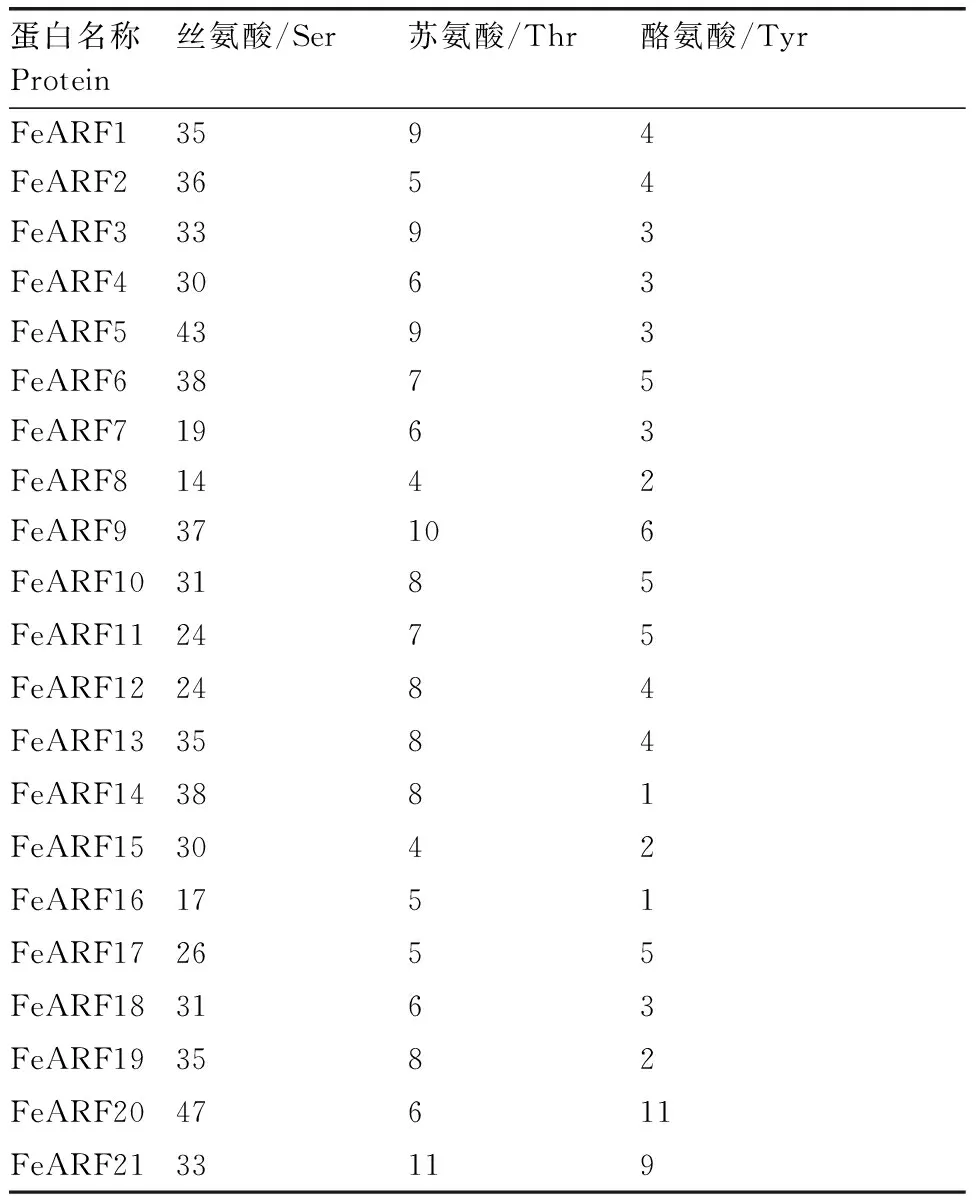
蛋白名称Protein丝氨酸/Ser苏氨酸/Thr酪氨酸/TyrFeARF13594FeARF23654FeARF33393FeARF43063FeARF54393FeARF63875FeARF71963FeARF81442FeARF937106FeARF103185FeARF112475FeARF122484FeARF133584FeARF143881FeARF153042FeARF161751FeARF172655FeARF183163FeARF193582FeARF2047611FeARF2133119
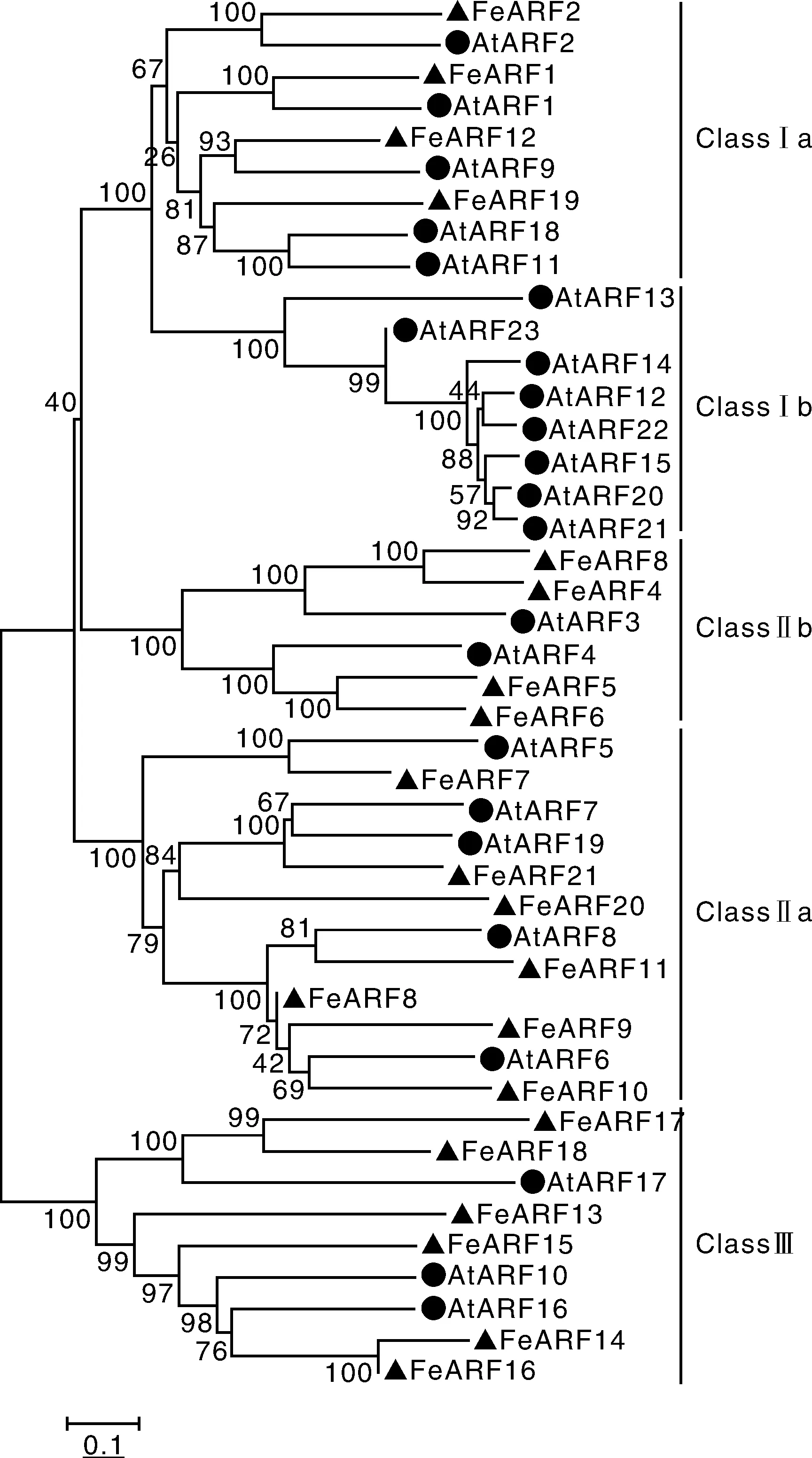
图3 荞麦和拟南芥ARF蛋白的进化关系Fig.3 Phylogenetic relationships of ARF proteins from buckwheat and Arabidopsis
3 讨论
ARF家族基因是生长素信号传导途径中的重要转录因子,它们接受上游传来的生长素信号,调控下游一系列生长素响应基因的表达,在植物生长发育过程中起着至关重要的作用。目前,ARF基因家族已在拟南芥、水稻、玉米、大豆、高粱、谷子、番茄等多种植物中被鉴定[1-2,19-20],但在荞麦中未见报道。本研究通过生物信息学方法从甜荞基因组数据库中共鉴定到21个荞麦ARF基因,这与拟南芥(23个)和水稻(25个)ARF基因数目相差不大[28],表明ARF基因在进化上是保守的。
通常,ARF蛋白具有3个保守的结构域,即N-端起DNA捆绑作用的B3结构域,中间具有激活/抑制作用的ARF结构域和C-端具有二聚作用的Aux /IAA结构域[3]。21个荞麦ARF蛋白都含有B3和ARF结构域,此外,FeARF1、FeARF2、FeARF10、FeARF12、FeARF14、FeARF19、FeARF20和FeARF21还含有Aux /IAA 结构域。这些结果表明,ARF蛋白的蛋白结构在不同植物中是高度保守的。
系统进化树分析表明,21个荞麦和23个拟南芥ARF蛋白可以分为Ⅰ、Ⅱ、Ⅲ三个亚家族,其中,Ⅰ亚家族可以进一步分为Ⅰa和Ⅰb两个家族,Ⅱ亚家族可以进一步分为Ⅱa和Ⅱb两个家族。值得注意的是,Ⅰb家族只包含拟南芥ARF蛋白;同样,在多种植物的进化分析中也发现Ⅰb家族只存在于拟南芥中[1-2,19],表明Ⅰb家族是拟南芥与其他植物发生分离后在进化过程中新获得的,可能具有其独特的功能。除Ⅰb家族外,大多数荞麦ARF蛋白与拟南芥ARF蛋白具有“一一对应关系”,表明这些蛋白在荞麦和拟南芥发生分离前已存在于二者的共同祖先中,同时也表明这些蛋白在进化中是高度保守的,可能具有拟南芥中相似的功能。此外,通过进化树分析发现了4个荞麦基因复制对(FeARF3/FeARF4、FeARF5/FeARF6、FeARF14/FeARF16、FeARF17/FeARF18),表明在荞麦和拟南芥发生分离后,基因复制事件已独立存在于荞麦基因组中。
通过对荞麦基因组中ARF基因家族的鉴定、进化等一系列生物信息学分析,为将来进一步克隆该家族基因和深入研究该基因家族的功能提供参考,为揭示该基因家族调控植物生长发育的机制提供理论依据。
[1] CHANDLER J W. Auxin response factors[J].PlantCellandEnvironment, 2016, 39(5): 1014-1028.
[2] 李慧峰, 冉昆, 何平, 等. 苹果生长素响应因子(ARF)基因家族全基因组鉴定及表达分析[J]. 植物生理学报, 2015, 51(7): 1045-1054.
LI H F, RAN K, HE P, et al. Genome-wide identification and expression analysis of auxin response factor(ARF) gene family in apple[J].PlantPhysiologyJournal, 2015, 51 (7): 1045-1054. (in Chinese with English abstract)
[3] LI S B, XIE Z Z, HU C G, et al. A review of auxin response factors (ARFs) in plants[J].FrontiersinPlantScience, 2016, 7: 47.
[4] ELLIS C M, NAGPAL P, YOUNG J C, et al. Auxin response factor1 and auxin response factor2 regulate senescence and floral organ abscission inArabidopsisthaliana[J].Development, 2005, 132 (20): 4563-4574.
[5] FAHLGREN N, MONTGOMERY T A, HOWELL M D, et al. Regulation of auxin response factor3 by TAS3 ta-siRNA affects developmental timing and patterning inArabidopsis[J].CurrentBiology, 2005, 16 (9): 939-944.
[6] WILMOTH J C, WANG S, TIWARI S B, et al. NPH4/ARF7 and ARF19 promote leaf expansion and auxin-induced lateral root formation[J].ThePlantJournal, 2005, 43 (1): 118-130.
[7] ZHOU G K, KUBO M, ZHONG R, et al. Overexpression of miR165 affects apical meristem formation, organ polarity establishment and vascular development inArabidopsis[J].PlantandCellPhysiology, 2007, 48 (3): 391-404.
[8] GARRETT J J, MEENTS M J, BLACKSHAW M T, et al. A novel, semi-dominant allele of MONOPTEROS provides insight into leaf initiationand vein pattern formation[J].Planta, 2012, 236 (1): 297-312.
[9] MARIN E, JOUANNET V, HERZ A, et al. miR390, Arabidopsis TAS3 tasiRNAs, and their auxin response factor targets define an autoregulatory network quantitatively regulating lateral root growth[J].ThePlantCell, 2010, 22 (4): 1104-1117.
[10] SCHLERETH A, MÖLLER B, LIU W, et al. Monopteros controls embryonic root initiation by regulating a mobile transcription factor[J].Nature, 2010, 464 (7290): 913-916.
[11] OKUSHIMA Y, FUKAKI H, ONODA M, et al. ARF7 and ARF19 regulate lateral root formation via direct activation of LBD/ASL genes in Arabidopsis[J].ThePlantCell, 2007, 19 (1): 118-130.
[12] TIAN C E, MUTO H, HIGUCHI K, et al. Disruption and overexpression of auxin response factor 8 gene of Arabidopsis affect hypocotyl elongation and root growth habit, indicating its possible involvement in auxin homeostasis in light condition[J].ThePlantJournal, 2004, 40 (3): 333-343.
[13] DING Z, FRIML J. Auxin regulates distal stem cell differentiation in Arabidopsis roots[J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 2010, 107 (26): 12046-12051.
[14] TABATA R, IKEZAKI M, FUJIBE T, et al. Arabidopsis auxin response factor6 and 8 regulate jasmonic acid biosynthesis and floral organ development via repression of class 1 KNOX genes[J].PlantandCellPhysiology, 2010, 51 (1): 164-175.
[15] WU M F, TIAN Q, REED J W. Arabidopsis microRNA167 controls patterns of ARF6 and ARF8 expression, and regulates both female and male reproduction[J].Development, 2006, 133 (21): 4211-4218.
[16] YANG J, TIAN L, SUN M X, et al. Auxin response factor17 is essential for pollen wall pattern formation in Arabidopsis[J].PlantPhysiology, 2013, 162 (2): 720-731.
[17] SCHRUFF M C, SPIELMAN M, TIWARI S, et al. The auxin response factor 2 gene of Arabidopsis links auxin signaling, cell division, and the size of seeds and other organs[J].Development, 2006, 133 (2): 251-261.
[18] GOETZ M, VIVIAN-SMITH A, JOHNSON S D, et al. Auxin response factor8 is a negative regulator of fruit initiation in Arabidopsis[J].ThePlantCell, 2006, 18 (8): 1873-1886.
[19] 赵艳, 翁巧云, 马海莲, 等. 谷子ARF基因家族的鉴定与生物信息学分析[J]. 植物遗传资源学报, 2016, 17 (3): 547-554.
ZHAO Y, WENG Q Y, MA H L, et al. Genome-wide identification and bioinformatics analysis of ARF gene family inFoxtailmilletSetariaitalic[J].JournalofPlantGeneticResources, 2016, 17 (3): 547-554. (in Chinese with English abstract)
[20] 江海洋, 魏巍, 刘艳,等. 高粱生长素反应因子(ARF)基因的全基因组分析与进化研究[J].安徽农业大学学报, 2010, 37(3):395-400.
JIANG H Y, WEI W, LIU Y, et al. Genome-wide analysis and evolution of the auxin response factor(ARF) gene family inSorghumbicolor[J].JournalofAnhuiAgriculturalUniversity, 2010, 37(3):395-400. (in Chinese with Englishabstract)
[21] YASUI Y, HIRAKAWA H, UENO M, et al. Assembly of the draft genome of buckwheat and its applications in identifying agronomically useful genes[J].DNAResearch, 2016, 23 (3): 215-224.
[22] FINN R D, MISTRY J, SCHUSTER-BÖCKLER B, et al. Pfam: clans, web tools and services[J].NucleicAcidsResearch, 2006, 34(S1): D247-D251.
[23] HORTON P, PARK KJ, OBAYASHI T, et.al. WoLF PSORT: protein localization predictor[J].NucleicAcidsResearch, 2007, 35: W585-W587.
[24] FINN R D, CLEMENTS J, EDDY S R. HMMER web server: interactive sequence similarity searching[J].NucleicAcidsResearch, 2011, 39 (S2): W29-W37.
[25] BAILEY T L, ELKAN C. The value of prior knowledge in discovering motifs with MEME [C]//Proceedings of the International Conference on Intelligent Systems for Molecular Biology, 1995, 3: 21-29.
[26] BLOM N, GAMMELTOFT S, BRUNAK S. Sequence and structure-based prediction of eukaryotic protein phosphorylation sites[J].JournalofMolecularBiology, 1999, 294(5): 1351-1362
[27] KUMAR S, NEI M, DUDLEY J, et al. MEGA: A biologist-centric software for evolutionary analysis of DNA and protein sequences[J].BriefBioinformatics, 2008, 9 (4): 299-306.
[28] WANG D, PEI K, FU Y, et al. Genome-wide analysis of the auxin response factors (ARF) gene family in rice (Oryzasativa)[J].Gene, 2007, 394 (1): 13-24.

