基于深度学习的皮肤影像分类
周航宁,谢凤英,姜志国,刘 洁,晋红中,孟如松,崔 勇
1北京航空航天大学宇航学院图像处理中心,北京 100191 2中国医学科学院 北京协和医学院 北京协和医院皮肤科, 北京 100730 3解放军空军总医院皮肤科,北京 100142 4中日友好医院皮肤病与性病科,北京 100029
皮肤影像包括皮肤镜、共聚焦激光扫描显微镜、皮肤高频超声、皮肤光学相干层析成像、皮肤太赫兹成像、皮肤光声成像、多光子激光断层成像技术及皮肤组织病理图像等。基于皮肤影像对各类皮肤疾病进行计算机辅助诊断具有客观、可重复的优点,可为医生提供量化的诊断特征,并对皮损类别进行预测,从而帮助医生对病情进行分析和判断[1]。
计算机辅助诊断系统大致可分为5个环节,包括皮肤影像采集、图像预处理[2]、图像分割[3]、特征提取[4]和分类识别[5]。由于皮肤疾病种类繁多,对皮损进行自动分类一直颇具挑战。传统方法往往需对原始图像进行繁杂的预处理,需要有经验的工程师手动设计特征提取器,选择合适的分类器进行分类。这类方法泛化能力不强,很难实现复杂的多分类任务。
随着大数据时代的到来和计算机硬件的巨大进步,深度学习技术尤其是卷积神经网络(convolutional neural network, CNN),在图像分类、检测等很多任务中相对传统模式识别方法取得了令人瞩目的突破[6- 9]。CNN本质上是一个多层、高度非线性的神经网络,可充分挖掘高维数据中存在的内在结构,自发地从原始数据中习得有用的特征表达。就图像分类任务而言,相对于传统方法多步骤、过程复杂的特点,CNN往往是“端到端”模型,即输入皮损图像,便能输出皮损类别。2017年1月,斯坦福大学人工智能实验室与斯坦福医学院合作(以下简称斯坦福团队)采用深度学习方法对皮肤镜和临床皮损图像进行自动分类,并在《自然》杂志上发表了相关研究成果[10],受到人们广泛关注。该团队的工作代表了皮肤影像自动分析领域的最新研究进展,本文将对该项工作进行介绍。
1 数据集
大量样本对于深度学习至关重要。没有充足的训练数据,CNN很难从训练样本中学习到合适的特征,网络也很容易陷入“过拟合”的困境。为了让CNN拥有强大的泛化能力,必须提供足够多的训练样本。斯坦福团队构建了一个庞大的皮肤影像数据库,影像数据达到了惊人的129 450幅,比以往同类任务所用的数据库大了数十倍甚至上百倍[11]。该数据库不仅数据量大,标签信息还十分精细,包含多达2032种不同疾病。庞大的数据库和细粒度的标签信息给CNN的训练提供了强大助推力。该团队数据来源包括ISIC皮肤病数据库、爱丁堡皮肤病数据库、斯坦福医院数据库和一些公开的皮肤病数据库。其中前三者的数据均已经过活检验证且被精细分类。在数据准备阶段,该团队提出了一种简称为PA(disease-partitioning algorithm)的自动递归算法,将2032种皮肤病依照图1所示的皮肤病分类树形结构图聚合成分布更均匀的757类(训练类),使每一类所包含的数据量均不超过1000幅,同时也不至于过小,在保证细粒度分类的同时让每一个类别均有充足的训练数据,更加有利于深度学习的训练。在图1的树形结构中,大分支分别对应良性皮损、恶性皮损和非肿瘤性皮损3个一级分类。3个一级分类中的每一类对应2~4个子分支,形成9个二级分类。每个二级分类进一步细分,逐步形成了最底层的叶子节点,而这些叶子节点则对应了具体的皮肤病名。
以往研究往往仅针对皮肤镜图像或组织学图像进行分类,这两类图像均是高度标准化的图像。而该团队构建的数据库包括 3374幅皮肤镜图像和126 076幅临床图像,临床图像在相对尺寸、角度、光照等很多方面均不统一[12],给分类造成了很大困难。传统模式识别方法很难处理临床图像的分类问题,而该团队训练的CNN使皮损分类达到了很高的精度。
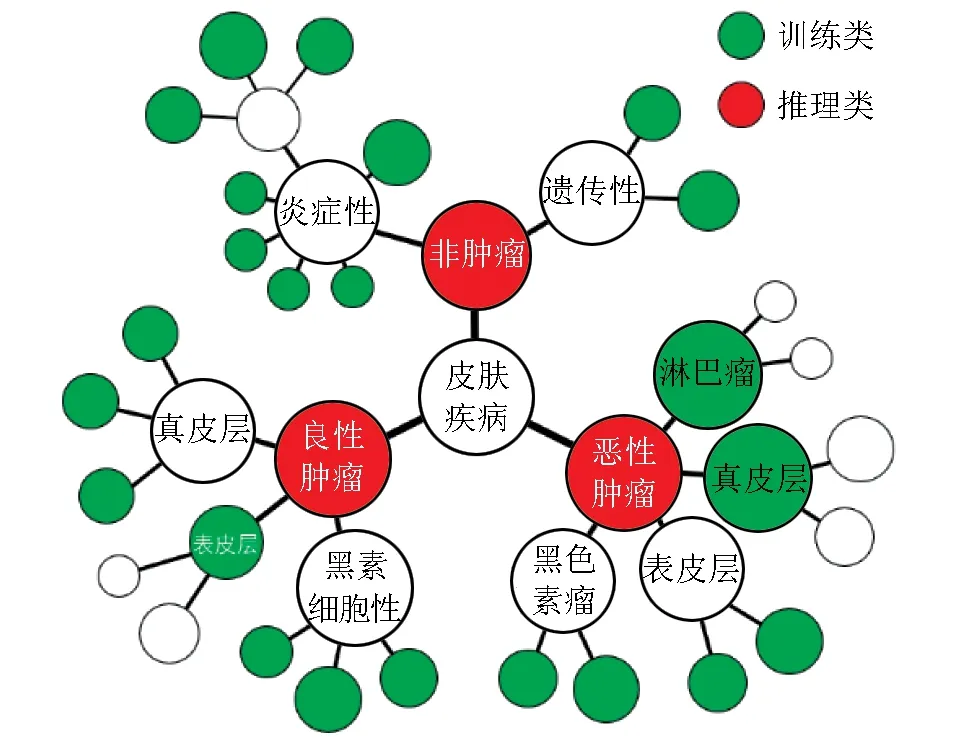
图1皮肤疾病分类树形结构示意图[10]
2 分类框架
除了数据集的构建,利用深度学习进行图像分类的另一个重要任务是分类网络的搭建。其对于分类精度起到了决定性作用。斯坦福团队选择了GoogLeNet Inception-v3[8]作为其分类网络并将迁移学习技术应用到自己的数据库上。网络结构如图2所示。
GoogLeNet Inception-v3是目前分类性能最强的CNN模型之一,通过ImageNet数据库中近130万幅的图像数据训练而成,具备极强的数据抽象能力、特征提取能力和图像分类能力。迁移学习技术可充分利用已训练好的网络参数,这比仅仅用自己的数据库从头开始训练精度更高。迁移学习的目标是将从一个应用场景中学到的知识,用来帮助实现新的应用场景中的学习任务。斯坦福团队便利用了这一技术,用约13万幅皮损图像对已经训练好的GoogLeNet Inception-v3网络进行参数微调,得到了最终用于皮损分类的CNN模型。
该团队对皮损图像进行了3分类和9分类实验。在训练网络时,将数据集划分为训练集、验证集和测试集,其中训练集和验证集包括127 463幅图像,测试集包括1942幅图像,且所有测试集图像均经过活检验证,这使该团队的实验结果具有很强的说服力。在训练CNN时,往往训练数据的标签类别数和分类的目标类别数一致,故要将皮损图像分成3类或9类,往往选择利用3类或9类图像标签对CNN进行训练。对于如何才能充分利用成百上千类的细粒度标签信息这一问题,该团队将分类树形结构图中每一个包含多个训练类的类别称为一个推理类,每一个推理类均能在分类树形结构图中找到对应节点(图1)。对于一幅输入图像,该网络可输出此图像属于757个训练类别中每一类的概率,要得到该图像属于某一推理类的概率,只需将其属于该推理类子类的概率求和即可,若子类也是推理类,则迭代该算法。该团队即是通过这种方式最终得到了3分类和9分类的结果,充分发挥了细粒度标签的优势,该算法被称作CNN-PA。
3 分类性能
斯坦福团队把CNN-PA的分类结果与使用3类或9类粗粒度标签进行训练得到的CNN及两名专业医生的分类结果进行了对比。结果表明,CNN-PA在3分类和9分类任务上的精度分别可达72.1%和55.4%,而CNN则是69.4%和48.9%,两名专业医生则分别是65.6%、53.3%和66.0%、55.0%,因此,CNN-PA在三者中拥有最好的分类能力。
该网络在许多分类任务上的准确率与专业皮肤科医生相当甚至更好。CNN-PA证明了该网络粗粒度分类的能力,而在细粒度分类任务中,该网络依然具有强大的分类能力。比如在区分角质形成细胞癌与脂溢性角化病、临床图像区分黑性色素瘤与良性色素痣、皮肤镜图像区分恶性黑色素瘤与良性色素痣这3个任务中,将网络分类性能与21名有专业资质的皮肤科医生进行对比,统计结果显示,对于每一个任务,超过91%的医生的分类敏感性和特异性在CNN分类网络之下,说明该网络对这3个任务的分类准确率也已超过大多数专业皮肤科医生。
以上两组试验表明,无论是3分类和9分类这样的粗粒度分类任务,还是判断具体皮肤病种类这样的细粒度分类任务,该团队设计的算法均能达到甚至超过专业医生的人眼判别水平。
4 未来应用空间
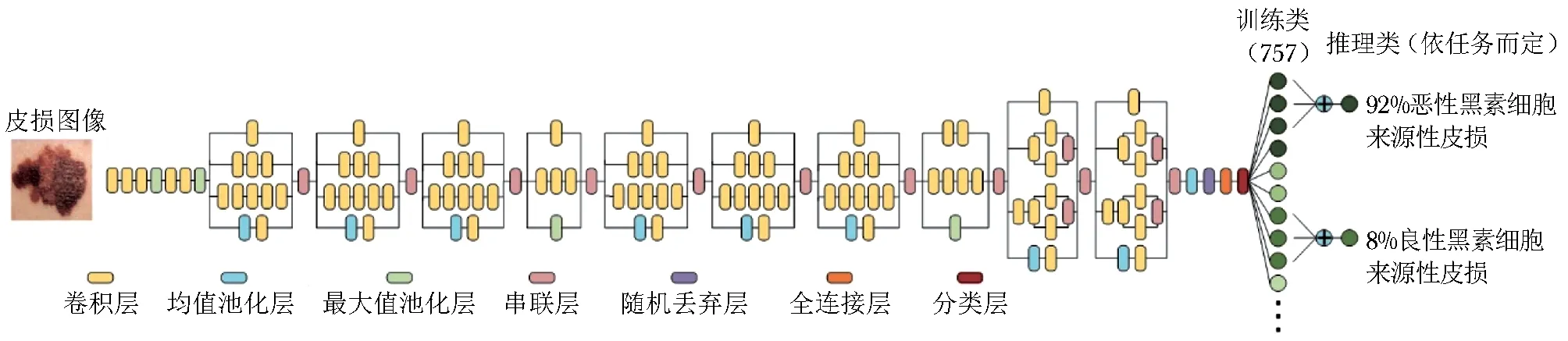
图2GoogLeNet Inception-v3结构图[10]
皮肤科医生在临床诊断过程中,需综合考虑皮损特征、位置以及患者年龄和性别等各种信息,而斯坦福团队当前的方法仅仅利用了皮肤镜图像和临床图像。除了皮肤镜和临床图像外,皮肤影像还包括共聚焦激光扫描显微镜、皮肤高频超声、皮肤光学相干层析成像、皮肤太赫兹成像、皮肤光声成像、多光子激光断层成像技术及皮损组织病理图像等等,其各具不同成像机理,可反映皮肤病变不同维度的性质。对于一些疑似病变,临床医生也通常会选择多种皮肤影像综合分析进而得到正确的诊断。而随着人工智能技术的发展,可把不同皮肤影像数据与患者个人信息相结合,采用深度学习的方法对这些多源、多维度的大数据进行融合分析,得到更为准确的自动分类结果,从而辅助医生作出更准确的诊断,并依此采取合适的方案为患者进行治疗。
预计到2021年,将会有大约63亿手机用户[13]。斯坦福团队设想,如果在移动端配备相应的软硬件,届时用手机进行低成本的皮肤病检测将成为可能。目前,CNN虽然功能强大,但巨大的存储和计算代价也使其实用性特别是在移动设备上的应用受到很大限制。不过目前已有学者进行有关网络压缩的研究[14],使用网络剪枝、权重量化和霍夫曼编码等技术对CNN进行压缩,其目标就是降低存储和计算消耗,使其可在移动设备上运行,相信这一快速、可扩展的方法未来将应用在手机,使皮肤病患者足不出户也能将自己的疾病信息与诊疗体系对接。
深度学习是一种数据驱动技术,对于同一分类网络,提供不同种类的训练样本,可实现不同分类任务。如能提供充足的训练图像数据,该技术可扩展至其他领域,比如眼科、耳鼻喉科、放射学和病理学等。可以预见的是,深度学习与皮肤病领域及其他医学图像领域的碰撞必将对临床诊断产生广泛而深刻的影响。
5 结语
斯坦福团队构建了庞大的数据库,利用深度学习技术结合皮肤病分类树形结构图以及巧妙设计的PA算法,在多个分类任务上达到了与专业皮肤科医生相当甚至更好的水平。代表了皮肤影像大数据分析的最新研究进展。基于皮肤影像对各类皮肤疾病进行计算机辅助诊断可以为医生提供重要的参考,具有很大的现实意义和研究价值。本文作者所在的北京航空航天大学图像处理中心实验室与北京协和医院、解放军空军总医院以及中日友好医院形成合作,将在皮肤影像大数据分析与计算机辅助诊断方面开展进一步的工作,以期推进黄色人种皮肤疾病的医疗诊断水平。
[1] Schmid-Saugeona P, Guillodb J, Thirana JP.Towards a computeraided diagnosis system for pigmented skin lesions [J]. Comput Med Imaging Graph, 2003, 27: 65- 78.
[2] Abbas Q, Celebi ME, García IF. Hair removal methods: a comparative study for dermoscopy images [J]. Biomed Signal Process Control, 2011, 6: 395- 404.
[3] Xie F, Bovik AC. Automatic segmentation of dermoscopy images using self-generating neural networks seeded by genetic algorithm [J]. Pattern Recognition, 2013, 46: 1012- 1019.
[5] Isasi AG, Zapirain BG, Zorrilla AM. Mendez. Melanomas Non-invasive Diagnosis Application Based on the ABCD Rule and Pattern Recognition Image Processing Algorithms [J]. Comput Biol Med, 2011, 41: 742- 755.
[6] Lecun Y, Bengio Y, Hinton G. Deep learning[J]. Nature, 2015, 521:436- 444.
[7] Krizhevsky A, Sutskever I, Hinton GE. ImageNet classifica-tion with deep convolutional neural networks[C]. Interna-tional Conference on Neural Information Processing Systems, 2012:1097- 1105.
[8] Szegedy C, Vanhoucke V, Ioffe S, et al. Rethinking the Inception Architecture for Computer Vision[J]. Computer Science, 2015:2818- 2826.
[9] Ren S, He K, Girshick R, et al. Faster R-CNN: towards real-time object detection with region proposal networks [J]. IEEE Trans Pattern Anal Mach Intell,2017,39:1137- 1149.
[10] Esteva A, Kuprel B, Novoa RA, et al. Dermatologist-level classification of skin cancer with deep neural networks[J]. Nature, 2017, 542:115.
[11] Masood A, Aljumaily AA. Computer aided diagnostic support system for skin cancer: a review of techniques and algorithms[J]. Int J Biomed Imaging, 2013, 2013:323268.
[12] Ramlakhan K, Shang Y. A mobile automated skin lesion classification system[C]. IEEE International Conference on TOOLS with Artificial Intelligence, 2011:138- 141.
[13] https://www.ericsson.com/res/docs/2016/ericsson-mobility-report- 2016.pdf (2016).
[14] Han S, Mao H, Dally WJ. Deep compression: compressing deep neural networks with pruning, trained quantization and huffman coding[J]. Fiber, 2016, 56:3- 7.

