DNA条形码技术及其在水生动物中的应用
王娜泠,胡则辉,卞光明,柴学军
(1.浙江海洋大学海洋与渔业研究所,浙江省海洋水产研究所,浙江舟山 316021;2.江苏省射阳县长荡镇农业技术推广服务中心,江苏盐城 224322)
随着环境的变化,水生动物面临着遗传多样性缺失、物种多样性枯竭等困境,对物种的快速检测、精准鉴定是保护水生动物多样性及其种质资源可持续利用的前提[1]。传统分类学主要依靠形态学及解剖学特征,受限于研究者经验、研究手段及研究对象形态近似等局限,难以快捷、有效地对水生动物进行精准分类,而DNA条形码技术利用生物一段标准DNA序列对生物条形码扫描即可识别物种,操作简便、鉴定快速准确、自动化水平高,对保护生物学和生物多样性研究具有重要意义[2-6]。
1 DNA条形码技术
1.1 DNA条形码技术发展历程
DNA条形码(DNA Barcoding)首先由HEBERT,et al[7]于2003年提出,即利用足够多的变异及已扩增的较短标准DNA序列片段,在生物种内特异性与种间多样性建立新的身份识别系统,从而快速、高效识别物种[8],随后HEBERT,et al[7]发现线粒体细胞色素氧化酶亚基I(cytochrome c oxidase I,COI)基因序列差异能够有效区分研究物种,提出建立以COI基因5'端648 bp序列多样性为基础的条形码鉴定系统。2004年,美国国立生物技术信息中心(National Biotechnology Information Center of the United States,NCBI)与生命条形码联盟(Consortium for the Barcode of Life,CBOL)合作将物种条形码标准DNA序列以及相关数据存储于GenBank中。随着2009年国际生命条形码计划实施,一个基于所有真核生物DNA条形码数据库自动鉴定系统逐步建立,截止2017年4月,生命条形码数据系统(Barcode of Life Data Systems,BOLD)数据库(http://www.barcodinglife.org/)已经收录超过538万条DNA条形码序列,其中正式记述动物物种17.7万个。此外,信息技术的发展为海量数据的处理提供了基础,推动了DNA条形码技术标准化应用,生命条形码协会和国际生命条形码计划还建立了许多针对各类群的DNA条形码数据库,如ABBI(All Birds Bar coding Initiative)(http://www.barcodingbirds.org/)、All-Leps(All Lepidopterans)(http://www.lepbarcoding.org/)、WoRMS(World Register of Marine Species)(http://www.marineSpecies.org/)等。
1.2 DNA条形码技术原理及特征
DNA条形码是由DNA序列4种碱基(A、T、G、C)不同的排列顺序构成,每个位点均有4种选择,该生物特有的DNA条形码标签,即15个位点便能够产生415种编码方式,是地球上现存生物物种数的10~100倍[9]。由于某些位点碱基受选择压力保持不变,可只考虑蛋白质编码基因,依据密码子的简并性,45个位点便可以形成10亿种不同序列。即使目前的一代测序技术,也能够精准的检测一段几百bp长度的DNA序列,故而,在理论上这段648 bp的DNA条形码足以鉴定所有物种。
作为DNA条形码基因区域应具备以下几个特征[10]:①积累足够多的变异,种内变异小,种间变异程度大,能够区分不同物种;②目标基因区具备相对保守性,便于引物通用性强,扩增成功率高;③必须是标准DNA片段,能够最大程度鉴别不同的分类群;④包含足够多的系统进化信息,能够精准地定位物种在分类系统中位置;⑤目标片段足够短,一个反应完成测序最佳,满足部分受损DNA样本扩增以及序列分析。鉴于以上条件,线粒体COI基因是水生动物分类和鉴定的理想DNA条形码区。主要是因为线粒体COI基因不含内含子,属于编码蛋白基因,能够通过翻译检测错误,几乎不发生重组、插入以及缺失等现象;其次,900 bp左右的蛋白编码基因(COI、Cyt b、ND4、ND5)中,COI基因进化速率适中,不会因为进化速率过快引起碱基二次突变而不能够区分远缘物种,几乎在所有动物种类COI基因5'端都能够找到作为扩增引物的通用序列[11],并且一般情况下水生动物的COI基因种间变异程度大于种内变异,HEBERT,et al[12]分析比较动物界11门13 320种亲缘关系较近同属物种的COI基因序列,发现种内差异多小于1%、极个别大于2%,而种间差异甚至高达11%以上,种内与种间差异之间存在形成明显的间隔区,是评价DNA条形码的重要指标;此外,相比较核基因以及其他蛋白基因,COI基因具有更多系统发育信息,更适合亲缘关系近的物种分类。故而,通常选用COI基因作为DNA条形码区,对于个别不能有效鉴定的物种,可使用其他基因片段辅助标记鉴别。
1.3 DNA条形码技术开发与分析
DNA条形码开发主要包括以下几个流程:首先,采集具有代表性、覆盖面广的群体样品,记录采集时间与位置,冷冻保存样品并提取样品DNA;然后设计引物对目的DNA进行PCR扩增,选择电泳条带清晰单一产物,进行DNA直接测序或连接质粒载体克隆测序;再次观察测序峰图,并对所得序列进行比对、人工校正,并通过MEGA、DNAsp等软件分析比较不同分类阶元的遗传距离(Kimura-2-parameter distance),构建Neighbour-jioning(N-J)或UPGMA系统树以及分析基因流情况等;最后将样品名称、DNA条形码、采集日期地点以及采集人员等样品数据提交至相关DNA条形码数据库(BOLD、ABBI、All-Leps以及WoRMS等)。流程图如图1。
1.4 DNA条形码技术优越性
生物表型特征具有一定可塑性,传统形态学分类方法对物种鉴定时易出现错误,而且此方法过于依赖分类学专家的识别能力,易忽略许多生物类群众普遍存在的隐存分类单元,形态检索表只能够与鉴别物种特定发育阶段相对应,无法对鉴定物种进行精准地识别,制约了生物多样性的研究。与之相比,DNA条形码技术具有以下优势:①对样品要求低,没有器官、组织特异性和完整性要求,微克级样品量、毛发、甚至某些死亡后的组织等均可提取DNA;②可利用生物DNA序列的稳定性与特异性,避免表型差异引起的鉴别错误,能够有效地鉴别形态差异微小的物种[10];③同一个体的DNA条形码序列在不同的生长阶段不会发生变化,这能够克服同一个体不同发育阶段表型差异显著而引起的误差,降低了鉴别工作量;④不同类群生物因生存环境等相似易出现表型特征相似、趋同进化现象,DNA条形码技术能够克服表型类似,从而鉴别出新种、隐存种,增加鉴定工作的准确性[13];⑤DNA条形码由四种碱基构成,表述明确,有效避免了因研究者语言表述不准确造成的歧义,且数据信息易于数据化管理,检索方便准确,能够实现大规模、高通量鉴定工作,推动物种鉴定以及进化研究的快速发展。两种分类方法主要特征比较见表1。
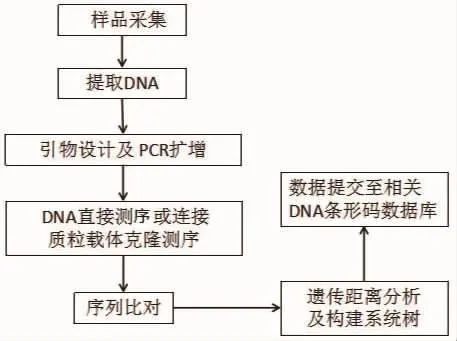
图1 DNA条形码开发流程图Fig.1 Process of DNA barcode development

表1 DNA条形码技术与传统分类法特征比较Tab.1 Comparison of DNA barcode technology and traditional taxonomy
2 DNA条形码技术在水生动物中应用
2.1 鱼类
目前,BOLD数据库中收录的鱼类DNA条形码大多来自COI基因,对鱼类进行分类鉴定、隐存种的发掘、系统发生研究等方面具有较大的应用价值。在国外,线粒体COI基因作为DNA条形码在淡水鱼(鲤鱼、鲫鱼、罗非鱼等)的物种鉴定中取得较好成效[14-20]。值得一提的是,CHAKRABORTY,et al[21]打破DNA条形码长片段(约600 bp)的常规,通过对1 367个线粒体COI基因序列(154 bp)分析,成功地界定了淡水鱼类中3个不同物种,发现该区域能够作为淡水鱼的物种特异性微型条形码。国外海水鱼类中,DNA条形码除了用于物种鉴别[22-23]之外,还用于对隐存种的发掘、系统发生研究等方面的研究[24-26]。随着生物分子学技术的发展,DNA条形码不再局限于线粒体COI基因,CHANG,et al[27]基于Cyt b片段有效地识别了台湾本土鳍刺类海水鱼类,并建立相关物种条形码参考数据库。FERNANDES,et al[28]利用线粒体COI和Cyt b作为条形码,将DNA微条形码与高分辨率溶解曲线(HRM)技术相结合,成功地鉴别了大西洋鳕鱼Gadus morhua、太平洋鳕鱼G.macrocephalus、阿拉斯加鳕鱼Theragra chalcogramma以及绿鳕。在DNA条形码技术较为成熟的基础上,BAMANIYA,et al[29]开发了31种印度重要的商业海洋观赏鱼DNA条形码。
在国内,鱼类线粒体COI基因被广泛用于DNA条形码的有效性验证及作为DNA条形码进行分类鉴别(表2)。当然,国内DNA条形码技术也不局限于线粒体COI基因。陈文炳等[30]利用线粒体16S rRNA基因进行鳗鱼的鉴定,成功检测出大量样品。陈文炳等[31]利用线粒体细胞色素b基因建立河豚鱼物种DNA鉴别技术,也证明了其可行性。
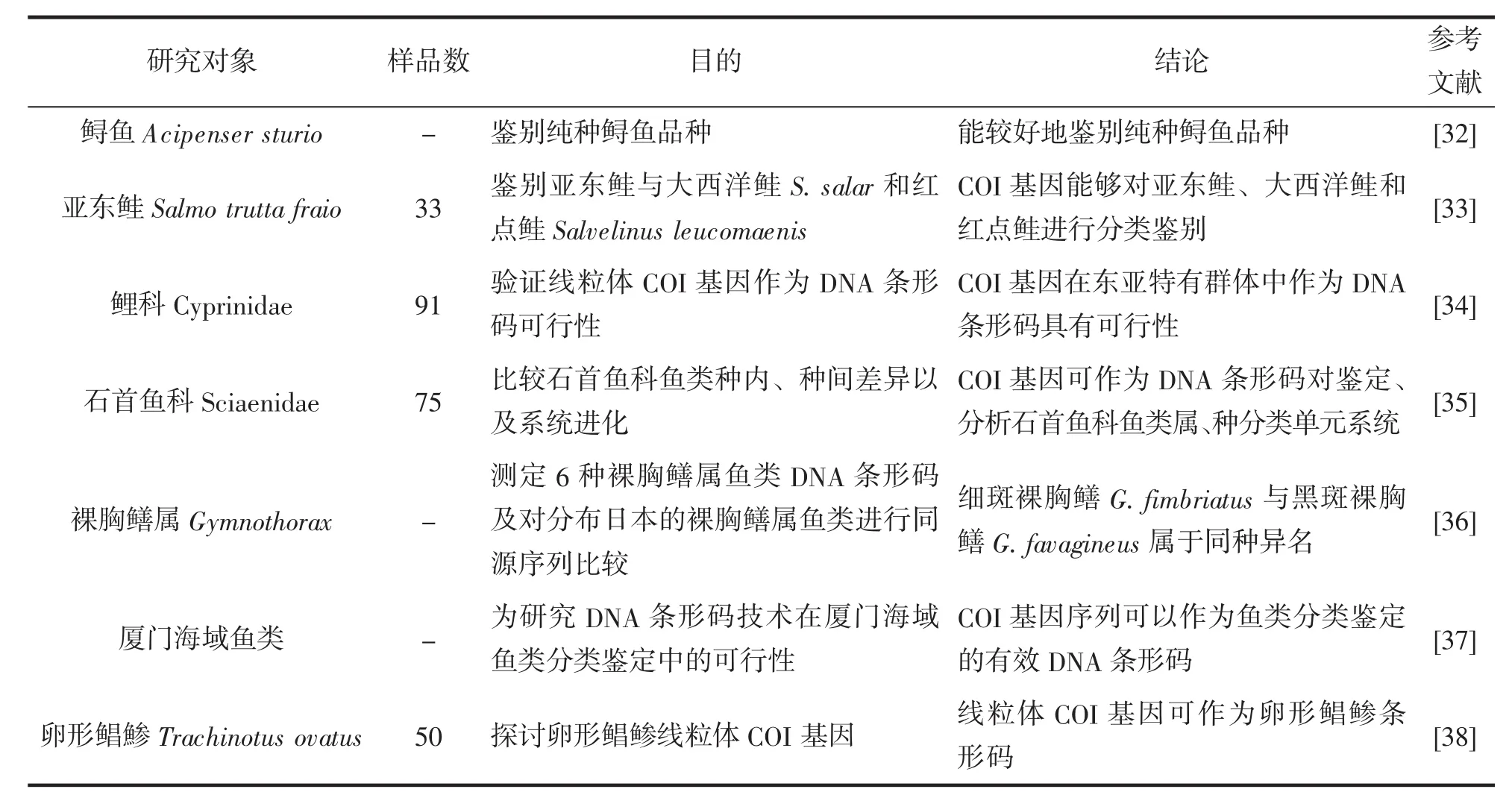
表2 国内基于线粒体COI基因的DNA条形码技术在鱼类中的应用Tab.2 The application of DNA barcoding technology on mitochondrial COI gene of fish in China
2.2 甲壳类
甲壳类DNA条形码研究相对鱼类更为完善,毋庸置疑线粒体COI基因被证明是理想的标准DNA条形码[39-42],也有学者尝试用其他区域基因作为DNA条形码对甲壳动物进行有效性验证,原帅等[43]测定了藤壶科线粒体COI、16S和12S的部分序列,并充分利用GenBank中藤壶科其它物种的相关基因序列,对其进行可行性和有效性鉴别,结果发现线粒体COI基因能更准确地鉴定藤壶科种间以及种内关系。
在小型甲壳动物桡足类物种中,BLANCOBERCIAL,et al[44]对63种桡足类物种的800条新条形码序列进行分析,并根据195个桡足类物种的1 381个条形码序列的数据集,检查了不同统计学方法对物种鉴定的可靠性和分辨率,发现DNA条形码技术在桡足类物种识别中具有重要价值,实现了DNA条形码高通量的应用。BTTGER-SCHNACKET,et al[45]对COI基因和12S基因比较分析,推测12S基因可能更适合作为DNA条形码对隆剑水蚤科进行有效鉴定。
90多种磷虾目中,由于其细微的形态特征及幼体的发育特征给鉴定带来极大的挑战。线粒体COI基因仍然能够作为DNA条形码对其进行物种鉴定、隐存种的发现及系统地理学方面的研究[46]。
2.3 贝类
贝类属于软体动物,目前国际上对贝类DNA条形码研究报道较少。大量利用DNA条形码对鱼类、甲壳类水生动物进行物种鉴定、隐存种的发掘等方面的研究启发了学者们利用同样的方法对形态相似、种类繁多的贝类进行鉴定。研究者们发现线粒体COI基因能够有效地对宝贝科Cypraeidae[47]、帽贝Lepetodrilus limpets[48]、双壳类[49]、四角蛤蜊 Mactra veneriformis[50]、蚌类[51]、缀锦蛤亚科 Tapetinae[52]、头足类[53]、贻贝科Mytilidae[54]、异齿亚纲[55]、骨螺科Muricidae和织纹螺科Nassariidae[56]等贝类进行物种鉴定。也有研究利用线粒体16S r RNA基因对贝类进行种类鉴定,李海涛等[57]采用DNA条形码技术对部分形态上难以鉴定的种类,如线缝摺塔螺Ptychobela suturalis和区系螺Funa sp.、锯齿巴非蛤Paphia gallus、西格织纹螺Nassarius siquijorensis、爪哇拟塔螺Turricula javana等,结果表明,DNA条形码技术能有效提高海洋贝类物种鉴定的准确性并发现隐存种。
2.4 其它水生动物
在其他水生动物中也有一些关于DNA条形码的报道。陈海燕[58]利用DNA条形码技术分析岩礁海藻附植动物,结果表明,鼠尾藻附植动物的年平均丰度、类群数、生物量均高于其他海藻附植动物,海藻附植动物的优势类群在不同月份和不同海藻之间均存在差异。律迎春[59]基于COI基因作为DNA条形码分析海参群体,发现海参的种间遗传距离显著大于种内遗传距离,同种海参不同个体在系统树种分别形成各自独立的分支。辛翠娜等[60]对采自中国海南、广东、云南以及缅甸的4科7个种18个龟鳖类个体的COI序列进行分析,证实了线粒体COI序列能够作为DNA条形码对龟鳖类进行物种鉴定。此外,张珰妮等[61]以COI和16S rRNA基因作为DNA条形码对北部湾北部的28种水螅水母进行了有效地物种鉴定。
3 展望
DNA条形码技术促进了水生动物分类研究,但若成为准确的分析手段还需进一步完善:首先,若研究的物种种内种间遗传差异较小,种内分化过高或种间分化较低,物种间界定不显著,则无法通过DNA条形码技术对物种进行准确鉴别[62];其次,DNA序列中存在假基因——非同义突变、提前出现终止密码子以及基因插入缺失等也会影响DNA条形码鉴定效果,SONG,et al[63]研究小龙虾假基因对DNA条形码鉴定的影响,发现假基因能够引起DNA条形码技术过高估计物种数目,在支系中存在特异性分布,无法完全剔除全部假基因;再次,通用引物获取难度较大,目的片段的获得需要利用大约20 bp长度的引物进行PCR扩增获得,获取合适的引物需要对物种的某区域基因深入了解,根据目、科、种、属逐级查找最合适条形码,而生物核基因、细胞器基因等不同DNA基因区段进化速率差异显著,难以找到某DNA序列来鉴定地球上所有水生动物,COI基因序列进化速率与物种所处的分类阶段都不是绝对对应,难以实现对水生动物进行完全鉴定,尤其对于多样性较高的热带物种[64];此外,许多实验研究取材数量较少,易低估种内差异,或没有考虑姊妹群而高估种间差异[65],对于普遍存在的共生、寄生物种,由于二者DNA提取分离难度较大,使得通用DNA条形码难以实现物种的有效区分。故而,进一步优化DNA提取方法、开发DNA条形码芯片、完善“微型条形码”鉴定技术、通过具体实践有针对性解决上述局限是DNA条形码发展的方向。
全面深入分清待分类物种的形态学特征是DNA条形码研究正确取样的前提[66],而DNA条形码是分类学新的发展动力,是电子技术便携性与信息存储功能的综合产物,能够分析物种分支来源、模拟物种进化趋势[67]。在传统分类学基础上发展出的DNA条形码技术,在与生物识别的DNA条形码相结合时发展出的高通量条形码技术(Metabarcoding),通过这些技术有机结合,将能够实现物种更加精准、科学地鉴别效果。
[1]卢 珊.常见水生动物与其环境DNA的定性与定量关系[D].南京:南京师范大学,2015.
[2]彭居俐,王绪桢,何舜平.DNA条形码技术的研究进展及其应用[J].水生生物学报,2008,32(6):916-919.
[3]宁淑萍,颜海飞,郝 刚,等.植物DNA条形码研究进展[J].生物多样性,2008,16(5):417-425.
[4]吴清楠.DNA条形码技术在常见水生动物分类中的应用[J].生物技术世界,2013(1):5-6.
[5]程新玮,赵焕新,宁 康,等.DNA条形码技术在中药质量评价中的研究进展[J].食品与药品,2013(4):295-299.
[6]高连明.DNA条形码在生物多样性编目与评价中的应用[J].生物多样性,2015,23(3):286-287.
[7]HEBERT P D N,CYWINSKA A,BALL S L,et al.Biological identification through DNA barcodes[J].Proceedings of the Royal Society B:Biological Sciences,2003,270(1512):313-321.
[8]任保青,陈之端.植物DNA条形码技术[J].植物学报,2010,45(1):1-12.
[9]赵广宇,李 虎,杨海林,等.DNA条形码技术在昆虫学中的应用[J].植物保护学报,2014,41(2):129-141.
[10]肖金花,肖 晖,黄大卫.生物分类学的新动向-DNA条形编码[J].动物学报,2004,50(5):852-855.
[11]焦明超,赵大显,欧阳珊,等.DNA条形码技术在生物分类学中的应用前景[J].湖北农业科学,2011,50(5):886-890.
[12]HEBERT P D N,RATNASINGHAM S,de WAARD J R.Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J].Proceedings Biological Sciences,2003,270(s1):S96-S99.
[13]李 琪,邹山梅,郑小东,等.DNA条形码及其在海洋生物中的应用[J].中国海洋大学学报:自然科学版,2010,40(8):43-47.
[14]HUBERT N,HANNER R,HOLM E,et al.Identifying Canadian Freshwater Fishes through DNA Barcodes[J].Plos One,2008,3(6):e2490.doi:10.137//journal.pone.0002490.
[15]BARMAN A S,SINGH M,PANDEY P K.DNA barcoding and genetic diversity analyses of fishes of Kaladan River of Indo-Myanmar biodiversity hotspot[J].Mitochondrial DNA A:DNA Mapp Seq Anal,2017:1-12.doi:10.1080/24701394.2017.1285290.
[16]DAHRUDDIN H,HUTAMA A,BUSSON F,et al.Revisiting the ichthyodiversity of Java and Bali through DNA barcodes:taxonomic coverage,identification accuracy,cryptic diversity and identification of exotic species[J].Molecular Ecology Resources,2016.doi:10.1111/1755-0998.12528.
[17]BECKER R A,SALES N G,SANTOS G M,et al.DNA barcoding and morphological identification of neotropical ichthyoplankton from the Upper Paraná and São Francisco[J].Journal of Fish Biology,2015,87(1):159-168.
[18]KARIM A,IQBAL A,AKHTAR R,et al.Barcoding of fresh water fishes from Pakistan[J].Mitochondrial DNA A:DNA Mapp Seq Anal,2016,27(4):2 685-2 688.
[19]KESKIN E,AǍDAMARS,TARKAN A S.DNA barcoding common non-native freshwater fish species in Turkey:low genetic diversity but high population structuring[J].Mitochondrial DNA A:DNA Mapp Seq Anal,2013,24(3):276-287.
[20]MARANAN J B D,BASIAO Z U,QUILANG J P.DNA barcoding of feral tilapias in Philippine lakes[J].Mitochondrial DNA A:DNA Mapp Seq Anal,2015:1-12.http://dx.doi.org/10.3109/19401736.2015.1089478.
[21]CHAKRABORTY M,DHAR B,GHOSH S K.Design of Character-based DNA Barcode Motif for species identification:A computational approach and its validation in fishes[J].Molecular Ecology Resources,2017,17(6).Doi:10.1111/1755-0998.12671.
[22]WARD R D,ZEMLAK T S,INNES B H,et al.DNA Barcoding Australia's Fish Species[J].Philosophical Transactions of the Royal Society B:Biological Sciences,2005,360(1462):1 847-1 857.
[23]BINEESH K K,GOPALAKRISHNAN A,AKHILESH K V,et al.DNA barcoding reveals species composition of sharks and rays in the Indian commercial fishery[J].Mitochondrial DNA A:DNA Mapp Seq Anal,2016.http://dx.doi.org/10.31091/19401736.2015.1137900.
[24]WARD R D,COSTA F O,HOLMES B H,et al.DNA barcoding of shared fish species from the North Atlantic and Australasia:minimal divergence for most taxa,but Zeus faber and Lepidopus caudatus each probably constitute two species[J].Aquatic Biology,2008,3(1):71-78.
[25]SCHLEI O L,WHITELEY A R,BROWN R J,et al.DNA barcoding of eight North American coregonine species[J].Molecular Ecology Resources,2008,8(6):1 212-1 218.
[26]ZEMLAK T S,WARD R D,CONNELL A D,et al.DNA barcoding reveals overlooked marine fishes[J].Molecular Ecology Resources,2009,9(s1):237-242.
[27]CHANG Chia-hao,SHAO Kwang-Tsao,LIN Han-yang,et al.DNA barcodes of the native ray-finned fishes in Taiwan[J].Molecular Ecology Resources,2017,17(4):796-805.
[28]FERMANDEST J,COSTA J,OLIVEIRA M B,et al.DNA barcoding coupled to HRM analysis as a new and simple tool for the authentication of Gadidae fish species[J].Food Chemistry,2017,230:49-57.
[29]BAMANIYA D C,PAVANKUMAR A,GIREESHBABU P,et al.DNA barcoding of marine ornamental fishes from India[J].Mitochondrial DNA,2016,27(5):3 093-3 097.
[30]陈文炳,缪婷玉,彭 娟,等.基于16SrRNA基因DNA条形码鉴定美洲鳗、欧洲鳗、日本鳗[J].食品科学,2017,38(4):283-289.
[31]陈文炳,林少华,邵碧英,等.河豚鱼Cyt b基因部分DNA序列分析与应用[J].食品科学,2012,33(20):227-232.
[32]吴文化.几种鲟鱼及杂交种DNA条形码研究及分子鉴别[D].呼和浩特:内蒙古农业大学,2010.
[33]孟 玮,杨天燕,海 萨,等.基于线粒体COI基因序列的亚东鲑DNA条形码研究[J].水产学杂志,2010,23(1):6-10.
[34]彭居俐.东亚特有鲤科类群的DNA条形码研究及其系统发育分析[D].武汉:中国科学院水生生物研究所,2007.
[35]柳淑芳,陈亮亮,戴芳群,等.基于线粒体COI基因的DNA条形码在石首鱼科(Sciaenidae)鱼类系统分类中的应用[J].海洋与湖沼,2010,41(2):223-232.
[36]齐兴柱,骆 剑,刘志亮,等.基于COⅠ序列的DNA条形码在中国南海裸胸鳝属鱼类中的应用[J].热带生物学报,2010,1(4):321-326.
[37]邢炳鹏,林汝榕,王彦国,等.基于COI基因的厦门海域鱼类DNA条形码鉴定[J].应用海洋学学报,2016(1):144-150.
[38]韦嫔媛,彭金霞,房振峰,等.卵形鲳鲹线粒体COI基因全长序列的克隆与分析[J].西南农业学报,2011,24(4):1 552-1 557.
[39]RADULOVICI A E,SAINTE-MARIE B,DUFRESNE F.DNA barcoding of marine crustaceans from the Estuary and Gulf of StLawrence:aregional-scaleapproach[J].MolecularEcologyResources,2009,9(s1):181-187.doi:10.1111/;1755-0998.2009.02643x.
[40]RAUPACH M J,KNEBELSBERGER T,LAAKMANN S,et al.DNA Barcoding of Amphipoda from the North Sea[R]//New frontiers in Monitoring European Biodiversity conference.22-29.sept.2011,Palermo,Italy.
[41]王敏晓,程方平,李超伦,等.基于线粒体COI片段序列的胶州湾浮游动物DNA条形码分析[J].海洋与湖沼,2011,42(5):702-710.
[42]KHALAJI-PIRBALOUTY V,RAUPACH M J.DNA barcoding and morphological studies confirm the occurrence of three Atarbolana(Crustacea:Isopoda:Cirolanidae)species along the coastal zone of the Persian Gulf and Gulf of Oman[J].Zootaxa,2016,4200(1):153-173.
[43]原 帅.围胸总目、短尾次目(甲壳动物亚门)DNA条形码研究[D].临汾:山西师范大学,2012.
[44]BLANCO-BERCIAL L,CORNILS A,COPLEY N,et al.DNA barcoding of marine copepods:assessment of analytical approaches to species identification[J].Plos Curr,2014,6(6):S122.Doi:10.1371/currents.tol.cdf8674881f87e360/d56643791626dz.
[45]BTTGER-SCHNACK R,MACHIDA R J.Comparison of morphological and molecular traits for species identification and taxonomic grouping of oncaeid copepods[J].Hydrobiologia,2011,666(1):111-125.
[46]BUCKLIN A,WIEBE P H,SMOLENACK S B,et al.DNA barcodes for species identification of euphausiids(Euphausiacea,Crustacea)[J].Journal of Plankton Research,2007,29(6):483-493.
[47]KNOWLTON N,WEIGT L A.New dates and new rates for divergence across the Isthmus of Panama[J].Proceedings of the Royal Society B:Biological Sciences,1998,265(1412):2 257-2 263.
[48]JOHNSON S B,WARÉN A,VRIJENHOEK R C.DNA Barcoding of Lepetodrilus Limpets Reveals Cryptic Species[J].Journal of Shellfish Research,2007,27(1):43-51.
[49]MIKKELSEN N T,SCHANDER C,WILLASSEN E.Local scale DNA barcoding of bivalves(Mollusca):a case study[J].Zoologica Scripta,2007,36(5):455-463.
[50]朱立静,陈淑吟,许晓风,等.四角蛤蜊江苏群体线粒体COI基因片段序列研究[J].江苏农业科学,2010(4):33-35.
[51]欧阳解秀,吴小平,欧阳珊,等.DNA条形码技术及其在蚌类保护生物学中的研究[J].中国畜牧兽医,2010,37(4):167-171.
[52]陈 军,李 琪,孔令锋,等.基于COI序列的DNA条形码在中国沿海缀锦蛤亚科贝类中的应用分析[J].动物学研究,2010,31(4):345-352.
[53]王 鹤,林 琳,柳淑芳,等.中国近海习见头足类DNA条形码及其分子系统进化[J].中国水产科学,2011,18(2):245-255.
[54]刘 君,李 琪,孔令锋,等.基于线粒体COⅠ的DNA条形码技术在贻贝科种类鉴定中的应用[J].水生生物学报,2011,35(5):874-881.
[55]于贞贞.异齿亚纲贝类DNA条形码与系统发生学研究[D].青岛:中国海洋大学,2014.
[56]邹山梅.新腹足目贝类DNA条形码系统构建及系统发育研究[D].青岛:中国海洋大学,2013.
[57]李海涛,张保学,高 阳,等.DNA条形码技术在海洋贝类鉴定中的实践:以大亚湾生态监控区为例[J].生物多样性,2015,23(3):299-305.
[58]陈海燕.用分子鉴定方法对岩礁海藻附植动物多样性和生态学的探索研究[D].青岛:中国海洋大学,2010.
[59]律迎春.基于DNA条形码的分子生物学方法鉴定海参种类的研究[D].青岛:中国海洋大学,2012.
[60]辛翠娜,王 莹,彭建军,等.DNA条形码在龟鳖类物种鉴定中的应用[J].林业实用技术,2010(4):12-14.
[61]张珰妮,郑连明,何劲儒,等.基于线粒体COI和16S片段序列的北部湾北部水螅水母DNA条形码分析[J].生物多样性,2015,23(1):50-60.
[62]BOYER S L,BAKER J M,GIRIBET G.Deep genetic divergences in Aoraki denticulata(Arachnida,Opiliones,Cyphophthalmi):a widespread“mite harvestman”defies DNA taxonomy[J].Molecular Ecology,2007,16(23):4 999-5 016.
[63]SONG Hojun,BUHAY J E,WHITING M F,et al.Many species in one:DNA barcoding overestimates the number of species when nuclear mitochondrial pseudogenes are coamplified[J].Proceedings of the National Academy of Sciences of the United States of America,2008,105(36):13 486-13 491.
[64]RUBINOFF D,CAMERON S,WILL K.A genomic perspective on the shortcomings of mitochondrial DNA for"barcoding"identification[J].Journal of Heredity,2006,97(6):581-594.
[65]DASMAHAPATRA K K,MALLET J.DNA barcodes:recent successes and future prospects[J].Heredity,2006,97(4):254-255.
[66]MEYER C P,PAULAY G.DNA barcoding:error rates based on comprehensive sampling[J].Plos Biology,2005,3(12):2 229-2 238.
[67]傅美兰,彭建军,王 莹,等.DNA条形码技术的应用与分析[J].河南师范大学学报:自然科学版,2010,38(4):118-122.

