作物纤维素合成酶家族序列研究与系统发育分析
张利慧,刘 欣,王晓峰,李积胜,陈少林
(西北农林科技大学 生命科学学院,陕西 杨陵 712100)
纤维素是维管植物细胞壁的主要成分,但在非维管植物、被囊类海洋生物和许多细菌中也有发现[1-3]。基于纤维素类的生物质能源是一种非常有前景的可再生能源[4-5]。细胞壁在植物生长发育和响应环境胁迫中发挥重要作用,因此纤维素合成机制的相关研究在多个学科中占重要地位[6-9]。
纤维素由定位于质膜上的纤维素合酶复合体(CESA)合成[10]。高等植物的CESA复合体在冷冻蚀刻电镜下呈现六瓣花状结构[11]。CESA4、CESA7、CESA8是拟南芥(Arabidopsis)次生壁合成必需的[12-15],而CESA1和CESA3是初生壁纤维素合成必需的[12-15]。CESA2、CESA5、CESA6、CESA9在初生壁纤维素合成中存在部分功能冗余[12-15]。据推测,CESA复合体中的每个蛋白单体合成一条β-(1,4)-D-葡糖苷链,再聚合形成纤维素微纤丝。CESA复合体与周质微管间存在直接的相互作用,在微管的导引下双向合成纤维素微纤丝[16]。纤维素合酶互作蛋白(CSI1)和纤维素合酶伴侣蛋白(CC)参与微管和纤维素合酶复合体的相互作用[17-18]。CC蛋白具有在盐胁迫条件下维持纤维素的合成的作用[18]。
据推测,植物CESAs在N端和C端分别有2个和6个跨膜区域 (图1)。拟南芥CESAs的N端由160~260个氨基酸残基组成,该区域在结构上又被划分成3个结构域(图1)[19],包括1个锌指结构域(Zn,大约50个氨基酸),含2个锌指模序(motif)和8个高度保守的半胱氨酸残基 (Cx2Cx12FxACx2Cx2PxCx2CxEx5Gx3Cx2C)。锌指结构域的上游是N末端(NT)结构域,约含8~38个氨基酸。锌指结构域的下游是N端的高变区(HVR1)。在2个跨膜区域之间的是中央结构域,其中含有另一个植物特有的高变区(HVR2)。2个高变区在直系同源中相对保守,因此HVR1和HVR2又分别被命名为物种特异区C-SR1和C-SR2[20]。中央结构域还含有另一个植物特有的保守区域(P-CR),以及3个保守的天冬氨酸残基和1个Q/RXXRW模序,它们可能在结合底物和催化过程中起重要作用(图1)。
蛋白磷酸化是进化的重要途径,为快速、可逆的调控蛋白质结构与功能提供了重要机制[21]。基于质谱技术的磷酸化蛋白组学研究表明,拟南芥等植物的CESAs中有多个磷酸化位点[22-26]。我们前期的模拟磷酸化(phosphomemetic)和去磷酸化(nonphosphorylable)的突变研究表明,拟南芥CESA1和CESA3的磷酸化是调控微管引导的纤维素合成与沉积以及组织特异的细胞各向异性生长的重要分子机制[27-28]。而CESA5的磷酸化则是光敏色素调控纤维素合成和下胚轴细胞伸长生长的重要机制[29]。磷酸化蛋白组学研究进一步表明, CESAs的磷酸化修饰响应植物生长发育及环境信号[22-26],调控微管介导的纤维素合成和沉积的关键机制[27-29]。
植物细胞的特殊形态通常与其行使的特定功能密切相关。纤维素生物合成对于植物细胞形态建成以及发挥特定功能至关重要。本研究以拟南芥CESA为对照,对杨树、葡萄、番茄、苜蓿以及小麦、大麦、玉米、水稻和大豆等13个物种的CESA家族成员进行了序列比对和系统发育分析,结合磷酸化蛋白组学数据的收集和整理,分析鉴定了磷酸化位点在CESA家族以及物种间的保守性。本研究提供了农林作物CESAs磷酸化位点演变的新见解,对于进一步了解农林作物的生长发育分子调控机制具有重要的科学理论意义。
1 材料与方法
1.1 序列信息
CESA序列来自于TAIR (http://www.arabidopsis.org/)、GeneBank (http://www.ncbi.nlm.nih.gov/genbank/) 和JGI (https://phytozome.jgi.doe.gov/pz/portal.html) 等数据库。本研究用到的基因组列于表1中。
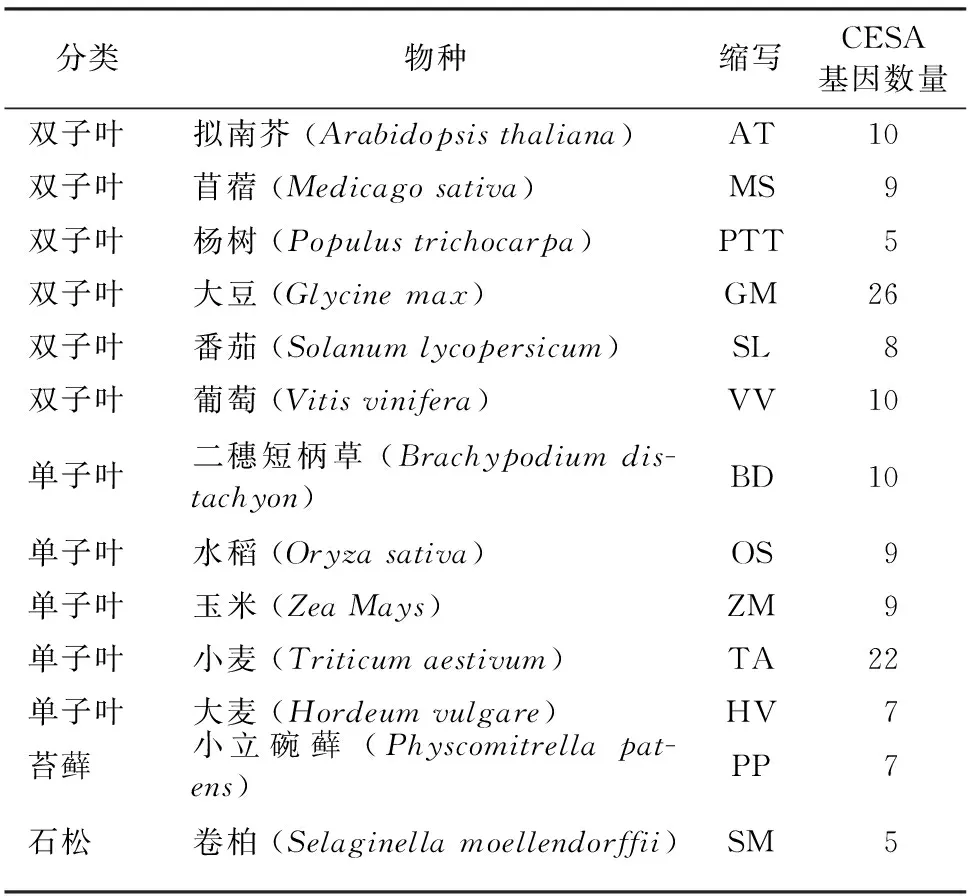
表1 基因组信息
1.2 系统发育树分析
CESAs的整合与分析借助于MUSCLE软件[30]。按照极大似然法构建CESA序列的进化树,由I.LETUNIC[31]等整理并展示出CESA家族序列在进化树中的分布。
1.3 磷酸化位点分析
从PhosPhAt和P3DB等磷酸化蛋白组学数据库中提取、整理和确认了拟南芥CESAs的磷酸化位点。表2为位置确定的CESAs磷酸化位点[22-26]。MEGA6.0用于测定磷酸化位点的保守程度[32]。每个磷酸化位点保守概率是按照丝氨酸/苏氨酸/络氨酸所占的百分比估算,该密度以热图的形式由Heml 1.0作标准化处理[31]。
2 结果与分析
2.1 CESA的系统发育树分析
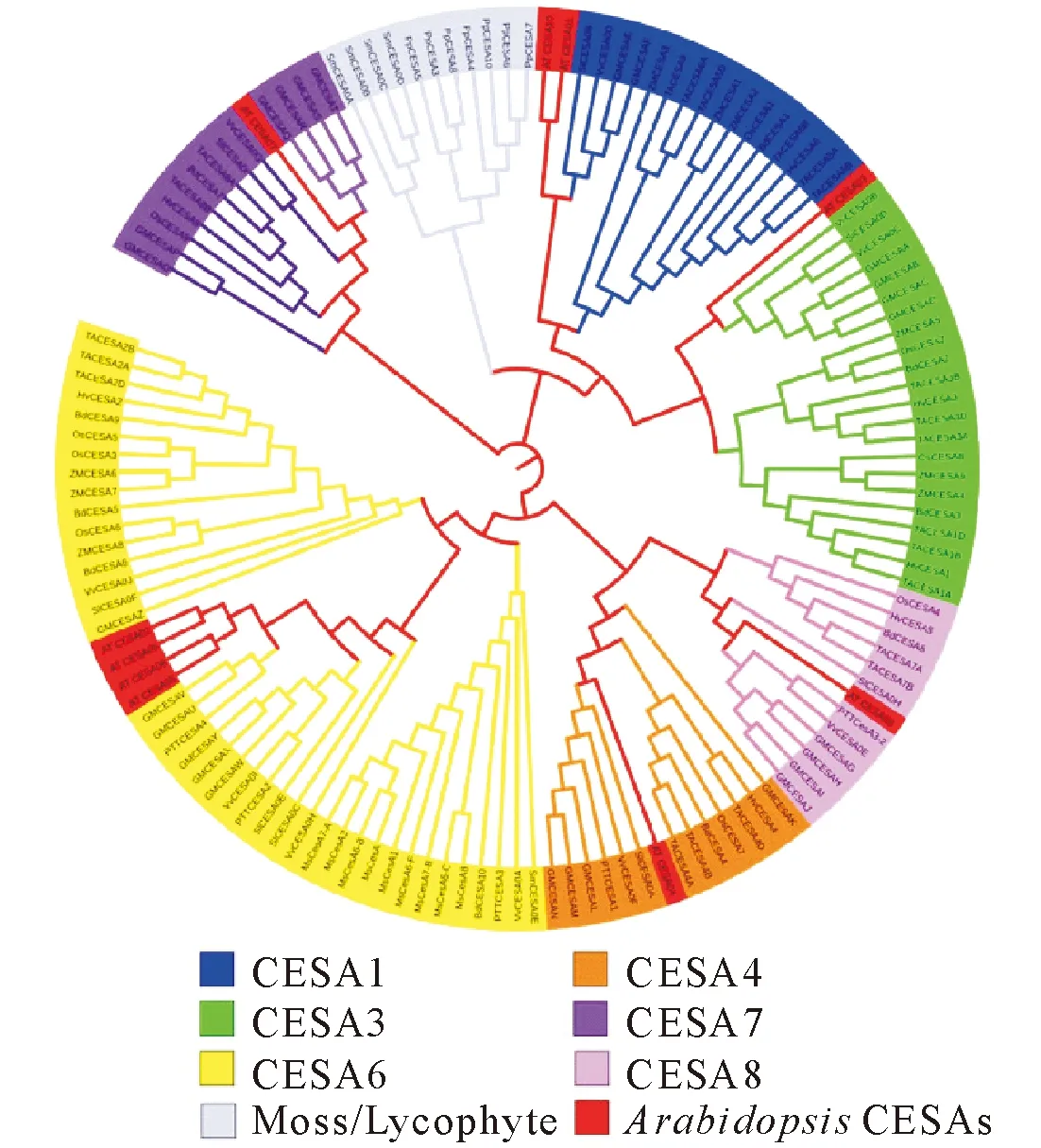
本研究以拟南芥以及小立碗藓和卷柏的CESA为对照,对杨树、葡萄、番茄、苜蓿以及小麦、大麦、玉米、水稻和大豆等作物的CESA家族成员进行了序列比对和系统发育分析。系统发育树分析结果表明,CESA蛋白家族聚为7大类,包括高等植物的CESA1、CESA3和CESA6家族以及CESA4、CESA7和CESA8家族。苔藓或石松等低等植物的CESA形成另一个家族(图2)。前期研究表明,参与初生壁或次生壁合成的纤维素合成酶复合体需要至少3种不同的CESA蛋白[12-15]。拟南芥的CESA1、CESA3和CESA6参与初生壁纤维素微纤丝的合成,而CESA4、CESA7和CESA8参与次生壁纤维素合成[12-15]。
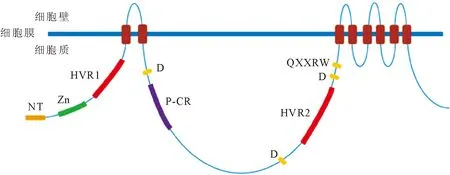
注:CESA蛋白包含8个跨膜区域(棕色短棒),1个N末端结构域(NT)和1个锌指结构域(Zn)。在N端的胞质区有1个高变区(HVR1),在中央胞质区有1个植物保守区域(P-CR)和另1个高变区(HVR2)。中央胞质区的保守氨基酸残基(D,D,D,QXXRW)被认为在与底物结合的过程中起重要作用,其次就是催化过程。
图1高等植物CESA蛋白的结构示意图
Fig.1 A schematic diagram of the higher plant CESA proteins

注:拟南芥CESAs中已鉴定出的磷酸化的丝氨酸位点和锌指结构域中保守的半胱氨酸均用灰色标出。
2.2 CESA蛋白序列分析及其磷酸化位点分布
高等植物的磷酸化位点主要集中在3个区域,分别是氮末端结构域(NT)以及高变区1(HVR1)和高变区2(HVR2)[22-26]。目前已发现的CESAs磷酸化位点主要集中于HVR1区域。例如,拟南芥CESA1和CESA3的HVR1区域分别含有8个和10个磷酸化位点[22-26]。在CESA6 和CESA6-like亚型(CESA2、5、9)中,只有CESA5被鉴定出在该区域含有磷酸化位点。参与次生壁合成的CESA4和CESA8的HVR1区域中也发现含有磷酸化位点(表2)。
NT区域只含有少量的磷酸化位点(图2)。拟南芥CESAs的NT结构域的磷酸化位点包括CESA1的S24位点,CESA3的S3位点以及CESA2、5、6、9的S11位点等(表2)[22-26]。序列比对分析结果表明,S11位点及其两端的序列在拟南芥的CESA6和CESA6-like(CESA2、5、9)亚基中相对保守。但是,相比于氮端的锌指结构域,不同CESAs的NT结构域的保守性相对较低(图2)。例如,相对于CESA1、CESA6以及CESA6-like,CESA3的NT结构域的部分序列缺失,导致S11位点的缺失(表2、图2),表明NT结构域对于CESA3的功能并非必需。然而,CESA3保留或获得了S3磷酸化位点。
除了NT和HVR1结构域外,另一个高变区HVR2也发现含有磷酸化位点,然而参与次生壁合成的CESA4、7、8的HVR2结构域中却没有发现磷酸化位点(图1、表2),表明初生壁和次生壁的纤维素合成调控机制可能有所不同。
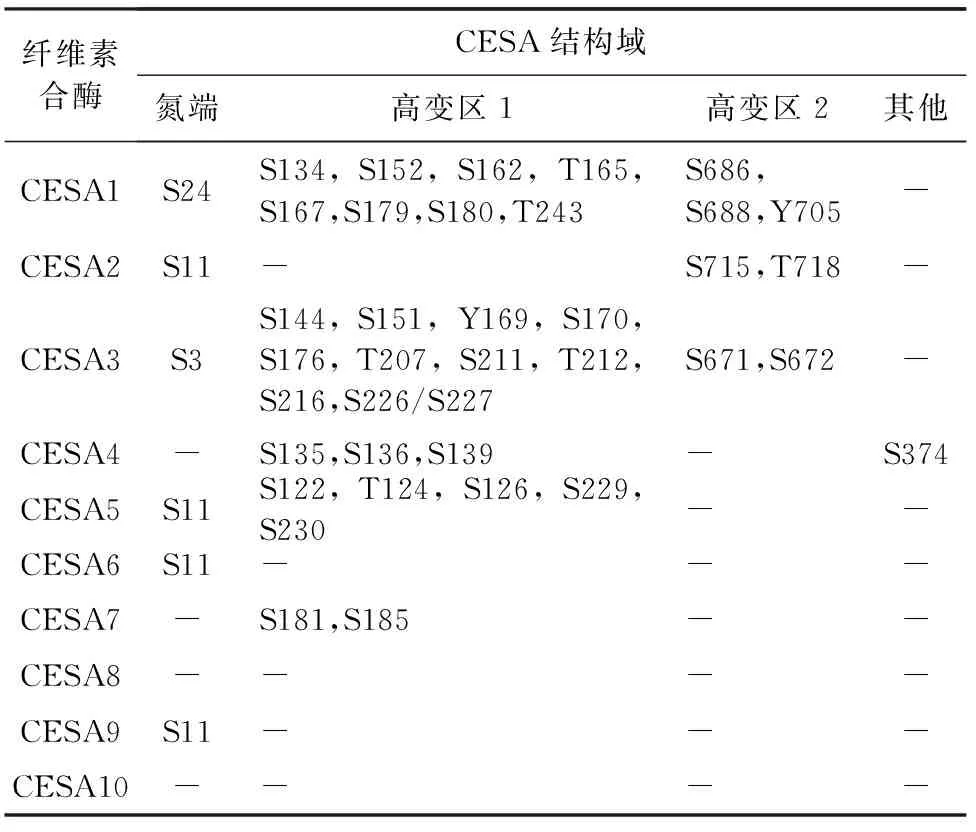
表2 拟南芥中已被鉴定出的磷酸化位点
注:拟南芥已鉴定出的CESAs磷酸化位点信息来源于PhospPHAte 4.0 (http://phosphat.uni-hohenheim.de/) 和 P3DB (http://p3db.org/)数据库。
2.3 CESA家族磷酸化位点的进化
拟南芥以及作物CESAs的磷酸化位点主要集中在NT、HVR1和HVR2结构域[22-26]。比较基因组学分析表明,这些结构域已存在于苔藓和石松类的CESAs中,但是细菌的CESAs则不含这些结构域,表明这些结构域对于植物细胞壁的纤维素合成乃至植物的生长发育可能具有重要功能,这一预测与近期的研究结果相一致[27-28]。
在苔藓植物小立碗藓和石松类植物卷柏中,分别存在着至少7条和5条CESA基因。在系统发育树中,这些序列分别形成了各自的分支(图3)。尽管小立碗藓的基因组中不存在与作物CESAs同源的序列,但是拟南芥CESAs中被鉴定的磷酸化位点却在小立碗藓CESAs中保守,包括CESA1的S152、S686、S688位点,CESA3的S144、S211、S671、T672位点,CESA 2、5、6和9的S11位点,CESA2的S715、T718位点,CESA5的S126位点以及CESA4的S135位点(图4)。另外,拟南芥CESA3的S151位点,CESA4的S139位点,CESA5的S122位点和CESA7的S185位点在小立碗藓的CESAs中部分保守。与作物CESAs相似的是,苔藓CESAs保守的丝氨酸/苏氨酸/络氨酸同样集中于NT、HVR1和HVR2结构域中。尤其是拟南芥CESA2的T718磷酸化位点,在苔藓CESA分支和作物CESA的6大分支中都保守(图4)。此外,拟南芥CESA1的S152位点,CESA2、5、6和9的S11位点,CESA2的S715位点,CESA3的S144、S211和T672位点,CESA4的S135位点,也在苔藓的CESAs中保守,但只在少数初生壁或次生壁的CESA分支里保守(图4)。另一方面,CESA1的S686、S688位点和CESA3的S671位点只在初生壁相关的CESA1或CESA3分支中保守,而CESA4的S139位点除了在苔藓CESA分支中相对保守之外,还在次生壁相关的CESA4和CESA8分支中保守。然而,拟南芥CESA5的S126磷酸化位点在苔藓CESA分支和CESA8分支中保守。
拟南芥CESAs中的一些磷酸化位点在苔藓的CESAs分支中并不保守,包括CESA1的S134、S162、S167、S180位点,CESA3的Y169、S176、T212、S216、S226位点,CESA5的T124、S229、S230位点,CESA4的S136、S374位点,以及CESA7的S181位点。这些氨基酸残基全都集中在HVR1区域,可能在作物CESA的进化中有重要作用。
3 结论与讨论
模拟磷酸化和去磷酸化突变研究表明[27-28],CESA1和CESA3的HVR1和HVR2区域的部分磷酸化位点的磷酸化和去磷酸化显著影响纤维素的生物合成以及细胞的伸长生长,表明HVR1和HVR2结构域的磷酸化在调节纤维素的合成和沉积以及细胞形态建成中发挥着重要作用[27-28]。通过系统进化分析,结合CESA磷酸化蛋白组学数据的收集和整理[34],本研究分析鉴定了CESA磷酸化位点在作物CESA家族中的保守性。结果表明,作物的初生壁CESA1、CESA3和CESA6-like家族的一些磷酸化位点在拟南芥CESA家族中以及苔藓和石松CESA家族中均高度保守,表明这些CESAs位点的磷酸化修饰可能是作物初生壁纤维素合成的一种相对保守和具有重要功能的分子调控机制。CESAs的N端的锌指结构域已被认定参与介导蛋白和蛋白的相互作用。例如,水稻CESA7锌指结构域的一个错义突变影响植物生长和纤维素的生物合成,表明该结构域对纤维素合酶复合体的结构和功能起重要作用[35]。CESAs的HVR1结构域以及NT结构域均靠近锌指结构域,可能对纤维素合酶复合体内蛋白互作有重要意义[36-37]。HVR1位点结构域以及NT结构域的磷酸化可能通过影响CESA复合体内蛋白互作,参与调控微管引导的纤维素合成与沉积[27-28]。
棉花CESA1中央结构域的计算机结构预测结果表明,在CESA装配成大的复合体时需要HVR2结构域的参与[38]。HVR2结构域的磷酸化可能是纤维素合酶复合体结构与功能的另一种调控机制[27-28]。模拟磷酸化和模拟去磷酸化突变研究结果表明,与CESA1和CESA3的HVR1结构域的磷酸化位点突变体的表型相似[27-28],CESA1的S686和S688位点的磷酸化也参与调控细胞的各向异性生长,说明位于HVR1和HVR2区域的磷酸化位点在调控纤维素生物合成的过程中有类似的功能[27-28],它们的调控功能是否有差异还有待进一步研究。
初生壁CESA基因在不同组织中的转录表达水平相对较高,且差异不大,被认为是管家(house-keeping)基因[10]。与初生壁的纤维素合成不同,转录水平调控对于次生壁的纤维素合成可能具有更为重要的作用[17]。参与次生壁纤维素合成的CESAs也有多个磷酸化位点[22-26],作物的CESA4家族的部分磷酸化位点在拟南芥CESA4家族以及苔藓和石松CESA家族中也具有高度保守性,表明除了转录水平调控外,次生壁CESA的磷酸化修饰可能同时是次生壁纤维素合成的重要分子调控机制。进一步研究CESA磷酸化在农林作物中的调控功能和机制,对于了解作物的生长发育和逆境响应以及提高作物产量,都具有非常重要的科学理论意义。
注:通过MUSCLE软件对多序列进行在线比对整合构树,圆形有根树由ITOL软件展示。CESA家族蛋白序列被分成多个分支,相当于拟南芥初生壁的CESA1、CESA3、 CESA6和次生壁的CESA4、CESA7、 CESA8。
图3陆生植物纤维素合酶的系统发育树
Fig.3 Phylogeny of the cellulose synthase family in land plants
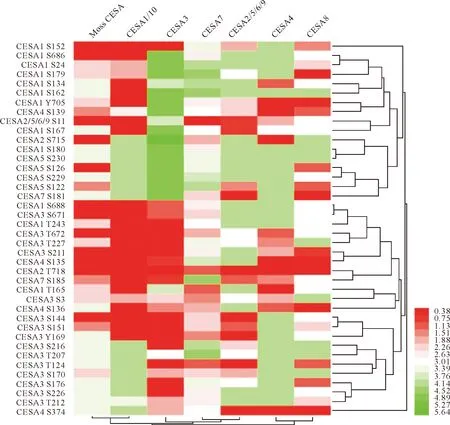
注:表2中拟南芥磷酸化位点在CESA的每个分支都计算了丝氨酸/苏氨酸的保守比例。通过Heml1.0对该比例进行标准化并绘制热图。
[1] NOBLES D R,ROMANOVICZ D K,BROWN R M.Cellulose in cyanobacteria,origin of vascular plant cellulose synthase [J].Plant Physiology,2001,127(2):529-42.
[2] NAKASHIMA K,YAMADA L,SATOU Y,etal.The evolutionary origin of animal cellulose synthase [J].Development Genes and Evolution,2004,214(2):81-88.
[3] ROBERTS A W,BUSHOVEN J T.The cellulose synthase (CESA) gene superfamily of the mossPhyscomitrellapatens[J].Plant Molecular Biology,2007,63(2):207-19.
[4] CARROLL A,SOMERVILLE C.Cellulosic biofuels[J].Annual Review of Plant Biology,2009,60(1):165-182.
[5] TILMAN D,SOCOLOW R,FOLEY J A,etal.Beneficial biofuels-the food,energy,and environment trilemma [J].Science,2009,325(5938):270-271.
[6] PAULY M,KEEGSTRA K,SONNEWALD U,etal.Plant cell wall polymers as precursors for biofuels [J].Current Opinion in Plant Biology,2010,13(3):305-311
[7] YOUNGS H,SOMERVILLE C.Development of feedstocks for cellulosic biofuels [J].F1000 Biology Reports,2012,4(1):10.
[8] 王国霞,耿晓东.秋冬季节泡桐顶芽形态及显微结构变化分析 [J].西北林学院学报,2017,32 (1):165-171.
WANG G X,GENG X D.Morphology and microstructure changes of terminal buds ofPaulowniain autumn and winter[J].Journal of Northwest Forestry University,2017,32(1):165-171.(in Chinese)
[9] 唐国建,杨金梅,王曙光,等.云龙箭竹纤维形态、化学成分及用作造纸原料可行性研究 [J].西北林学院学报,2015,30 (4):240-245.
TANG G J,YANG J M,WANG S G,etal.Fiber morphologies,chemical components ofFargesiapapyriferaand feasibility for paper making[J].Joural of Northwest Forestry University,2015,30 (4):240-245.(in Chinese)
[10] SOMERVILLE C.Cellulose synthesis in higher plants [J].Annual Review of Cell & Developmental Biology,2006,22(1):53-78.
[11] SAXENA I M,BROWN R M.Cellulose biosynthesis:current views and evolving concepts [J].Annals of Botany,2005,96(1):9-21.
[12] ATANASSOV I I,PITTMAN J K,TURNER S R.Elucidating the mechanisms of assembly and subunit interaction of the cellulose synthase complex ofArabidopsissecondary cell walls [J].Journal of Biological Chemistry,2009,284(6):3833-41.
[13] JR H J,HAMMUDI M B,TIEN M.The Arabidopsis cellulose synthase complex:a proposed hexamer of CESA trimers in an equimolar stoichiometry [J].Plant Cell,2014,26(12):4834-42.
[14] DESPREZ T,JURANIEC M,CROWELL E F,etal.Organization of cellulose synthase complexes involved in primary cell wall synthesis in Arabidopsis thaliana [J].Proceedings of the National Academy of Sciences of the United States of America,2007,104(39):15572-15577.
[15] PERSSON S,PAREDEZ A,CARROLL A,etal.Genetic evidence for three unique components in primary cell-wall cellulose synthase complexes in Arabidopsis [J].Proceedings of the National Academy of Sciences of the United States of America,2007,104(39):15566-15570.
[16] PAREDEZ A R,SOMERVILLE C R,EHRHARDT D W.Visualization of cellulose synthase demonstrates functional association with microtubules [J].Science,2006,312(5779):1491-5.
[17] GU Y,KAPLINSKY N,BRINGMANN M,etal.Identification of a cellulose synthase-associated protein required for cellulose biosynthesis [J].Proceedings of the National Academy of Sciences of the United States of America,2010,107(29):12866-71.
[18] ENDLER A,KESTEN C,SCHNEIDER R,etal.A mechanism for sustained cellulose synthesis during salt stress[J].Cell,2015,162(6):1353-64.
[19] KUMAR M,TURNER S.Plant cellulose synthesis:CESA proteins crossing kingdoms[J].Phytochemistry,2015,112(1):91-9.
[20] CARROLL A,SPECHT C D.Understanding plant cellulose synthases through a comprehensive investigation of the cellulose synthase family sequences[J].Front Plant Sci.,2011,2(5):5-11
[21] HUNTER T.Why nature chose phosphate to modify proteins [J].Phil.Trans.R.Soc.B,2012,367(1602):2513-2516.
[22] NÜHSE T S,STENSBALLE A,JENSEN O N,etal.Phosphoproteomics of theArabidopsisplasma membrane and a new phosphorylation site database [J].Plant Cell,2004,16(9):2394-405.
[23] GRIMSRUD P A,OS D D,WENGER C D,etal.Large-scale phosphoprotein analysis in medicago truncatula roots provides insight into in vivo kinase activity in legumes [J].Plant Physiology,2010,152(1):19-28.
[24] FACETTE M R,SHEN Z,BJÖRNSDTTIR F R,etal.Parallel proteomic and phosphoproteomic analyses of successive stages of maize leaf development [J].Plant Cell,2013,25(8):2798-812.
[25] XUE L,WANG P,WANG L,etal.Quantitative measurement of phosphoproteome response to osmotic stress in arabidopsis based on Library-Assisted eXtracted Ion Chromatogram (LAXIC) [J].Molecular & Cellular Proteomics Mcp,2013,12(8):2354-69.
[26] ROITINGER E,HOFER M,KÖCHER T,etal.Quantitative phosphoproteomics of the ataxia telangiectasia-mutated (ATM) and ataxia telangiectasia-mutated and rad3-related (ATR) dependent DNA damage response in Arabidopsis thaliana [J].Molecular & Cellular Proteomics Mcp,2015,14(3):556-571.
[27] CHEN S,SOMERVILLE C R.Mutations of cellulose synthase (CESA1) phosphorylation sites modulate anisotropic cell expansion and bidirectional mobility of cellulose synthase [J].Proceedings of the National Academy of Sciences of the United States of America,2010,107(40):17188-93.
[28] CHEN S,JIA H,ZHAO H,etal.Anisotropic cell expansion is affected through the bidirectional mobility of cellulose synthase complexes and phosphorylation at two critical residues on CESA3 [J].Plant Physiology,2016,171(1):242-250.
[29] BISCHOFF V,DESPREZ T,MOUILLE G,etal.Phytochrome regulation of cellulose synthesis in [J].Current Biology Cb,2011,21(21):1822-1827.
[30] EDGAR R C.Muscle:multiple sequence alignment with high accuracy and high throughput [J].Nucleic Acids Research,2004,32(5):1792-1797.
[31] LETUNIC I,BORK P.Interactive tree of life v2:online annotation and display of phylogenetic trees made easy [J].Nucleic Acids Research,2011,39:475-478.
[32] TAMURA K,STECHER G,PETERSON D,etal.Mega6:molecular evolutionary genetics analysis version 6.0 [J].Molecular Biology & Evolution,2013,30(12):2725-2729.
[33] DENG W,WANG Y,LIU Z,etal.HemI:a toolkit for illustrating heatmaps [J].Plos One,2013,9(11):111988.
[34] VAN WIJK K J,FRISO G,WALTHER D,etal.Meta-analysis of arabidopsis thaliana phospho-proteomics data reveals compartmentalization of phosphorylation motifs [J].Plant Cell,2014,26(6):2367-2389.
[35] WANG D,QIN Y,FANG J,etal.A Missense mutation in the zinc finger domain of OsCESA7 deleteriously affects cellulose biosynthesis and plant growth in rice [J].Plos One,2016,11(4):153993.
[36] Timmers J,Vernhettes S,Desprez T,etal.Interactions between membrane-bound cellulose synthases involved in the synthesis of the secondary cell wall [J].Febs Letters,2009,583(6):978-982.
[37] VANDAVASI V G,PUTNAM D K,ZHANG Q,etal.A structural study of CESA1 catalytic domain ofArabidopsiscellulose synthesis complex:evidence for CESA trimers [J].Plant Physiology,2015,170(1):123.-135
[38] SETHAPHONG L,HAIGLER C H,KUBICKI J D,etal.Tertiary model of a plant cellulose synthase [J].Proceedings of the National Academy of Sciences of the United States of America,2013,110(18):7512-17.

