全基因组关联定位籼稻种质资源外观和加工品质QTL
方雅洁 朱亚军 吴志超 陈 凯 申聪聪 石英尧徐建龙,,4,*
全基因组关联定位籼稻种质资源外观和加工品质QTL
方雅洁1,2朱亚军3吴志超2陈 凯3申聪聪3石英尧1,*徐建龙2,3,4,*
1安徽农业大学, 安徽合肥 230036;2中国农业科学院作物科学研究所, 北京 100081;3中国农业科学院农业基因组研究所, 广东深圳 518210;4中国农业科学院深圳生物育种创新研究院, 广东深圳 518210
以400份籼稻变异丰富的种质资源材料在2个环境下考察外观与加工品质性状表型, 利用404k高密度SNP基因型进行全基因组关联分析, 挖掘影响外观与加工品质性状的重要位点。结果表明, 粒形和籼米的外观和加工品质密切相关, 垩白性状降低稻米的精米率和整精米率, 除粒形外, 籼稻的外观和加工品质明显受到环境影响。全基因组关联分析共鉴定到39个QTL, 其中17个为新位点, 6个新位点(、、、和)在2个环境下均被检测到, 暗示这6个位点在水稻外观与加工品质上可能是不依赖于环境稳定表达的QTL。此外, 21个控制稻米外观品质的位点很可能具一因多效。结合前人研究结果, 我们推论、和在籼稻外观和加工品质性状上扮演着关键性角色。研究结果为克隆新的外观与加工品质基因, 以及通过分子手段加速培育优质高产籼稻新品种提供了指导信息。
籼稻种质资源; 外观与加工品质; 全基因组关联分析; 数量性状座位
自从20世纪60年代初水稻矮秆基因利用和70年代末杂交水稻的推广以来, 大幅度提高了水稻的产量。但是随着改革开放我国经济快速发展和人民生活水平不断提高, 国内外市场竞争日趋激烈, 对水稻品质的要求也逐渐提高, 育种家们开始将超高产与优质育种结合起来。
稻米品质包括外观品质、加工品质、蒸煮品质和营养品质, 其中外观与加工品质是影响稻米市场销售的重要因素。稻米外观品质涉及米粒的形状、大小、透明度和垩白(又称心白、腹白)大小等, 一般通过粒长、粒宽、长宽比、垩白度、垩白粒率等指标来描述。加工碾磨品质主要包括糙米率、精米率和整精米率3个指标。其中整精米率和垩白粒率是优质稻育种中最重要也是最难改良的2个指标[1-3]。
稻米品质性状大多受多基因控制, 以往利用双亲本衍生群体定位了大量影响品质性状的QTL[1,4-6]。迄今, 在水稻粒形方面有[7]、[8-9]、[10]、[11-12]、[13]等基因被克隆,[14]和[15]被精细定位。对于垩白性状, 已有[16]、[17]、[18]、[19]等基因被克隆,被精细定位[20]。最近, Ren等[21]将控制糙米率的基因精细定位到第10染色体上39.5 kb区间。许多粒形基因存在一因多效作用, 如和是2个主效的控制粒宽和粒重的基因, 在增加粒重的同时也增加了籽粒垩白[7,22]。
起源于不同地域的种质资源中往往蕴藏着大量有利等位基因[23], 基于变异丰富的种质资源的全基因组关联分析(genome-wide association study, GWAS)是当前有利基因挖掘方面的热门方法, 其以群体的LD (linkage disequilibrium)和基因组中数以百万计的SNP (single nucleotide polymorphism)为基础, 进行表型与基因型关联分析, 定位与目标性状相关的基因组区域, 不但可以挖掘目标位点的最优等位基因, 而且可以实现主效基因的精细定位[3,24]。迄今已有不少利用GWAS策略开展控制水稻复杂性状基因定位的报道[25], 但在稻米外观和加工品质性状上的研究尚不多。邱先进等[26]利用籼稻种质资源检测到42个和44个与垩白粒率和垩白度显著关联的位点, 其中2个性状分别有21个和19个位点在2个以上环境下同时被检测到。Qiu等[22]利用272份全球籼稻微核心种质检测到38个外观和加工品质相关的位点, 其中影响多个性状的位点()、()、()、()和在多个环境下被定位到。Wang等[3]利用27k SNP对258份种质资源材料进行GWAS分析, 检测到72个与加工质量、外观质量显著相关的QTL, 进一步用更高密度的SNP实现了3个粒形QTL的精细定位。
为了更有利地挖掘有利基因, 在充分考虑生育期和起源地多样化的基础上, 本研究从3000份水稻重测序项目(3k Rice Genome Project, 3k RGP)中选取400份遗传多样性丰富的籼稻材料, 在海南三亚、广东深圳2个环境下考察外观与加工品质性状表型, 结合404k高密度SNP标记, 通过GWAS方法挖掘影响水稻外观与加工品质的重要位点, 以期为水稻分子辅助选育优质品种克隆重要位点提供指导性信息。
1 材料与方法
1.1 试验材料
400份籼稻资源来自45个国家或地区, 包括中国128份(32%), 印度68份(17%), 东南亚89份(22%), 其他亚洲地区44份(11%), 欧洲、美洲、非洲和澳大利亚合计59份(15%), 未知来源的12份(3%)(图1)。
1.2 田间设计
分别在位于海南三亚的中国农业科学院作物科学研究所南繁基地(2015冬)和广东深圳的中国农业科学院深圳农业基因组研究所基地(2016夏)进行田间试验。采用2重复随机区组设计。每个重复中, 每个材料种植3行, 每行10株, 株行距统一为20 cm × 25 cm, 其中中间行的中间8株设定为表型测定目标单株。三亚播种日期为2015年11月30日, 抽穗期变化范围为66~90 d, 平均抽穗期为78 d, 深圳播种日期为2016年6月15日, 抽穗期变化范围为75~101 d, 平均抽穗期为87 d。常规田间管理, 注意防倒伏。
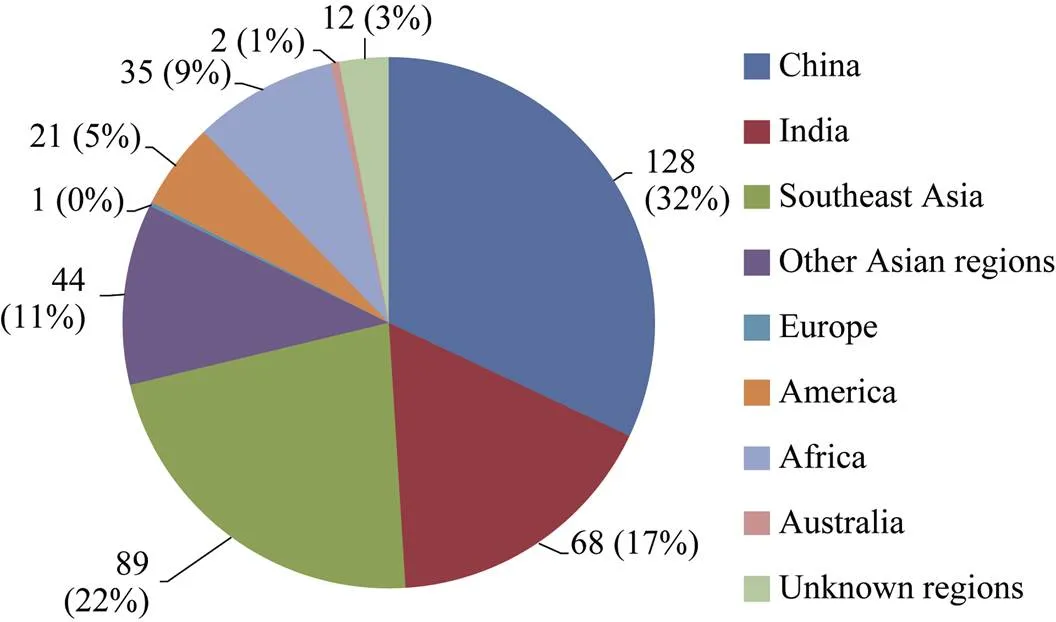
图1 400份籼稻种质资源的分布
1.3 表型鉴定
抽穗期前后去除个别异常或病弱单株。待种子成熟后, 混收中间行的中间8株。收获后自然晾干, 室温储藏3个月后按照国标优质稻谷GB/T17891- 1999标准考察其品质相关性状表型数据。先取20 g干净饱满稻谷利用砻谷机(JLGL-45)研磨成糙米, 再用Pearlist型实验用精米机(日本Kett公司)碾磨成精米, 依次记录各测定样本的糙米、精米和整精米重量, 然后分别计算糙米率(brown rice rate, BRR, %)、精米率(milled rice rate, MRR, %)、整精米率(head milled rice rate, HMRR, %), 最后利用大米外观品质检测分析系统(万深SC-E型)检测每个品种的粒长(grain length, GL, mm)、粒宽(grain width, GW, mm)、长宽比(grain length-width ratio, GLWR)、垩白粒率(percentage of grain with chalkiness, PGWC, %)、垩白度(degree of endosperm chalkiness, DEC, %)。以2个重复的平均值作为单个材料相应性状的表型值。
1.4 SNP筛选
3K RGP项目原始测序数据已在GigaScience公开[27]。所选400份材料平均测序深度约15×。首先使用BWA软件“mem”算法将测序数据与参考基因组日本晴IRGSP 1.0比对, 使用samtools、GATK、Picardtools等对短序列比对结果进行处理得到单核苷酸多态性(SNP)数据, 进而分别剔除稀有等位基因标记(基因型频率<5%)、缺失标记数多于20%样本数的标记和相关性大于>0.95的标记, 最终获得高质量高密度的404k SNP标记[28]。
1.5 数据分析
1.5.1 表型数据分析 用Microsoft Excel 2007整理数据, 以2个重复的平均值作为相应性状的表型值计算单个环境下的群体变幅和变异系数, 用SAS 9.2软件中的Corr程序分析同一环境下不同性状间的相关性和同一性状不同环境下的相关性。
1.5.2 群体结构和连锁不平衡分析 使用plink软件“--indep-pairwise 50 10 0.5”参数过滤掉临近的连锁较强的SNP后, 再使用EIGENSOFT软件进行主成分分析(Principal Component Analysis, PCA), 以PC1和PC2揭示群体的遗传结构。使用TASSEL软件(5.2.30版本)构建亲缘矩阵(kinship)。使用缺失率<20%, MAF>0.1的SNP数据, 利用plink v1.90软件“--r2--ld-window-kb 1000--ld-window 99999--ld- window-r2 0”参数计算全基因组水平1 Mbp区域内SNP之间的2, 进行连锁不平衡衰减(LD decay)分析。选用极显著(<0.001)的位点对, 绘制LD衰减散点图, 并绘制拟合曲线, 以LD衰减至起始值一半时所对应的物理距离作为LD衰减距离[23]。
1.6 关联分析定位QTL
使用缺失率<20%, MAF>0.05的404 k高质量高密度SNP基因型数据, 应用TASSEL version 5.2.30软件中的混合线性模型(Mixed Linear Model, MLM)结合kinship矩阵与PCA对2个环境8个性状进行全基因组关联分析。选择-value<1E–5或<1E–6为关联分析显著性阈值, 利用R语言中的qqman包绘制曼哈顿图和QQ plot图展示关联分析的结果。
2 结果与分析
2.1 供试材料在2个环境下加工与外观品质性状的表现
由表1可知, BRR、MRR和HMRR在广东深圳的变异范围稍大于海南三亚环境, PGWC和DEC在深圳的变异范围稍小于三亚环境, GL、GW和GLWR在2个环境下变异范围相对稳定。相关性分析也表明, GL、GW、GLWR、PGWC和DEC两个环境下的相关系数分别为0.93、0.89、0.88、0.61和0.63, 均为极显著相关, 而BRR、MRR、HMRR两个环境下的相关系数较低, 仅为0.18、0.11和0.13。这表明, 粒形性状遗传稳定受环境影响小, 而BRR、MRR、HMRR、PGWC和DEC易受种植环境的影响。
2.2 供试材料不同性状间的相关性
粒形性状GL与GW之间呈极显著负相关, 加工品质性状BRR、MRR和HMRR两两之间呈极显著正相关, 外观品质PGWC与DEC呈显著正相关(表2)。粒形与垩白性状极显著相关, 即粒长增长能显著降低籽粒垩白度和垩白率, 而粒宽则增加籽粒垩白度和垩白率(表2)。GL与HMRR显著或极限著负相关, 但与BRR和MRR没有相关性, 而粒宽与加工品质性状的相关性较为复杂, 明显受环境的影响(表2)。2个环境下垩白性状与BRR没有相关性, 但与MRR和HMRR显著或极显著负相关, 且受到环境的影响(表2)。
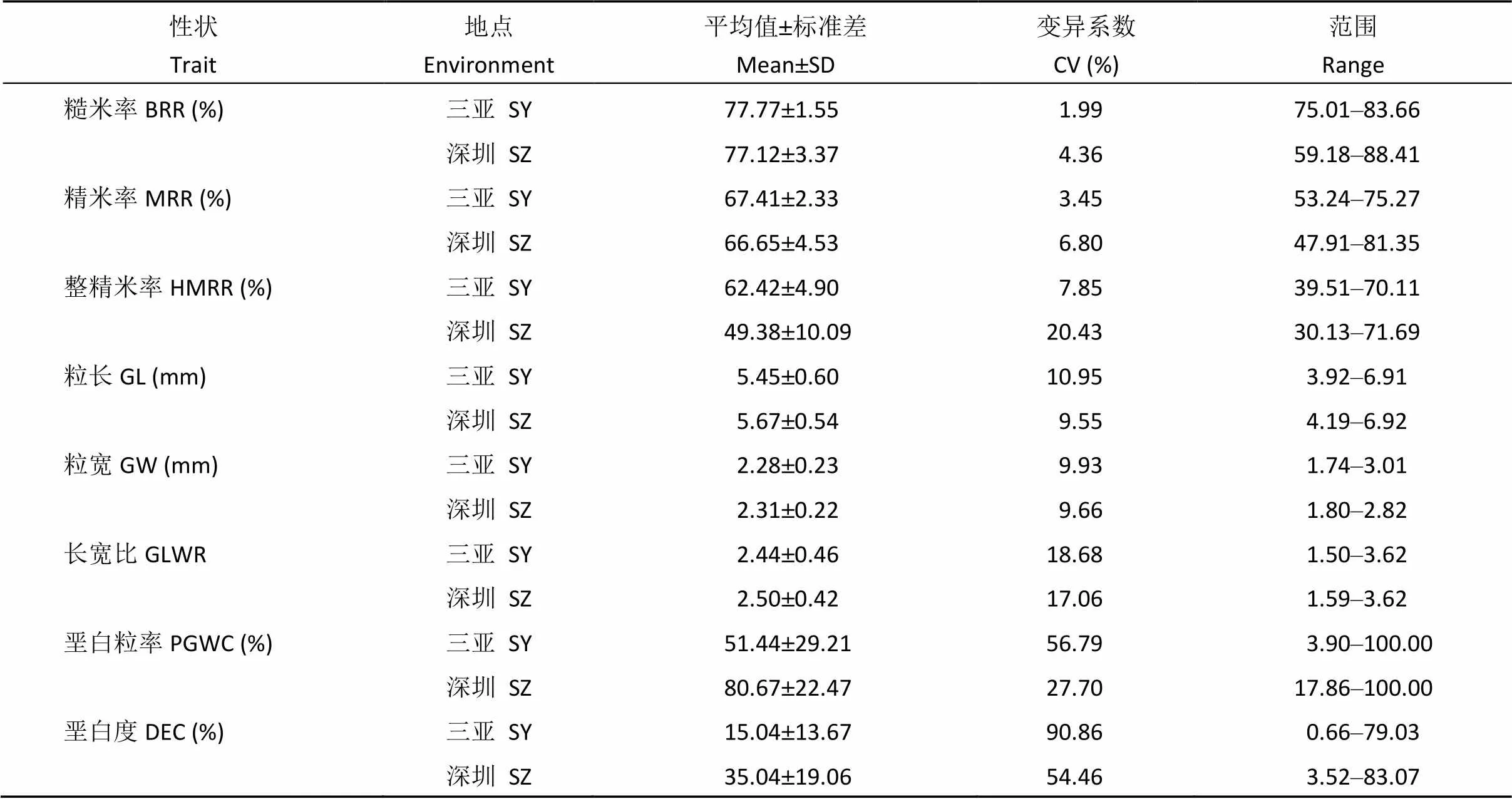
表1 籼稻种质资源在2个环境下的加工和外观品质性状表现
BRR: brown rice rate; MRR: milled rice rate; HMRR: head milled rice rate; GL: grain length; GW: grain width; GLWR: grain length-width ratio; PGWC: percentage of grain with chalkiness; DEC: degree of endosperm chalkiness.
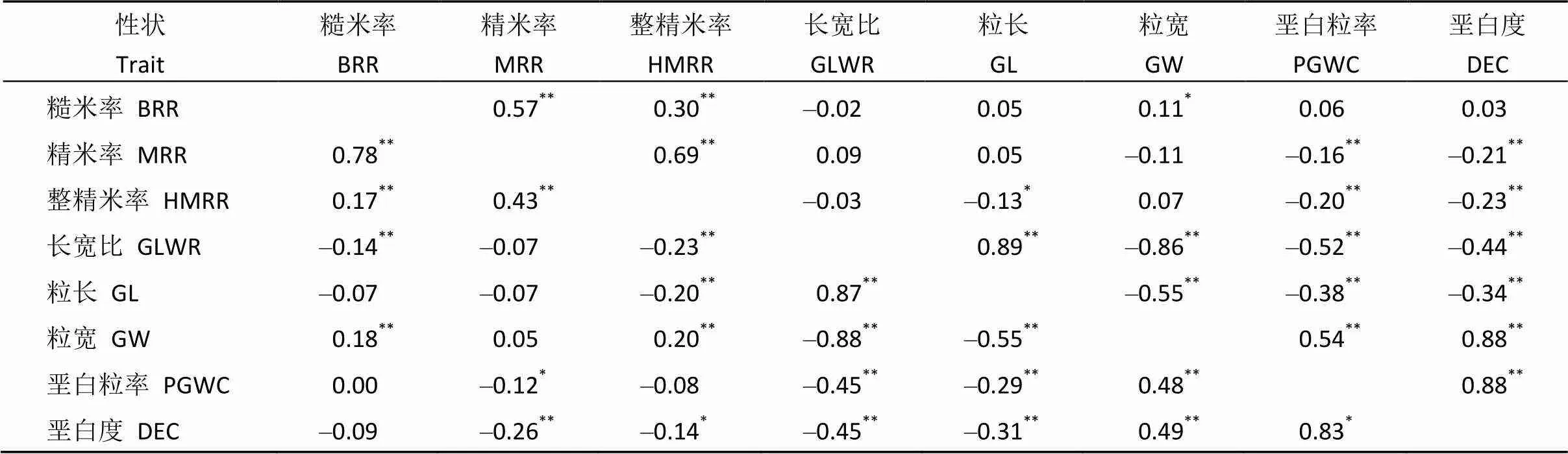
表2 籼稻种质资源外观和加工品质性状在2个环境下的相关系数
左下角为2015年冬季海南三亚数据, 右上角为2016年夏季广东深圳数据。*和**表示在5%和1%水平上差异显著。
Data in below left of diagonal are from winter season in Sanya of Hainan in 2015 while ones in upper right of diagonal are from summer season in Shenzhen of Guangdong in 2016. BRR: brown rice rate; MRR: milled rice rate; HMRR: head milled rice rate; GL: grain length; GW: grain width; GLWR: grain length-width ratio; PGWC: percentage of grain with chalkiness; DEC: degree of endosperm chalkiness.*and**denote significant difference at the 5% and 1% probability levels; respectively.
2.3 LD和连锁不平衡分析
群体的PC1和PC2分别是24.3%和14.4% (图2), 表现出轻微的群体结构。连锁不平衡分析显示群体的2临界值为0.14, 表现出随物理距离(kb)增加而下降的趋势(图3)。以连锁不平衡(LD)降低至原始值的50% (2>0.2)作为其阈值, 群体的物理距离为98 kb。
2.4 全基因组关联分析
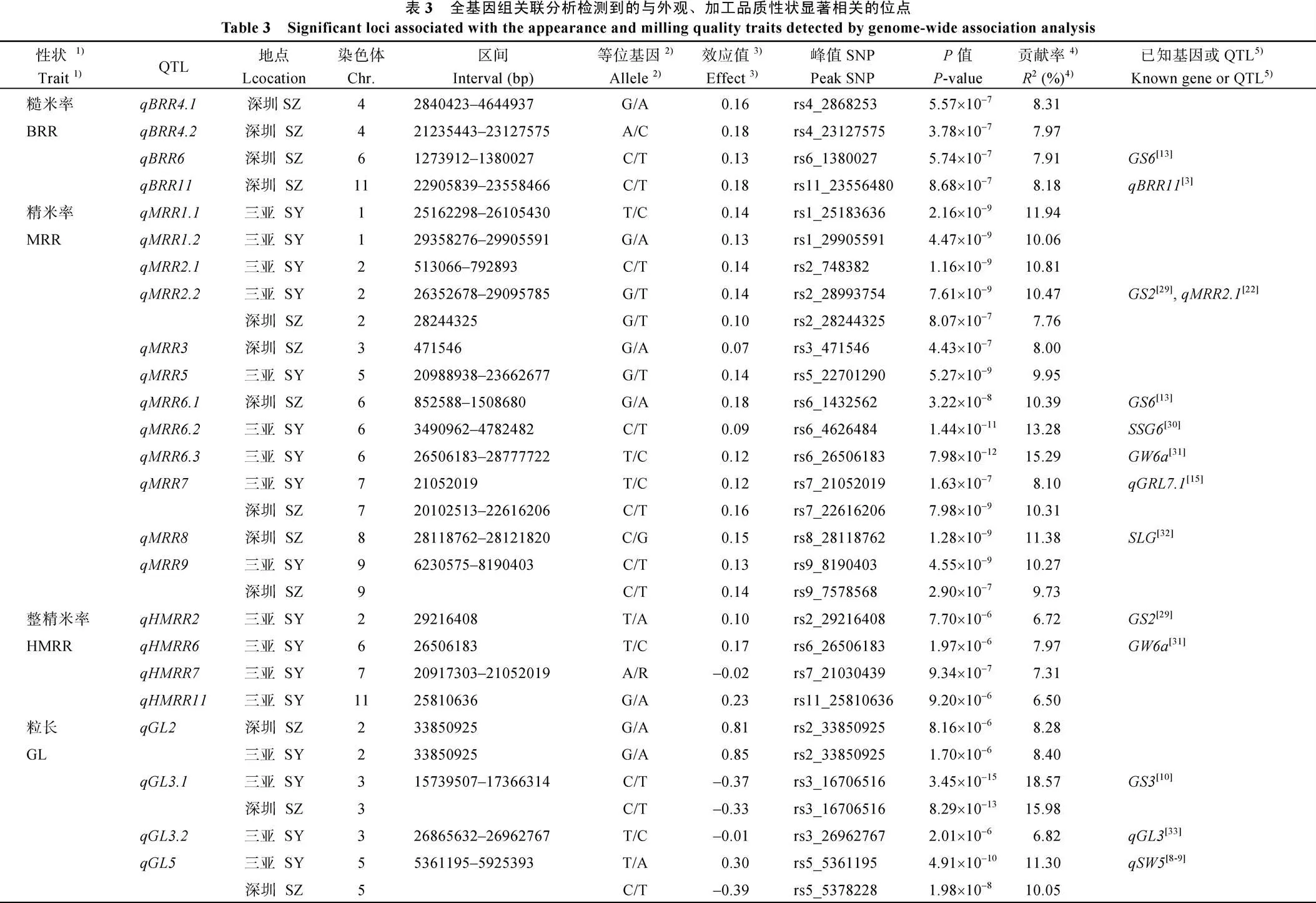
使用TASSEL 5.2.30中的MLM模型(考虑群体结构和kinship矩阵)对404k SNP标记与表型值进行全基因组关联分析。在2个环境中, 总共检测到39个与外观和加工品质相关的QTL, 其中控制BRR、MRR、HMRR、GL、GW、GLWR、PGWC和DEC位点分别为4、12、4、5、4、6、2和2个, 分布在除第12染色体外的其余11条染色体上(表3和图4)。
对于粒形性状, 深圳和三亚分别定位到12个和15个位点, 其中, 控制GL的4个位点(和)、控制GW的2个位点(和)和控制GLWR的6个位点(、、、、和)在2个环境下均被定位到(表3和图4)。这12个位点中,、、、和为主效位点, 2个环境下的贡献率为9.04%~20.91%。、、和两个环境下的平均贡献率分别为8.30%、6.41%、7.06%、6.48%、6.49%和6.60%。在上述控制粒形的位点中,和定位区间重叠,、和定位区间重叠,定位区间重叠,和定位区间重叠,和定位区间重叠, 暗示控制这些性状的重叠位点可能由相同的基因控制。
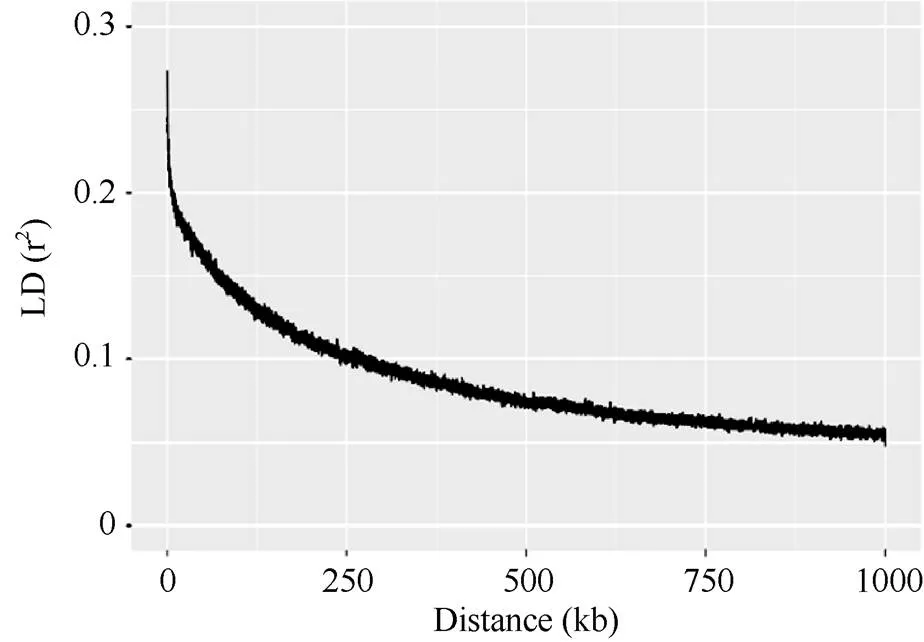
图2 群体的LD衰减图
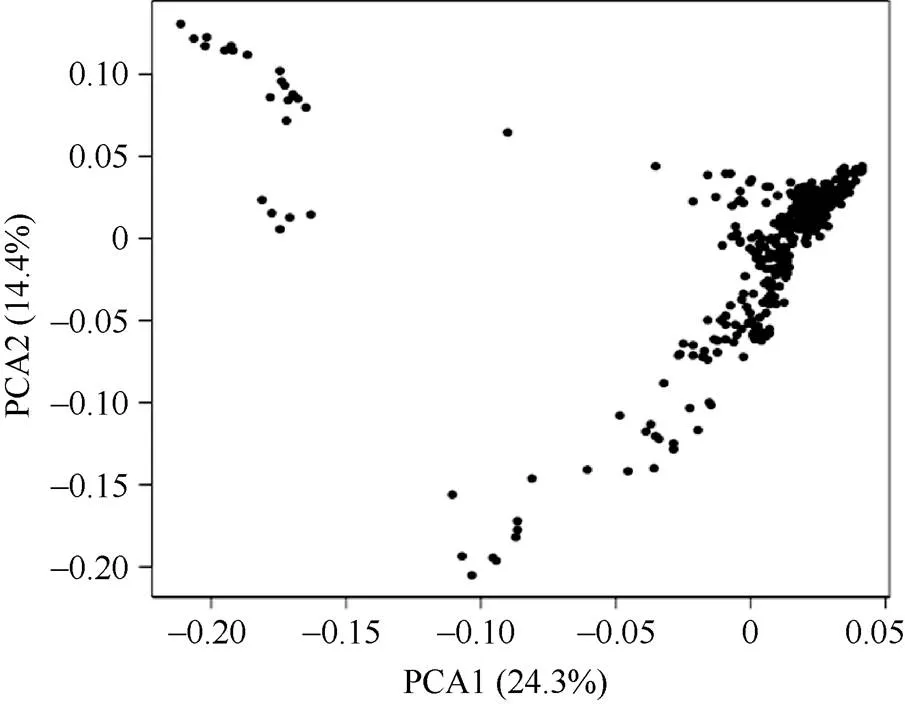
图3 400份种质的主成分分析
群体最大变异的2个主成分分别图示于X轴和Y轴。
The two principal components (PCs) capturing the largest amount of variation are shown as X- and Y-axis, respectively.
2个环境中共检测到20个控制加工品质性状BRR、MRR和HMRR的位点, 分布于除第10和12外的其余染色体上, 其中控制BRR的位点4个, 控制MRR位点12个, 控制HMRR位点4个(表3和图4)。在这些位点中,、和在2个环境中均被定位到, 分别位于第2、第7和第9染色体上, 2个环境下的平均贡献率分别为9.12%、9.21%和10.00%。在仅1个环境下检测到的17位点中,、、、、、和等7个位点的贡献率超过10% (表3和图4)。对于不同加工品质性状而言, 位于第6染色体短臂的和被定位到重叠区间, 位于第6染色体长臂的和被定位到重叠区间, 位于第2染色体长臂的和被定位到相邻位点, 位于第7染色体长臂的和被定位到重叠区间, 暗示控制这些性状的重叠位点可能存在基因的一因多效作用。
影响稻米垩白的QTL在2个环境中检测到4个。其中控制PGWC的2个位点和分别位于第5染色体短臂和第11染色体长臂, 控制DEC的2个位点和分别位于第2染色体长臂和第5染色体短臂。在这4个位点中,和在深圳和三亚2个环境下均被检测到, 2个环境下的平均贡献率分别为9.24%和7.63%, 且这2个QTL位于重叠区间。实际上, 在所有定位的控制外观和加工品质相关的39个位点中, 位于第5染色体短臂5 007 414~5 925 393 bp最大区间内包含5个控制粒形和垩白相关性状的位点(、、、和), 表明该区间内存在一个效应很大的一因多效基因, 是影响粒形和外观品质的重要基因组区域(表3和图4)。
3 讨论
3.1 稻米品质相关性状间的复杂相关性
不同地域的人群对稻米品质有不同的喜好, 但稻米品质却是受多基因控制的数量性状, 明显受生长环境的影响[34-35]。本研究表明, 3个加工品质指标(糙米率、精米率和整精米率)之间在不同环境下的相关性稳定, 彼此间均存在显著正相关; 3个粒形性状(粒长、粒宽和长宽比)之间的相关性也稳定, 粒长和长宽比存在显著正相关, 但两者与粒宽均为显著负相关; 2个重要的外观品质(垩白粒率和垩白度)之间的显著正相关在不同环境下也很稳定。上述同一类型性状之间稳定的相关性在不同的研究中均有报道,而且受环境的影响较小[1-2,22,36]。
各性状缩写见表1; SY表示海南三亚环境, SZ表示广东深圳环境。
Abbreviations of traits correspond with those given in Table 1. SY stands for Sanya environment whereas SZ for Shenzhen environment.
不同类型性状间的相关表现得比较复杂。一般而言, 长粒有助于降低垩白但增加了整精米率的损失度, 宽粒则反之[1-3]。本研究中观察到3个粒形性状与3个加工品质性状之间相关不紧密, 虽然在三亚环境下整精米率和糙米率与不同粒形性状存在显著相关, 但相关系数偏小, 而在深圳条件下基本上不存在相关性。粒形性状与外观品质之间的相关性相对稳定, 而且不受环境条件影响, 表现为长粒减小垩白粒率和垩白度, 粒宽则增加这2个垩白相关性状。3个加工品质性状2个外观品质性状之间的相关性在三亚和深圳2个环境表现一致, 即糙米率与垩白粒率和垩白度不相关, 但精米率和整精米率与垩白粒率和垩白度均显著负相关。由于本研究的种质资源材料来源相当广泛, 由此得出的粒形与加工品质之间的相关性与以往报道的结果[2,37]不完全一致。从而暗示我们, 粒形与不同加工品质之间的相关性是多样化的, 而且受环境影响较大, 不同粒形都有可能存在精米率和整精米率高的材料。因此, 通过选择适当的亲本配组, 在后代群体中完全有可能选育到长粒形但精米率和整精米率高的个体。
3.2 QTL定位结果的比较
本研究在2个环境下共检测到影响品质相关性状的39个QTL, 其中17个位点在2个环境下均被检测到。将已报道的外观和加工品质QTL比对到日本晴基因组物理图谱(IRGSP 1.0)上, 以置信区间重叠或位置邻近(500 kb范围内)视为相同QTL, 我们发现22个位点与已克隆或定位的控制稻米粒形和垩白的基因定位在同一或非常临近的区间, 而、、、、和六个QTL为新位点且在2个环境下均被检测到, 同时我们也发现21个位点其定位区间存在交叉, 如和区间重叠,、、、、五个QTL位于包含的热点区间内等。是一个控制水稻籽粒宽度的主效基因, 位于第5染色体短臂5 362 625~5 370 506 bp (IRGSP 1.0)区间, 其在籼稻的等位基因减小粒形而在日本晴的等位基因增加粒宽[8]。有报道表明,同时控制GL、GLWR以及PGWC和DEC[3,9,22], 暗示这几个位点可能与一因多效。最近, 位于座位上的一个控制粒形大小的等位基因[38]被克隆。显而易见, 包含和的第5染色体热点区域(5 007 414~5 925 393 bp)在籼稻粒形大小和垩白高低形成中起关键作用。此外, 鉴定到和所在区间包括已克隆粒形基因型[29]和未克隆QTL[22];1和所在区间包括已克隆粒形[10]和未克隆QTL[3,22];和所在区间包括已克隆粒形[13];和所在区间包括已克隆粒形[31]等。除了上述已有基因被克隆的染色体区段外, 我们也发现精米率位点与整精米率位点定位区间存在重叠。上述影响稻米品质性状的主效QTL与以往在不同群体和环境下检测到的基因或QTL定位在同一区间, 表明这些QTL具有遗传背景和环境的独立性, 具有育种应用价值, 其与已知基因的关系还有待精细定位后方能知晓。不可否认, 对于垩白性状而言, 本研究鉴定到的位点偏少, 如已克隆基因[16]、[17]未被检测到,[20]所在区间关联SNP有峰值但未能超过-value<1E–5 (图4)。一方面这可能与数量性状遗传易受环境影响有关, 也可能与所采用的试验材料不同有关。如我们曾采用两套不同的籼稻种质资源群体开展籼稻外观和加工品质有利基因挖掘试验[3,22], 同样未检测到[16]和[17], 仅[20]被Wang等[3]定位在相似区间。有意思的是, 在上述2个自然群体均检测到和, 且发现它们调控多个外观和加工品质性状, 这和本研究的结果非常相似。因此, 我们推断和所在热点区域以及可能在籼稻稻米外观和加工品质中扮演者关键性角色。
3.3 对稻米品种育种的启示
本研究结果表明, 粒形与不同加工品质性状之间的相关性呈现多样化, 而且这种相关性受环境影响, 不同粒形都有可能存在精米率和整精米率高的材料。因此, 通过选择适当的亲本配组, 在后代群体中完全有可能选育到粒形长且精米率和整精米率高的个体。
当前, 遗传育种领域的科技工作者开始对效应较小或微效等级的位点或基因表现出浓厚兴趣, 认为克隆并功能上解析该类型位点或基因能为分子设计育种或全基因组选择育种提供更为充分的条件[39]。本研究除定位到与以往克隆基因或定位的QTL位于同一或非常临近区间的QTL外, 还发现了17个控制稻米外观和加工品质的新位点, 其中6个在2个环境下同时被检测到, 包括、、、、和, 在2个环境下的平均贡献率从6.48%到10.00%不等, 多数属微效QTL, 但其对水稻外观与加工品质的影响不受或较少受环境条件的影响。因此, 通过不同位点有利等位基因的合理配置, 采用全基因组分子标记辅助选择技术可以实现品种外观和加工品质的分子改良。本研究结果为克隆新的控制外观与加工品质基因以及通过分子手段加速培育优质高产籼稻新品种提供了更多有价值的信息。
4 结论
籼稻的外观和加工品质性状之间存在不同程度的相关, 品质相关性状的表现及彼此间的相关性不同程度地受到环境的影响。关联分析共鉴定到39个影响粒形、加工和外观品质相关性状的QTL, 其中6个新位点(、、、、和)在2个环境下均被检测到, 另有22个位点与以往定位或克隆到的相关品质性状基因/QTL位于相同或邻近区间, 一因多效可能是这些重叠区间内相关品质性状的遗传基础。本研究结果为进一步克隆新的控制外观与加工品质基因以及通过分子手段加速培育优质高产籼稻新品种提供了有利信息。
致谢: 感谢本实验室曹国亮、朱双兵、金国啸、吴景林等同学帮助调查田间性状。
[1] 梅德勇, 朱玉君, 樊叶杨. 籼稻稻米碾磨与外观品质性状的QTL定位. 遗传, 2012, 34: 1591–1598 Mei D Y, Zhu Y J, Fan Y Y. Mapping QTL for rice milling and appearance quality traits inrice.(Beijing), 2012, 34: 1591–1598 (in Chinese with English abstract)
[2] 王丹英, 章秀福, 朱智伟, 陈能, 闵捷, 姚青, 严建立, 廖西元. 食用稻米品质性状间的相关性分析. 作物学报, 2005, 31: 1086–1091 Wang D Y, Zhang X F, Zhu Z W, Chen N, Min J, Yao Q, Yan J L, Liao X Y. Correlation analysis of rice grain quality characteristics., 2005, 31: 1086–1091 (in Chinese with English abstract)
[3] Wang X Q, Pang Y L, Wang C C, Chen K, Zhu Y J, Shen C C, Ali J, Xu J L, Li Z K. New candidate genes affecting rice grain appearance and milling quality detected by genome-wide and gene-based association analyses., 2017, 7: 1998
[4] Xu J L, Yu S B, Luo L J, Zhong D B, Sanchez A, Mei H W, Khush G S, Li Z K. Molecular dissection of the primary sink size and its related traits in rice., 2004, 123: 43–50
[5] 周立军, 江玲, 翟虎渠, 万建民. 水稻垩白的研究现状与改良策略. 遗传, 2009, 31: 563–572 Zhou L J, Jiang L, Zhai H Q, Wan J M. Current status and strategies for improvement of rice grain chalkiness.(Beijing), 2009, 31: 563–572 (in Chinese with English abstract)
[6] Qiu X J, Chen K, Lv W K, Qu X X, Zhu Y J, Xing D Y, Yang L W, Fan F J, Yang J, Xu J L. Examining two sets of introgression lines reveals background-independent and stably expressed QTL that improve grain appearance quality in rice (L.)., 2017, doi: 101007/s00122-017-2862-z
[7] Song X J, Huang W, Shi M, Zhu M Z, Lin H X. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase., 2007, 39: 623–630
[8] Shomura A, Izawa T, Ebana K, Ebitani T, Kanegae H, Konishi S, Yano M. Deletion in a gene associated with grain size increased yields during rice domestication., 2008, 40: 1023–1028
[9] Yan S, Zou G H, Li S J, Wang H, Liu H Q, Zhai G W, Guo P, Song H M, Yan C J, Tao Y Z. Seed size is determined by the combinations of the genes controlling different seed characteristics in rice., 2011, 123: 1173–1181
[10] Fan C C, Xing Y Z, Mao H L, Lu T T, Han B, Xu C G, Li X H, Zhang Q F., a major QTL for grain length and weight and minor QTL for grain width and thickness in rice, encodes a putative transmembrane protein.2006, 112: 1164–1171
[11] Wang Y X, Xiong G S, Hu J, Jiang L, Yu H, Xu J, Fang Y X, Zeng L J, Xu E B, Xu J, Ye W J, Meng X B, Liu R F, Chen H Q, Jing Y H, Wang Y H, Zhu X D, Li J Y, Qian Q. Copy number variation at thelocus contributes to grain size diversity in rice., 2015, 47: 944–948
[12] Wang S K, Li S, Liu Q, Wu K, Zhang J Q, Wang S S, Wang Y, Chen X B, Zhang Y, Gao C X, Wang F, Huang H X, Fu X D. Theregulatory module determines grain shape and simultaneously improves rice yield and grain quality., 2015, 47: 949–954
[13] Sun L J, Li X J, Fu Y C, Zhu Z G, Tan L B, Liu F X, Sun X Y, Sun X W, Sun C Q., a member of the GRAS gene family, negatively regulates grain size in rice., 2013, 55: 938–949
[14] Guo L B, Ma L L, Jiang H, Zeng D L, Hu J, Wu L W, Gao Z Y, Zhang G Z, Qian Q. Genetic analysis and fine mapping of two genes for grain shape and weight in rice., 2009, 51: 45–51
[15] Singh R, Singh A K, Sharma T R, Singh A, Singh N K. Fine mapping of grain length QTLs on chromosomes 1 and 7 in Basmati rice (L.)., 2012, 21: 157–166
[16] Li Y B, Fan C H, Xing Y Z, Yun P, Luo L J, Yan B, Peng B, Xie W B, Wang G W, Li X H, Xiao J H, Xu C G, He Y Q.encodes a vacuolar H(+)-translocating pyrophosphatase influencing grain chalkiness in rice., 2014, 46: 398–404
[17] Wang E T, Wang J J, Zhu X D, Hao W, Wang L Y, Li Q, Zhang L X, He W, Lu B R, Lin H X, Ma H, Zhang G Q, He Z H. Control of rice grain-filling and yield by a gene with a potential signature of domestication., 2008, 40: 1370–1374
[18] Zhang Y, Verhoeff N I, Chen Z, Chen S, Wang M, Zhu Z, Ouwerkerk P B F. Functions ofin regulation of., 2015, 89: 229–242
[19] Ryoo N, Yu C, Park C S, Baik M Y, Park I M, Cho M H, Bhoo S h, An G, Hahn T R, Jeon J S. Knockout of a starch synthase genecauses white-core floury endosperm in rice (L.)., 2007, 26: 1083–1095
[20] Zhou L J, Chen L M, Jiang L, Zhang W W, Liu L J, Liu X, Zhao Z G, Liu S J , Zhang L J, Wang J K, Wan J M. Fine mapping of the grain chalkiness QTLin rice (L.)., 2009, 118: 581–590
[21] Ren D Y, Rao Y H, Huang L C, Leng Y J, Hu J, Lu M, Zhang G H, Zhu L, Gao Z Y, Dong G J, Guo L B, Qian Q, Zeng D L. Fine mapping identifies a new QTL for brown rice rate in rice (L.)., 2016, 9: 4–14
[22] Qiu X J, Pang Y L, Yuan Z H, Xing D Y, Xu J L, Dingkuhn M, Li Z K, Ye G Y. Genome-wide association study of grain appearance and milling quality in a worldwide collection ofrice germplasm., 2015, 10: e0145577
[23] Zhao K Y, Tung C W, Eizenga G C, Wright M H, Ali M L, Price A H, Norton G J, Islam M R, Reynolds A, Mezey J, McClung A M, Bustamante C D, McCouch S R. Genome-wide association mapping reveals a rich genetic architecture of complex traits in., 2011, 2: 467–478
[24] 唐富福, 徐非非, 包劲松. 全基因组关联分析在水稻遗传育种中的应用. 核农学报, 2013, 27: 598–606 Tang F F, Xu F F, Bao J S. Application of genome-wide association analysis in rice., 2013, 27: 598–606 (in Chinese with English abstract)
[25] 侯青青, 司丽珍, 黄学辉, 韩斌. 水稻复杂性状研究的新途径: 水稻重要农艺性状全基因组关联分析. 生命科学, 2016, 28: 1250–1257 Hou Q Q, Si L Z, Huang X H, Han B. Progress on genome-wide association study of important agronomic traits in rice., 2016, 28: 1250–1257 (in Chinese with English abstract)
[26] 邱先进, 袁志华, 陈凯, 杜斌, 何文静, 杨隆维, 徐建龙, 邢丹英, 吕文恺. 用全因组关联分析解析籼稻垩白的遗传基础. 作物学报, 2015, 41: 1007–1016 Qiu X J, Yuan Z H, Chen K, Du B, He W J, Yang W L, Xu J L, Xing D Y, Lyu W K. Genetic dissection of grain chalkiness inmini-core germplasm using genome-wide association method., 2015, 41: 1007–1016 (in Chinese with English abstract)
[27] The 3,000 Rice Genomes Project. The 3,000 rice genomes project., 2014, 3: 7–13
[28] Alexandrov N, Tai S, Wang W S, Mansueto L, Palis K, Fuentes R R, Ulat V J, Chebotarov D, Zhang G Y, Li Z K. SNP-Seek database of SNPs derived from 3000 rice genomes., 2015, 43: D1023
[29] Hu J, Wang Y X, Fang Y X, Zeng L J, Xu J, Yu H P, Shi Z T, Pan J J, Zhang D, Kang S J, Zhu L, Dong G J, Guo L B, Zeng D L, Zhang G H, Xie L H, Xiong G S, Li J Y, Qian Q. A rare allele ofenhances grain size and grain yield in rice., 2015, 8: 1455–1465
[30] Matsushima R, Maekawa M, Kusano M, Tomita K, Kondo H, Nishimura H, Crofts N, Fujita N, Sakamoto W. Amyloplast membrane protein SUBSTANDARD STARCH GRAIN6 controls starch grain size in rice endosperm., 2016, 170: 1445–1459
[31] Song X J, Kuroha T, Ayano M, Furuta T, Nagai K, Komeda N, Segami S, Miura K, Ogawa D, Kamura T, Suzuki T, Higashiyama T, Yamasaki M, Mori H, Inukai Y, Wu J Z, Kitano H, Sakakibara H, Jacobsen S E, Ashikari M. Rare allele of a previously unidentified histone H4 acetyltransferase enhances grain weight, yield, and plant biomass in rice., 2015, 112: 76–81
[32] Feng Z M, Wu C Y, Wang C M, Roh J, Zhang L, Chen J, Zhang S Z, Zhang H, Yang C Y, Hu J L.controls grain size and leaf angle by modulating brassinosteroid homeostasis in rice., 2016, 67: erw204
[33] Zhang X J, Wang J F, Huang J, Lan H X, Wang C L, Yin C F, Wu Y Y, Tang H J, Qian Q, Li J Y. Rare allele ofassociated with grain length causes extra-large grain and a significant yield increase in rice., 2012, 109: 21534–21543
[34] Tan Y F, Xing Y Z, Li J X, Yu S B, Xu C G, Zhang Q F. Genetic bases of appearance quality of rice grains in Shanyou 63, an elite rice hybrid.2000, 101: 823–829
[35] Zhao X Q, Zhou L J, Ponce K, Ye G Y. The usefulness of known genes/QTLs for grain quality traits in anpopulation of diverse breeding lines tested using association analysis., 2015, 8: 29–42
[36] 张云康, 林榕辉, 闵捷, 吴戍君, 朱智伟, 葛进平. 浙江水稻品种资源的品质研究. 作物品种资源, 1992, (4): 23–25 Zhang Y K, Lin R H, Min J, Wu S J, Zhu Z W, Ge J P. The research of germplasm on rice quality in Zhejiang province., 1992, (4): 23–25 (in Chinese)
[37] 徐正进, 陈温福, 马殿荣, 吕英娜, 周淑清, 刘丽霞. 稻谷粒形与稻米主要品质性状的关系. 作物学报, 2004, 30: 894–900 Xu Z J, Chen W F, Ma D R, Lyu Y N, Zhou S Q, Liu L X. Correlations between rice grain shapes and main qualitative characteristics., 2004, 30: 894–900 (in Chinese with English abstract)
[38] Duan P G, Xu J S, Zeng D L, Zhang B L, Geng M F, Zhang G Z, Huang K, Huang L J, Xu R, Ge S, Qian Q, Li Y H. Natural variation in the promoter ofcontributes to grain size diversity in rice., 2017, doi: http://dxdoiorg/101016/jmolp2017 03009
[39] Xu Y B, Lu Y K, Xie C X, Gao S B, Wan J M, Prasanna B M. Whole-genome strategies for marker-assisted plant breeding., 2012, 29: 855–855
[40] Feng Y, Lu Q, Zhai R R, Zhang M C, Xu Q, Yang Y L, Wang S, Yuan X P, Yu H Y, Wang Y P, Wei X H. Genome wide association mapping for grain shape traits inrice., 2016, 244: 819–830
[41] Nagata K, Ando T, Nonoue Y, Mizubayashi T, Kitazawa N, Shomura A, Matsubara K, Ono N, Mizobuchi R, Shibaya T, Ogiso-Tanaka E, Hori K, Yano M, Fukuoka S. Advanced backcross QTL analysis reveals complicated genetic control of rice grain shape in across., 2015, 65: 308–318
Genome-wide Association Study of Grain Appearance and Milling Quality in a Worldwide Collection ofRice Germplasm
FANG Ya-Jie1,2, ZHU Ya-Jun3, WU Zhi-Chao2, CHEN Kai3, SHEN Cong-Cong3, SHI Ying-Yao1,*, and XU Jian-Long2,3,4,*
1Anhui Agricultural University, Hefei 230036, Anhui, China;2Institute of Crop Science, Chinese Academy of Agricultural Sciences, Beijing 100081, China;3Agricultural Genomics Institute, Chinese Academy of Agricultural Sciences, Shenzhen 518210, Guangdong, China;4Shenzhen Institute of Breeding & Innovation, Chinese Academy of Agricultural Sciences, Shenzhen 518120, Guangdong, China
Appearance and milling quality are two crucial properties of rice affecting their market acceptability. Understanding the genetic basis of rice grain quality could improve the efficiency of breeding for high quality. Here we carried out a genome-wide association analysis using the 404k SNP genotype and data of grain appearance and milling quality for a diverse panel of 400accessions selected from 3k Rice Genome Project collected in two environments. These were closely correlation between grain shape and quality (appearance and milling) ofrice. The chalk trait reduced milled rice rate and high milled rice rate. In addition to grain shape, the phenotype value of appearance and milling quality traits was obviously affected by environments. Total of 39 QTL were detected which was significantly associated with grain appearance and milling quality in two environments and 17 of them were new QTL. Six new QTL (,,,,, and) with minor or small effect were considered as stably expressed QTL because they were simultaneously identified in two environments. In addition, we found 21 QTL probably having multiple-effect in appearance and milling quality ofrice. Combined with the results of previous studies, we concluded that,, andplay an important role in affecting appearance and milling quality. This research provides valuable information for cloning QTL controlling appearance and milling quality and breeding rice with high yield and good quality by molecular techniques.
germplasms; appearance and milling quality; GWAS; quantitative trait loci/locus (QTL)
2017-04-05;
2017-09-10;
2017-09-28.
10.3724/SP.J.1006.2018.00032
通信作者(Corresponding authors): 徐建龙, E-mail: xujlcaas@126.com; 石英尧, E-mail: shiyy123@163.com
E-mail: fangyajie2016@126.com, Tel: 0755–28315619
本研究由国家高技术研究发展计划(863计划)项目(2014AA10A601), 农业部引进国际先进农业科学技术(948)项目(2016-X16), 深圳孔雀团队计划(20130415095710361)和中国农业科学院科技创新工程团队项目资助。
This study was supported by the National High-Tech Research & Development Program (863 Program) (2014AA10A601), the Introduction of International Advanced Agricultural Science and Technology of Ministry of Agriculture (948) (2016-X16), ShenZhen Peacock Plan (20130415095710361), and the Scientific and Technological Innovation Project of Chinese Academy of Agricultural Sciences.
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20170928.1458.012.html

